采后黄瓜在冷驯化处理过程中的转录组变化
2024-03-28王斌杨盼迪王玉昆蒋园园
王斌 杨盼迪 王玉昆 蒋园园


摘 要 采后黄瓜是冷敏性果菜类蔬菜,在低温贮藏时易发生冷害。前期研究结果表明,冷驯化处理通过诱导采后黄瓜耐冷性,减少冷害发生。为探究冷驯化处理诱导的转录组学变化,以采后黄瓜为试材,分析冷驯化处理期间的转录组变化。与贮藏前(0 h)相比,在冷驯化处理12 h和72 h时,分别鉴定到1 870和3 550个差异表达基因。基因表达验证结果表明,RT-qPCR和转录组结果高度一致,证明转录组测序数据的准确性和可靠性。GO富集分析结果显示,冷驯化处理诱导的差异表达基因主要富集在氧化还原过程、细胞膜组分和转录因子活性3个GO途径中,表明冷驯化处理通过调节细胞膜组分、细胞内氧化还原状态,增强冷藏黄瓜耐冷性。进一步分析发现,104个转录因子基因响应冷驯化低温,差异表达的转录因子主要是ERF、bZIP、WRKY和HSF家族,表明转录因子介导的转录调控在冷驯化诱导的耐冷性中发挥重要作用。研究结果为采后黄瓜诱导耐冷性提供了新见解,有助于加深对冷驯化诱导耐冷性分子机理的认识,为耐冷黄瓜培育提供了重要基因资源。
关键词 采后黄瓜;耐冷性;冷驯化;转录组学分析;转录因子
黄瓜(Cucumis sativus L.)又名青瓜、刺瓜、胡瓜等,是葫芦科黄瓜属1 a生蔓生或攀援草本植物,最初起源于喜马拉雅山南麓的热带雨林地区,现在世界各地均有广泛栽种,对低温的耐受性较差[1]。黄瓜是重要的果菜类蔬菜,因营养丰富、口感清脆,深受消費者喜爱[2]。但采后黄瓜含水量很高,组织鲜嫩,采后呼吸作用仍比较旺盛,常温条件下易失水萎蔫而腐烂变质,贮藏期很短[3]。低温贮藏可有效抑制病原微生物的生长繁殖,降低采后果蔬的呼吸作用和乙烯释放速率[4-5]。因此,在生产上,通常采用低温贮运的方式,延长采后黄瓜的贮藏期[6]。但采后黄瓜对低温特别敏感,低于10 ℃贮藏时就会发生冷害[7-8]。产生冷害的采后黄瓜在货架期很快腐烂变质,导致商品性显著降低,给果蔬经营者带来很大的经济损失[9],严重制约采后黄瓜产业发展。
近年来,植物耐冷性研究已从生理生化转向分子生物学研究,且对植物响应并耐受低温胁迫的分子机制研究取得了很大进步[10-12]。关于植物耐受低温胁迫的分子机制,在模式植物拟南芥(Arabidopsis thaliana)中研究的比较多,分子调控机制研究的也相对清楚[13-14]。在拟南芥中的研究证实,CBF(C-repeat binding transcription factor/dehydrate responsive element binding factor,CBF/DREB)转录因子是调控植物耐冷性的关键因子,低温处理激活CBF转录活性,随后识别并与冷响应相关基因(COR)启动子中的 CRT/DRE(C-repeat /dehydration responsive element)元件相结合,直接调控靶基因的表达[15-16]。拟南芥基因组中含有4个CBF基因,低温处理能迅速诱导4个CBF基因的表达,过表达CBF基因能显著增强转基因拟南芥的耐冷性[17-18]。由于物种特异的原因,不同植物耐受低温胁迫的分子机制可能存在明显差异。此外,拟南芥等模式植物没有严格意义上的肉质果实,与具有果实器官的园艺作物差异很大,离体的采后果实耐冷机制是否与仍处于生长状态的植物相似,仍值得研究。
随着第二代测序技术和生物信息学的联合应用,转录组测序(RNA sequencing,RNA-seq)已成为研究植物耐冷分子机理的重要方法[19-20]。Xie等[21]利用Illumina nextseq 2 500 测序平台,对低温处理的地瓜果实进行转录组测序,发现低温处理显著诱导18 681个基因的表达,下调 21 983个基因的表达。低温处理诱导的差异表达基因功能主要与细胞膜系统、抗氧化酶、碳水化合物代谢和激素代谢有关,推测地瓜可能通过调节激素平衡和提高抗氧化活性,以适应低温胁迫[21]。通过分析苦瓜叶片在低温处理后的转录组变化,发现了大量与低温胁迫相关的代谢途径,且鉴定出了10个潜在的具有低温抗性的MYB、WRKY和NAC等转录因子基因[22]。采后果蔬病理与分子生物学课题组利用RNA-seq结合生物信息学分析的方法,鉴定了与黄瓜 GR-RBP3(Glycine-rich RNA-binding protein 3)表达趋势一致的MYB转录因子基因,随后证明MYB62直接调控 GR-RBP3表达[8]。这些研究表明,转录组测序是揭示采后黄瓜耐冷性分子机理的可靠技术手段。
采后果蔬病理与分子生物学课题组前期证实,适宜时长和温度的冷驯化处理,能有效诱导采后黄瓜耐冷性,延缓冷害发生[9,23-27],但冷驯化诱导采后黄瓜耐冷性的分子机理仍未探明。分析采后黄瓜响应冷驯化处理的转录组学变化,能为采后黄瓜诱导耐冷性分子机理提供新见解[23]。
本研究以采后黄瓜果实为试材,分析冷驯化处理过程中的转录组变化,并鉴定与采后黄瓜耐冷性密切相关的转录因子基因,以期加深人们对采后黄瓜诱导耐冷性的理解,为黄瓜分子育种提供重要基因资源。
1 材料与方法
1.1 材料与处理
供试黄瓜品种为‘翠夏,父母本信息不详,种子由广东现代种业发展有限公司提供。黄瓜果实在适宜成熟度时采收,采收后立即运回实验室。按照如下标准挑选采后黄瓜样品:果实长度约为30 cm,纵切面直径约3 cm,果皮表面无明显虫害和药害症状,果皮表面的果刺没有明显脱落,果形笔直,两端粗细均一。
将挑选好的黄瓜分成3组,代表3个生物学重复,每组30根。装在塑料框中,用塑料薄膜保鲜袋密封包装。先将采后黄瓜在10 ℃的低温恒温培养箱中贮藏72 h,之后再转移至5 ℃贮藏,低温培养箱的相对湿度为95%~98%。在10 ℃贮藏0 h、12 h和72 h时,取样用于转录组测序分析。收集黄瓜果皮作为分析样品,用市售普通去皮刀削下果皮,果皮厚度约1 mm。立即用液氮速冻处理,并冻存于-80 ℃冰箱。
1.2 试验方法
1.2.1 冷藏黄瓜冷害评估指标 冷害指数(Chilling injury index,CII)、相对电导率(Relative electrical conductivity,REC)和PSⅡ原初光能转化效率(Fv/Fm)3个指标反映采后黄瓜的冷害严重程度。CII、REC和Fv/Fm测定方法见文献[23,26]。
1.2.2 总RNA提取、文库构建和RNA-seq 将冻存样品在液氮中研磨,使用天根生化科技(北京)有限公司的RNAprep Pure多糖多酚植物总RNA提取试剂盒(产品编号:DP441)提取黄瓜果皮总RNA。分别使用Agilent Bioanalyzer 2 100和Nano DropTM仪器检测RNA样品的浓度和质量,运用Illumina TruSeqTM RNA Sample Preparation Kit试剂盒构建cDNA文库。ABI Step One Plus Real-Time PCR 系统定量cDNA文库的有效浓度(>2 nM),库检合格后,利用Illumina next seq 2 500 测序平台测序[23],测序工作由北京百迈客生物科技有限公司完成。
1.2.3 生物信息学分析和差异表达基因筛选 转录组测序数据下机后,采用HISAT(Hierarchical indexing for spliced alignment of transcripts)方法[28],以黄瓜基因组数据(http://www.cucurbitgenomics.org/organism/20)作為参考基因组,进行序列比对及后续分析。采用DESeq2法比较样品组间的基因表达差异[29],获得差异表达基因集。
利用NCBI 非冗余蛋白质数据库(ftp://ftp.ncbi.nih.gov/blast/db/)、GO (gene ontology)(http://www.geneontology.org/)、大型蛋白质家族数据库(http://pfam.xfam.org/)、蛋白质直系同源数据库(http://www.ncbi.nlm.nih.gov/COG/)、KEGG(kyoto encyclopedia of genes and genomes)(http://www.genome.jp/kegg)、蛋白质序列注释数据库Swiss-Prot(http://www.uniprot.org/)等公共数据库注释基因[30]。利用 Blast2GO(https://www. blast2go.org/) 进行差异表达基因的GO 注释,通过P值(P< 0.05) 的 Benjamini-Hochberg矫正方法确定差异表达基因中显著富集的GO分类,使用Cytoscape (http://www.cytoscape. org/)注释通路。
采用FPKM(每千个碱基的转录每百万映射读取的片段数)计算基因的表达量[22,31]。差异表达基因(Differentially expressed genes,DEGs)的筛选标准为:差异倍数(Fold change,FC)≥ 2.5,且错误发现率(False discovery rate,FDR)<0.01。
1.2.4 差异表达基因的表达验证 采用荧光定量PCR(qRT-PCR)法验证差异表达基因的表达水平。所用引物信息见表1,以黄瓜ACTIN作为内参基因。
1.3 数据整理与分析
使用Excel 2016软件记录和整理数据,通过Tbtools软件绘制差异表达基因的表达热图[32]。
2 结果与分析
2.1 冷驯化处理对采后黄瓜冷害的影响
图1显示,采后黄瓜经10 ℃冷驯化处理72 h(3 d)后,冷害指数从0 d 时的0增加到12 d的 0.70,仅增加了0.70。相对电导率从0 d 时的 14.86%增加到12 d的27.38%,仅增加了 12.52%。Fv/Fm值在整个贮藏期间缓慢下降,从0 d 时的0.71下降到12 d的0.56,仅降低了0.15。
2.2 转录组测序数据质量评估
为分析采后黄瓜在冷驯化处理期间的转录组变化,选取0 h、12 h和72 h 3个时间点的黄瓜果皮样品进行转录组测序。转录组测序共获得 53.82 Gb的高质量数据(Clean data),基因组比对效率为89.43%~93.91%,总碱基对数平均值为7 111 873 467 bp,GC平均含量为 44.39%,Q20平均值为97.56%(表2)。这些结果表明,RNA seq数据质量很高,完全满足生物信息学分析的要求。
将DEGs在NR、GO、KEGG等数据库中比对,得到对应的注释信息。0 h 与 12 h的比较组共注释到1 870个差异表达基因,0 h 与 72 h的比较组共注释到3 550个差异表达基因,12 h 与 72 h的比较组共注释到1 818个差异表达基因(表3)。各数据库的注释结果如表3所示。
2.3 冷驯化处理12 h期间的转录组变化
与冷驯化处理前(0 h)相比,冷驯化处理12 h期间共鉴定到1 870个DEGs(图2-A)。其中,906个DEGs表达上调,964个DEGs表达下调,下调表达的DEGs数量稍多于上调表达的DEGs数量(图2-A)。说明在基因表达层面,冷驯化迅速诱导了采后黄瓜对低温的适应反应。GO通路富集分析结果显示,在生物过程类别中,氧化还原过程(oxidation-reduction process)、转录调控(regulation of transcription)和代谢过程(metabolic process)是3个富集DEGs数量最多的GO途径,氧化还原过程富集度最高;在细胞组分类别中,DEGs主要富集在膜中间组分(integral component of membrane)亚类;在分子功能类别中,DEGs主要富集在转录因子活性(transcription factor activity)、血红素结合(heme binding)和铁离子结合(iron ion binding)亚类中(图2-B),转录因子活性富集度最高。这些结果表明,氧化还原过程和转录调控可能在采后黄瓜适应低温胁迫过程中至关重要。此外,冷驯化诱导细胞膜组分变化,可能是采后黄瓜响应低温的重要方式。
2.4 冷驯化处理72 h期间的转录组变化
与冷驯化处理前(0 d)相比,冷驯化处理72 h期间共鉴定到3 550个DEGs,且DEGs的数量明显多于12 h(图3-A),表明冷驯化处理持续强化了采后黄瓜对低温胁迫的适应能力。其中,冷驯化72 h处理诱导1 370个DEGs表达,抑制2 180个DEGs表达,下调表达的DEGs数量多于上调表达(图3-A)。
GO富集结果显示,在生物过程类别中,差异表达基因主要富集在氧化还原过程、转录调控和细胞氧化剂解毒(cellular oxidant detoxification)3个亚类中,氧化还原过程亚类中富集的DEGs最多;在细胞组分类别中,DEGs主要富集在膜中间组分、质膜(plasma membrane)和胞外区域(extracellular region)3个亚类,膜中间组分亚类的富集度最高;在分子功能类别中,DEGs主要富集在金属离子结合(metal ion binding)、血红素结合和蛋白激酶活性(protein kinase activity)亚类中,在金属离子结合亚类富集的基因数量最多(图3-B)。这些结果再次表明,提高抗氧化解毒能力可能是采后黄瓜耐冷性增强的重要原因。另外,较长时间的冷驯化处理可能激活了蛋白激酶活性,导致蛋白质构象和酶活性发生变化,从而更好地适应低温环境。[FL)]
2.5 冷驯化处理72 h与12 h间的转录组差异
冷驯化处理72 h与12 h相比,72 h的冷驯化处理上调626个DEGs表达,下调1 192个DEGs表达,下调表达的DEGs多于上调表达 (图4-A),表明持续的冷驯化处理抑制一些生物过程,避免物质过度消耗,以维持更长时间的生命活动。
在生物过程类别中,DEGs主要富集在氧化还原过程、转录调控和生长素激活的信号途径(auxin-activated signaling pathway)3个亚类中。同样,氧化还原过程亚类中富集的DEGs最多;在细胞组分类别中,膜中间组分富集了最多的DEGs;在分子功能类别中,DEGs主要富集在转录因子活性、血红素结合和铁离子结合3个亚类,转录因子活性亚类富集的基因数量最多(图 4-B)。这些结果表明,经过持续的冷驯化处理,采后黄瓜的抗氧化解毒活性被强化,以应对持续的胁迫低温对采后黄瓜造成的氧化损伤。[FL)]
2.6 冷驯化处理对氧化还原过程有关差异表达基因表达的影响
在生物过程分类中,由于氧化还原过程中富集的差异表达基因数量最多,表明氧化还原过程在冷驯化诱导的耐冷性中发挥重要作用。为此,重点分析了富集与氧化还原过程有关DEGs在冷驯化处理期间的表达模式。共有381个DEGs的功能与氧化还原过程有关,其表达模式总体分为3种类型。一些DEGs的表达被冷驯化处理持续诱导,表达量随处理时间增加而增加(图5)。比如CsaV3_1G011780基因,在0 h的表达量为 1.78,12 h的表达量为2.96,72 h的表达量为 19.05。一些DEGs只被特定时长的冷驯化处理所诱导(图5)。比如CsaV3_3G012260基因只在处理12 h时被诱导表达,而CsaV3_7G030830基因只在72 h时被诱导表达。另一些DEGs被冷驯化处理所抑制,有些基因在12 h时抑制作用更强,有些在72 h时抑制作用更强(图5)。比如CsaV3_2G017780基因在72 h的抑制作用强于12 h,而CsaV3_5G001900基因在12 h的抑制作用更强。這些结果表明,在冷驯化处理期间,与氧化还原过程有关基因的表达模式十分复杂,相关基因精准并特异地参与采后黄瓜适应低温胁迫的过程。值得注意的是,上调表达的差异表达基因主要是活性氧清除酶编码基因,比如抗坏血酸过氧化物酶、过氧化氢酶、过氧化物酶、谷胱甘肽过氧化物酶、谷胱甘肽巯基转移酶、超氧化物歧化酶和脯氨酸脱氢酶等,表明冷驯化处理提高了采后黄瓜的抗氧化解毒活性。
2.7 冷驯化处理对膜组分有关差异表达基因表达的影响
在细胞组分类别中,冷驯化处理导致的DEGs主要富集在细胞膜组分亚类中,表明冷驯化处理过程中膜组分发生了显著变化。冷驯化诱导细胞膜组分改变,可能是采后黄瓜响应低温信号并适应低温胁迫的重要机制。为此,重点分析了冷驯化处理对膜组分有关DEGs表达模式的影响。共筛选出1 126个DEGs的功能与细胞膜组分有关,表达模式总体分为2种类型。一些基因的表达被冷驯化处理所诱导,另一些基因的表达被冷驯化处理抑制(图6)。比如,CsaV3_1G027990基因在72 h的表达量显著高于12 h 和0 h,且12 h的表达量高于0 h,而CsaV3_2G029640基因在0 h的表达量显著高于12 h和72 h时。这些结果表明,冷驯化处理显著影响细胞膜组分,在冷驯化处理期间,细胞膜组分会根据细胞所处环境不断调整优化,以更好地适应低温胁迫。
2.8 响应冷驯化处理的转录因子鉴定
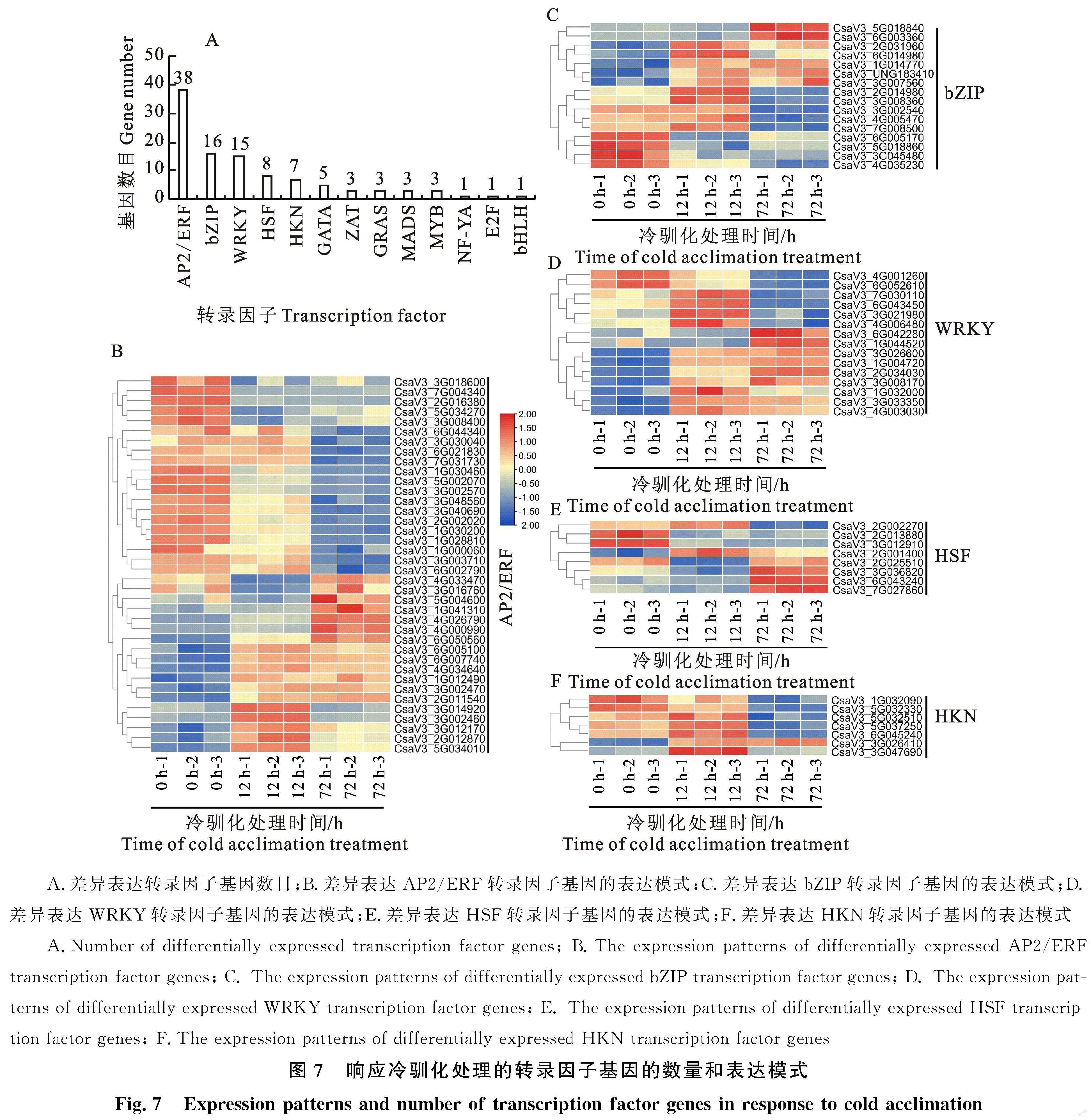
GO富集分析结果显示,与转录调控有关的DEGs被显著富集,表明冷驯化通过调节转录调控活性,被激活或抑制的转录因子,进一步调控下游耐冷相关基因表达(图2-B~图4-B)。为此,从DEGs中筛选了响应冷驯化处理的转录因子基因,并分析了主要转录因子家族基因在冷驯化处理过程中的表达模式。共有104个差异表达的转录因子基因响应冷驯化处理,主要是AP2/ERF、bZIP、WRKY、HSF、HKN、GATA等转录因子家族(图7-A)。其中,38个是AP2/ERF家族基因,16个是bZIP家族基因,15个是WRKY家族基因,8个是HSF家族基因,7个是HKN家族基因,5个是GATA家族基因,ZAT、GRAS、MADS和MYB家族各有3个,CBF/NF-YA、E2F和bHLH家族各有1个(图7-A)。其次,重点分析了AP2/ERF、bZIP、WRKY、HSF和HKN家族基因在冷驯化处理期间的表达模式。
冷驯化处理下调20个 AP2/ERF基因的表达。3个 AP2/ERF基因(CsaV3_4G003470、CsaV3_3G016760和CsaV3_5G004600)在12 h表达下调,却在72 h时又表达上调。冷驯化处理显著上调另外15个 AP2/ERF基因的表达 (图7-B)。
12 h 和72 h的冷驯化处理显著上调7个bZIP基因的表达。5个bZIP基因的表达只被12 h的冷驯化处理所诱导,而72 h的冷驯化处理却抑制他们的表达。冷驯化处理显著抑制其余4个bZIP基因的表达(图7-C)。
冷驯化处理抑制2个WRKY基因(CsaV3_4G001260和CsaV3_6G051260)的表达,且72 h的抑制作用更强烈。4个WRKY基因的表达在冷驯化处理12 h时显著上调,但在72 h时显著下调。12 h 和72 h的冷驯化处理显著上调其余9个WRKY基因的表达(图7-D)。
共有8个HSF基因在冷驯化处理过程中差异表达。其中,CsaV3_2G002270基因在12 h时的表达量显著高于0 h和72 h,CsaV3_2G025510基因在12 h时的表达量显著低于0 h和72 h。冷驯化处理显著下调2个HSF基因(CsaV3_2G 013880和CsaV3_3G012910)的表达,显著上调另外4个HSF基因的表达(图7-E)。
冷驯化处理共导致7个HKN基因的表达量发生显著变化(图7-F)。其中,12 h和72 h的冷驯化处理显著下调2个HKN基因(CsaV3_1G032090和CsaV3_5G032330)的表达,显著诱导CsaV3_3G026410基因的表达。另外4个HKN基因在处理12 h时表达上调,在72 h时表达显著下调(图7-F)。
这些结果说明,转录因子参与了冷驯化诱导的采后黄瓜耐冷性,且表达模式具有明显的时空特异性特征。也进一步说明,转录因子之间紧密配合,相互协调,复杂地调控采后黄瓜耐冷性。
2.9 冷驯化诱导差异表达的转录因子互作分析
转录因子不仅可以单独调控靶基因表达,且他们之间还可以相互作用,协同调控靶基因表达[33-34]。为探究冷驯化诱导的转录因子之间是否存在潜在的相互作用关系,在百迈客公司的在线云平台(http://www.biocloud.net/),预测了转录因子之间的相互作用。结果显示,共发现7个转录因子之间可能存在相互作用关系。AAI5(abscisic acid-insensitive 5)(CsaV3_7G008500)与GAI1(gibberellin-inducible factor)(CsaV3_1G031060)互作(圖8)。ZAT9(zinc finger protein)(CsaV3_3G007370)与NF-YA9(nuclear transcription factor Y subunit A-9)(CsaV3_7G031020)、GATA8(GATA transcription factor 8)(CsaV3_6G036510)、GATA9(CsaV3_3G023720)互作(图8)。GATA9与ZAT10(CsaV3_4G034970)之间也存在相互作用关系 (图8)。由此可以看出,冷驯化诱导的转录因子之间存在相互作用关系,协同调控采后黄瓜耐冷性。是否其他转录因子之间还存在相互作用关系,仍需进一步分析验证。
2.10 qRT-PCR验证
为了验证转录组测序数据的准确性和可靠性,随机挑选9个差异表达基因,利用qRT-PCR法检测在冷驯化处理期间的表达变化,结果如图9所示。在转录组测序结果中, ER2和 MYB301的表达量在冷驯化处理12 h时急剧增加,72 h时又有所下降(图9)。与0 h相比,在冷驯化处理12 h和72 h时, EI3、 ACO1、 ERF113、 MYB36和 MYB4的表达显著上调, AS1和 ACS1的表达却显著下调(图9)。qRT-PCR分析的结果显示,9个代表性差异表达基因的表达模式与RNA-seq结果总体一致(图9),表明转录组测序数据是可靠的。冷驯化处理诱导或抑制其表达,预示着它们在采后黄瓜耐冷性中可能具有重要的调控 作用。
3 讨 论
采后果蔬的寿命与贮藏环境温度有关,贮藏温度越高,贮藏寿命越短;相反,贮藏温度越低,贮藏期越长[35],但过低的贮藏温度会引发冷害。采后黄瓜贮藏在10 ℃以下低温,就会发生冷害[6]。在本研究中,采后黄瓜在10 ℃冷驯化处理72 h,再贮藏在5 ℃低温,缓解了采后黄瓜冷害,与本课题组之前的研究结果一致[9,27],再次证实冷驯化处理是有效控制冷藏黄瓜冷害的采后处理技术。
在冷驯化处理期间,许多DEGs的表达水平发生了显著变化,且下调表达的DEGs数量明显多于上调表达的差异表达基因数量,表明冷驯化抑制了一些代谢反应,避免采后黄瓜对低温胁迫作出过度反应。GO富集结果显示,DEGs主要富集在氧化还原过程、细胞膜中间组分和转录调控类别中,表明冷驯化通过改变细胞膜组分,调节氧化还原状态和转录活性,复杂地协调相关防御途径,最终诱导采后黄瓜产生系统抗冷性。这与在采后番茄和番木瓜中低温处理诱导的主要GO通路类似[36-37]。
氧化还原动态平衡对植物正常生长发育至关重要[38]。在逆境胁迫条件下,植物组织内活性氧代谢伴随着氧化还原状态变化而变化,从而引起细胞内代谢和相关基因表达的改变,为适应新环境变化而作出代谢调整,达到新的氧化还原平衡[39]。相比于低温胁迫,冷驯化是一种相对温和的低温处理[40]。本研究中,冷驯化处理显著影响大量与氧化还原过程有关基因的表达,且特定基因的表达模式具有明显的时空特异性,表明冷驯化处理通过调节冷藏黄瓜的氧化还原动态平衡,赋予采后黄瓜耐冷性,以更好地适应低温胁迫。此外,冷驯化处理上调的与氧化还原过程有关的DEGs主要是活性氧清除酶编码基因,表明冷驯化处理激活了冷藏黄瓜的抗氧化系统,提高了对活性氧自由基的清除能力,这可能是冷驯化诱导采后黄瓜耐冷性增强的重要原因之一,这些结果与本课题组之前的结果相互印证[9]。
细胞膜是细胞生命活动赖以延续的基础,当植物发生冷害后,细胞膜选择透过性增大,流动性降低[41]。本研究中,冷驯化处理的采后黄瓜,在胁迫低温条件下贮藏时,相对电导率缓慢增加,表明冷驯化处理有助于维持细胞膜完整性,避免细胞膜透性增大而有毒物质大量渗漏,造成细胞中毒。冷驯化处理诱导的DEGs显著富集在细胞膜组分分类中,表明细胞膜组分在冷驯化处理期间发生了显著变化。在冷驯化处理过程中,细胞膜组分不断调整优化,从而维持细胞膜较高流动性和完整性,以适应更低的胁迫温度。总之,冷驯化处理通过影响细胞膜组分相关基因的表达,保持细胞膜完整性和流动性,增强了冷藏黄瓜对低温胁迫的耐受性,这与笔者之前的结果是一致的[9,23,27]。
许多研究表明, WRKY、 AP2/ERF、 bZIP、 HSF、 MYB等转录因子在调节植物低温胁迫应答反应中起着重要调控作用[42-46]。本研究中,104个转录因子基因在冷驯化处理过程中表达发生了显著变化。同一家族的转录因子基因在冷驯化处理期间的表达模式不同,表明特定转录因子特异性地调控采后黄瓜耐冷反应。许多转录因子基因表达被冷驯化处理诱导,表明这些转录因子与采后黄瓜耐冷性正相关。被冷驯化处理抑制表达的转录因子基因,可能负调控采后黄瓜耐冷性。HSF转录因子通常响应热激处理,热处理诱导的采后黄瓜耐冷性与HSF表达密切相关[47]。本研究中,冷驯化处理影响8个HSF基因的表达,表明除热处理外,低温处理也可诱导HSF表达,HSF表达可能与冷驯化诱导的采后黄瓜耐冷性密切相关。冷驯化诱导的转录因子之间还存在相互作用关系,表明转录因子之间可能通过相互作用形成复合体,协同调控冷藏黄瓜耐冷性。综上,这些结果为耐受低温胁迫的黄瓜新种质培育提供了重要基因资源。
4 结 论
冷驯化处理诱导的差异表达基因主要富集在氧化还原过程、细胞膜中间组分和转录调控活性GO类别中,表明冷驯化处理通过调节采后黄瓜氧化还原活性和细胞膜组分,增强采后黄瓜耐冷性,以适应低温胁迫。此外,许多 AP2/ERF、WRKY、bZIP、HSF等转录因子家族基因的表达在冷驯化处理期间发生了明显变化,表明这些转录因子参与了采后黄瓜诱导耐冷性的调控,可在今后深入研究这些转录因子基因的功能。总之,本研究通过分析冷驯化处理期间的转录组学变化,从转录水平上阐述了冷驯化诱导采后黄瓜耐冷性的潜在机制。
参考文献 Reference:
[1] 梁肇均,陈 林,王 瑞,等. 廣东黄瓜育种研究进展[J].广东农业科学,2021,48(9):22-31.
LIANG ZH J,CHEN L,WANG R,et al. Research progress of cucumber breeding in Guangdong province[J].Guangdong Agricultural Sciences,2021,48(9):22-31.
[2] 高珏晓,王俊英,梁增文,等. 5种鲜食黄瓜感官与品质评价初探[J].蔬菜,2022(5):11-16.
GAO Y X,WANG J Y,LIANG Z W,et al. Preliminary study on sensory and quality evaluation of five types of fresh cucumber[J].Vegetables,2022(5):11-16.
[3] 王 斌,武春爽,何金明,等. CsCAT3克隆与其在冷驯化诱导采后黄瓜耐冷性中的作用初探[J].核农学报,2021, 35(10):2267-2276.
WANG B,WU CH SH,HE J M,et al. Cloning of CsCAT3 and its role in pre-storage cold acclimation-induced chilling tolerance in harvested cucumber[J].Journal of Nuclear Agricultural Sciences,2021,35(10):2267-2276.
[4] 张 敏,解 越. 采后果蔬低温贮藏冷害研究进展[J]. 食品与生物技术学报,2016,35(1):1-11.
ZHANG M,XIE Y. Research progress of chilling injury on post-harvest fruits and vegetables stored at low temperature[J].Journal of Food and Biotechnology,2016,35(1):1-11.
[5] KASHASH Y,HOLLAND D,PORAT R. Molecular mechanisms involved in postharvest chilling tolerance of pomegranate fruit[J].Journal of the Science of Food and Agriculture,2019,99(13):5617-5623.
[6] 王 斌,朱世江. 阶段降温对冷藏黄瓜耐冷性的诱导作用[J].江苏农业学报,2020,36(4):1028-1035.
WANG B,ZHU SH J. Induction of chilling tolerance in cold-stored cucumbers by slowly cooling treatment[J].Jiangsu Journal of Agricultural Sciences,2020,36(4):1028-1035.
[7] YANG H Q,WU F H,CHENG J Y. Reduced chilling injury in cucumber by nitric oxide and the antioxidant response[J].Food Chemistry,2011,127(3): 1237-1242.
[8] 王 斌,武春爽,湯冰琳,等. 黄瓜果实 CsMYB62克隆及其对 CsGR-RBP3表达的调控[J].核农学报,2022,36(5):907-917.
WANG B,WU CH SH,TANG B L,et al. Cloning of CsMYB62 and its regulations on CsGR-RBP3 expression in cucumber fruit[J].Journal of Nuclear Agricultural Sciences,2022,36(5):907-917.
[9] WANG B,ZHU S J. Pre-storage cold acclimation maintained quality of cold-stored cucumber through differentially and orderly activating ROS scavengers[J].Postharvest Biology and Technology,2017,129:1-8.
[10] GUO X,LIU D,CHONG K. Cold signaling in plants: Insights into mechanisms and regulation[J].Journal of Integrative Plant Biology,2018,60(9):745-756.
[11] SHI Y T. Molecular regulation of CBF signaling in cold acclimation[J].Trends in Plant Science,2018,23(7):623-637.
[12] DING Y L,SHI Y T,YANG S H. Molecular regulation of plant responses to environmental temperatures[J].Molecular Plant,2020,13(4):544-564.
[13] DING Y L,SHI Y T,YANG S H. Advances and challenges in uncovering cold tolerance regulatory mechanisms in plants[J].New Phytologist,2019,222(4):1690-1704.
[14] PAREEK A,KHURANA A,SHARMA A K,et al. An overview of signaling regulons during cold stress tolerance in plants[J].Current Genomics,2017,18(6):498-511.
[15] RITONGA F N,CHEN S. Physiological and molecular mechanism involved in cold stress tolerance in plants[J].Plants,2020,9(5):560.
[16] BARRERO-GIL J,SALINAS J. Gene regulatory networks mediating cold acclimation:the CBF pathway[J].Advances in Experimental Medicine and Biology,2018,1081:3-22.
[17] GILOMOUR S J,SEBOLT A M,SALAZAR M P,et al. Overexpression of the Arabidopsis CBF3 transcriptional activator mimics multiple biochemical changes associated with cold acclimation[J].Plant Physiology,2000, 124(4):1854-1865.
[18] JIANG B C,SHI Y T,ZHANG X Y,et al. PIF3 is a negative regulator of the CBF pathway and freezing tolerance in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2017,114(32):E6695-E6702.
[19] CAO X,WU Z,JIANG F L,et al. Identification of chilling stress-responsive tomato microRNAs and their target genes by high-throughput sequencing and degradome analysis[J].BMC Genomics,2014,15(1):1-16.
[20] TANG S W,XIAN Y J,WANG F,et al. Comparative transcriptome analysis of leaves during early stages of chilling stress in two different chilling-tolerant brown-fiber cotton cultivars[J].PLoS One,2021,16(2):e0246801.
[21] XIE Z Y,ZHOU Z L,LI H M,et al. High through put sequencing identifies chilling responsive genes in sweetpotato (Ipomoea batatas Lam.) during storage[J].Genomics,2019,111(5):1006-1017.
[22] 杜文丽,陈中钐,许端祥,等. 低温胁迫下苦瓜叶片转录组差异基因分析及生理响应特征[J].核农学报,2021, 35(2):338-348.
DU W L,CHEN ZH SH,XU R X,et al. Physiological response and differentially expressed genes analysis of transcriptome in Momordica charantia L.leaf under cold stress[J].Journal of Nuclear Agricultural Sciences,2021, 35(2):338-348.
[23] WANG B,WU C,WANG G,et al. Transcriptomic analysis reveals a role of phenylpropanoid pathway in the enhancement of chilling tolerance by pre-storage cold acclimation in cucumber fruit[J].Scientia Horticulturae,2021,288:110282.
[24] WANG B,SHEN F,ZHU S J. Proteomic analysis of differentially accumulated proteins in cucumber (Cucumis sativus) fruit peel in response to pre-storage cold acclimation[J].Frontiers in Plant Science,2018,8:2167.
[25] WANG B,WANG G,SHEN F,et al. A Glycine-rich RNA-binding protein,CsGR-RBP3,is involved in defense responses against cold stress in harvested cucumber (Cucumis sativus L.) fruit[J].Frontiers in Plant Science,2018,9:540.
[26] WANG B,WANG G,ZHU S J. DNA damage inducible protein 1 is involved in cold adaption of harvested cucumber fruit[J].Frontiers in Plant Science,2020,10:1723.
[27] 王 斌. 冷馴化诱导采后黄瓜抗冷性的生理与分子机理[D]. 广州: 华南农业大学,2018.
WANG B. Physiological and molecular mechanisms of chilling tolerance induced by cold acclimation in postharvest cucumber[D]. Guangzhou: South China Agricultural University,2018.
[28] KIM D,LANGMEAD B,SALZBERG S L. HISAT: a fast spliced aligner with low memory requirements[J].Nature Methods,2015,12(4):357-360.
[29] LOVE M I,HUBE W,AANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J].Genome Biology,2014,15(12):550.
[30] 蒋 彧,何俊蓉. 国兰叶色突变体根状茎差异表达基因分析[J].核农学报,2022,36(3):497-508.
JIANG X,HE J R. Analysis of differential expression genes in rhizome of leaf color mutant of Chinese orchid[J].Journal of Nuclear Agricultural Sciences,2022, 36(3):497-508.
[31] MUHAMMAD I I,KONG S L,AKMAR ABDULLAH S N,et al. RNA-seq and ChIP-seq as complementary approaches for comprehension of plant transcriptional regulatory mechanism[J].International Journal of Molecular Sciences,2020,21(1):167.
[32] CHEN C J,CHEN H,ZHANG Y,et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194-1202.
[33] ZENG J K,LI X,XU Q,et al. EjAP2-1,an AP2/ERF gene,is a novel regulator of fruit lignification induced by chilling injury,via interaction with EjMYB transcription factors[J].Plant Biotechnology Journal,2015,13(9):1325-1334.
[34] GE H,ZHAGN J,ZHANG Y J,et al. EjNAC3 transcriptionally regulates chilling-induced lignification of loquat fruit via physical interaction with an atypical CAD-like gene[J].Journal of Experimental Botany,2017,68(18):5129-5136.
[35] LI Y L,ZHAO Y T,ZHANG Z C,et al. Near-freezing temperature storage improves shelf-life and suppresses chilling injury in postharvest apricot fruit (Prunus armeniaca L.) by regulating cell wall metabolism[J].Food Chemistry,2022,387:132921.
[36] HUNTER D A,NAPIER N J,ERRIDGE Z A,et al. Transcriptome responses of ripe cherry tomato fruit exposed to chilling and rewarming identify reversible and irreversible gene expression changes[J].Frontiers in Plant Science,2021,12:685416.
[37] WU Q X,LI Z W,CHEN X,et al. Comparative metabolites profiling of harvested papaya (Carica papaya L.) peel in response to chilling stress[J].Journal of the Science of Food and Agriculture,2019,99(15):6868-6881.
[38] MHAMDI A,VAN BREUSEGEM F. Reactive oxygen species in plant development[J].Development,2018,145(15):dev164376.
[39] 王 勇. 酸棗适应干旱的生理特征和氧化还原特征物质研究[D]. 北京: 中国科学院研究生院(教育部水土保持与生态环境研究中心),2016.
WANG Y. Research on physiological characteristics and redox characteristic substances of wild jujube adapting to drought[D]. Beijing: Graduate School of Chinese Academy of Sciences (Water and Soil Conservation and Ecological Environment Research Center of the Ministry of Education),2016.
[40] HINCHA D K,ZUTHER E. Introduction: Plant cold acclimation and winter survival[J].Methods in Molecular Biology,2020,2156:1-7.
[41] LYONS J M. Chilling injury in plants[J].Annual Review of Plant Physiology,1973,24:445-466.
[42] ZHANG Y,YU H J,YANG X Y,et al. CsWRKY46,a WRKY transcription factor from cucumber,confers cold resistance in transgenic-plant by regulating a set of cold-stress responsive genes in an ABA-dependent manner[J].Plant Physiology and Biochemistry,2016,108:478-487.
[43] IMEN K,SANDRA G,LIU M C,et al. Ethylene Response Factors (ERF) are differentially regulated by different abiotic stress types in tomato plants[J].Plant Science,2018,274:137-145.
[44] ZHANG L N,ZHANG L C,XIA C,et al. A novel wheat bZIP transcription factor,TabZIP60,confers multiple abiotic stress tolerances in transgenic Arabidopsis[J].Physiologia Plantarum,2015,153(4):538-554.
[45] ANDRASI N,PETTKO-SZANDTNER A,SZABADOS L. Diversity of plant heat ahock factors:Regulation,interactions and functions[J].Journal of Experimental Botany,2021,72(5):1558-1575.
[46] WANG Y,LI X Y,LI C X,et al. The regulation of adaptation to cold and drought stresses in Poa crymophila Keng revealed by integrative transcriptomics and metabolomics analysis[J].Frontiers in Plant Science,2021,12:631117.
[47] 陳 珊,王晓晨,邝健飞,等. 黄瓜果实耐冷性与CsHSFs基因表达关系的研究[J].华南农业大学学报,2015, 36(5):85-91.
CHEN SH,WANG X CH,KUANG J F,et al. The expressions of CsHSFs gene in relation to chilling tolerance of cucumber fruits[J].Journal of South China Agricultural University,2015,36(5):85-91.
Transcriptomic Changes of Postharvest Cucumber during Cold Acclimation
Abstract Postharvest cucumber is a kind of cold-sensitive fruit vegetable,which is subjected to chilling injury during cold storage. The results of previous studies showed that cold acclimation treatment could induce the chilling tolerance of postharvest cucumber and reduce the occurrence of chilling injury. In order to explore the transcriptomic changes induced by cold acclimation,transcriptomic changes during cold acclimation were analyzed. Compared with that before storage (0 h),cold acclimation significantly affected the differential expression of 1 870 and 3 550 genes at 12 h and 72 h,respectively. The expression verification results of representative differentially expressed genes (DEGs) showed that the results obtained by qRT-PCR and RNA-seq were consistent in general,which conformed the accuracy and reliability of RNA-seq data. The results of GO enrichment analysis showed that the DEGs induced by cold acclimation were mainly enriched in three GO pathways: oxidation-reduction process,integral component of membrane and transcription factor activity,suggesting that cold acclimation enhanced the chilling tolerance of cucumber fruit by regulating oxidation-reduction status and cell membrane component. Further analysis showed that 104 transcription factor genes were responded to cold acclimation,and differentially expressed transcription factors were mainly ERF,bZIP,WRKY and HSF families,indicating that transcription regulation mediated by transcription factor played an important role in cold-acclimation-induced chilling tolerance in cucumber fruit. The results provide new insights into the induction of chilling tolerance in harvest cucumbers,and help to deepen the understanding of the molecular mechanisms of chilling tolerance induced by cold acclimation,and identify important gene resources for the cultivation of cold-tolerant cucumber.
Key words Harvested cucumber; Chilling tolerance; Cold acclimation; Transcriptomic analysis; Transcriptional factor
