水禽细小病毒选择压力与基因重组分析
2023-12-25汪宏才商雨郭云清曾哲张蓉蓉温国元罗青平
汪宏才 商雨 郭云清 曾哲 张蓉蓉 温国元 罗青平

摘要:通过对临床水禽细小病毒进行PCR检测、基因组测序和进化分析,确定分离毒株的基因型。进一步通过Paml、RDP软件对分离株的基因组进行选择压力和基因重组分析,确定分离株的演化趋势。结果表明,分离的3株水禽细小病毒均属于新型鹅细小病毒,分别命名为YiCH株、AnQ株和XiY株,基因组长度分别为5 056、5 056、5 068 bp。3个分离株与新型鹅细小病毒M15株亲源关系最近,其中XiY株两端ITR区域191~196位、4 878~4 883位均有6个碱基插入。对分离株进行选择压力分析,分离株VP蛋白检测出3个正选择位点,分别为116Q、261A、485S。重组分析显示,分离株YiCH株和AnQ株都存在重组现象,以XiY株为主要亲本株、GPV 06-0329株为次要亲本株重组而成。
关键词:水禽细小病毒;新型鹅细小病毒;分离鉴定;选择压力;基因重组
中图分类号:S855.3 文献标识码:A
文章编号:0439-8114(2023)11-0191-07
DOI:10.14088/j.cnki.issn0439-8114.2023.11.033 开放科学(资源服务)标识码(OSID):
Selection pressure and gene recombination analysis of waterfowl parvovirus
WANG Hong-cai, SHANG Yu, GUO Yun-qing, ZENG Zhe, ZHANG Rong-rong, WEN Guo-yuan, LUO Qing-ping
(Institute of Animal Husbandry and Veterinary Sciences/Key Laboratory of Animal Bacterial Disease Prevention and Control Formulations of the Ministry of Agriculture and Rural Affairs/Hubei Key Laboratory of Animal Embryo and Molecular Breeding,Hubei Academy of Agricultural Sciences,Wuhan 430064,China)
Abstract: By conducting PCR detection, genome sequencing, and evolutionary analysis on clinical waterfowl parvovirus, the genotype of the isolated strain was determined.Further selection pressure and gene recombination analysis were conducted on the genome of the isolated strains using Paml and RDP software to determine the evolutionary trend of the isolated strains. The results showed that the three strains of waterfowl parvovirus isolated belonged to the novel goose parvovirus, named YiCH strain, AnQ strain, and XiY strain, with genome lengths of 5 056, 5 056, and 5 068 bp, respectively. The three isolates had the closest phylogenetic relationship with the novel goose parvovirus M15 strain, with 6 base insertions at positions 191~196 and 4 878~4 883 in the ITR region at both ends of the XiY strain. Selection pressure analysis was conducted on the isolated strains, and three positive selection sites were detected for the VP protein of the isolated strains, namely 116Q, 261A, and 485S. Recombination analysis showed that both the YiCH and AnQ strains exhibited recombination phenomenon, with XiY strain as the main parent strain and GPV 06-0329 strain as the secondary parent strain.
Key words: waterfowl parvovirus; novel goose parvovirus; isolation and identification; select pressure; gene recombination
水禽細小病毒(Waterfowl parvovirus,WPV)主要包括鹅细小病毒(Goose parvovirus,GPV)、番鸭细小病毒(Mute duck parvovirus,MDPV)和鸭细小病毒(Duck parvovirus,DPV)。该病毒对鹅、番鸭、鸭等水禽影响较大,在中国、法国、匈牙利、德国、以色列、美国等都有发病报道。病毒呈球形或六角形,无囊膜,基因组为单链、线性DNA,长度约5 kb。病毒基因组编码VP1、VP2和VP3 3种衣壳蛋白,其中VP3蛋白是主要结构蛋白,能诱导机体产生中和抗体。该病毒不仅能感染雏鹅或雏番鸭,而且引起麻鸭等水禽发病,导致短喙和生长迟缓[1]。对雏鸭的危害严重,死亡率超过60%。湖北省农业科学院畜牧兽医研究所禽病实验室从多地送检的病死鸭组织中检测出WPV。临床发病日龄为1月龄左右,鸭喙发育不良,表现为上喙短、下喙长、舌头异常突出。解剖可见肝脏轻微出血,其他组织无肉眼可见病变。
鹅细小病毒最早由中国专家方定一[2]于1962年使用鹅胚分离获得,随后世界各国相继报道了该病[3,4]。近年来,水禽细小病毒不断出现变异和重组,病原宿主谱逐渐扩大,临床症状也出现变化。由最初的下痢、肝脏出血、肠道栓塞等症状转变为短喙和生长迟缓[5]等症状,在生产上造成重大经济损失。早在1970年,法国骡鸭出现了大量短喙和侏儒综合征(Short beak and dwarfism syndrome,SBDS)病例[6]。2015年以来,中国也出现了一种新型鹅细小病毒(NGPV),导致侏儒综合征[7]。国内有学者认为,鹅源细小病毒的变种可能是引起鸭侏儒综合征的主要病原[8-10]。侏儒综合征的病原对多种樱桃谷鸭和骡鸭具有高致病性,全基因组序列之间没有显著差异[11],但感染其他品种鸭后却不一定出现侏儒综合征症状[12]。鸭SBDS的病原体与GPV密切相关,与目前已知的所有禽源细小病毒不同,应该是一种新型鸭细小病毒(NDPV)[13]。国内外学者对GPV的基因变异、基因重组等开展了相关研究,发现了SBDS是由GPV变异株引起,GPV通过基因重组等,由鹅向鸭进行适应性演化,造成鸭发病,生产性能下降。引起基因变异的主要原因包括选择压力和基因重组2个方面。本研究通过对湖北省、安徽省等地区鸭源细小病毒的分离鉴定,分析GPV的流行趋势,病毒变异情况,并通过生物信息学方法对病毒进行选择压力、基因重组等方面分析,旨为WPV的演化趋势以及防控提供参考。
1 材料与方法
1.1 材料
1.1.1 样品 疑似GPV感染的病料来自湖北省、安徽省3个發病鸭场,由湖北省农业科学院畜牧兽医研究所禽病实验室保存。发病鸭群日龄在21~42 d,死亡率在5%~12%,使用氟苯尼考、多西环素等抗生素治疗无明显效果。临床症状以拉稀、死亡和短喙为主。解剖病变主要为肝脏轻度出血、脾脏萎缩、肾脏出血。初步诊断为细小病毒感染。
1.1.2 试剂 DNA提取试剂盒为AxyPrep体液病毒DNA/RNA小量试剂盒;PCR试剂为Vazyme公司的2×Rapid Taq Master Mix;DL2000 DNA Marker、pMD18-T载体、DH5α感受态细胞购自宝日医生物技术(北京)有限公司。Gel Extraction Kit琼脂糖凝胶回收试剂盒、Plasmid Mini Kit I小量提取试剂盒购自奥美嘉生物技术公司。
1.1.3 引物设计 根据GenBank数据库中公布的鸭、番鸭、鹅细小病毒全基因组序列,通过Lasergene软件中 MegAlign程序进行多重比对,使用Primer premier 6.0软件在保守区域设计了1对检测引物和2对测序引物,扩增Rep基因和VP基因,反向末端重复序列(ITR)片段的扩增参考文献[14]的方法。引物由生工生物工程(上海)股份有限公司合成。引物序列信息见表1。
1.2 方法
1.2.1 病料的处理 将收集的病鸭肝、脾、肠道等疑似病变组织按照1∶5(V∶V)的比例加入pH 7.4磷酸盐缓冲液(PBS)进行组织匀浆,然后置于-20 ℃冻融3次,6 000 r/min离心15 min,经0.22 μm过滤器过滤除菌后,置于-80 ℃冰箱备用。
1.2.2 核酸的提取和PCR扩增 按照“1.2.1”项方法处理样品,使用AxyPrep体液病毒DNA/RNA小量试剂盒,按照说明书的方法提取DNA,然后以PV-F/R引物对3个不同地区的鸭病料组织进行PCR鉴定。PCR反应体系:Master Mix 12.5 μL、ddH2O 8.5 μL、上下游引物各1.0 μL、DNA模板2.0 μL,合计25.0 μL。按以下条件进行PCR反应:95 ℃预变性5 min;95 ℃变性15 s、52 ℃退火15 s、72 ℃延伸15 s,共35个循环;72 ℃延伸5 min。PCR产物用1.0%琼脂糖凝胶进行电泳,观察是否出现阳性条带。
1.2.3 测序片段扩增及序列测定 以“1.2.2”项中提取的DNA为模板,扩增ITR区域、Rep基因、VP基因。PCR反应体系:Master Mix 25.0 μL、ddH2O 17.0 μL、上下游引物各2.0 μL、DNA模板4.0 μL,合计50.0 μL。按以下条件进行PCR反应:95 ℃预变性5 min;95 ℃变性20 s、52 ℃退火20 s、72 ℃延伸45 s,共35个循环;72 ℃延伸10 min。由于ITR区域具有复杂的发卡结构,按以下条件进行PCR反应:95 ℃预变性5 min;95 ℃变性20 s、55 ℃退火30 s、72 ℃延伸20 s,共35个循环。PCR产物用1.0%琼脂糖凝胶进行电泳,观察是否出现阳性条带。将PCR产物回收纯化后,连接至pMD18-T载体,通过DH5α感受态细胞转化,经氨苄西林筛选和菌液PCR鉴定后,取阳性菌液送至生工生物工程(上海)股份有限公司测序。
1.2.4 测序结果的组装与拼接 使用Lasergen软件SeqMan程序进行组装拼接,以FM株为参考株,得到全基因组序列。根据病料来源,分别命名为YiCH株、AnQ株和XiY株。
1.2.5 进化分析 登录Genbank数据库,选择背景清晰、不同时空分布且具有代表性的GPV、MDPV和NGPV全基因组序列,合计31条。使用MEGA软件,选择最大似然法(Maximum likelihood,ML)建立进化树,自展值(Bootstrap method)设为500。参考毒株信息见表2。
1.2.6 选择压力分析 按文献[15]的方法,使用Paml软件中Codeml程序,分析Rep蛋白和VP蛋白所承受的正选择压力位点。结果通过Datamonkey网站(http://www.datamonkey.org/)在线验证,选择位点模式Episodic下MEME(Mixed
1.2.7 重组分析 使用ClustalX、MEGA等软件,将参考序列和分离株核酸序列进行多重比对后,使用RDP软件[16],采用RDP、GENECONV、Chimaera、MaxChi、BootScan、SiScan和3Seq 7种检测模式,对分离株的全基因组序列进行重组分析,选择7种检测方法均支持的毒株为重组毒株。
2 结果与分析
2.1 检测引物PCR扩增结果
3个样品使用检测引物PV-F/R进行PCR扩增,1.0%琼脂糖凝胶电泳检测,均在800 bp处出现电泳条带(图1),条带大小与预期相符,表明被检测组织中含有细小病毒。
2.2 测序片段的扩增
使用引物Rep-F/R扩增Rep基因,产物大小为 2 000 bp;使用引物VP-F/R扩增VP基因,产物大小为2 400 bp。PCR扩增后,1.0%琼脂糖凝胶电泳检测,出现预期大小的条带(图2)。ITR区域分段扩增后,分别得到188 bp短片段和476 bp 长片段2个条带(图3)。这些产物将用于后续的测序分析。
2.3 测序结果的组装与拼接
将3株病毒的测序结果使用Lasergen软件SeqMan组装拼接后,得到全基因组序列。YiCH株、AnQ株和XiY株全基因组大小分别为5 056、5 056、5 068 bp。其中XiY株在ITR区域191~196位、 4 878~4 883位均有6个碱基插入(图4)。
2.4 进化分析
参照文献[17]对Rep基因、VP3基因进行分子进化和系统发育分析,碱基替代方法选用Maximum likelihood,系统发育检验采用Bootstrap方式,替代次数为500次。结果显示,根据Rep基因绘制的系统发生树如图5所示,AnQ株、YiCH株和XiY株处于同一进化分支,且与新型鹅细小病毒M15株亲缘关系较近。基于VP3基因绘制的系统发生树如图6所示,3个分离株同样处于同一分支,与sdlc01株、QH15株、SDLY1512株亲缘关系较近。结果表明,AnQ株、YiCH株和XiY株属于新型鹅细小病毒。
2.5 分离株选择压力分析
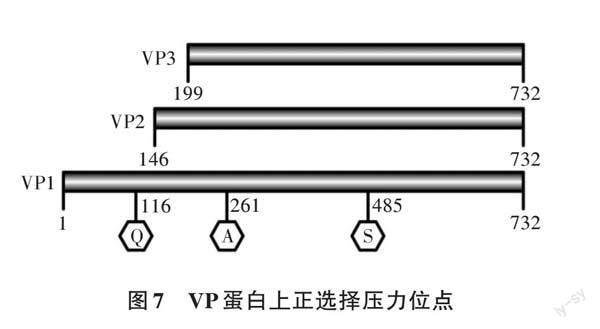
在Rep蛋白上未检测出正选择压力位点,在VP蛋白上检测出3个正选择压力位点(表3、图7),分别位于VP1蛋白第116位Q氨基酸、261位A氨基酸和485位S氨基酸,其中261A、485S同时也位于VP2和VP3蛋白上。進一步说明鹅细小病毒VP1、VP2和VP3 3个蛋白都受到正选择压力作用,更容易产生适应性进化,推测也是跨种传播的主要原因。
2.6 分离株重组分析
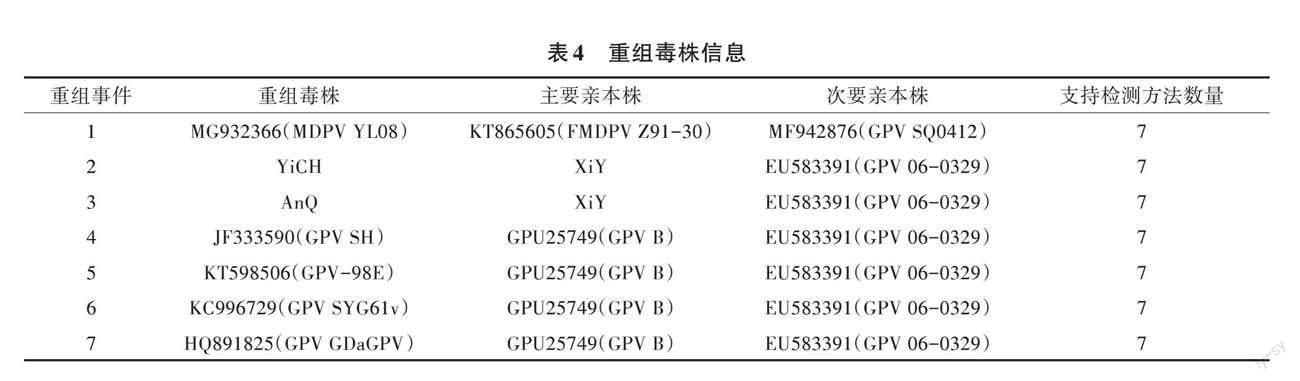
参照文献[18]的方法,对Rep基因、VP基因以及ITR区域进行重组分析,其中ITR区域、Rep基因无检出,VP基因共检测出14个重组事件。其中同时支持7种检测方法的重组事件有7个(表4),由分离株参与的重组事件有2个,分别为重组事件2和重组事件3。对重组事件2和重组事件3采用RDP方法进行验证,YiCH株和AnQ株在VP区域111~541 bp处检测到重组信息(图8)。结果显示,YiCH株、AnQ株都是重组毒株,以XiY株为主要亲本株、GPV 06-0329株为次要亲本株重组而成。
3 讨论
经典GPV感染主要引起肠道病变。近年来,随着病毒的变异和重组,出现以鸭短喙、瘫痪、侏儒为主要症状的新型鹅细小病毒,给中国的水禽养殖造成严重的影响。MDPV和GPV在宿主谱、致病性方面也存在显著差异。GPV既能感染鹅,也能感染番鸭,而MDPV只能感染番鸭,不能感染鹅,且早期MDPV和GPV在血清学方面无交叉反应[19]。随着NDPV的出现,水禽细小病毒出现跳跃式进化,衣壳蛋白上2个氨基酸发生变化,可能是引起NDPV宿主转移的原因。自然选择压力驱动水禽细小病毒不断产生适应性改变;构成NDPV的基因组中,阳性选择位点主要位于结构蛋白上[20]。疫苗加速病毒的变异频率。
细小病毒重组现象比较普遍,经典MDPV在演化过程中,强毒株和疫苗株在感染同一宿主时发生重组,导致新的毒株产生,MDPV ZW株就是由经典的MDPV和GPV疫苗株重组产生的[21]。2种不同基因型的GPV在自然界中容易进行重组,并产生新的变异毒株,如SDLY1602 被证明是一种重组毒株,是由GPV疫苗株82-0321v为主要亲本和GPV野生毒株GDaGPV为次要亲本重组而成,因此NGPV常被认为是GPV的一个分支[22]。而在福建骡鸭中发现一株重组M15株,是由GPV和MDPV组成的天然重组体[23]。但番鸭细小病毒也可引起SBDS,2008年福建省骡鸭养殖场和台湾省白鸭养殖场发现了由MDPV感染引起的SBDS临床病例[24]。
不同物种之间细小病毒重组现象也常有发生[25-27],本研究中YiCH株就是以XiY株为主要亲本,GPV 06-0329株为次要亲本重组而成。重组区域主要位于结构蛋白区域,而非结构蛋白区域和两端ITR区域未检测出重组事件。GPV 06-0329株于2006年从台湾省鹅中分离,接种1日龄番鸭能100%引起死亡[28],属于强毒株。随后,安徽省[29]、广东省[30]等地相继分离到台湾省06-0329株。而主要亲本株XiY株则是近年在湖北省分离得到,二者重组后得到YiCH株和AnQ株,说明在同一地区,细小病毒产生重组是普遍现象,且随着引种和活禽交易,病毒易向周边地区扩散。
GPV通过自然选择压力和基因重组,产生适应性进化,可以在自然界中逐渐演变为NGPV。阳性选择压力主要作用于VP结构蛋白,通过改变蛋白质结构进行宿主迁移。基因重组则是水禽细小病毒进化的另一种方式,重组的主要区域位于VP蛋白区域,GPV、MDPV参与重组过程,并通过重组产生新的基因型,从而扩大宿主范围。
参考文献:
[1] SOLIMAN M A, ERFAN A M, SAMY M, et al. Detection of novel goose parvovirus disease associated with short beak and dwarfism syndrome in commercial ducks[J]. Animals, 2020,10(10): 1833.
[2] 方定一. “小鵝瘟”的介绍[J]. 中国畜牧兽医, 1962(8): 19-20.
[3] 傅宏庆, 姚志兰, 王永娟, 等. 鸭短喙型小鹅瘟病毒SBDS-GPV JS01株的分离鉴定及其对雏鸭致病性的研究[J]. 中国兽医科学, 2020,50(10): 1286-1293.
[4] ISIDAN H, TURAN T, ATASOY M O, et al. Molecular analysis of goose parvovirus field strains from a Derzsys disease outbreak reveals local European-associated variants[J]. Archives of virology, 2021,166(7): 1931-1942.
[5] WAN C, LIU R, CHEN C, et al. Novel goose parvovirus in domestic Linwu sheldrakes with short beak and dwarfism syndrome, China[J]. Transboundary and emerging diseases, 2019,66(5): 1834-1839.
[6] PALYA V, ZOLNAI A, BENYEDA Z, et al. Short beak and dwarfism syndrome of mule duck is caused by a distinct lineage of goose parvovirus[J]. Avian pathology, 2009,38(2): 175-180.
[7] LI D L, ZHANG L D, CHEN S H, et al. Detection and molecular characterization of two genotypes of goose parvoviruses isolated from growing period geese and cherry valley ducks in China[J]. Avian diseases, 2019,63(3): 411-419.
[8] YU K X, MA X L, SHENG Z Z, et al. Identification of goose-origin parvovirus as a cause of newly emerging beak atrophy and dwarfism syndrome in ducklings[J]. Journal of clinical microbiology, 2016,54(8): 1999-2007.
[9] ZHAO G W, HUANG T T, WU D, et al. Diagnosis and characterization of duck beak atrophy and dwarfism syndrome in Chongqing of China[J]. Veterinary research forum, 2019,10(2): 169-172.
[10] CHEN S L, WANG S, CHENG X X, et al. Isolation and characterization of a distinct duck-origin goose parvovirus causing an outbreak of duckling short beak and dwarfism syndrome in China[J]. Archives of virology, 2016,161(9): 2407-2416.
[11] BIAN G Z, MA H B, LUO M P, et al. Identification and genomic analysis of two novel duck-origin GPV-related parvovirus in China[J]. BMC veterinary research, 2019,15(1): 1833-1839.
[12] XIAO S F, CHEN S L, CHENG X X, et al. The newly emerging duck-origin goose parvovirus in China exhibits a wide range of pathogenicity to main domesticated waterfowl[J]. Veterinary microbiology, 2017,203: 252-256.
[13] LI C F,LI Q,CHEN Z Y,et al. Novel duck parvovirus identified in cherry valley ducks (anas platyrhynchos domesticus), China[J]. Infection, genetics and evolution, 2016,44: 278-280.
[14] 贾婧宇,秘清灵,李勇霖,等. 3株樱桃谷鸭源新型鹅细小病毒的分离及分子特征解析[J]. 中国动物传染病学报,2022,30(2):7-13.
[15] 汪宏才, 温国元, 张腾飞, 等. 新城疫病毒阳性选择区域氨基酸与毒力相关性研究[J]. 中国家禽, 2018,40(21): 26-30.
[16] MARTIN D P, MURRELL B, GOLDEN M, et al. RDP4: Detection and analysis of recombination patterns in virus genomes[J]. Virus evolution, 2015,1(1): vev003.
[17] 傅秋玲,程龍飞, 万春和, 等. 半番鸭胚中鹅细小病毒的分离鉴定及其基因组分子特征分析[J]. 畜牧兽医学报,2017,48(11): 2148-2156.
[18] MARTIN D P, VARSANI A, ROUMAGNAC P, et al. RDP5:A computer program for analyzing recombination in, and removing signals of recombination from, nucleotide sequence datasets[J]. Virus evolution, 2021,7(1): 1-3.
[19] 程由铨, 胡奇林, 陈少莺, 等. 番鸭细小病毒和鹅细小病毒生化及基因组特性的比较[J]. 中国兽医学报, 2001(05): 429-433.
[20] FAN W, SUN Z, SHEN T, et al. Analysis of evolutionary processes of species jump in waterfowl parvovirus[J]. Frontiers in microbiology, 2017,8: 421.
[21] WANG J, LING J, WANG Z, et al. Molecular characterization of a novel Muscovy duck parvovirus isolate: Evidence of recombination between classical MDPV and goose parvovirus strains[J]. BMC veterinary research, 2017,13(1): 1238-1246.
[22] LI P, LIN S, ZHANG R, et al. Isolation and characterization of novel goose parvovirus-related virus reveal the evolution of waterfowl parvovirus[J]. Transboundary and emerging diseases, 2018,65(2): 284-295.
[23] WANG S, CHENG X X, CHEN S L, et al. Identification of a novel goose parvovirus (GPV) recombinant associated with short beak and dwarfism syndrome in Mainland China, 2015[J]. Infection, genetics and evolution, 2016,41: 289-291.
[24] FU Q L, HUANG Y, WAN C H, et al. Genomic and pathogenic analysis of a Muscovy duck parvovirus strain causing short beak and dwarfism syndrome without tongue protrusion[J]. Research in veterinary science, 2017,115: 393-400.
[25] WANG S, CHENG X X, CHEN S Y, et al. Genetic characterization of a potentially novel goose parvovirus circulating in Muscovy duck flocks in Fujian Province, China[J]. The Journal of veterinary medical science, 2013,75(8): 1127-1130.
[26] ZHU Y, ZHOU Z, HUANG Y, et al. Identification of a recombinant Muscovy duck parvovirus (MDPV) in Shanghai, China[J]. Veterinary microbiology, 2014,174(3-4): 560-564.
[27] SHEN H, ZHANG W, WANG H, et al. Identification of recombination between Muscovy duck parvovirus and goose parvovirus structural protein genes[J]. Archives of virology, 2015,160(10): 2617-2621.
[28] SHIEN J H, WANG Y S, CHEN C H, et al. Identification of sequence changes in live attenuated goose parvovirus vaccine strains developed in Asia and Europe[J]. Avian pathology, 2008,37(5): 499-505.
[29] 王 浩, 田先举, 张 烁, 等. 2株鹅细小病毒全基因组特征分析[J]. 畜牧兽医学报, 2013,44(4): 602-609.
[30] 黄冠雄. 2015-2016年广东省小鹅瘟病毒分子流行病学调查[D]. 广州:仲恺农业工程学院, 2017.
