TaqMan-MGB探针实时荧光定量PCR快速检测光滑念珠菌
2016-03-10杨靖娴邵冬华梁国威
郭 毅,杨靖娴,邵冬华,刘 静,梁国威
(北京大学航天临床医学院 检验科,北京100049)
TaqMan-MGB探针实时荧光定量PCR快速检测光滑念珠菌
郭毅,杨靖娴,邵冬华,刘静,梁国威*
(北京大学航天临床医学院 检验科,北京100049)
摘要:目的采用TaqMan-MGB探针作为检测信号,建立快速检测光滑念珠菌的实时荧光定量PCR方法。方法以光滑念珠菌核糖体RNA编码基因(rDNA)中的内转录间隔区2(ITS2)作为靶基因,设计特异性引物和TaqMan-MGB探针。在梯度光滑念珠菌纯菌液标本中评价所建方法灵敏度和重复性;在102株光滑念珠菌临床分离株、非光滑念珠菌的其他4种念珠菌临床分离株、7种细菌临床分离株和人基因组的DNA中评价所建方法特异度;并在模拟光滑念珠菌血症的血标本中初步探讨其临床应用价值。结果所建方法对光滑念珠菌纯菌液的检测下限为101CFU/ml,相应的批内、批间变异系数(CV)分别为1.68%和2.84%。对102株光滑念珠菌临床分离株检出率为100%;对非光滑念珠菌无交叉反应,显示该方法特异性为100%。对模拟光滑念珠菌血症血标本的最低检测下限为101CFU/ml。结论所建方法具有特异性和灵敏度高、重复性好的特性,适于临床上光滑念珠菌血症的快速检测。
关键词:光滑念珠菌;荧光定量PCR;内转录间隔区2(ITS 2) ;TaqMan-MGB探针;血流感染
(ChinJLabDiagn,2016,20:0178)
目前念珠菌中光滑念珠菌的临床分离率仅次于白色念珠菌,可达到19%-26.7%[1,2]。由于光滑念珠菌对唑类抗真菌剂广泛耐药[3,4],而且对棘白菌素和多烯类药物不敏感的菌株也呈逐渐增多趋势[5],所以早期确诊由光滑念珠菌引起血流感染对临床诊疗具有重要的指导意义。
文献显示,多数PCR方法侧重于针对念珠菌属或者白色念珠菌菌种的检测[6],但对于光滑念珠菌的检测方法则较为少见,而采用TaqMan-MGB探针作为实时荧光定量PCR检测信号检测光滑念珠菌的方法未见报道。
因此,本研究的目的是以光滑念珠菌核糖体RNA编码基因(rDNA)中的内转录间隔区2(ITS2)作为靶基因,设计特异性引物和TaqMan-MGB探针,建立光滑念珠菌的实时荧光定量PCR检测方法,并对该方法的应用价值进行初步评价。
1材料与方法
1.1实验材料(1)标准菌株:光滑念珠菌 (ATCC 2001)1株,购自中国普通微生物菌种保藏中心;(2)临床分离菌株:来自航天中心医院微生物室菌种保存库,包括102株光滑念珠菌、135株白色念珠菌、61株热带念珠菌、24株近平滑念珠菌、12株克柔念珠菌、24株大肠埃希菌、24株肺炎克雷伯菌、24株变形杆菌、24株粘质沙雷菌、24株铜绿假单胞菌、24株鲍曼不动杆菌以及24株金黄色葡萄球菌。以上菌株经微生物鉴定仪(VitekⅡ,生物梅里埃公司,法国)鉴定确认;(3)血液标本10例,来自健康志愿者。
1.2临床分离菌株及人基因组DNA提取(1)菌株DNA提取:采用热休克法提取菌株DNA。具体步骤:菌悬液100℃加热15 min,-80℃冷冻10 min,重复3次,12 000 r/min 离心15 min,上清即为菌株DNA粗提液,-80℃保存备用。(2)人基因组DNA提取:按照血液基因组DNA提取试剂盒(天根生化科技有限公司,中国)操作步骤,从健康志愿者血液标本中提取人基因组DNA,-80℃保存备用。
1.3光滑念珠菌梯度菌液配制和DNA提取(1)光滑念珠菌计数:采用改良牛鲍式血球计数板,按标准操作规程计数。(2)梯度菌液配制和提取:将光滑念珠菌(ATCC2001)计数后的107CFU/ml的菌液进行10倍梯度稀释,取稀释后的菌液各100 μl,分别注入900 μl DNA提取液Ⅰ中(中山大学达安基因股份有限公司.中国)。按上述热休克法提取DNA后,-80℃保存备用。
1.4光滑念珠菌梯度模拟感染血标本的配制及其DNA提取(1)将光滑念珠菌(ATCC2001)计数后的107CFU/ml的菌液进行10倍梯度稀释,取稀释后的菌液各100 μl,分别注入900 μl EDTA抗凝的健康人血液中,制成梯度模拟感染血标本。(2)DNA提取:上述标本中加入500 μl细胞裂解液CL(天根生化科技有限公司,中国),室温10 min,12 000 r/min 离心15 min后弃上清;之后向沉淀加入1 ml Polaris细胞裂解液[7](500 mM Na2CO3和1% SDS),室温5 min,12 000 r/min离心15 min,弃上清。400 μl DNA提取液Ⅰ重悬沉淀后,按热休克法提取DNA,上清液经过滤柱,50 μl 洗脱,-80℃ 保存备用。
1.5引物和探针设计在Pubmed的Nucleotide数据库中检索ATCC 2001光滑念珠菌ITS2序列(GenBank:JQ070075.1),通过比对筛选种内变异率小和种间特异性高的序列,使用Primer 5.0软件设计特异性引物和TaqMan-MGB探针。上游引物:5’- GCC ATA TCA GTA TGT GGG ACA CG-3’、下游引物5’-GTA TTA ACC CCC GCC GCT-3’,TaqMan-MGB探针:5’-VIC-CAA ACG AGC AGC AGA TTA AT-NFQMGB-3’,扩增片段长度为80 bp。
1.6实时荧光定量PCR反应体系与扩增条件PCR反应体系为20 μl,包括模板DNA 2 μl,上下游引物各2 μl(0.5 μmol/L)(北京赛百盛基因技术有限公司,中国),TaqMan-MGB探针1 μl(0.25 μmol/L)(ABI公司,美国),2.5×Master Mix 8 μl和20×Probe Enhancer solution 1 μl(天根生化科技有限公司,中国),水1 μl。循环参数为95 ℃ 2 min,95 ℃ 15 s 和60 ℃ 1 min,进行45个循环。循环阈值(Ct值)设定在0.03,基线设定为自动设定,测定仪器为ABI 7500荧光定量PCR仪(ABI公司,美国)。

2结果
2.1所建方法检测灵敏度评价以梯度为106-101 CFU/ml的光滑念珠菌菌液提取的DNA为模板,对各水平DNA同时进行10次PCR反应,显示皆有特异性扩增曲线,其中101 CFU/ml 菌液所对应的Ct值为34.89±0.59。显示本方法最低可从101 CFU/ml的菌液中检出光滑念珠菌,见图1。浓度为106-101 CFU/ml纯菌液标本的PCR扩增标准曲线的斜率=-3.163,R2=0.990。
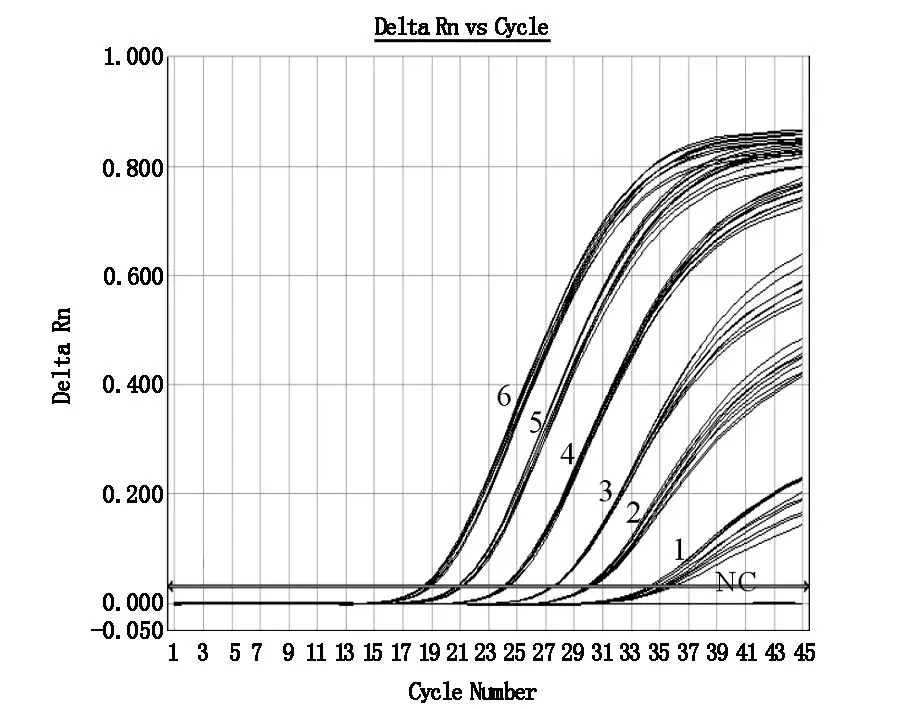
图1光滑念珠菌标准菌株纯菌液梯度PCR扩增曲线图中6-1代表浓度为106-101 CFU/ml光滑念珠菌纯菌液;NC代表阴性对照。
2.2所建方法检测重复性评价对106-101CFU/ml 的光滑念珠菌各梯度菌液的DNA分别进行10次批内和批间的重复性检测。批内CV值的变化范围为0.21%-1.68%,其中101CFU/ml对应的V值最大(1.68%)。批间CV值的变化范围为1.15%-2.84%,101CFU/ml浓度对应的CV值最大(2.84%),详见表1。

表1 所建方法检测各梯度菌液DNA的批内、批间Ct值
2.3检测方法特异度评价采用所建方法检测不同来源DNA(具体目录见本文“材料与方法 1.实验材料”部分),以评价其特异性。其中102株光滑念珠菌临床分离菌株全部检出(检出率为100%)。其它非光滑念珠菌来源的DNA检测结果均为阴性,显示该方法对于检测光滑念珠菌的特异度为100%。
2.4梯度念珠菌模拟感染血标本检测情况对含菌量为106-101CFU/ml的光滑念珠菌模拟感染血标本DNA进行检测(平行重复3次),结果显示对于血流感染标本的检测下限为101CFU/ml(Ct值为37.87±1.05),见图2。含菌量106-101CFU/ml模拟感染血标本的PCR标准曲线的斜率=-3.26,R2=0.998。

图2光滑念珠菌标准菌株模拟感染血标本扩增曲线图中6-1代表含菌量为106-101CFU/ml光滑念珠菌模拟感染血标本,NC代表阴性对照。
3讨论
本研究选择光滑念珠菌rDNA基因中ITS2区作为扩增靶序列,设计特异性引物和TaqMan-MGB探针,成功建立了实时荧光定量PCR检测光滑念珠菌的方法。该方法对于光滑念珠菌纯菌液的检出下限为101CFU/ml,相应批内和批间变异率分别为1.68%和2.84%。特异性评价显示,102株光滑念珠菌临床分离株检出率为100%,且与研究所纳入的其它念珠菌、细菌以及人基因组DNA标本无交叉反应。另外,通过对模拟感染血标本的检测,证实该方法对于血流感染中光滑念珠菌的检测下限也可达到101CFU/ml。
ITS2序列位于真核生物5.8S和28S rDNA之间,在种间高度特异[8],可作为设计种特异性引物和探针的位点。本研究以光滑念珠菌 ATCC 2001菌株的ITS2序列作为比对标准序列。首先,在Pubmed数据库进行种间序列比对(nucleotide blast),显示光滑念珠菌ITS2序列与人基因组、细菌库、真菌库、病毒库、衣原体库、支原体库、立克次氏体库皆无显著重叠序列,特别是对于同光滑念珠菌表型、生化反应等特性相似的C.nivariensis和C.bracarensis也能作出很好的区分。实际检测结果也显示本方法对光滑念珠菌以外的各种生物来源DNA皆无显著性扩增曲线。其次,进行光滑念珠菌种内ITS2序列比对,筛选种内变异少的序列区间作为引物和探针的靶序列。通过对数据库中的116条光滑念珠菌ITS2参考序列的比对,显示本研究设计的引物和探针序列中均无种内变异,据此计算的光滑念珠菌理论检出率为100%。我们采用所建方法对102株光滑念珠菌临床分离株进行了检测,检出率为100%,未出现漏检的情况。
TaqMan-MGB探针是在TaqMan探针3′端增加MGB (Minor Groove Binder,小沟结合物)修饰的一种水解探针,其特性包括:(1)显著提高探针与模板结合的Tm值,15个碱基的探针可以提高 Tm 达18℃,允许设计更短的探针;(2)探针3′端-1 ~ -7位碱基内的单一碱基错配可抑制探针与模板的结合,显著提高探针特异性;(3)荧光本底低,提高信噪比[9]。利用上述特性,本研究设计的TaqMan-MGB探针长度为20 bp,除了避开了光滑念珠菌ITS2序列种内碱基变异外,还同时增加了光滑念珠菌种间区分能力,提高了检测特异度。在光滑念珠菌纯菌液中,本方法的检测下限可达到101CFU/ml,优于Foongladda 等在18S rDNA上设计引物和采用普通水解探针的检测结果(525 CFU/ml)[10]。而且浓度为101CFU/ml光滑念珠菌纯菌液的批内、批间的CV值分别为1.68%和2.84%,重复性良好,可满足常规临床要求(CV值<5%)。
血液中的血细胞成分是否完全去除可显著影响PCR检测的灵敏度。如有报道称血红素会造成DNA聚合酶的解离,从而影响PCR反应的进行[11];而白细胞的残留也会使得大量人基因组DNA释放到样本中,干扰引物和探针与模板,特别是与低浓度模板的配对。本实验借鉴Loonen AJ[7]等人所采用的细胞裂解方法,结合实验结果等具体情况,改良出一套适合本实验室的红细胞和白细胞去除流程,详见“实验方法”中的(4)。实验显示,我们对光滑念珠菌模拟感染的DNA检测的灵敏度可达到101CFU/ml,优于Lehmann LE[12]等人采用高分辨溶解曲线原理所建立检测方法(含菌量为102CFU/ml时,检出率为100%;含菌量为30 CFU/ml时,检出率为75%),也优于Roche公司SeptiFast试剂盒对于血流中光滑念珠菌的检测下限(102CFU/ml)[13]。另外,与Loffler 等[14]所述红、白细胞裂解法相比,还具有耗时短、成本低、试剂配制简易等优点。
综上所述,本研究所述的基于光滑念珠菌ITS2序列,采用TaqMan-MGB探针作为检测信号的荧光定量PCR方法,具有检测灵敏度和特异性高,重复性好以及检测时间短等优点,显著优于临床常规的血培养方法,适于临床光滑念珠引起的血流感染的快速鉴定。
参考文献:
[1]Ericsson J,Chryssanthou E,Klingspor L,et al.Candidaemia in Sweden:a nationwide prospective observational survey[J].Clin Microbiol Infect,2013,19(4):E218.
[2]Pfaller M,Neofytos D,Diekema D,et al.Epidemiology and outcomes of candidemia in 3648 patients:data from the Prospective Antifungal Therapy (PATH Alliance(R)) registry,2004-2008[J].Diagn Microbiol Infect Dis,2012,74(4):323.
[3]Wisplinghoff H,Ebbers J,Geurtz L,et al.Nosocomial bloodstream infections due to Candida spp.in the USA:species distribution,clinical features and antifungal susceptibilities[J].Int J Antimicrob Agents,2014,43(1):78.
[4]Taj-Aldeen SJ,Kolecka A,Boesten R,et al.Epidemiology of candidemia in Qatar,the Middle East:performance of MALDI-TOF MS for the identification of Candida species,species distribution,outcome,and susceptibility pattern[J].Infection,2014,42(2):393.
[5]Dudiuk C,Gamarra S,Leonardeli F,et al.Set of classical PCRs for detection of mutations in Candida glabrata FKS genes linked with echinocandin resistance.J Clin Microbiol,2014,52(7):2609.
[6]Schell WA,Benton JL,Smith PB,et al.Evaluation of a digital microfluidic real-time PCR platform to detect DNA of Candida albicans in blood[J].Eur J Clin Microbiol Infect Dis,2012,31(9):2237.
[7]Loonen AJ,Bos MP,van Meerbergen B,et al.Comparison of pathogen DNA isolation methods from large volumes of whole blood to improve molecular diagnosis of bloodstream infections[J].PLoS One,2013,8(8):e72349.
[8]Lott TJ,Kuykendall RJ,Reiss E.Nucleotide sequence analysis of the 5.8S rDNA and adjacent ITS2 region of Candida albicans and related species[J].Yeast,1993,9:1199.
[9]Kutyavin IV,Afonina IA,Mills A,et al.3’-minor groove binder-DNA probes increase sequence specificity at PCR extension temperatures[J].Nucleic Acids Res,2000,28:655.
[10]Foongladda S,Mongkol N,Petlum P,et al.Multi-probe real-time PCR identification of four common Candida species in blood culture broth[J].Mycopathologia,2014,177(5-6):251.
[11]Gosiewski T,Brzychczy-Wloch M,Pietrzyk A,et al.Evaluation of the activity of thermostable DNA polymerases in the presence of heme,as a key inhibitor in the real time PCR method in diagnostics of sepsis[J].Acta Biochim Pol,2013,60(4):603.
[12]Lehmann LE,Hunfeld KP,Emrich T,et al.A multiplex real-time PCR assay for rapid detection and differentiation of 25 bacterial and fungal pathogens from whole blood samples[J].Med Microbiol Immunol,2008,197(3):313.
[13]Westh H,Lisby G,Breysse F,et al.Multiplex real-time PCR and blood culture for identification of bloodstream pathogens in patients with suspected sepsis[J].Clin Microbiol Infect,2009,15(6):544.
[14]Loffler J,Hebart H,Schumacher U,et al.Comparison of different methods for extraction of DNA of fungal pathogens from cultures and blood[J].J Clin Microbiol,1997,35(12):3311.
Rapid detection ofCandidaglabrataby using a TaqMan-MGB probe-based quantitative real-time PCR assayGUOYi,YANGJing-xian,SHAODong-hua,etal.(DepartmentofClinicalLaboratory,PekingUniversityAerospaceSchoolofClinicalMedicine,Beijing100049,China)
Abstract:ObjectiveTo develop a TaqMan-MGB probe-based quantitative real-time PCR assay for rapid identification ofCandidaglabrata(C.glabrata).MethodsThe internal transcribed spacer 2 (ITS2) on ribosomal DNA (rDNA) ofC.glabratawas used as a target for designing specific primers and TaqMan-MGB probe.The sensitivity and reproducibility of the present method were evaluated with serial diluted samples ofC.glabrata.The specificity of the method was validated in DNA samples from clinical isolates of 102 strains ofC.glabrata,4 other species of non-glabrataCandida,7 species of bacteria and human blood specimen,respectively.We also evaluated the clinical application values of the assay using simulatedC.glabratacandidemia in blood samples.ResultsThe lowest counts ofC.glabratathat the assay can measure accurately was 101CFU/ml,and its coefficients of variability (CV) of intra-assay and inter-assay were 1.68% and 2.84%,respectively.The detectable rate was 100% for 102 clinical isolates ofC.glabrata.No cross reaction by the assay were observed indicating the specificity was 100%.For simulated infection blood samples,the detection limit ofC.glabratawas 101CFU/ml by the assay.ConclusionThe newly developed method was a highly sensitive and specific assay,which could be applied to rapid diagnosis of bloodstream infections induced byC.glabrata.
Key words:Candidaglabrata;Real-time PCR;Internal Transcribed Spacer 2(ITS2);TaqMan-MGB probe;Bloodstream infections
(收稿日期:2015-11-10)
文献标识码:A
中图分类号:Q78
文章编号:1007-4287(2016)02-0178-04
*通讯作者
基金项目:北京大学航天临床医学院科研基金(YN201322)
