基于生物信息学分析白血病抑制因子受体与乳腺癌预后的相关关系
2020-09-15田甜邓飞唐金海
田甜, 邓飞, 唐金海
白血病抑制因子(leukemia inhibitory factor,LIF)是一种具有广泛活性的细胞因子。白血病因子抑制受体(leukemia inhibitory factor receptor,LIFR)又称CD118,它与糖蛋白130(glycoprotein 130,gp130)的跨膜和细胞质区域密切相关,并可以通过与gp130形成复合物以激活Janus激酶/信号转导与转录激活子(the janus kinase/signal transducers and activators of transcription, JAK/STAT)、丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)和磷脂酰肌醇3-激酶(phosphatidylinositol kinase 3-kinase,PI3Ks)信号通路[1-4]。其中JAK/STAT信号通路是多种细胞因子的共同传导途径,一旦激活,JAK就会使STAT磷酸化,当STAT的第705个酪氨酸(Tyr)被磷酸化后,STAT可以形成二聚体入核,进一步结合辅助因子和协助转录因子启动转录。JAK/STAT信号传导途径也是激活炎性反应的上游信号传导,被认为是炎性疾病的治疗目标[5-8]。同样的,JAK还可以激活含SH2结构域的酪氨酸蛋白磷酸酶2(SHP2)或PIK3通过MAPK或PI3K-AKT信号通路参与炎性反应[9]。研究结果表明,LIFR/STAT3信号传导可以赋予扩散至骨髓的乳腺癌细胞休眠表型,且该途径的失活可以原本处于休眠状态的乳腺癌细胞下调休眠、静止状态,而缺氧可以下调乳腺癌中LIFR的表达水平[10]。一项在印度的调查研究显示,乳腺癌中普遍存在LIFR表达缺失现象,76.64%的乳腺癌病例中出现了LIFR启动子的甲基化,且LIFR启动子甲基化与LIFR的蛋白表达水平下降相关[11]。LIFR还可以在MCF7细胞中充当肿瘤抑制因子,在SUM159人乳腺癌细胞中作为乳腺癌肺转移抑制因子[12-13]。本研究通过生物信息学,分析LIFR在不同癌症类型中的表达,并进一步验证LIFR与乳腺癌预后的相关关系。
1 资料与方法
1.1 LIFR表达水平分析 使用Oncomine数据库(https://www.oncomine.org/resource/login.html)[14-15]分析LIFR在多种癌症类型中癌组织和正常组织中的mRNA表达水平。TIMER(tumor immune estimation resource,cistrome.shinyapps.io/timer)是一个可以全面系统分析肿瘤与免疫相互作用的数据库,它提供了包括基因表达、临床结果、体细胞突变和体细胞拷贝数变化的在线分析[16-17],再通过TIMER数据库Diff Exp模块分析LIFR在不同癌症类型中癌组织与正常组织的mRNA表达水平。
1.2 肿瘤突变负荷与微卫星不稳定性相关性分析UCSC Xena(http://xena.ucsc.edu/)[18]下载TCGA数据库中33种癌症类型的突变数据,利用perl脚本根据肿瘤突变负荷定义计算每兆碱基中有多少个碱基发生了突变,即为每个样品的TMB值。通过spearman检验计算肿瘤突变负荷与LIFR基因表达相关性。同样通过spearman方法计算微卫星不稳定性与LIFR基因表达相关性。结果以R“fmsb”包绘制雷达图表示(***P<0.001;**P<0.01;*P<0.05)。
1.3 LIFR与乳腺癌肿瘤微环境相关性分析 利用“ESTIMATE”R包计算乳腺癌各个样品的免疫打分和基质打分,将得到的肿瘤微环境打分文件与基因表达文件取交集,通过计算spearman相关系数,以P<0.001为过滤标准将符合标准的结果利用“ggplot2”、“ggpubr”和“ggExtra”R包进行可视化。
1.4 LIFR对乳腺癌的预后分析 使用PrognoScan(http://www.abren.net/PrognoScan/)[19]评估LIFR对预后的影响,PrognoScan结果显示LIFR表达高低与患者预后指标的关系,如总生存期(overall survival, OS)、疾病特异性生存期(disease free survival,DSS)、无复发生存期(relapse free survival,RFS)和无远处转移生存期(distant metastasis free survival,DMFS)。阈值设定为Cox P<0.05。Kaplan Meierplotter数据库能够评估21种癌症类型的54k基因对预后的影响。Kaplan Meierplotter中的数据集包括乳腺癌(n=6 234),卵巢癌(n=2 190),肺癌(n=3 452)和胃癌(n=1 440)。通过Kaplan-Meier plotter(http://kmplot.com/analysis)[20]分析乳腺癌、卵巢癌、肺癌和胃癌中LIFR的表达与生存的相关性。
1.5 TISIDB数据库分析LIFR在乳腺癌免疫亚型、分子亚型中的分布情况 通过TISIDB数据库(http://cis.hku.hk/TISIDB/)[21]亚型模块分析LIFR在乳腺癌免疫亚型、分子亚型中的分布情况,结果以小提琴图表示。
1.6 乳腺癌中LIFR启动子甲基化水平分析 为进一步分析LIFR在乳腺癌中下调的可能原因,我们通过UALCAN(http://ualcan.path.uab.edu/index.html)进行分析。UALCAN可以方便地访问公开的癌症组学数据(包括TCGA和MET500),可以通过启动子甲基化评估基因表达的表观遗传调控[22]。UALCAN数据库通过β值指示DNA甲基化的水平,范围从0(未甲基化)到1(完全甲基化)。不同的β截止值来表示高甲基化[β值:0.7~0.5]或低甲基化[β值:0.3~0.25][23-24]。
1.7 LIFR在乳腺癌中的表达与作用 通过TRizol法提取细胞中总RNA,用分光光度计OD-1000+Spectrophotometer测定RNA浓度及纯度后,采用诺维赞HiScript Q RT SuperMix for qPCR试剂盒进行逆转录,采用诺维赞SYBR qPCR Master Mix (High ROX Premixed)进行RT-qPCR,对比高侵袭性乳腺癌细胞系MDA-MB-231与低侵袭性乳腺癌细胞系MCF-7中LIFR表达水平,所有操作均按照说明书完成。LIFR过表达质粒(pcDNA LIFR)及空载体质粒(pcDNA 3.1)采购自江苏凯基生物技术股份有限公司,LIFR小干扰RNA(siRNA LIFR)及对照(siRNA NC)采购自广州锐博生物科技有限公司,采用美国Invitrogen公司的Lipofectamine 3000转染试剂,进行转染后行细胞划痕实验于4倍镜下拍照,对比0、12、24、48以及72小时划痕愈合情况并统计。
1.8 预测LIFR上游miRNA 通过miRTarBase(http://mirtarbase.mbc.nctu.edu.tw/)[25-26]、TargetScan_7.2(http://www.targetscan.org/vert_72/)[27-28]、microRNA.org(http://www.microrna.org/microrna/home.do)[29-30]、miRDB(http://mirdb.org/index.html)[31-32]数据库分析预测LIFR上游miRNA,并利用Venny 2.1绘制韦恩图。构建野生型和突变型质粒,分别与miR-27a模拟物mimics miR-27a及阴性对照mimics NC共转染入MDA-MB-231细胞,进行双荧光素酶报告基因实验。将转染后的细胞接种于96孔白色酶标板中,每孔约1×105细胞。按照Dual-Glo©Stop & Glo©Substrate:Dual-Glo©Stop &Glo©Substrate Buffer=100∶1的比例配置Dual-Glo©Stop & Glo©Reagen;按照每孔50 μl培养基加入50 μl Dual-Glo©Luciferase Reagent,室温避光孵育10 min后测量各组萤火虫荧光值;避光条件下,每孔加入50 μl Dual-Glo©Stop & Glo©Reagent;室温孵育10 min后测量各组海肾荧光值;计算萤火虫荧光值与海肾荧光值比值。
2 结果
2.1 LIFR在泛癌中的mRNA表达水平 为了研究LIFR在不同类型肿瘤组织与正常组织中的表达差异,利用Oncomine数据库分析不同肿瘤类型的LIFR的mRNA表达水平,结果显示,除中枢系统癌症与淋巴瘤外,与正常组织相比,癌组织LIFR的表达普遍较低(图1A)。进一步,我们使用TIMER数据库在线分析了LIFR在多种恶性肿瘤组织与其邻近的正常组织中LIFR的表达差异,如图1B所示。相比于正常组织,LIFR的表达在膀胱尿路上皮癌(bladder urothelial carcinoma,BLCA)、浸润性乳腺癌(breast invasive carcinoma,BRCA)、胆管癌(cholangiocarcinoma,CHOL)、结肠癌(colon adenocarcinoma,COAD)、食管癌(esophageal carcinoma,ESCA)、头颈部鳞状细胞癌(head and neck squamous cell carcinoma,HNSC)、肾嫌色细胞癌(kidney chromophobe,KICH)、肾透明细胞癌(kidney renal clear cell carcinoma,KIRC)、肾乳头状细胞癌(kidney renal papillary cell carcinoma,KIRP)、肝细胞癌(liver hepatocellular carcinoma,LIHC)、肺腺癌(lung adenocarcinoma,LUAD)、肺鳞状细胞癌(lung squamous cell carcinoma,LUSC)、前列腺癌(prostate adenocarcinoma,PRAD)、直肠腺癌(rectum adenocarcinoma,READ)、胃腺癌(stomach adenocarcinoma,STAD)、甲状腺癌(thyroid carcinoma,THCA)、子宫内膜癌(uterine corpus endometrial carcinoma,UCEC)中显著下调。
2.2 LIFR表达与肿瘤突变负荷及微卫星不稳定性
利用UCSC Xena网站下载TCGA数据库中33种肿瘤类型的突变数据,进一步研究LIFR的基因表达水平与肿瘤突变负荷的相关性。结果显示,LIFR表达水平与乳腺癌、结肠癌、淋巴样肿瘤弥漫性大B细胞淋巴瘤(lymphoid neoplasm diffuse large b-cell lymphoma,DLBC)、食管癌、急性髓性白血病(acute myeloid leukemia,LAML)、脑低度胶质瘤(brain lower grade glioma,LGG)、肝细胞癌、胰腺癌(pancreatic adenocarcinoma,PAAD)、前列腺癌、恶性间叶肿瘤(sarcoma,SARC)、胃腺癌、甲状腺癌、胸腺瘤(thymoma,THYM)、葡萄膜黑色素瘤(uveal melanoma,UVM)的肿瘤突变负荷具有相关性,其中与急性髓性白血病、恶性间叶肿瘤和胸腺瘤为正相关,其余皆为负相关(图2A)。分析LIFR表达与微卫星不稳定性相关性,结果表明,LIFR表达水平与乳腺癌、淋巴样肿瘤弥漫性大B细胞淋巴瘤、头颈部鳞状细胞癌、前列腺癌、皮肤黑色素瘤(skin cutaneous melanoma,SKCM)、胃腺癌和甲状腺癌皆为负相关(图2B)。
2.3 LIFR与乳腺癌肿瘤微环境的相关性 利用“ESTIMATE”R包计算乳腺癌样品的免疫打分与基质打分后与LIFR的基因表达文件取交集并将结果输出,结果发现,LIFR的表达量和浸润性乳腺癌肿瘤微环境中的基质细胞含量成正相关,即乳腺癌中LIFR表达量越高肿瘤微环境中基质细胞所占成分越大(图3)。
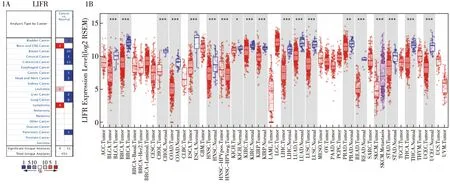
1A:Oncomine数据库中的正常组织与不同癌症中LIFR表达水平比较;1B:TIMER数据库分析TCGA数据集中不同肿瘤类型的LIFR表达水平。***表示P<0.001;**表示P<0.01;*表示P<0.05图1 不同类型肿瘤中LIFR的表达水平
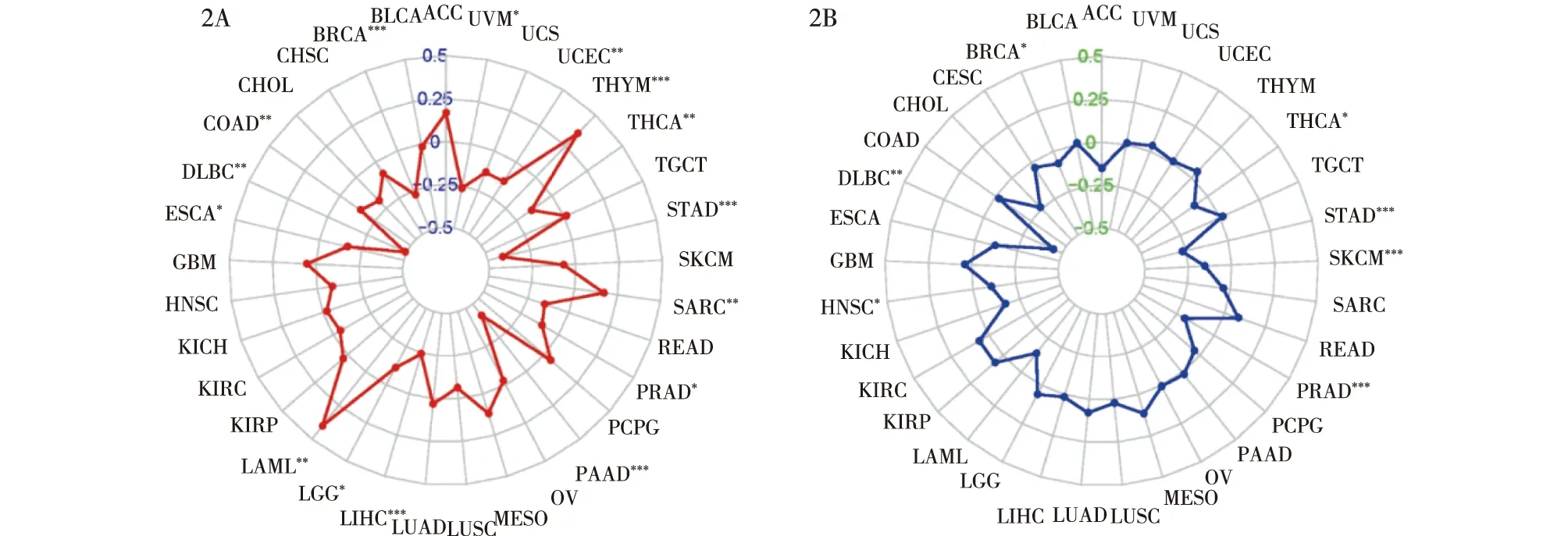
2A:LIFR表达与肿瘤突变负荷相关系数雷达图;2B:LIFR表达与微卫星不稳定性相关系数雷达图。***表示P<0.001;**表示P<0.01;*表示P<0.05图2 LIFR表达与肿瘤突变负荷及微卫星不稳定性相关性

图3 LIFR表达量与乳腺癌肿瘤微环境的相关性
2.4 LIFR对乳腺癌的预后影响 通过PrognoScan评估LIFR表达水平高低对患者生存率的影响可以发现,LIFR的表达主要影响脑肿瘤、乳腺癌、结肠癌和肺癌,高LIFR表达提示患者预后较好。其中我们重点分析了LIFR对乳腺癌的预后价值(表1,图4),结果显示,高表达LIFR乳腺癌患者有较好的总生存期、肿瘤无远处转移生存期和无复发生存期。通过Kaplan-Meier plotter数据库,基于Affymetrix微阵列进一步分析LIFR对乳腺癌的预后价值,结果显示,LIFR低表达与乳腺癌(OSHR=0.55,95%CI=0.4~0.76,P=0.002;DMFSHR=0.68,95%CI=0.49~0.94,P=0.02;RFSHR=0.66,95%CI=0.56~0.77,P=1.3e-07)的不良预后有关。
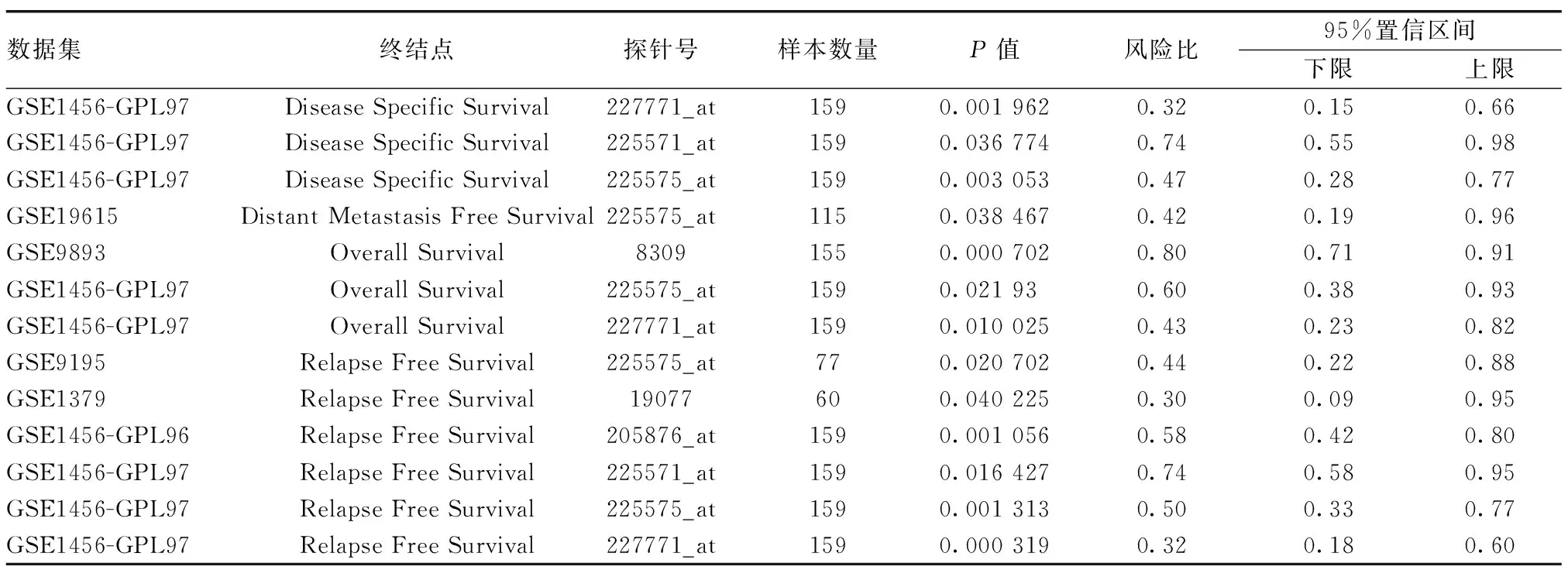
表1 LIFR表达水平与乳腺癌预后指标的相关性
2.5 免疫和分子亚型中LIFR基因的表达 通过TISIDB数据库所汇总的跨TCGA数据集的各种癌症LIFR的基因表达情况及与免疫和分子亚型的关系见图5。
2.6 乳腺癌中LIFR启动子甲基化水平 通过UALCAN分析LIFR在乳腺癌中启动子甲基化水平发现,LIFR在乳腺癌中表现为低甲基化。乳腺癌不同亚型、分期、组织学分类、淋巴结转移分期、年龄的LIFR启动子甲基化差异情况见图6。
2.7 LIFR在乳腺癌中低表达并抑制乳腺癌细胞迁移能力 细胞实验证实,LIFR在高侵袭性乳腺癌细胞系MDA-MB-231细胞中的基因表达水平显著低于低侵袭性乳腺癌细胞系MCF7细胞,二者具有统计学差异(图7A)。进一步通过在MDA-MB-231细胞中进行转染,干扰或过表达LIFR的水平后进行划痕实验,结果显示,干扰LIFR表达后,在第42与第72小时的划痕愈合速度明显快于对照组(siRNA NC),而过表达LIFR后,在第42与第72小时划痕愈合速度慢于转染空白质粒组(pc DNA 3.1)(图7B、7C),说明LIFR能够抑制乳腺癌细胞的迁移能力。实验所用引物序列见表2。

表2 实验所用引物序列
2.8 LIFR是miR-27a的直接靶点 为了探究LIFR与微小RNA(microRNA,miRNA)的关系,通过对miRTarBase、TargetScan_7.2、microRNA.org和miRDB数据库预测取交集绘制韦恩图,发现,miR-27a为LIFR的上游靶点(图8A);构建野生型和突变型质粒,进行双荧光素酶报告基因实验,结果表明,miR-27a可以直接与LIFR的3’UTR区域靶向结合(图8B)。
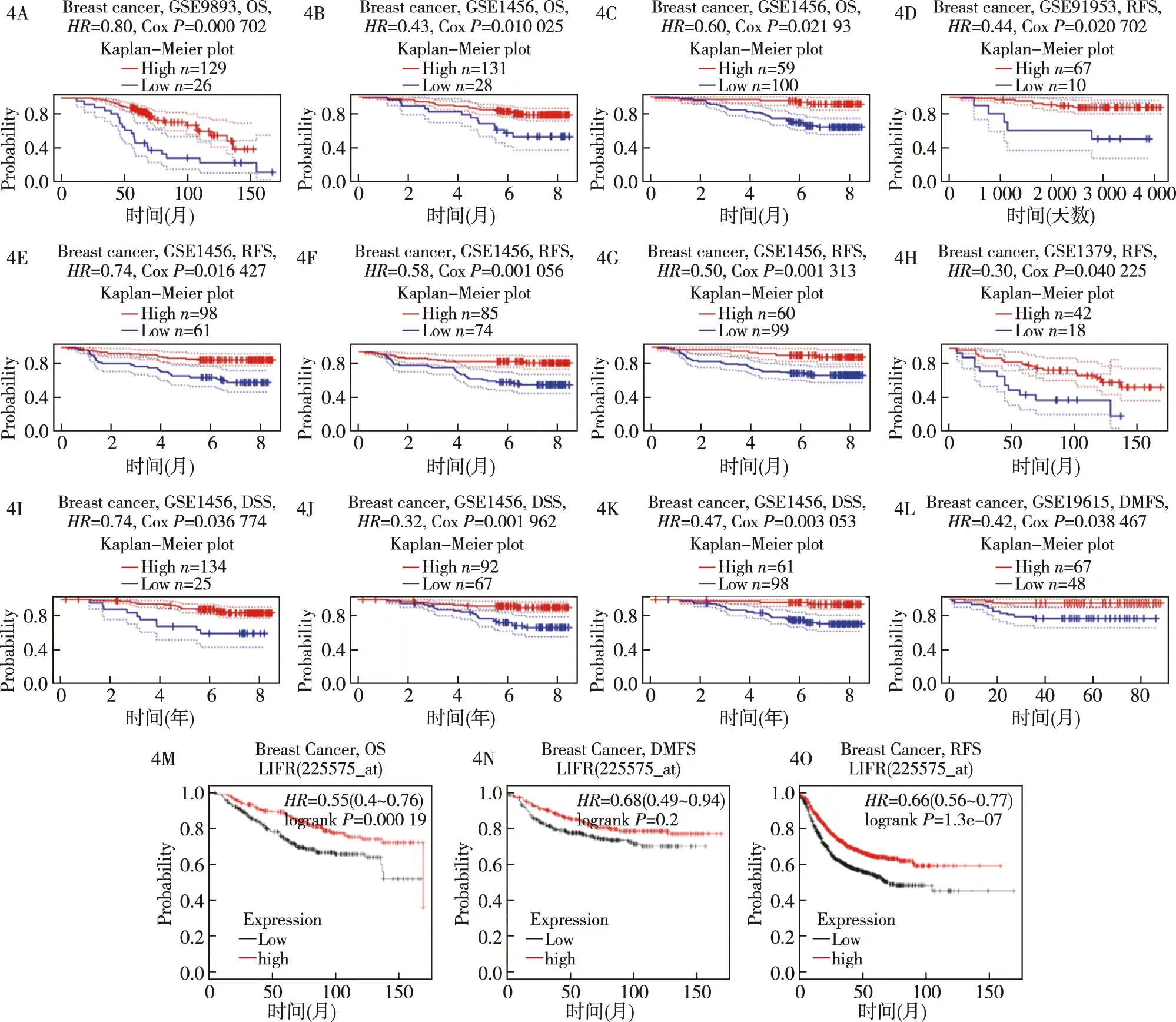
注:PrognoScan(4A~4L)和Kaplan-Meier plotter数据库(4M~4O);4A~4C:LIFR表达水平高低与乳腺癌OS;4D~4H:LIFR表达水平高低与乳腺癌RFS;4I~4K:LIFR表达水平高低与乳腺癌DSS;4L:LIFR表达水平高低与乳腺癌DMFS;4M~4O乳腺癌OS、DMFS、RFS曲线图4 Kaplan-Meier生存曲线显示LIFR表达水平高低与乳腺癌预后关系
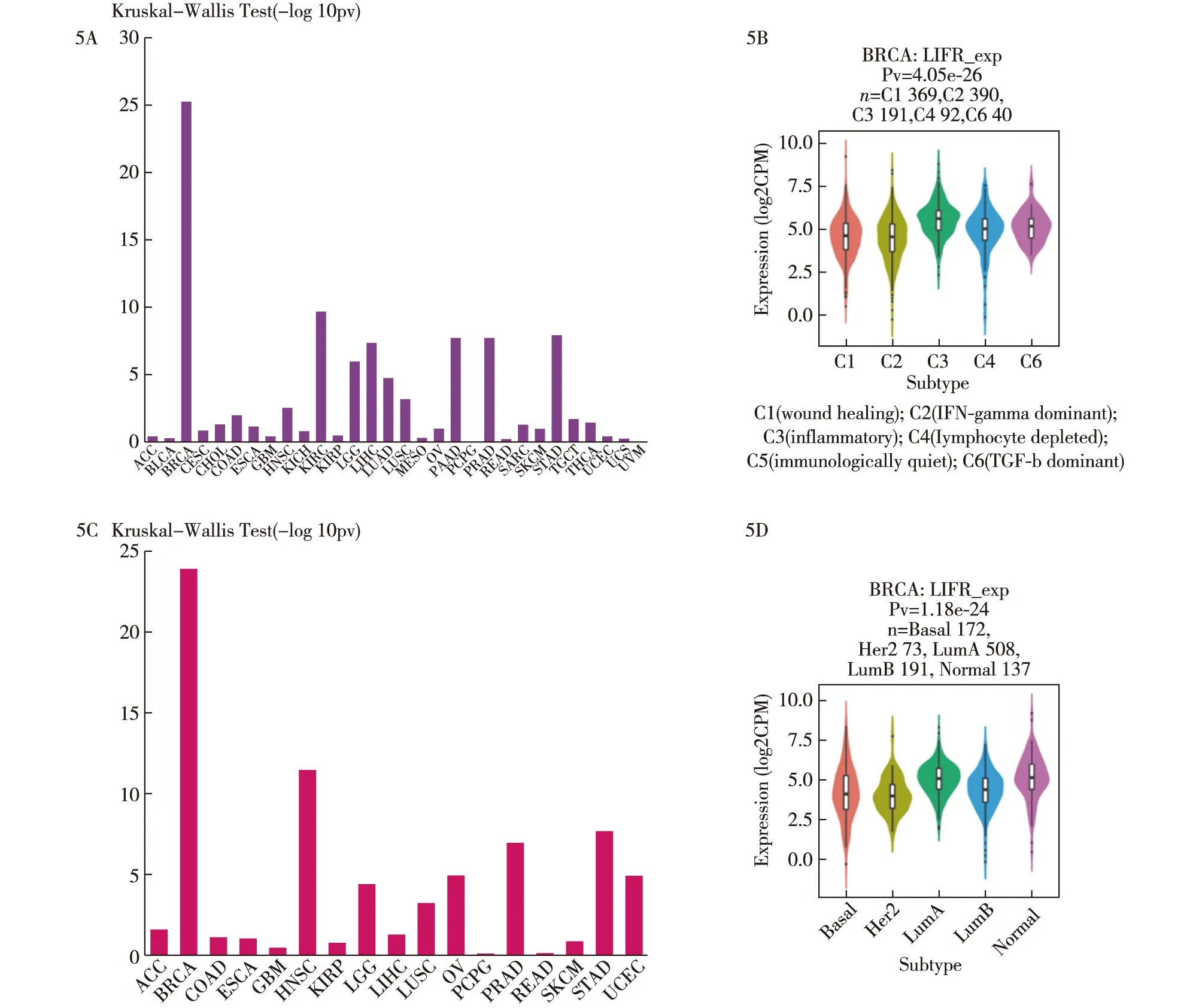
5A:LIFR表达与不同肿瘤类型免疫亚型的关系;5B:乳腺癌不同免疫亚型中LIFR的基因表达水平;5C:LIFR表达与不同类型肿瘤分子亚型的关系;5D:LIFR在乳腺癌不同亚型中的基因表达水平图5 TISDB数据库分析LIFR的基因表达水平与免疫和分子亚型的关系
3 讨论
本研究通过Oncomine数据库以及TIMER数据库在线分析发现,LIFR在多种癌症中表达下调。已有研究证明,LIFR能够抑制胰腺癌[33]、肺腺癌[34]、肝癌[35]、胃癌[36]、乳腺癌[13]等多种癌症的转移抑制因子,且与食管癌治疗中耐药具有相关性[37]。通过PrognoScan以及Kaplan-Meier plotter数据库绘制Kaplan-Meier生存曲线,结果发现LIFR主要与脑肿瘤、乳腺癌、结肠癌、肺癌、胃癌以及卵巢癌的预后有关。对乳腺癌,LIFR主要影响了乳腺癌患者的总生存期、无复发生存期和无远处转移生存期,乳腺癌中LIFR高表达往往提示患者具有更好的预后,LIFR的表达缺失往往提示患者的预后较差。细胞实验结果同样证实乳腺癌细胞中高表达的LIFR能够抑制细胞迁移能力,而低表达LIFR后乳腺癌细胞迁移能力则明显增强。进一步分析LIFR表达水平与肿瘤突变负荷及微卫星不稳定性的相关性后发现,LIFR表达水平与乳腺癌、结肠癌、淋巴样肿瘤弥漫性大B细胞淋巴瘤、食管癌、脑低度胶质瘤、肝细胞癌、胰腺癌、前列腺癌、胃腺癌、甲状腺癌、葡萄膜黑色素瘤的肿瘤突变负荷负相关(即LIFR表达水平越高,每兆碱基中突变的总数越少),与急性髓性白血病、恶性间叶肿瘤和胸腺瘤为正相关。肿瘤突变负荷(tumor mutation burden,TMB)即肿瘤组织每兆碱基中突变的总数,是指一份肿瘤样本中相对的基因突变数量。目前来说,TMB越高,肿瘤被T细胞识别的可能性越大。乳腺癌中LIFR表达缺失,降低了机体对肿瘤细胞的免疫效应,这可能是LIFR对乳腺癌的作用机制之一。肿瘤与免疫的相关研究是近年来的热点。根据治疗策略,肿瘤的免疫治疗分为两类。第一类是增强免疫激活机制,通过调节免疫系统来增强对肿瘤的免疫反应,这种方法往往效果不理想,还会引发一系列副作用。第二类是免疫正常化,修复肿瘤微环境(tumor microenvironment,TME)中的抗肿瘤免疫力,最典型的为细胞程序性死亡(PD)途径,该途径在TME中上调抑制了效应T细胞的抗肿瘤效应,阻断该途径能够有效改善多种肿瘤类型的抗肿瘤免疫应答。实验结果表明,非小细胞肺癌中TMB和PD-L1表达是两个独立的变量,但是却具有相似的预测能力,而将TMB和PD-L1表达均纳入多变量预测模型时,可获得更好的免疫治疗预测能力,具备更佳的临床指导价值[38]。PD-L1在Her-2阳性乳腺癌患者中具有独立的不良预后影响[39-41]。相当一部分Her-2阳性乳腺癌病例中肿瘤浸润淋巴细胞(TILs)、PD-L1升高,这可能增加机体抗肿瘤反应的发生概率,使得这一部分患者有更大的概率从免疫治疗中获益[42]。
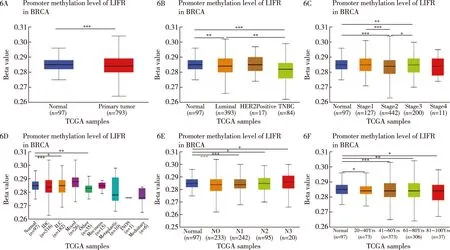
6A:不同组织类型中;6B:不同亚型中;6C:不同分期中;6D:不同组织学分类中;6E:不同淋巴结转移分期中;6F:不同年龄分期中。***表示P<0.001;**表示P<0.01;*表示P<0.05图6 乳腺癌中LIFR启动子甲基化水平
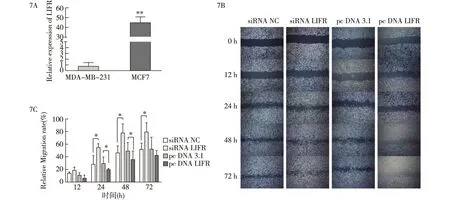
7A:LIFR mRNA在高侵袭性乳腺癌细胞系MDA-MB-231与低侵袭性乳腺癌细胞系MCF7之间的表达差异;7B、7C:划痕实验比较干扰(siRNA LIFR)或过表达(pcDNA LIFR)后乳腺癌细胞迁移能力的变化,测量第0、12、24、48和72小时划痕面积并统计图7 LIFR在乳腺癌中低表达并抑制乳腺癌细胞迁移能力

8A:通过miRTarBase、TargetScan_7.2、microRNA.org和miRDB数据库预测LIFR的上游靶点,结果显示miR-27a为共同预测结果;8B:双荧光素酶报告基因实验证实miR-27a可以直接靶向LIFR的3’UTR区域图8 LIFR是miR-27a的下游靶基因
未来免疫介导的抗肿瘤机制可能会在肿瘤治疗中发挥更大的作用,通过研究肿瘤发生发展过程中的关键基因,寻找其与免疫的关联,将使免疫治疗的作用进一步放大,为癌症患者带来更多益处。
