circFAT1对胶质母细胞瘤细胞生物过程的机制研究
2023-11-08张立群乌日吉木斯郑晓明汪琳韩玉秀张薇阎涛
张立群 乌日吉木斯 郑晓明 汪琳 韩玉秀 张薇 阎涛
摘要:目的 探討circFAT1吸附miR-1182调控转化生长因子(TGF)-β/Smad信号通路对胶质母细胞瘤(GBM)增殖、侵袭、上皮-间充质转化(EMT)的调控作用及机制。方法 收集50例GBM样本和5例非瘤脑组织(NBT)样本,随机抽取3例GBM和3例NBT样本进行组织RNA测序。体外培养GBM细胞系U87、U251、T98G、LN229及星形胶质细胞系NHA,通过qRT-PCR检测组织和细胞系中circFAT1表达。通过RNase R选择性降解线性转录本以明确circFAT1成环特性。将GBM细胞分为sh-对照组和sh-circFAT1组,采用慢病毒进行转染。转染后集落形成实验、Transwell实验、Western blot实验分别检测细胞增殖、侵袭及EMT能力。荧光原位杂交(FISH)检测circFAT1在GBM细胞内定位,通过双萤光素酶报告基因和RNA免疫共沉淀(RIP)实验验证circFAT1和miR-1182的相互作用。Western blot实验验证敲低circFAT1后或抑制miR-1182表达对TGF-β/Smad信号通路的调控作用。结果 circFAT1在GBM组织和细胞中表达增加(P<0.05)。RNase R可减少线性FAT1表达(P<0.05),而不影响circFAT1。敲除circFAT1抑制了GBM细胞的增殖、侵袭和EMT(P<0.05)。双萤光素酶报告基因和RIP实验证实circFAT1靶向结合miR-1182。敲低circFAT1后TGFB2、p-SMAD2、p-SMAD3蛋白表达下降,抑制miR-1182表达后GFB2、p-SMAD2、p-SMAD3蛋白表达升高。结论 敲除circFAT1可抑制GBM细胞增殖、侵袭和EMT,其作用机制可能与吸附miR-1182调控TGF-β/Smad信号通路有关。
关键词:胶质母细胞瘤;细胞增殖;转化生长因子β;微RNAs;circFAT1;上皮间充质转化
中图分类号:R739.41文献标志码:ADOI:10.11958/20230224
The mechanisms of circFAT1 on the biological process of GBM cells
ZHANG Liqun WURI Jimusi ZHENG Xiaoming WANG Lin HAN Yuxiu ZHANG Wei YAN Tao
1 Tianjin Neurological Institute, Tianjin Medical University General Hospital, Tianjin 300052, China;
2 Department of Obstertrics and Gynecology, Tianjin First Central Hospital
Corresponding Author E-mail: taoyan@tmu.edu.cn
Abstract: Objective To investigate the regulation and mechanism of circFAT1 on proliferation, invasion and epithelial-mesenchymal transition (EMT) of glioblastoma (GBM). Methods Fifty GBM samples and 5 non-tumor brain tissue (NBT) samples were collected, and 3 GBM and 3 NBT samples were randomly selected for tissue RNA sequencing. The expression levels of circFAT1 in U87, U251, T98G, LN229 GBM cell lines and NHA astrocyte lines were detected by qRT-PCR. The cyclic properties of circFAT1 were determined by selective degradation of linear transcripts by RNase R. GBM cells were divided into the sh-control group and the sh-circFAT1 group. Cell proliferation, invasion and EMT were detected by colony formation assay, transwell assay and Western blot assay after lentivirus transfection. Fluorescence in situ hybridization (FISH) was used to detect the localization of circFAT1 in GBM cells, and the interaction between circFAT1 and miR-1182 was verified by dual-luciferase reporter and RNA immunocoprecipitation (RIP) assay. Finally, Western blot assay was performed to verify the regulatory effect of circFAT1 knockdown or inhibition of miR-1182 expression on TGF-β/Smad signaling pathway. Results The expression of circFAT1 was increased in GBM tissue and cells (P<0.05). RNase R reduced linear FAT1 expression without affecting circFAT1 (P<0.05). Knockout of circFAT1 inhibited the proliferation, invasion and EMT of GBM cells (P<0.05). Dual-luciferase reporter and RIP assay confirmed that circFAT1 targeting on miR-1182. The protein levels of TGFB2, p-SMAD2 and p-SMAD3 were decreased after circFAT1 knockdown, while protein expressions of TGFB2, p-SMAD2 and p-SMAD3 increased after inhibiting the expression of miR-1182 (all P<0.05). Conclusion Knockout of circFAT1 can inhibit proliferation, invasion and EMT of GBM cells, and its mechanism may be related to the regulation of TGF-β/Smad signaling pathway by sponge miR-1182.
Key words: glioblastoma; cell proliferation; transforming growth factor beta; microRNAs; circFAT1; epithelial-mesenchymal transition
膠质母细胞瘤(glioblastoma,GBM)是癫痫的诱发因素之一[1],患者的预后与癫痫的临床表现有关[2]。研究表明,GBM患者经过手术联合系统放化疗后的中位生存时间也低于15个月[3]。因此,进一步研究GBM发生发展的分子机制,寻找GBM诊断和预后的生物标志物和治疗靶点具有重要意义。环状RNA(circRNA)已被证实与细胞的病理生理过程相关[4]。研究表明,多种circRNAs通过不同的分子机制参与癌症的发展过程[5]。但环状RNA脂肪非典型钙黏蛋白1(circRNA FAT atypical cadherin 1,circFAT1)在肿瘤特别是GBM中的作用及相应机制尚不明确。转化生长因子β(transforming growth factor β,TGF-β)通路因其在肿瘤早期抑制肿瘤细胞增殖,而在肿瘤晚期通过调节上皮-间充质转化(epithelial-mesenchymal transition,EMT)、血管生成、免疫逃逸、侵袭和转移促进肿瘤的发展而备受关注。研究表明,TGF-β信号通路促进细胞增殖与EMT进程,从而促进GBM的发展[6]。本研究旨在观察敲除circFAT1对GBM细胞增殖、侵袭、EMT以及TGF-β通路的影响,为探求GBM诊治靶点提供新的思路与方向。
1 材料与方法
1.1 一般材料
1.1.1 胶质瘤细胞及组织样本
选取2019年6月—2022年1月天津医科大学总医院神经外科患者经手术切除的50例GBM和5例非瘤脑组织(NBT)标本,所有组织样本均经病理证实,冻存于-80 ℃以备后续研究。所有患者均签署书面知情同意书。本研究方案经天津医科大学总医院伦理委员会批准(批件号:2019-SR-479)。本研究所用GBM细胞系U87、U251、T98G、LN229,星形胶质细胞系NHA和工具细胞HEK293T均购自中国科学院上海细胞库。
1.1.2 主要试剂与仪器
高糖Dulbecco改良Eagles(DMEM)培养基、胎牛血清(FBS)购自美国Sigma公司;TRIzol法RNA提取试剂盒购自美国Thermo Fisher公司;RNA反转录试剂盒、高敏qRT-PCR试剂盒购自中国Vazyme公司;RiboMinus真核细胞试剂盒购自德国Qiagen公司;Agilent DNA 1000测序芯片购自美国Agilent公司;EntransterTM-R4000转染试剂购自中国英格恩公司;核糖核酸酶R(RNase R)购自美国Epicentre公司;兔抗人钙黏蛋白E(E-cadherin)、钙黏蛋白N(N-cadherin)、波形蛋白(Vimentin)、转化生长因子β2(transforming growth factor beta 2,TGFB2)、磷酸化SMAD2(p-SMAD2)、磷酸化SMAD3(p-SMAD3)蛋白抗体购自英国Abcam公司,兔抗人GAPDH和小鼠抗人β-actin抗体购自美国cell signaling technology公司。辣根过氧化物酶标记的山羊抗兔、山羊抗小鼠二抗购自美国Thermo Fisher公司。双萤光素酶报告基因试剂盒购自美国Promega公司;Transwell小室(含基质胶)购自美国Corning公司。荧光原位杂交(fluorescence in situ hybridization,FISH)试剂盒、sh-circFAT1、sh-ctrl、miR-1182 inhibitor、miR-NC inhibitor购自中国RiboBio公司。RNA免疫共沉淀(RIP)试剂盒、人源argonaute2(ago2)抗体、小鼠IgG抗体购自美国Millipore公司。野生型circFAT1、突变型circFAT1报告质粒、miR‐1182 mimics、miR‐NC、sh-circFAT1及sh-对照序列均由中国锐博生物构建。NanoDrop 2000分光光度计购自美国Thermo-Fisher公司;7900HT PCR系统购自美国Applied Biosystems公司;Agilent 2100生物分析仪购自美国Agilent公司。
1.2 实验方法
1.2.1 RNA测序(RNA-seq)分析
采用TRIzol试剂从随机抽取的3例GBM组织和3例NBT组织中提取总RNA[7],采用NanoDrop 2000分光光度计对RNA进行质控及定量检测。通过琼脂糖凝胶电泳检测RNA完整性及是否存在gDNA污染。在构建RNA-seq文库之前,先用RiboMinus真核细胞试剂盒去除核糖体RNA。测序文库由Agilent 2100生物分析仪通过使用Agilent DNA 1000芯片进行测定。
1.2.2 细胞培养
所有细胞均在含10% FBS的DMEM培养基中培养,细胞培养环境为37 ℃、5%CO2。
1.2.3 RNA提取及qRT-PCR检测
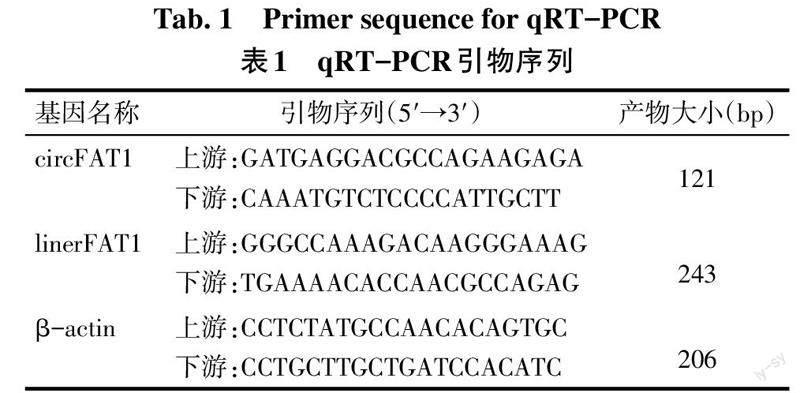
使用TRIzol试剂从细胞或组织中提取总RNA[7]。根据试剂盒说明书反转录为cDNA。根据高敏qRT-PCR试剂盒说明书配制反应体系。然后在7900HT系统上进行qRT-PCR反应,反应条件:95 ℃预变性10 min;95 ℃变性10 s,65 ℃退火15 s,72 ℃延伸15 s,共计40个循环。每个样本设置3个复孔,并通过2-ΔΔCt法计算基因相对表达量,β-actin作为内参对照。引物序列见表1。选取circFAT1表达水平最高的细胞系用作后续研究。
1.2.4 GBM细胞中circFAT1表达稳定性鉴定
将GBM细胞按是否经过RNase R处理分为对照组和RNase R组,按1.2.3方法提取细胞总RNA。各组分别取2 ?g总RNA,RNaseR组加入6 U的RNase R,37 ℃消化20 min,对照组加入等量PBS。消化后的RNA根据试剂盒说明书进行纯化,按1.2.3方法通过qRT-PCR检测circFAT1及线性FAT1(linerFAT1)表达。引物序列见表1。
1.2.5 sh-circFAT1及miR‐1182抑制剂(inhibitor)转染
细胞转染方案同本课题组前期研究方案[7]。将GBM细胞分为sh-对照组、sh-circFAT1-1组、sh-circFAT1-2组及sh-circFAT1-3组。将细胞铺于6孔板中,待细胞贴壁后将50 pmol shRNA溶于Opti-MEM培养基(200 ?L)中,与EntransterTM-R4000转染试剂充分混合后转染48 h。通过qRT-PCR验证转染效率,选取效率最高的shRNA进行后续实验。miR‐1182 inhibitor转染:将细胞铺于6孔板中,待融合度达60%~70%、细胞贴壁后将质粒(根据需要3~5 ?g)溶于Opti-MEM培养基(200 ?L)中,与EntransterTM-R4000转染试剂充分混合后转染48 h。
1.2.6 集落形成实验检测细胞增殖
将GBM细胞分为sh-对照组及sh-circFAT1组。转染目的RNA 48 h后,将2组细胞分别接种于含10% FBS的DMEM培养基中培养11 d。所得细胞集落用PBS洗涤2次,4%多聚甲醛固定10 min,0.1%结晶紫染色30 min。普通摄像机拍照后使用Image J软件计数集落数目。
1.2.7 Transwell实验检测细胞侵袭
细胞分组同1.2.6。使用由聚碳酸酯滤器(8 μm孔径;0.33 cm2)组成的直径为6.5 mm的Transwell小室进行实验。转染目的RNA 48 h后,将GBM细胞(3×104个)转移至含有无血清培养基的上层小室中,下层小室加入含10%FBS的DMEM培养基。37 ℃孵育48 h后,去除上层未侵袭的细胞,剩余细胞用4%多聚甲醛固定20 min。用0.1%结晶紫染色,用Nikon ECLIPSE E800荧光倒置显微镜观察。
1.2.8 Western blot检测EMT能力
细胞分组同1.2.6。用免疫沉淀缓冲液裂解细胞,提取总蛋白,用BCA法测定蛋白浓度。使用10%浓度的SDS-PAGE进行蛋白电泳分离蛋白。220 mA恒流转膜90 min。将PVDF膜放入5%脱脂奶粉中封闭1 h;一抗稀释后(E-cadherin、N-cadherin、Vimentin稀释比例为1∶1 000,GAPDH稀释比例为1∶2 000),4 ℃与PVDF膜共同孵育过夜,二抗稀释后(1∶2 000),与PVDF膜在37 ℃摇床孵育1 h。ECL化学发光液均匀涂抹于PVDF膜,BIO-RAD化学发光凝胶成像仪拍照。
1.2.9 FISH检测circFAT1在細胞中的分布
通过FISH检测试剂盒(RiboBio)检测[7]。使用0.5%Triton X-100对经甲醛固定的细胞行透性处理,并与特异性探针在37 ℃下杂交过夜。用4,6-二氨基-2-苯基吲哚(DAPI)标记细胞核。荧光图像由蔡司LSM 700共聚焦显微镜拍摄得到。
1.2.10 RIP
RIP实验方案同本课题组先前研究方案[7]。制备GBM细胞裂解物,用含磁珠的RIP缓冲液孵育,磁珠与人源ago2抗体结合,按1.2.3方法通过qRT-PCR检测免疫沉淀RNA表达。裂解物组(Input)作为阳性对照,IgG抗体组作为阴性对照,抗ago2(anti-Ago2)抗体组检测circFAT1富集水平。
1.2.11 双萤光素酶报告基因检测
萤光素酶测定方案同同本课题组先前研究方案[7]。从DIANA数据库(http://diana.imis.athena-innovation.gr/)预测circFAT1与miR-1182的结合位点,并将野生型(WT)或突变型(MUT)circFAT1报告质粒、miR‐1182 mimics或miR‐NC和海肾萤光素酶质粒共转染至U87细胞。使用双萤光素酶报告基因试剂盒检测所有荧光素酶活性。萤火虫萤光素酶活性通过海肾萤光素酶活性进行均一化。
1.2.12 Western blot检测TGF-β信号通路蛋白表达
将GBM细胞分为A组(转染sh-对照序列+miR-NC inhibitor)、B组(转染sh-circFAT1+miR-NC inhibitor)、C组(转染sh-对照序列+miR-1182 inhibitor)、D组(转染sh-circFAT1+miR-1182 inhibitor)。按1.2.7方法检测TGMB2、p-SMAD2、p-SMAD3、β-actin蛋白表达水平,以β-actin为内参蛋白,计算TGMB2、p-SMAD2、p-SMAD3相对表达量。
1.3 统计学方法
采用GraphPad Prism 7软件进行数据分析。计量数据用均数±标准差(x±s)表示,2组间比较采用t检验,多组间比较采用单因素方差分析,组间多重比较采用LSD-t检验。P<0.05为差异有统计学意义。
2 结果
2.1 circFAT1在GBM组织和细胞中的表达情况
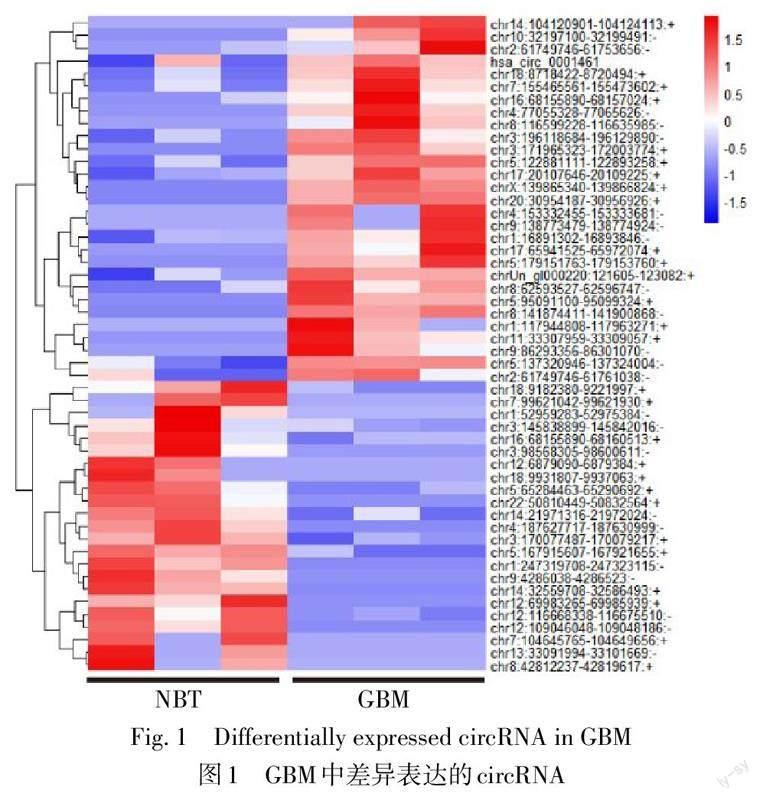
组织间circRNA测序显示,在GBM中共有29个表达上调的circRNA(|log2| fold change≥1且P<0.05),其中包含has_circ_0001461(即circFAT1[8]),见图1。NHA、U87、LN229、T98G、U251细胞系中circFAT1表达水平分别为1.00±0.02、3.99±0.25、2.01±0.05、1.89±0.18和2.83±0.11,差异有统计学意义(F=31.514,P<0.01)。U87细胞系中circFAT1表达水平最高,用作后续研究。GBM组织中circFAT1表达水平高于NBT组织(4.17±1.10 vs. 1.24±0.41,t=12.091,P<0.01)。
2.2 circFAT1的環状结构及表达稳定性鉴定
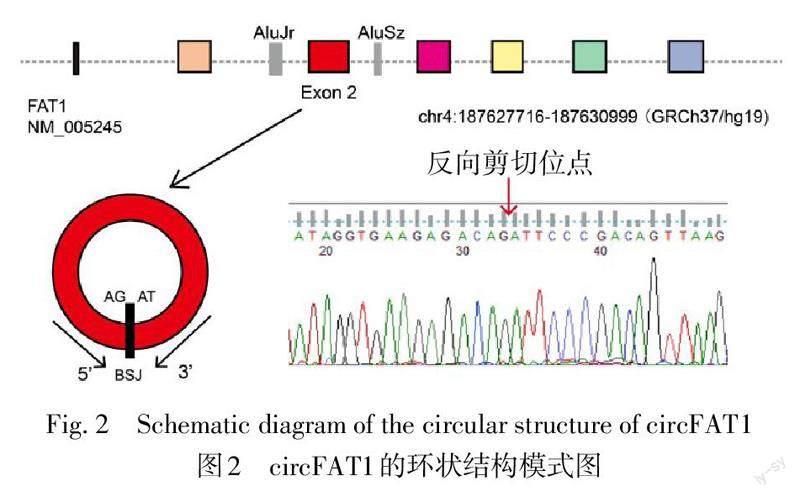
circFAT1是通过FAT1基因第2外显子与两侧内含子中的Alu元件的反向剪接而产生,见图2。linerFAT1在对照组和RNase R组中相对表达量分别为1.02±0.01和0.26±0.01,差异有统计学意义(t=7.963,P<0.01),而circFAT1的表达量分别为0.95±0.00和1.03±0.01,差异无统计学意义(t=1.061,P>0.05)。
2.3 敲除circFAT1基因抑制GBM细胞的增殖、侵袭和EMT能力
sh-对照组、sh-circFAT1-1组、sh-circFAT1-2组及sh-circFAT1-3组circFAT1相对表达量分别1.00±0.01、0.15±0.00、0.34±0.02、0.65±0.01,差异有统计学意义(n=3,F=48.082,P<0.05)。其中sh-circFAT1-1序列敲除效率最高,用于后续实验。与sh-对照组相比,sh-circFAT1组U87细胞集落数量和侵袭率下降,E-cadherin表达增高,N-cadherin和Vimentin表达降低,见图3、表2。
2.4 双萤光素酶报告基因验证circFAT1下游靶点
circFAT1在GBM细胞中定位于细胞质。RIP实验显示,与阴性对照相比,anti-Ago2可显著富集circFAT1,差异有统计学意义(F=549.152,P<0.01)。双萤光素酶报告基因显示,与miR-NC组相比,miR-1182 mimic组可降低含circFAT1-WT报告载体细胞的萤光素酶活性(t=6.726,P<0.01),对含circFAT1-MUT报告载体细胞的萤光素酶活性无明显影响(t=0.555,P>0.05)。见图4。
2.5 circFAT1对TGF-β/Smad信号通路的影响
与A组比较,B组TGFB2、p-SMAD2、p-SMAD3表达量下降,而C组中相应蛋白表达升高;与C组比较,D组TGFB2、p-SMAD2、p-SMAD3表达量下降;与B组比较,D组TGFB2、p-SMAD2、p-SMAD3表达量升高(均P<0.05),见图5、表3。
3 讨论
胶质瘤是中枢神经系统的原发性肿瘤,成人的发病率最高,占颅内肿瘤的50%[9],其中GBM约占胶质瘤的46.6%[10],其发病率呈逐年上升趋势。目前,GBM患者的平均生存时间仅为15个月左右,即使在接受标准化胶质瘤治疗方案(即手术切除加术后放化疗)后效果亦不显著[11]。因此,有必要进一步研究GBM发生发展的潜在机制,改进现有治疗方案,以改善患者预后。
circRNA是通过前体mRNA的反向剪接而形成[12],其核心形成机制包括单外显子环化、多外显子环化及协同环化[13]。circRNA的形成机制主要包括套索驱动环化、RNA结合蛋白驱动环化和反向剪接[14]。尽管circRNA的表达量远不如其线性转录本,但由于其存在共价闭环结构且缺乏游离末端,使其可免受RNase R的降解作用,进而具有高度稳定性及保守性[15]。因此,circRNA可用于识别不同的肿瘤类型[16]。circRNA表达具有组织特异性和发育阶段特异性,在癌症的发生和发展中起着重要作用[17-18]。环状RNA瘙痒E3泛素蛋白连接酶(circRNA itchy E3 ubiquitin protein ligase,circITCH)在食管癌中的表达明显低于癌旁组织。此外,环状RNA-7(circRNA 7,ciRS-7)在神经母细胞瘤和胶质瘤中广泛表达[19]。circRNAs在肿瘤中的差异表达提示它们可能在肿瘤进展中起调节作用,可作为诊断和预后评估的潜在生物标志物[20]。本研究对GBM和NBT样本进行circRNAs测序分析,发现circFAT1在GBM组织中异常高表达。本研究还发现,在RNase R处理下,circFAT1具有较高的表达稳定性,敲除circFAT1可抑制GBM细胞的增殖和侵袭。这些结果表明,circFAT1在GBM的侵袭性生长过程中起重要作用。
目前,circRNA的主要作用机制包括以下3点:(1)circRNA作为miRNA“分子海绵”发挥作用。(2)circRNA与蛋白质结合相互作用。(3)circRNA可直接进行翻译,得到相应蛋白进而发挥作用。circRNA的亚细胞定位决定其自身的生物学功能[21]。本研究发现,circFAT1主要在细胞质中表达。circRNA已被证明可通过定向结合任意miRNA发挥作用,从而形成复杂的调控网络[22-23]。通过生物信息学分析发现,miR-1182可能是circFAT1的潜在靶点。miR-1182过表达可以抑制卵巢癌的转移和增殖以及非小细胞肺癌的进展,表明其可能作为多种肿瘤结局的预测指标,并可作为潜在的诊断和治疗靶点[24]。本研究结果表明,circFAT1可作为miR-1182的分子海绵吸附miR-1182,进而参与miR-1182在GBM中的功能调控。进一步的通路研究表明,敲除circFAT1可下调TGF-β通路相关蛋白TGFB2、p-SMAD2、p-SMAD3的表达,抑制该通路活化,而TGF-β信号通路的活化已被证实在多种恶性肿瘤中起着重要的促进作用[25]。
综上所述,敲低circFAT1可抑制TGF-β信号通路激活,延缓GBM细胞的增殖和EMT过程。因此,circFAT1可能是GBM的潜在生物标志物和治疗靶点。但circFAT1/miR-1182轴在GBM中的下游基因尚未被阐明,并且circFAT1的功能仍需进一步的体内研究进行验证。
参考文献
[1] HUBERFELD G,VECHT C J. Seizures and gliomas-towards a single therapeutic approach[J]. Nat Rev Neurol,2016,12(4):204-216. doi:10.1038/nrneurol.2016.26.
[2] HATCHER A,YU K,MEYER J,et al. Pathogenesis of peritumoral hyperexcitability in an immunocompetent CRISPR-based glioblastoma model[J]. J Clin Invest,2020,130(5):2286-2300. doi:10.1172/jci133316.
[3] FURNARI F B,FENTON T,BACHOO R M,et al. Malignant astrocytic glioma:genetics,biology,and paths to treatment[J]. Genes Dev,2007,21(21):2683-2710. doi:10.1101/gad.1596707.
[4] SPIZZO R,ALMEIDA M I,COLOMBATTI A,et al. Long non-coding RNAs and cancer:a new frontier of translational research?[J]. Oncogene,2012,31(43):4577-4587. doi:10.1038/onc.2011.621.
[5] CHEN L,SHAN G. CircRNA in cancer:fundamental mechanism and clinical potential[J]. Cancer Lett,2021,505:49-57. doi:10.1016/j.canlet.2021.02.004.
[6] ZHANG G,FENG W,WU J. Down-regulation of SEPT9 inhibits glioma progression through suppressing TGF-β-induced epithelial-mesenchymal transition (EMT)[J]. Biomed Pharmacother,2020,125:109768. doi:10.1016/j.biopha.2019.109768.
[7] ZHANG L,WANG H,XU M,et al. Long noncoding RNA HAS2-AS1 promotes tumor progression in glioblastoma via functioning as a competing endogenous RNA[J]. J Cell Biochem,2020,121(1):661-671. doi:10.1002/jcb.29313.
[8] FANG J,HONG H,XUE X,et al. A novel circular RNA,circFAT1(e2),inhibits gastric cancer progression by targeting miR-548g in the cytoplasm and interacting with YBX1 in the nucleus[J]. Cancer Lett,2019,442:222-232. doi:10.1016/j.canlet.2018.10.040.
[9] YANG Y,GAO X,ZHANG M,et al. Novel role of FBXW7 circular RNA in repressing glioma tumorigenesis[J]. J Natl Cancer Inst,2018,110(3):304-315. doi:10.1093/jnci/djx166.
[10] LIU J,ZHAO K,HUANG N,et al. Circular RNAs and human glioma[J]. Cancer Biol Med,2019,16(1):11-23. doi:10.20892/j.issn.2095-3941.2018.0425.
[11] SONG X,ZHANG N,HAN P,et al. Circular RNA profile in gliomas revealed by identification tool UROBORUS[J]. Nucleic Acids Res,2016,44(9):e87. doi:10.1093/nar/gkw075.
[12] HE Q,ZHAO L,LIU Y,et al. circ-SHKBP1 regulates the angiogenesis of U87 glioma-exposed endothelial cells through miR-544a/FOXP1 and miR-379/FOXP2 pathways[J]. Mol Ther Nucleic Acids,2018,10:331-348. doi:10.1016/j.omtn.2017.12.014.
[13] ZHU L P,HE Y J,HOU J C,et al. The role of circRNAs in cancers[J]. Biosci Rep,2017,37(5):BSR20170750. doi:10.1042/bsr20170750.
[14] SHANG Q,YANG Z,JIA R,et al. The novel roles of circRNAs in human cancer[J]. Mol Cancer,2019,18(1):6. doi:10.1186/s12943-018-0934-6.
[15] MA S,KONG S,WANG F,et al. CircRNAs:biogenesis,functions,and role in drug-resistant Tumours[J]. Mol Cancer,2020,19(1):119. doi:10.1186/s12943-020-01231-4.
[16] SALZMAN J,CHEN R,OLSEN M,et al. Cell-type specific features of circular RNA expression[J]. PLoS Genet,2013,9(9):e1003777. doi:10.1371/journal.pgen.1003777.
[17] ASHWAL-FLUSS R,MEYER M,PAMUDURTI N R,et al. circRNA biogenesis competes with pre-mRNA splicing[J]. Mol Cell,2014,56(1):55-66. doi:10.1016/j.molcel.2014.08.019.
[18] HANAN M,SOREQ H,KADENER S. CircRNAs in the brain[J]. RNA Biol,2017,14(8):1028-1034. doi:10.1080/15476286.2016.1255398.
[19] YANG Y,LI J M,WANG X,et al. Analyzing the interactions of mRNAs,miRNAs,lncRNAs and circRNAs to predict competing endogenous RNA networks in glioblastoma[J]. J Neurooncol,2018,137(3):493-502. doi:10.1007/s11060-018-2757-0.
[20] LI F,ZHANG L,LI W,et al. Circular RNA ITCH has inhibitory effect on ESCC by suppressing the Wnt/β-catenin pathway[J]. Oncotarget,2015,6(8):6001-6013. doi:10.18632/oncotarget.3469.
[21] ZHANG H,ZHU L,BAI M,et al. Exosomal circRNA derived from gastric tumor promotes white adipose browning by targeting the miR-133/PRDM16 pathway[J]. Int J Cancer,2019,144(10):2501-2515. doi:10.1002/ijc.31977.
[22] ZHOU C,MOLINIE B,DANESHVAR K,et al. Genome-wide maps of m6A circRNAs identify widespread and cell-type-specific methylation patterns that are distinct from mRNAs[J]. Cell Rep,2017,20(9):2262-2276. doi:10.1016/j.celrep.2017.08.027.
[23] LEI M,ZHENG G,NING Q,et al. Translation and functional roles of circular RNAs in human cancer[J]. Mol Cancer,2020,19(1):30. doi:10.1186/s12943-020-1135-7.
[24] HOU X S,HAN C Q,ZHANG W. MiR-1182 inhibited metastasis and proliferation of ovarian cancer by targeting hTERT[J]. Eur Rev Med Pharmacol Sci,2018,22(6):1622-1628. doi:10.26355/eurrev_201803_14569.
[25] ROD?N L,GONZ?LEZ-JUNC? A,INDA M M,et al. Active CREB1 promotes a malignant TGFβ2 autocrine loop in glioblastoma[J]. Cancer Discov,2014,4(10):1230-1241. doi:10.1158/2159-8290.CD-14-0275.
(2023-02-22收稿 2023-05-16修回)
(本文編辑 魏杰)
