嗜热菌ToPif1解旋酶与G四链体的互作活性位点预测及鉴定
2022-11-09张玲玲戴阳雪奚绪光
张玲玲,戴阳雪,奚绪光
(西北农林科技大学 生命科学学院,陕西 杨凌 712100)
富含鸟苷酸的DNA序列可以折叠成G四链体(G4),它们是通过Hoogsteen键连接在一起并通过单价阳离子进一步稳定的核酸二级结构[1]。生物信息学研究表明,人类基因组高度富含潜在的G4形成序列[2],这些序列存在于DNA启动子、复制起点、减数分裂双链断裂热点、核糖体DNA和端粒区域[3]。G4形成可以调节重要细胞过程,包括DNA复制、转录和翻译[4];但稳定的G4结构会作为物理屏障阻碍复制叉的运动,并导致基因组不稳定。细胞中进化有专门的解旋酶特异性调控并解旋G4二级结构,揭示解旋酶解旋G4的分子机制至关重要。
在细胞中,G4结构在折叠和展开状态之间动态波动[5],而G4的展开主要由G4解旋酶调控。目前,已报道的G4解旋酶包括RecQ家族[6]、DExH/D家族RNA解旋酶[7]以及本文研究对象Pif1家族DNA解旋酶[8]。根据解旋酶的基序及易位方向,Pif1被归类为超家族1B解旋酶。遗传和生化研究表明,Pif1广泛参与细胞代谢过程,包括维持线粒体DNA[9]、负调控端粒酶[10]、参与冈崎片段处理[11]以及在断裂诱导复制过程中结合聚合酶Polδ并修复相关的DNA合成[12]。Pif1还可以通过有效解旋G4而抑制由G4基序引发的基因组不稳定,防止细胞出现复制叉损伤、异常表观遗传沉默、总染色体重排等现象[13]。此外,最近研究发现,Pif1解旋酶在哺乳动物细胞的断裂诱导复制(BIR)中具有关键作用,Pif1依赖性BIR对肿瘤抑制很重要,推测Pif1可以作为抗癌靶点[14]。
相比于单链DNA、双链DNA和带有部分单链的双链DNA底物,Pif1解旋酶优先结合G4 DNA,表明其具有G4作用特异性[15]。Pif1与G4互作的相关研究表明,当Pif1蛋白浓度较高时,酿酒酵母Pif1先以单体形式解开G4结构,再在ss/dsDNA连接处发生二聚化,以二聚体形式解开下游的dsDNA[16];当Pif1浓度较低时,它以单体形式解开G4结构,且呈现出重复展开、重折叠的解旋机制[17-18]。Hou等利用单分子技术研究发现,Pif1以反复连续形式解开G4结构,并有G三链体中间态出现[19]。目前还没有在原子水平上研究Pif1解旋G4活性氨基酸位点的相关报道,而这才是药物靶点设计的重要依据。
考虑到解旋酶的热稳定性影响其在体内较高温度下与G4的互作,本研究选用嗜热细菌ThemusoshimaiPif1(ToPif1)为研究对象,在确定ToPif1在体外具有耐热性以及在高温条件下还保持有效解旋活力的前提下,重点探讨ToPif1参与G4 DNA结合与解旋的关键氨基酸位点,为Pif1家族解旋酶相关抗癌药物的靶点设计提供重要实验依据。
1 材料与方法
1.1 材料与试剂
pET15b-Sumo-ToPif1质粒、pET15b-Sumo载体、E.coli2984克隆菌株及E.coliC2566H表达菌株感受态细胞均由课题组前期制备保存;蛋白质SusPif1、BsPif1、Sumo蛋白酶由课题组前期纯化并保存;PrimeSTAR HS DNA聚合酶及EcoRⅠ、XhoⅠ限制性核酸内切酶购自NEB公司;T4 DNA连接酶购自宝生物工程(大连)有限公司;Ni-NTA亲和层析柱、Heparin纯化柱购自GE公司;其他试剂均为进口分析纯。
1.2 DNA底物制备
核酸底物由生工生物工程(上海)有限公司合成,底物序列如表1所示。采用退火缓冲液(pH为7.5的25 mmol/L Tris-HCl、100 mmol/L NaCl)将底物稀释至2 μmol/L,95 ℃水浴加热5 min后缓慢降至室温,于-20 ℃保存备用。

表1 核酸底物序列Tab.1 Nucleic acid substrate sequences
1.3 ToPif1的嗜热特性检测
利用圆二色谱(circular dichroism,CD)技术检测ToPif1、嗜热菌Sulfurihydrogenibiumsp.YO3AOP1 Pif1(SusPif1)及常温拟杆菌Pif1(BsPif1)蛋白质结构的耐热性。当蛋白质CD峰谱的典型特征峰发生变化时,所对应的温度即为蛋白质变性温度,该温度越高表明蛋白质的耐热性越强。
采用快速停留荧光共振能量转移(stopped-flow FRET)技术测定ToPif1、SusPif1在37 ℃和50 ℃条件下的解旋活性,分析ToPif1在高温下的解旋活力。
1.4 ToPif1-G4互作预测、突变体载体构建及原核表达纯化
根据ToPif1晶体结构[20],利用AlphaFold2模拟ToPif1解旋酶与G4结构的位点特异性互作,确定空间位置上可能与G4结构结合的碱性氨基酸位点。将ToPif1的潜在互作位点分别突变为不带电荷的丙氨酸,并通过原核表达系统表达纯化,得到8个单点突变体蛋白。
以pET15b-Sumo-ToPif1质粒为模板,采用PCR方法构建8个单点突变体表达载体,进行菌落PCR和EcoRⅠ、XhoⅠ酶切鉴定并测序。将构建正确的8个突变体重组质粒分别转化到表达菌株E.ColiC2566H感受态中,于18 ℃条件下诱导表达。使用裂解缓冲液(pH 7.5的20 mmol/L Tris-HCl、500 mmol/L NaCl、5%甘油、5 mmol/L咪唑)重悬菌体,利用低温高压和超声波裂解细胞。裂解液经12 000 r/min高速离心后,使用Ni-NTA亲和层析柱初步纯化,利用Sumo蛋白酶切除Sumo-His6标签。酶切后的蛋白溶液进行低盐缓冲液(pH 7.5的20 mmol/L Tris-HCl、50 mmol/L NaCl、5%甘油、2 mmol/L EDTA)透析,并采用Heparin柱进一步纯化,使用AKTA-purifier系统进行梯度洗脱。利用10% SDS-PAGE检测突变体蛋白质的纯度,30 kDa超滤管浓缩蛋白,于-80 ℃保存备用。
1.5 ToPif1突变体的ATP水解活性测定
利用ATPase/GTPase Activity Assay Kit(Sigma-Aldrich,美国)测定ToPif1及其突变体的ATP水解活性,建立标准曲线并在同一波长下检测ToPif1及其突变体的吸光度。反应体系包括400 mmol/L ATP、1 mmol/L S18H12底物、100 nmol/L蛋白质,反应时间为30 min,空白对照中仅包含相同浓度的ATP和DNA底物S18H12。根据标准曲线计算突变体通过水解ATP产生的磷酸量,以单位时间内产生的磷酸量表示蛋白质的ATP水解速率,即ATP水解活性。测定重复3次,结果以平均值±标准差的形式进行表示。
1.6 G4构型检测及ToPif1突变体对G4的结合活性测定
利用MOS450/AF-CD光学系统(Bio-Logic,法国)检测G4CEB及G4Tel的构型,在缓冲液(pH 7.5的20 mmol/L Tris-HCl、50 mmol/L NaCl)中检测浓度为2 μmol/L的G4底物,记录220~320 nm之间的光谱信号。利用Infinite F200/M200型多功能酶标仪测定ToPif1及其突变体对FAM荧光标记G4CEB、G4Tel的结合活性。反应缓冲液为pH 7.5的20 mmol/L Tris-HCl、50 mmol/L NaCl、2 mmol/L MgCl2,反应体系包含5 nmol/L FAM荧光标记的DNA底物、0.2 mmol/L ADP·AlF4和不同浓度的ToPif1突变体蛋白。反应体系在37 ℃条件下孵育2 min,测定各向异性值,测定重复3次,结果以平均值±标准差的形式进行表示。利用如下公式计算得到平衡解离常数Kd:
Δr=Δrmax×P/(Kd+P)。
式中:Δr表示各向异性值;Δrmax表示各向异性值的最大差值;P表示蛋白质浓度。Kd值越小,表示蛋白质和底物的亲和力越高。
1.7 ToPif1突变体的解旋活性测定
利用stopped-flow FRET技术检测ToPif1及其突变体对双链DNA(S26D17)和G4底物(S12-G4CEB-D12和S26-G4Tel-D12)的解旋活性(底物序列见表1)。反应缓冲液为pH 7.5的20 mmol/L Tris-HCl、50 mmol/L NaCl、2 mmol/L MgCl2,反应体系包含4 nmol/L底物、100 nmol/L蛋白质和1 mmol/L ATP。检测解旋动力学时采用双注射器模式,蛋白质和底物混合后在37 ℃条件下孵育5 min并注射到S3通道,ATP单独孵育5 min并注射到S4通道,反应通过二者快速瞬时结合由ATP激发蛋白解旋活性。通过监测荧光信号得到解旋反应曲线,计算解旋速率和幅度以表征蛋白质的解旋活性。每个反应测定3次,结果以平均值±标准差的形式进行表示。
2 结果
2.1 ToPif1解旋酶的嗜热性质
根据蛋白质二级结构的峰谱检测结果(图1),对比ToPif1、SusPif1、BsPif1结构的耐热性。结果显示,源于嗜热菌的ToPif1和SusPif1的二级结构在温度高于80 ℃以上时才发生变性,而常温菌BsPif1在48 ℃时就已经发生变性,表明ToPif1和SusPif1的蛋白质结构在体外条件下具有很强耐热性,该结果与李丹等的研究结果一致[21]。对比嗜热菌ToPif1、SusPif1在37 ℃和50 ℃条件下的解旋活性(图2),发现SusPif1在50 ℃条件下完全丧失解旋活力,而ToPif1仍保持有一定的解旋活力,说明ToPif1解旋酶具有独特的嗜热性质,可以以ToPif1为研究对象分析嗜热细菌Pif1解旋酶结合、解旋G4结构的关键氨基酸位点。

图1 ToPif1、SusPif1和BsPif1的变温CD结果Fig.1 Variable temperature CD results of ToPif1, SusPif1 and BsPif1注:网络版为彩图。

图2 ToPif1、SusPif1在37 ℃和50 ℃条件下的解旋曲线(a)与解旋速率及幅度(b)Fig.2 Unwinding curves (a) and unwinding rates and amplitudes (b) of ToPif1 and SusPif1 at 37 ℃ and 50 ℃注:网络版为彩图。
2.2ToPif1与G4 DNA的互作位点预测
根据ToPif1晶体结构[20],采用AlphaFold2对ToPif1解旋酶和G4结构的互作位点进行预测,结果如图3所示。在空间位置上可能会与G4结构发生相互作用的碱性氨基酸位点有8个,分别为R135、R139、R150、R159、K329、R355、R392、R419。图3a中的各结构域颜色与图3b一致。
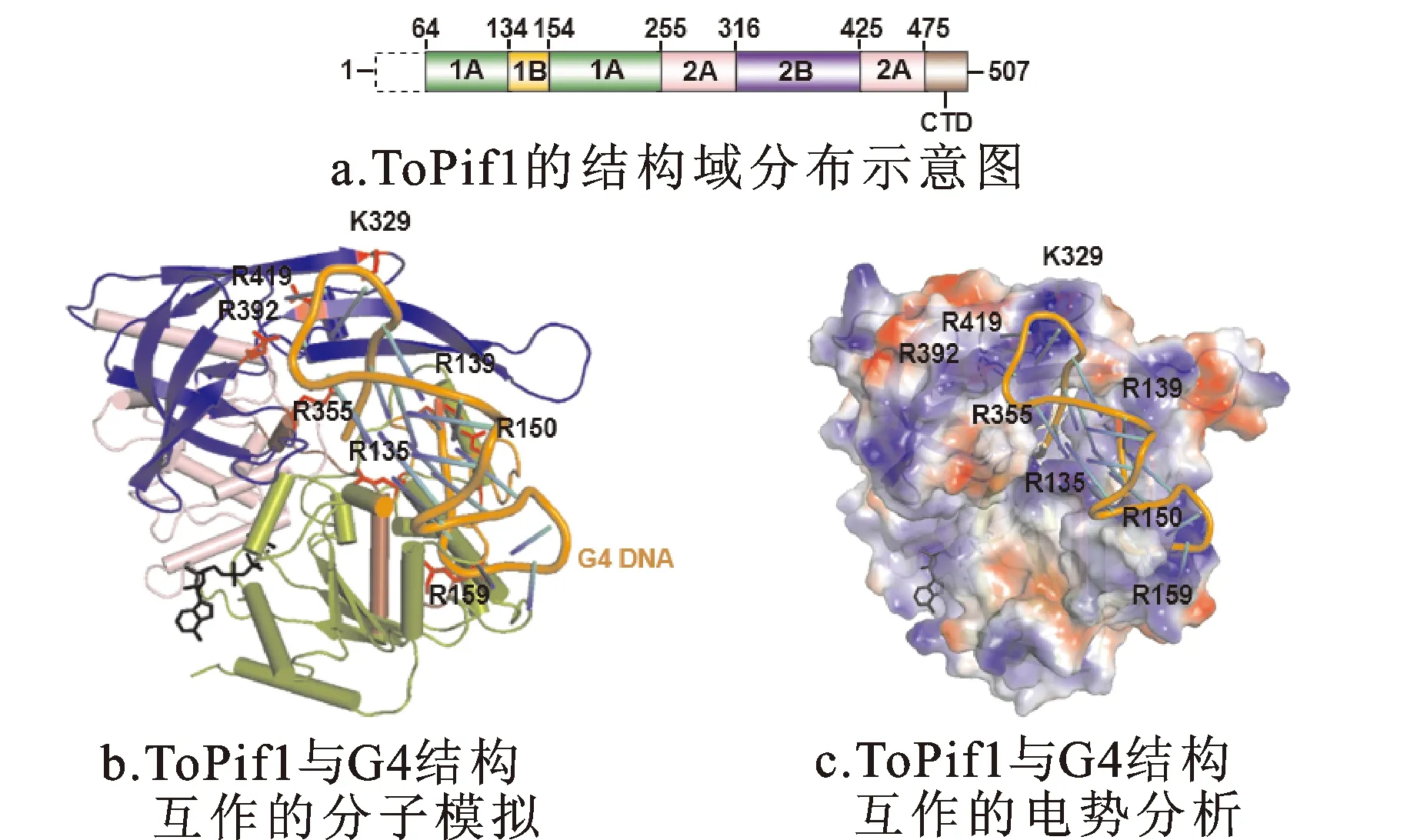
图3 ToPif1与G4结构位点特异性互作的预测结果Fig.3 Prediction results of site-specific interaction between ToPif1 and G4 structure注:网络版为彩图。
2.3 ToPif1突变体的蛋白纯化
将预测的8个潜在互作位点分别突变为丙氨酸,并构建到pET15b-Sumo载体中重组表达。图4为各突变体的纯度检测结果,所有蛋白质的纯度都高于95%,且可溶性良好、性质稳定,表明这些位置的单点突变对蛋白质的表达与纯化没有影响,可用于后续实验。

图4 ToPif1突变体的蛋白纯化结果Fig.4 The protein purification results of ToPif1 mutants
2.4 ToPif1突变体的ATP水解活性
为验证突变体设计对ToPif1解旋酶活性的影响,对ToPif1野生型及其突变体的ATP水解速率进行检测,结果如图5所示。与野生型相比,8个单点突变体蛋白的ATP水解速率均没有发生较大变化,表明这些位点的单点突变不会破坏ToPif1解旋酶的基本ATP水解活性。
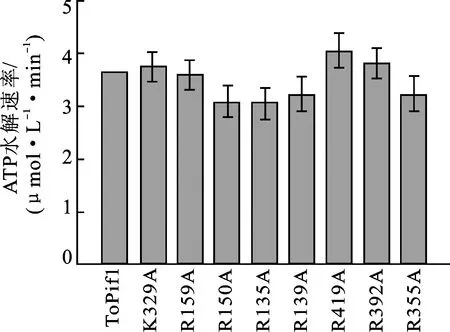
图5 ToPif1及其突变体的ATP水解活性Fig.5 ATP hydrolysis activities of ToPif1 and its mutants
2.5 ToPif1突变体对不同构型G4 DNA的结合活性
采用圆二色谱技术检测G4CEB和G4Tel的构型,结果如图6所示,G4CEB是平行构型,G4Tel是反平行构型。对ToPif1及其突变体与G4CEB和G4Tel的结合活性进行测定,结果如图7所示。由蛋白质与G4 DNA底物结合的Kd值可以看出:对于平行构型的G4CEB底物, 突变体R139A、R355A的Kd值远大于ToPif1野生型,表明R139、R355位点是ToPif1结合G4CEB的重要氨基酸位点;而R135、R150、R159、R355是ToPif1结合反平行构型G4Tel底物的关键氨基酸位点。以上结果说明ToPif1结合不同构型G4所涉及的氨基酸位点不同。对比分析发现,R355位点对G4CEB和G4Tel的识别均至关重要,表明R355具有识别平行和反平行G4的双重功能。

图6 G4CEB和G4Tel的CD谱图Fig.6 CD spectrogram of G4CEB and G4Tel

图7 ToPif1及其突变体与G4的结合活性Fig.7 Binding activities of ToPif1 and its mutants with G4注:网络版为彩图。
2.6 ToPif1突变体的解旋活性
Pif1解旋酶发挥解旋功能需要ssDNA作为加载链,因此设计G4底物的5′端带有一段ssDNA,3′端带有一段较短的dsDNA,以模拟G4结构导致的复制叉停滞状态。本研究中共设计了2种带有不同构型G4的解旋底物,分别为S12-G4CEB-D12和S26-G4Tel-D12。通过对比ToPif1野生型和突变体的解旋活性差异,确定ToPif1上影响不同构型G4底物解旋的特异性氨基酸位点。同时,为明确突变体对上述底物解旋活性减弱的途径(通过影响G4结构还是其下游的dsDNA),进一步测定了突变体对S26D17的解旋活性,结果如表2所示。图8展示了对S12-G4CEB-D12和S26-G4Tel-D12底物解旋均具有较大影响的突变体R135A、R139A和R355A的解旋活性。由表2可以看出,与ToPif1野生型相比,上述3个突变体对S12-G4CEB-D12和S26-G4Tel-D12的解旋速率都明显降低。值得注意的是,突变体R139A对S26D17的解旋活性大大减弱,而其余突变体则与野生型相差不大。这表明R139是影响dsDNA解旋的氨基酸位点,而R135和R355是影响平行构型G4CEB和反平行构型G4Tel解旋的重要氨基酸位点,2个位点在ToPif1解旋不同构型G4 DNA时都发挥着关键作用。此外,R150位点仅对S12-G4CEB-D12解旋具有显著影响(图9),表明该位点只参与平行构型G4的解旋。
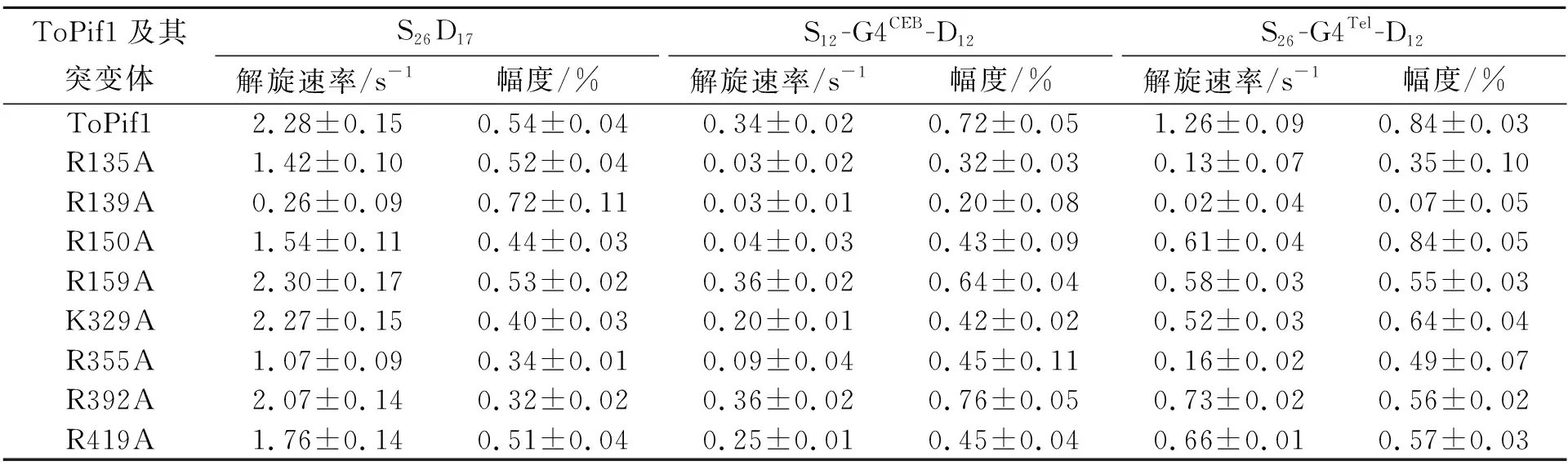
表2 ToPif1及其突变体对dsDNA和G4底物的解旋速率及幅度Tab.2 Unwinding rates and amplitudes of ToPif1 and its mutants on dsDNA and G4 substrates

图8 ToPif1及其突变体对S12-G4CEB-D12和S26-G4Tel-D12的解旋活性Fig.8 Unwinding activities of ToPif1 and its mutants on S12-G4CEB-D12 and S26-G4Tel-D12注:网络版为彩图。

图9 R150A对S12-G4CEB-D12的解旋活性Fig.9 Unwinding activities of R150A to S12-G4CEB-D12注:网络版为彩图。
3讨论
本研究首先通过热变温CD技术检测了嗜热菌ToPif1、SusPif1及常温拟杆菌BsPif1蛋白质二级结构的耐热性,发现嗜热菌ToPif1、SusPif1的二级结构在温度高于80 ℃时才发生变性,这与李丹等报道的嗜热性黄石热脱硫弧菌Pif1(TyPif1)解旋酶的二级结构具有耐高温特性类似[21]。此外,通过stopped-flow FRET技术测定ToPif1、SusPif1解旋酶在37 ℃及50 ℃条件下的解旋活性,发现50 ℃时SusPif1彻底丧失解旋活性,而ToPif1可以保持一定的解旋活性,表明ToPif1解旋酶具有独特的嗜热性质。
ToPif1是目前唯一解析出结构的嗜热细菌Pif1解旋酶,将ToPif1与目前已知结构的所有非嗜热Pif1(BsPif1[22]、ScPif1[23]、Human Pif1[24])进行比对(图10a),发现2B结构域的角度存在显著差异。其中,ToPif1与Human Pif1呈现较为闭合的状态,而ScPif1和BsPif1呈现向上打开的状态,闭合状态会使蛋白质的三维结构更加致密,这可能是ToPif1结构具有嗜热性的原因之一。此外,2B结构域是解旋的关键区域[20],该结构域的闭合状态可能与解旋的动力学机制存在关联。结合ToPif1高温环境下的解旋活性,有理由认为ToPif1的嗜热特性与2B结构域的闭合状态相关。进一步比对上述4个Pif1的氨基酸序列,发现ToPif1与其他非嗜热Pif1差异较大的区域为150~155和289~294区域。分析2个区域的结构可以看出,150~155区域在人类和酵母中呈现α螺旋,而在细菌中则为无规则卷曲,说明ToPif1在该区域与细菌的Pif1结构较为类似(图10b),这与ToPif1来源于嗜热菌的物种类型相符;ToPif1在289~294区域为α螺旋结构,而其他非嗜热Pif1在该区域均为无规则卷曲(图10c),由于该位置处于较为保守的2A结构域内,推测该区域的差异可能与ToPif1的嗜热特性相关。

图10 ToPif1与非嗜热性Pif1的整体结构(a)、150~155区域(b)及289~294区域(c)对比Fig.10 Comparison of the overall structures (a), regions 150~155 (b) and regions 289~294 (c) of ToPif1 and non-thermophilic Pif1注:网络版为彩图。
近年来,随着G4结构与人类疾病的密切关联性被逐渐揭示,G4特异性解旋酶也被广泛研究。目前,研究最为成熟的G4解旋酶之一是超家族Ⅱ(SF Ⅱ)的DHX36,它对平行构型的G4结构具有偏好性[25]。另有研究报道嗜热厌氧杆菌Ana.Pif1解旋酶的核心结构域蛋白对G4底物具有很高的亲和力[26]。本研究通过测定ToPif1解旋酶对平行构型G4CEB和反平行构型G4Tel的结合活性,发现ToPif1对平行及反平行构型的G4 DNA都具有很高的亲和力,表明该蛋白结合G4时对其构型不具有偏好性。然而,在ToPif1解旋S12-G4CEB-D12和S26-G4Tel-D12底物时,ToPif1对G4Tel的解旋速率远大于G4CEB,表明Pif1解旋酶可能偏好于解开反平行构型的G4 DNA。由此可见,ToPif1对G4结构的结合及解旋性质与DHX36存在较大差异,SF Ⅰ和SF Ⅱ的G4解旋酶可能具有不同的G4识别与解旋机制。
先前研究揭示了DHX36结合及解旋G4结构的机理[27],DHX36通过N末端的18个氨基酸结构域(RSM基序)识别平行G4结构,而RSM基序通过覆盖外部G-四分体并利用3个带正电荷的氨基酸与带负电荷磷酸基团的静电作用夹住G4[28]。本研究成功预测并验证了ToPif1结合与解旋G4 DNA的关键氨基酸位点,为揭示Pif1蛋白解旋G4结构的机理提供了参考。通过测定ToPif1野生型及其突变体与G4CEB、G4Tel的结合活性,发现参与不同构型G4结合的关键位点存在差异,但R355位点对平行和反平行构型G4的结合均至关重要。 通过ToPif1野生型与突变体之间的G4解旋活性差异,证明了R135和R355是ToPif1解旋G4CEB和G4Tel的关键氨基酸位点,R355位点在ToPif1解旋酶结合、解旋平行和反平行构型G4 DNA的过程中都发挥着关键作用。对比不同物种Pif1解旋酶的氨基酸序列(图11)可以看出,R135不具有保守性,但R355在多个细菌甚至酵母和人类中都高度保守,推测该位点是Pif1蛋白家族解旋G4 DNA的保守性氨基酸位点。

图11 细菌、酵母和人类的Pif1序列比对Fig.11 Sequence alignment results of Pif1 in bacteria, yeast and human注:ESPript网站预测。网络版为彩图。
4 结论
本研究首次证明了嗜热菌ToPif1在体外高温条件下仍可保留蛋白构象及活力的嗜热特性,为后续在体内进行G4-Pif1的互作研究提供了热稳定性较强的研究材料。利用AlphaFold2预测出8个ToPif1与G4 DNA互作的特异性位点,并通过原核表达系统纯化得到8个ToPif1的单点突变体蛋白,8个位点的单点突变均未显著影响ToPif1的ATP水解活力。各向异性实验及ToPif1解旋活力测定表明,R355位点在ToPif1与G4结构的结合中具有重要功能,而R135和R355位点显著影响G4CEB和G4Tel的解旋,这不仅为Pif1解旋酶与G4结构的互作研究提供了思路,也为揭示嗜热细菌Pif1解旋G4结构的机理提供了参考。
