单分子技术研究T 7解旋酶的解旋与换链∗
2018-06-19陈泽马建兵黄星榞贾棋徐春华张慧东陆颖
陈泽 马建兵 黄星榞 贾棋 徐春华张慧东 陆颖‡
1 引 言
解旋酶是一类在细胞内广泛存在的分子马达蛋白,种类繁多.不同解旋酶具有不同的功能及特性,在脱氧核糖核酸(DNA)复制、转录、修复、重组以及端粒稳定等诸多方面都发挥着不可忽视的作用[1−3].人体内的诸多过程都涉及数量庞大的生物分子,常规的实验方法仅能检测初始态和终态的平均效应结果,不仅仅忽略了更为关键的中间过程,也缺少对于生物个体差异性和多样性的分析.单分子技术是近些年发展起来的一种先进实验技术,能够用于针对单个生物分子的研究,以得到常规技术无法得到的诸多重要信息[4].近些年来,单分子技术越来越多地运用到解旋酶的相关研究中,使得科研人员对于解旋酶的功能和机制的认知越来越清晰.解旋酶在解旋过程中进行的换链现象是其功能中非常重要的一项,这种换链现象对于解旋酶而言,能够在其遭遇各类DNA损伤时,避免发生停滞而导致复制体崩解,乃至细胞死亡;同时,能够以此方式消除结合在DNA上的内源性中间产物及单链DNA结合蛋白,确保解旋过程的顺利进行[5];最后,换链现象还能够直接参与到同源重组的过程中,并发挥作用.因此,深入研究解旋酶进行换链的机制具有重要意义和价值.
目前运用单分子技术的相关研究表明,诸如Pif1[6],RecQ[7],BLM[5]及UvrD[8]等非环状解旋酶在解旋过程中是存在换链现象的.研究Pif1换链过程的文献[6]提及,其发生换链的原因在于Pif1可能与3′-尾链之间存在一个相互作用,这种相互作用会将Pif1从5′-链拉至3′-链, 并在此链上继续行走,而解开部分的单链DNA退火重新形成双链DNA.对于六聚体解旋酶而言,因其6个子单位形成环状结构,人们总是直观地认为这种结构会稳定地存在于单链DNA上而无法自由脱落下来.基于这种猜想,科研人员认为六聚体解旋酶是通过从单链DNA的端口处直接穿入的方式进行加载,并进一步结合在单链DNA上进行行走.如果是这种加载方式,六聚体解旋酶无疑是无法发生换链过程的.然而,不论是磁镊(magnetic tweezers,MT)实验还是光镊实验[9,10],单链DNA的两端均被固定,不存在所谓的自由端口.而在溶液中已形成六聚体的解旋酶依旧能够直接加载在单链DNA上进行行走和解旋,说明这种加载方式并不准确.因此,本文以此作为切入点,以T7解旋酶为研究对象,力求揭示六聚体解旋酶是否能够进行换链的问题.
本文主要采用的单分子技术为单分子荧光共振能量转移(single-molecule fl uorescence resonance energy transfer,smFRET)和MT.荧光共振能量转移(FRET)是一种通过两个荧光基团之间无辐射的偶极耦合的相互作用来进行能量转移的方式.能量通常由供体荧光基团(短波长)传递给受体荧光基团(长波长).本文用于smFRET的仪器装置为常规的全内反射显微系统,为消除其余因素对于信号的影响,生物分子被固定在玻片表面.MT技术是将单个生物分子通过相应修饰后使其一端连接在玻片表面,另一端连接在超顺磁性小球表面.磁球位于外磁场中而受到恒定磁力的作用,通过改变磁场强度(平移或者旋转装置上磁铁)可以实现拉伸或旋转磁球等操纵,从而实现对于单个生物分子的力学操纵.本文采用常规的纵向MT开展研究.
通过运用smFRET和MT技术,本文能够实时地观测DNA底物被T7解旋酶解开的动态过程,进而能够得到解旋过程的具体信息,便于分析解旋酶的解旋机制.运用smFRET技术研究解旋反应能够观测的解旋长度较短(<10 bp),但是其精度非常高,对于精确研究较小尺度内的解旋过程和机制有得天独厚的优势.本文研究的主要问题是六聚体解旋酶的换链现象,因此采用smFRET技术能够更精确地观测此过程;运用MT技术研究解旋反应能够观测的解旋长度较大,但是其精度比不上smFRET技术.本文运用MT技术在大尺度上观测这种换链现象是否依旧存在,并进一步分析施加力的作用是否会对其产生影响.因此,本文同时采用了smFRET和MT两种技术进行实验,以达到研究目的.
解旋实验通过改变荧光标记DNA的方法以及改变GC含量和脱氧胸苷三磷酸(dTTP)浓度等实验条件,深入地探究了T7解旋酶是否如单体解旋酶一样存在换链过程.实验结果表明T7解旋酶的解旋工作需要3′-尾链的参与,且DNA序列中GC含量的提高能够导致其更易发生回退;在所有的回退现象中存在着一种缓慢回退的现象,我们推测该现象为六聚体解旋酶的换链过程,并提出其模型.
2 实验方法和材料
2.1 玻片的清洗与修饰
先后用丙酮、甲醇、浓硫酸和过氧化氢混合液(体积比7:3)及乙醇钠对FRET实验用盖玻片和载玻片进行彻底清洗,盖玻片需要进一步进行硅烷化和聚乙二醇(PEG,购于Laysan Bio,Inc.公司)修饰,具体方法参照文献[11];MT实验用盖玻片和载玻片清洗方法同于FRET实验用玻片,后期需要进一步用Sigmacote(购于Sigma-Aldrich公司)修饰,具体方法参照文献[12].
2.2 DNA设计
单分子FRET实验用DNA共5种:一种是不存在3′-尾链的双链DNA底物,于双链岔口标记Cy3和Cy5荧光分子,序列中GC含量设计为70%;一种是3′-尾链为单链结构的双链DNA底物,标记方法与前相同,序列中GC含量设计为70%;另外3种3′-尾链均为双链但留有1 nt单链结构的双链DNA底物,Cy3标记在双链上第8位,Cy5标记在岔口,序列中GC含量分别设计为50%,70%和100%.所有DNA底物均通过在一条链尾端修饰生物素连接在玻片上.DNA底物均购于生工生物工程(上海)股份有限公司,详细制备方法见文献[13].MT实验用DNA是轮廓长度为0.5µm带有270 bp发卡结构的双链DNA,详细制备方法见文献[6].
2.3 蛋白质及缓冲液
T7 gp4解旋酶蛋白由陆军军医大学毒理学研究所张慧东课题组纯化完成,具体方法见文献[14,15].蛋白纯化完成后,在盛有储存液(50 mM(1 M=1 mol/L)K-phosphate,0.1 mM乙二胺四乙酸(EDTA),0.1 mM二硫苏糖醇(DTT)及50%甘油)的烧杯中进行透析,并于−30◦C密封保存.实验所用的缓冲液包括:T50缓冲液(50 mM NaCl及20 mM Tris-HCl,pH值为8.0),反应缓冲液(10 mM MgCl2,50 mM NaCl,5 mM DTT及20 mM Tris-HCl,p H值为8.0)以及磷酸缓冲盐溶液(PBS缓冲液)(20 mM Na2HPO4,150 mM NaCl,p H值为8.0).缓冲液配置后需要进行高温消毒,再经孔径为0.2µm的过滤器过滤,最后于−20◦C密封保存.
2.4 解旋实验方法
MT实验中,首先取磁球原液10µL,加入20µL PBS缓冲液并用移液枪吹打清洗,随后用磁铁吸引磁球去除上清液,反复操作3次,最后稀释至20µL.随后,取1µL 100 p M的DNA底物(带有270 bp发卡结构)与磁球稀释液充分混匀1 min.将上述混合液泵入实验槽中,孵育10 min使得磁球充分连接在玻片表面,并冲走多余未连接磁球.具体的磁球尺寸、修饰方法及操作方法见文献[6].通过检测连接磁球上的DNA底物为单根且较为稳定后,开始进行下一步解旋实验.首先将高浓度gp4单体解旋酶与2 mM d TTP混匀孵育10 min,使其充分形成六聚体结构.随后在上述混合液中加入T50缓冲液及DTT,通过恒流泵泵入槽中,孵育10 min使得T7解旋酶充分结合在DNA底物上,且因没有镁离子而无法启动解旋反应.当孵育完成后,泵入T50缓冲液充分冲掉槽中多余的解旋酶,确保后续为单个解旋酶进行反应.最后,泵入含镁离子的反应缓冲液,实验浓度d TTP及DTT启动解旋反应,收集实验数据;smFRET实验中,首先取1µL链霉亲和素原液(1 mg/mL),用T50缓冲液稀释至100µL,充分混匀后加入实验槽中孵育5 min.随后冲走多余链霉亲和素,加入约100 pM带荧光标记的DNA底物孵育5 min.随后冲走多余DNA,观察DNA连接情况良好后进行下一步解旋实验,具体的原料来源、修饰方法及操作方法见文献[13].后续解旋实验的步骤与MT实验的步骤保持一致,只是需要在启动解旋反应并收集数据的泵入反应液中加入抗荧光淬灭体系(0.8%D-葡萄糖,1 mg/mL葡糖氧化酶,0.4 mg/mL过氧化氢酶和1 mM水溶性维生素E).
3 结果与讨论
3.1 T 7解旋酶的解旋反应
T7解旋酶是5′-3′方向行走的解旋酶,因此正常的解旋反应需要足够长的5′-尾链用于解旋酶结合.有研究表明,T7解旋酶的解旋反应还需要3′-尾链的存在,认为其与3′-尾链之间存在着某种相互作用[16,17],并发现3′-尾链的单链部分长度必须大于7 nt时,T7解旋酶才能够正常进行解旋反应.那么T7解旋酶是怎样与3′-尾链上DNA进行相互作用的呢?为此,本文设计了一组DNA底物进行实验,其5′-尾链均有30 nt的单链以供解旋酶结合,3′-尾链分别设计为不存在尾链、存在30 nt单链的尾链及存在29 bp双链及1 nt单链的尾链结构,如图1(a),(c),(e)所示.
FRET解旋实验使用的DNA底物均为带有荧光标记的双链DNA,Cy3标记在3′-链,Cy5标记在解旋酶结合的5′-链上.实验是通过532 nm激光激发Cy3发出荧光,并通过能量转移而使得Cy5同样发出荧光.因此,随着解旋酶解开双链DNA,Cy3和Cy5之间距离增大,能量转移效率(FRET值)随之降低.当解旋酶完全解开双链DNA,即解旋到头时,Cy3标记的3′-链会因为双链部分完全打开而脱落,导致Cy3荧光强度和Cy5荧光强度均迅速降低至0,由此现象来判断解旋酶是否解旋到头[18].
通过解旋实验,发现T7解旋酶在无3′-尾链结构参与的条件下无法进行解旋反应,如图1(b)所示,Cy3和Cy5之间距离一直保持不变,FRET值维持为1.说明以往研究中提及的解旋酶与3′-尾链之间存在相互作用的结论是成立的.当缺乏这种相互作用时,解旋酶就无法进行解旋[16,17].3′-尾链设计为单链DNA和双链DNA结构的实验结果如图1(d)和图1(f)所示,当3′-尾链为单链DNA时,因Cy3和Cy5距离很近,初始FRET值为1.随着解旋反应的进行,Cy3和Cy5的距离逐渐变大而导致Cy3荧光强度不断增加,Cy5荧光强度不断降低,FRET值随之不断降低,最终完全解开双链;当3′-尾链为双链DNA时,初始FRET值约为0.9,随着解旋反应的进行与前者有着相似的现象.
本文的实验结果说明3′-尾链的存在对于T7解旋酶进行解旋反应尤为重要,即此解旋酶行使其解旋功能需要DNA底物为叉形结构.由于3′-尾链为单链或双链结构的实验结果并无本质不同,本文推测T7解旋酶或与3′-尾链上磷酸根结构域存在着某种相互作用.
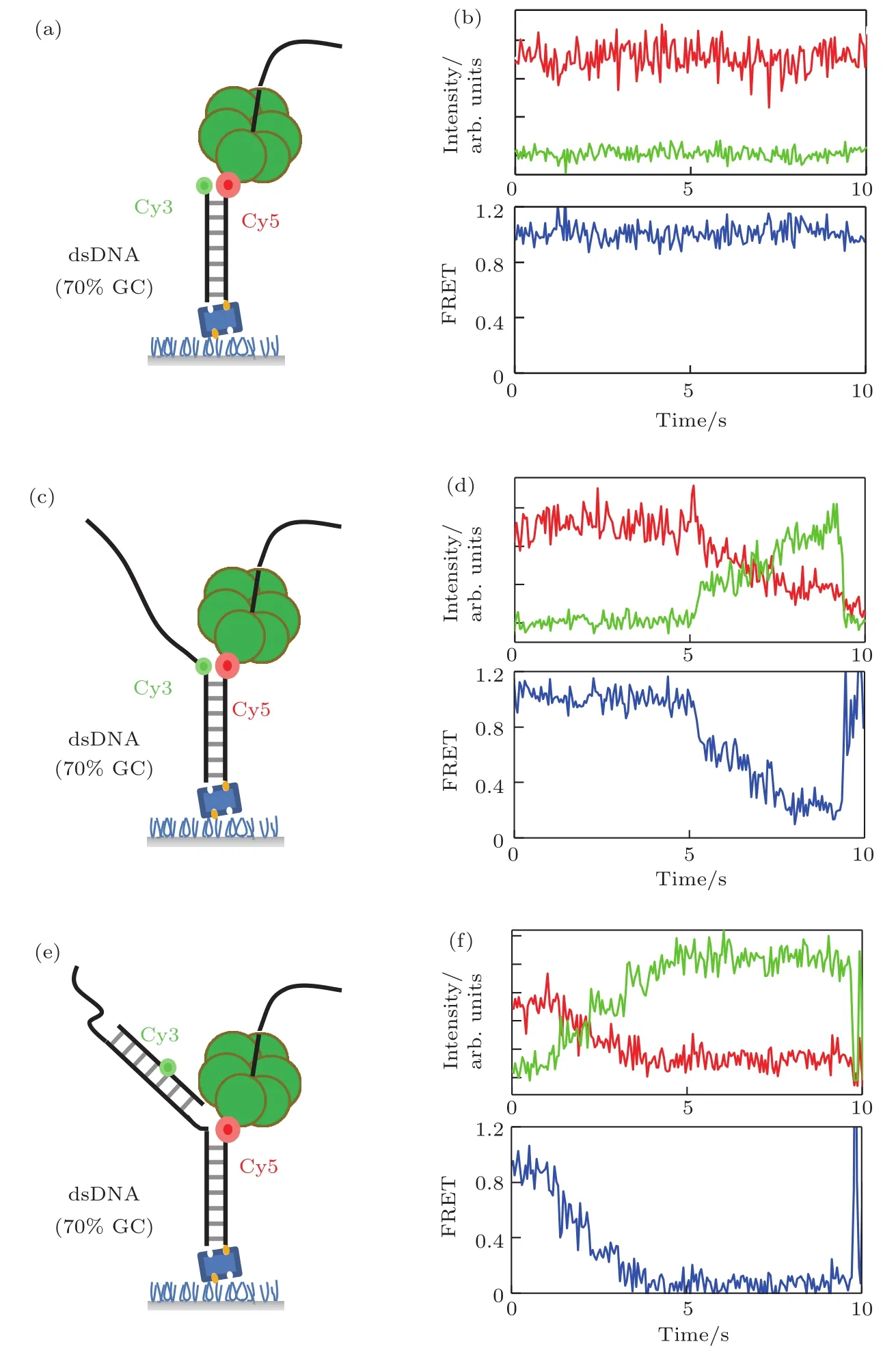
图1 不同3′-尾链结构DNA底物的sm FRET解旋现象 (a)不存在3′-尾链结构时DNA底物的实验示意图;(b)对应(a)图DNA底物的T 7解旋酶解旋数据;(c)3′-尾链为单链DNA结构时DNA底物的实验示意图;(d)对应(c)图DNA底物的T7解旋酶解旋数据;(e)3′-尾链为双链DNA结构时DNA底物的实验示意图;(f)对应(e)图DNA底物的T 7解旋酶解旋数据Fig.1.Observation of DNA with diff erent 3′-tail structures unwinding with smFRET assay:Schematic diagram of the smFRET experiment in DNA substrate with diff erent 3′-tail structures(a),(c)and(e);smFRET-time traces showing from three diff erent structures of DNA,respectively(b),(d)and(f).
3.2 序列中GC含量对T 7解旋酶解旋反应的影响
已有研究表明,解旋部分的DNA序列中GC含量越高,T7解旋酶的解旋速率越慢[18,19].然而,人们并没有关注到这种GC含量的提高是否会影响其解旋长度.常规实验方法受限于技术并不能解决此问题,而以往FRET实验所采用的荧光标记手段,当解旋长度很短时并不能检测到相应FRET值的变化.基于此,本文在实验设计序列中包含50%,70%和100%GC含量的3种DNA底物,采用上述改良的荧光标记方法如图1(c)所示,运用sm-FRET技术进行T7解旋酶的解旋实验,解旋数据和解旋是否到头的判定方法与图1标准保持一致.
本文在设计实验用DNA底物时参考最近运用smFRET技术研究T7解旋酶的文献[18].如文献[18]中所描述的,当DNA序列中GC含量为35%时,解旋速率为8 bp/s;而GC含量为80%时,其解旋速率仅为4 bp/s,这也是其使用的最大GC含量的DNA底物.由文献[18]可知,提高GC含量能够更加明显地观测解旋酶进行解旋的中间过程.因此,本文在设计实验时,为降低实验中的解旋速率以便更加精确地评估其解旋过程,同时为设计一组GC含量变化梯度比较其解旋长度的变化情况,决定将实验中DNA序列的GC含量确定为50%,70%和100%.
另一方面,文献[18]中提及降低d TTP浓度能够降低解旋速率,本文实验通过改变d TTP浓度梯度,发现100µM d TTP浓度条件下能够明显降低解旋酶的解旋速率,便于观测解旋中间过程,且不至于明显影响解旋酶的解旋比例.因此,本文解旋实验均在100µM d TTP浓度条件下进行.
由图2结果可知,当序列中GC含量为50%时,T7解旋酶解旋到头的实验数据比例约为78%,解旋中途回退的比例约为22%;当序列中GC含量为70%时,T7解旋酶解旋到头的实验数据比例约为61%,解旋中途回退的比例约为39%;当序列中GC含量为100%时,T7解旋酶解旋到头的实验数据比例仅约为21%,解旋中途回退的比例约为79%.由此可知,GC含量从50%提高至100%时,会明显提高解旋酶在解旋过程中发生回退的概率,导致其解旋长度降低.
在解旋过程中,这种回退现象发生的前提是解旋酶无法继续进行解旋反应而发生暂停或停滞.然而,这种暂停或停滞现象并不能稳定维持,解旋酶会进一步失去与单链DNA之间的相互作用.此时,解旋酶或直接从单链DNA上脱落下来,解开部分单链自由退火形成双链;或解旋酶虽然缺失与单链DNA的相互作用,但并未脱落,解开部分单链仍然自由退火而形成双链,解旋酶滑退回初始位置.从实验数据上看,当DNA序列为全GC时,T7解旋酶大多数都无法解旋到头.
因此,我们认为DNA序列中GC含量高,不仅会影响解旋酶的解旋速率,还会导致其更易发生暂停或停滞,进而发生回退现象,使得解旋长度明显降低.分析其原因,可能是解旋酶在解旋过程中不仅受到当前解旋位置碱基对之间氢键作用力的限制,后续DNA序列的稳定程度也会对其解旋过程造成影响.当DNA序列中GC含量非常高,乃至为GC含量为100%时,这种稳定程度可能会使得解旋酶难以克服而无法继续往前解旋.
3.3 T 7解旋酶在解旋过程中的回退现象
3.2 节提及当GC含量为100%时,T7解旋酶在解旋过程中大多数无法解旋到头而在中途发生回退.通过进一步分析数据,发现大多数的回退现象是瞬时回去的过程,如图3(a)所示.但是,实验数据中还存在一种缓慢回退的现象,如图3(b)所示.这里对于瞬时回退和缓慢回退两种现象的界定在于前者在回到初始位置的过程中并不存在停顿或过程,分析仅为一步完成;而后者在回到初始位置的过程中存在诸多的停顿或过程,从实验数据上来看,可以观测到许多的数据点存在于这一回退过程中,分析为多步完成.因此,瞬时回退现象并不代表其是一瞬间回到初始位置,相对于缓慢回退来讲,这里的瞬时指的是回退过程中不存在中间步骤,而缓慢指的是回退过程中存在诸多中间步骤.
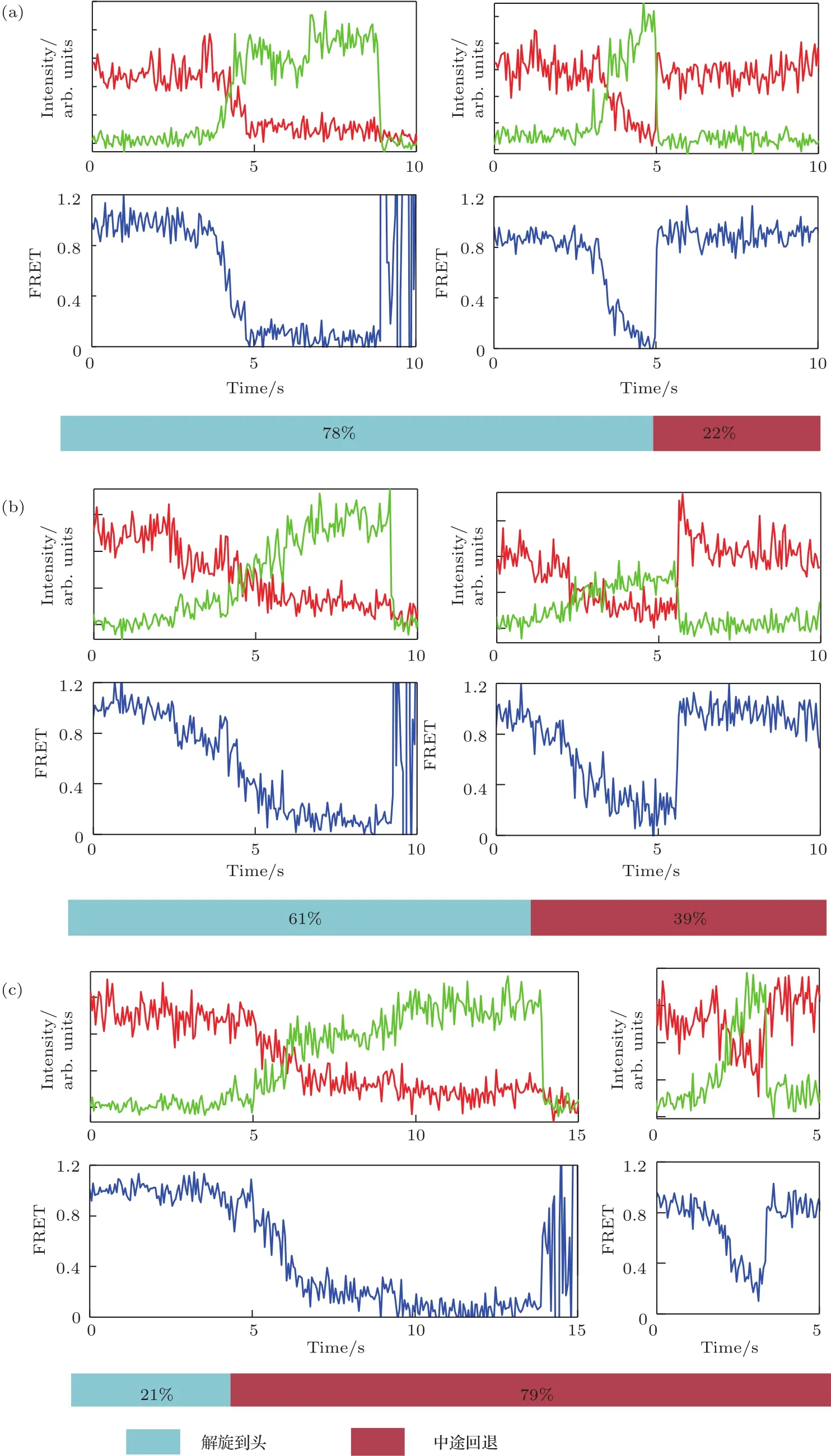
图2 三种不同GC含量DNA底物的T 7 gp4解旋现象 (a)DNA底物GC含量为50%;(b)DNA底物GC含量为70%;(c)DNA底物GC含量为100%Fig.2.Analysis of unwinding processivity of T7 gp4 helicase in DNA substrate with diff erent GC contents:(a)Helicase in DNA substrate with 50%GC content;(b)helicase in DNA substrate with 70%GC content;(c)helicase in DNA substrate with 100%GC content.
通过进一步统计数据可知,所有的实验数据中存在瞬时回退现象的比例约为81%,而存在缓慢回退现象的比例约为19%.对于前者而言,当T7解旋酶无法继续解旋而发生暂停或停滞时,因无法稳定维持此状态,解旋酶或从单链DNA上脱落而解开部分单链因退火压力而自由形成双链;或虽失去与单链DNA的相互作用但继续留在5′-链上,解开部分单链自由退火而形成双链,解旋酶滑退回初始位置.这两个过程都非常快而导致观察几乎是瞬时的,不存在中间步骤.同时,因这两个过程仅仅是T7解旋酶的状态存在区别,DNA底物的状态是一致的,而FRET实验的观测对象是荧光标记DNA,所以无法区分这两种情况,均算入瞬时回退现象中.在图3(a)的时间曲线中,随着T7解旋酶解开双链,FRET值逐渐降低至约0.1.此时,解旋酶因无法继续解旋而在5′-链上滑退回初始位置或直接从DNA底物上脱落下来,解开双链DNA迅速退火重新形成双链DNA而导致FRET值恢复初始值.图3(a)所示实验现象在文献[9]中也有所提及.而对于少数情况下解旋酶发生缓慢回退的现象,以往并没有研究发现.
通过分析相关文献,发现早期研究中,如果改变DNA底物迫使T7解旋酶必须由3′到5′方向进行解旋时,完全无法观察到解旋现象[20−23].因此,T7解旋酶仅能从5′到3′方向进行单向的行走和解旋,而完全无法从3′到5′方向进行行走和解旋,将这种现象称为T7解旋酶的极性,也称为单向性.此外,这种极性不仅仅存在于T7解旋酶,经过研究诸多解旋酶都被证实存在极性,仅能沿着单向进行行走和解旋.因此,反观本文的实验数据,如果这种缓慢回退的现象发生在5′-链,表明T7解旋酶是从3′到5′方向进行行走,这种现象在以往的研究中完全未被观察到,且不符合公认的解旋酶具有极性的结论.因此,本文认为这种缓慢回退的现象是发生在3′-链上,而不可能发生在5′-链上. 换而言之,这一现象预示着T7解旋酶换到了对面的3′-链上,即这个六聚体解旋酶在解旋过程中发生了换链现象.
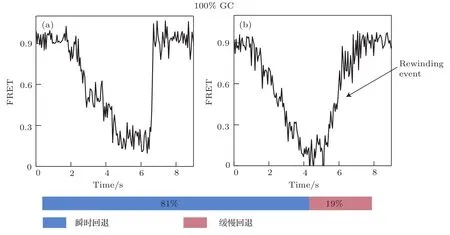
图3 T 7解旋酶的两种回退现象(100%GC) (a)瞬时回退;(b)缓慢回退Fig.3.Two diff erent backward movements of T 7 helicase(100%GC):(a)Rapid rewinding process;(b)slow rewinding process.
3.4 运用M T技术研究T 7解旋酶的缓慢回退现象
基于上述结果和猜想,进一步设计MT实验进行研究,DNA连接方式如图4(a)所示.d TTP浓度及序列中GC含量均保持与FRET实验一致,实验方法参照文献[18]中的步骤,确保均由单个解旋酶进行解旋反应.MT实验中使用的DNA底物解旋部分发卡结构长度为270 bp,随着解旋酶解旋反应的进行,发卡结构逐渐被打开,检测到磁球高度发生相应变化,由此来判断是否为解旋数据.通过计算解旋酶解开的发卡结构长度是否到达270 bp,来判断解旋酶是否完成解旋反应,即解旋到头.
如图4(b)所示,当T7解旋酶解旋到头,即完全解开发卡结构DNA时,其会沿着3′-链继续行走回到初始位置.从数据上来看,这种行走并不是瞬时过程,而是有一定中间步骤的过程.其速度大于在5′-链上进行解旋的速度,可能是由于DNA双链退火压力加快了这一行走速度.当T7解旋酶并没有完全打开发卡结构DNA而停止解旋时,从数据上看多数情况下会瞬时回到初始位置,而少数情况下是一种缓慢回退的过程,如图4(c)和图4(d)所示.运用MT技术进行解旋实验得到的未解旋到头的数据中,瞬时回退现象所占比例约为76%,缓慢回退现象所占比例约为24%,基本与FRET实验得到的结果保持一致.这种未解旋到头且发生缓慢回退的现象区别于图4(b)中的现象,其并不是因为解旋到头而直接移动到3′-链上沿着5′到3′方向行走.如果此时解旋酶依旧位于5′-链上,这种缓慢回退的行走方式并不符合T7解旋酶仅能由5′到3′单向移动的单向性或极性[16,20−25].另一方面,不论是本文实验还是已发表文献中的实验[9,10],已形成环状六聚体的T7解旋酶都能够直接结合在没有5′和3′端口的单链DNA上开始行走和解旋,说明其也能以相同的方式从此单链DNA上脱落下来.同时,MT实验中观测到的这种缓慢回退的现象与FRET实验中观测到的现象基本保持一致,相互印证.由此可知,这种缓慢回退的现象是因为T7解旋酶在解旋过程中发生了换链现象,且这种换链现象与DNA底物以及施加其上的力没有关系,它是T7解旋酶的一种本征性质.
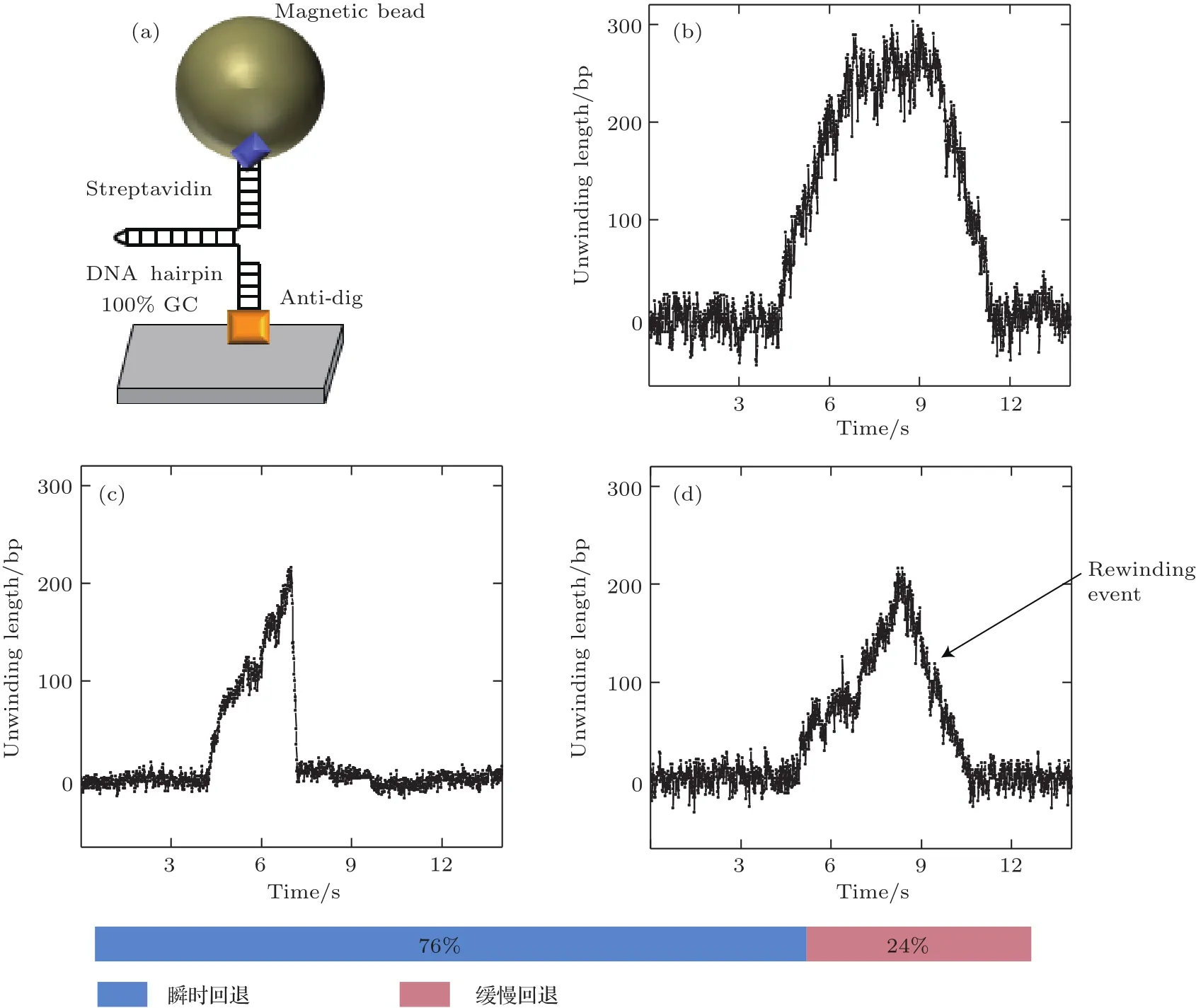
图4 MT实验研究T 7解旋酶的缓慢回退现象 (a)单分子MT实验示意图;(b)T 7解旋酶在解旋到头时,缓慢行走的实验现象;(c)T7解旋酶在未解旋到头时,瞬时回退的实验现象;(d)T7解旋酶在未解旋到头时,缓慢回退的实验现象Fig.4.The slow rewinding process of T 7 helicase by magnetic tweezers:(a)Schematic diagram of the magnetic tweezers assay;(b)slow translocation phenomenon of T 7 helicase after unwinding the whole DNA hairpin;(c)rapid rewinding process of T 7 helicase during unwinding;(d)slow rewinding process of T 7 helicase during unwinding.
3.5 T 7解旋酶换链机制的模型
研究表明,T7解旋酶可能是以一种开环的方式结合在单链DNA上,而非通常认知的环状解旋酶整体从5′端口进入[26].基于此,T7解旋酶在解旋过程中也存在很大可能性发生开环而直接从单链DNA上解离而脱落下来.从本文实验结果和以往的文献结论来看[16,17],T7解旋酶的解旋反应必须有3′-尾链的参与,推测解旋酶在解旋过程中可能与3′-尾链之间存在某种相互作用.综上所述,我们推测这种缓慢回退的现象是T7解旋酶在解旋过程中发生暂停或停滞时,进一步发生换链现象而转移至3′-链上,沿着5′到3′的方向进行行走回到初始位置.
基于此,本文进一步提出了T7解旋酶进行换链过程的模型,如图5所示.图5(a)表示的是T7解旋酶在解旋过程中发生暂停或停滞时无法继续进行解旋的状态.我们推测解旋酶与3′-尾链之间存在一个相互作用位点,用红色圆点进行标示.当T7解旋酶并不发生开环现象时,它不能从单链DNA上脱落下来而继续留在链上.此时,解旋酶和单链DNA均处于完全自由状态,解开部分的单链DNA会自由退火形成双链DNA,解旋酶会因受到退火压力的作用而沿着5′-链快速滑退回到初始位置,如图5(b)所示,实验数据中表现为瞬时回退现象;当T7解旋酶在发生暂停或停滞的同时发生开环现象而完全从单链DNA上脱落下来时,解开部分单链自由退火形成双链DNA,实验数据中同样表现为瞬时回退现象,因采用的单分子技术检测的对象为DNA底物而无法与图5(b)中的情况进行区分;当这种开环现象的发生使得解旋酶从5′-链上脱落,但是仍然保持着与3′-链之间的相互作用时,这种相互作用会使得解旋酶从5′-链上转移到3′-链上,如图5(c)所示;当T7解旋酶重新稳定结合在3′-链上时,图5(c)会过渡到图5(d),解旋酶会进一步在3′-链上沿着5′到3′方向开始行走过程.这一现象相对于单链DNA自由退火过程来说是一个慢速过程,实验数据中表现为缓慢回退现象.
因此,本文动态观测到六聚体解旋酶——T7解旋酶的换链过程,并提出其模型.相信此研究结果将推进对于六聚体解旋酶功能和特性的理解,并进一步完善对其分子机制的研究.
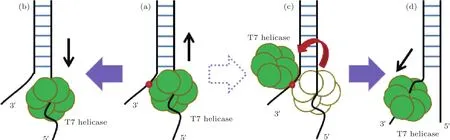
图5 T 7解旋酶换链过程的模型 (a)T 7解旋酶在解旋过程中发生停滞的状态,红色点表示与3′-尾链存在相互作用位点;(b)T 7解旋酶发生瞬时回退或从单链DNA上脱落的状态;(c)T7解旋酶发生开环,并借助相互作用重新结合到3′-链上;(d)T 7解旋酶稳定地结合在3′-链,并缓慢进行行走回到初始位置Fig.5.Model for the switching strand process of T 7 helicase:(a)T 7 helicase stop unwinding during its unwinding process,the red dot means the interaction site between T 7 helicase and 3′-tail;(b)the state that T 7 helicase instantaneously slipping back to initial position on the 5′-strand or dissociating from the 5′-strand;(c)the process that T 7 helicase open the ring-shaped structure and rebinding to the 3′-strand by the interaction with it;(d)the state that T 7 helicase slowly translocating to the initial position on the 3′-strand.
4 结 论
采用smFRET和MT技术对T7解旋酶在解旋过程中解旋和换链问题进行了研究.首先通过设计不同3′-尾链结构的DNA底物验证了T7解旋酶进行解旋反应需要3′-尾链的参与,但是并不受其结构为单链DNA或双链DNA的影响.同时,通过改变DNA序列中的GC含量,发现序列中GC含量的提高会导致T7解旋酶在解旋过程中更容易发生暂停或停滞,进而从DNA底物上脱落或滑退回初始位置,降低其解旋长度;通过分析解旋酶的回退现象,发现大多数情况下为瞬时回退的过程,少数情况下为缓慢回退的过程;通过进一步设计MT实验,研究发现这种缓慢回退现象的存在,并由此推测出这种现象可能为T7解旋酶在解旋过程中发生的换链现象.最后,提出了T7解旋酶进行换链过程的模型.
[1]Dillingham M S 2011 Biochem.Soc.Trans.39 413
[2]Jankowsky E 2011 Trends Biochem.Sci.36 19
[3]Bernstein K A,Gangloff S,Rothstein R 2010 Annu.Rev.Genet.44 393
[4]Zhao D Y,Liu SY,Gao Y 2018 Acta Biochim.Biophys.Sin.146 1093
[5]Klaue D,Kobbe D,Kemmerich F,Kozikowska A,Puchta H,Seidel R 2013 Nat.Commun.4 2024
[6]Li J H,Lin W X,Zhang B,Nong D G,Ju H P,Ma J B,Xu C H,Ye F F,Xi X G,Li M,Lu Y,Dou S X 2016 Nucleic Acids Res.44 4330
[7]Wang S,Qin W,Li J H,Lu Y,Lu K Y,Nong D G,Dou S X,Xu C H,Xi X G,Li M 2015 Nucleic Acids Res.43 3736
[8]Dessinges M N,Lionnet T,Xi X G,Bensimon D,Croquette V 2004 Proc.Natl.Acad.Sci.USA 101 6439
[9]Sun B,Johnson D S,Patel G,Smith B Y,Pandey M,Patel S S,Wang M D 2011 Nature 478 132
[10]Johnson D S,Bai L,Smith B Y,Patel S S,Wang M D 2007 Cell 129 1299
[11]Zhao Z Y,Xu C H,Li J H,Huang X Y,Ma J B,Lu Y 2017 Acta Phys.Sin.66 188701(in Chinese)[赵振业,徐春华,李菁华,黄星榞,马建兵,陆颖 2017物理学报 66 188701]
[12]Wang S,Zheng H Z,Zhao Z Y,Lu Y,Xu C H 2013 Acta Phys.Sin.62 168703(in Chinese)[王爽,郑海子,赵振业,陆越,徐春华2013物理学报62 168703]
[13]Lin W X,Ma J B,Nong D G,Xu C H,Zhang B,Li J H,Jia Q,Dou S X,Ye F F,Xi X G,Lu Y,Li M 2017 Phys.Rev.Lett.119 138102
[14]Zhang H,Lee SJ,Zhu B,Tran N Q,Tabor S,Richardson C C 2011 Proc.Natl.Acad.Sci.USA 108 9372
[15]Zhang H,Tang Y,Lee S J,Wei Z,Cao J,Richardson C C 2016 J.Biol.Chem.291 1472
[16]Matson S W,Tabor S,Richardson C C 1983 J.Biol.Chem.258 14017
[17]Ahnert P,Patel S S 1997 J.Biol.Chem.272 32267
[18]Syed S,Pandey M,Patel S S,Ha T 2014 Cell Rep.6 1037
[19]Donmez I,Patel S S 2008 EMBO J.27 1718
[20]Jeong Y J,Levin M K,Patel SS 2004 Proc.Natl.Acad.Sci.USA 101 7264
[21]Patel S S,Picha K M 2000 Annu.Rev.Biochem.69 651
[22]Morris P D,Raney K D 1999 Biochem.38 5164
[23]Tabor S,Richardson C C 1981 Proc.Natl.Acad.Sci.USA 78 205
[24]Hacker K J,Johnson K A 1997 Biochem.36 14080
[25]Korhonen J A,Gaspari M,Falkenberg M 2003 J.Biol.Chem.278 48627
[26]Ahnert P,Picha K M,Patel S S 2000 EMBO J.19 3418
