RNA解旋酶在细胞中的功能研究进展*
2019-04-15陈艳玲
陈艳玲
(家蚕基因组生物学国家重点实验室,农业农村部蚕桑生物学与遗传育种重点实验室,西南大学生物技术学院,重庆 400715)
最先提出解旋酶(helicase)这一术语是在20世纪70年代末期,解旋酶一词用来描述以依赖ATP(腺苷-三磷酸)方式解开DNA双链的一种酶[1]。直到1988年,第一次有团队发现RNA病毒编码的类似解旋酶的蛋白可以在病毒复制过程中解开RNA链,其作用类似于解开DNA双链的解旋酶,因此首次提出“RNA解旋酶”这一词[2]。1993年Gorbalenya和Koonin提出了一种基于保守序列的系统化的解旋酶分类法[2],发现包含特征性解旋酶基序的蛋白质数量急剧增加。因此,根据序列特征将解旋酶分为几种超家族,解旋酶超家族又可进一步分为蛋白质家族[2],而根据最新的解旋酶系统学把解旋酶分成了6个超家族SF1~SF6。
细胞中含有大量的RNA分子,RNA在编码、基因的表达和调控等多种生物学过程中发挥的重要作用越来越受到人们的关注。大多数RNA分子是单链线状,但具有生物活性的RNA会在某些区域碱基互补配对,而自身折叠形成高度结构化的二级、三级结构。在体内,RNA的结构以及RNA-蛋白质复合物,即核糖核蛋白(RNP)和一切生物大分子的结构一样是处于动态平衡过程中,在其功能周期要经历广泛的构象变化。而RNA伴侣蛋白,特别是超家族SF2 RNA解旋酶蛋白,经常促进这些折叠步骤和构象转变。SF2解旋酶的两个最大家族,即DEAD-box和DEAH-box蛋白,共享进化上保守的解旋酶核心,但通过不同的机制解开RNA双螺旋。
RNA解旋酶不仅是最大的酶类之一,而且在RNA代谢中尤其是在真核生物中几乎无处不在。本质上,真核RNA代谢的每个方面都涉及几种不同的RNA解旋酶,通常来自各种解旋酶家族和超家族[3]。例如在酿酒酵母中核糖体的生物发生中至少涉及21种RNA解旋酶,在mRNA前体剪接中至少有8种RNA解旋酶起作用。大量的RNA解旋酶涉及其它过程包括核RNA的输出、mRMA翻译(尤其是翻译起始)和RNA衰变等等。
本文首先介绍了RNA解旋酶的分类和结构,并针对SF2超家族的DEAD-box和DEAH-box蛋白近年的研究进展进行概述,主要体现在其解旋机制以及细胞功能方面的研究。这为进一步了解RNA代谢的分子基础提供依据和参考,同时也能为更多地探索RNA解旋酶的调控机制提供重要线索。
1 解旋酶的结构
1.1 解旋酶的分类
根据蛋白质序列、组织和功能相似性,将解旋酶分为6个超家族(SF)[2,4]。所有解旋酶是都具有P环的核苷-三磷酸(NTP)酶,包含典型的Walker A和B位点,其用于NTP结合和水解。RNA解旋酶与DNA解旋酶密切相关,所有真核RNA解旋酶均属于超家族1和2(SF1和SF2),它们不能形成环结构[5]。而来自超家族SF3~SF6的解旋酶则可形成环结构,并存在于细菌中(例如Rho[6]和病毒[7])。值得注意的是大多数RNA解旋酶是SF2超家族成员,只有少数属于SF1超家族。所有SF2蛋白共享一个保守的解旋酶核心,并由两个RecA样结构域Domain1(D1)和Domain2(D2)组成(图1A),它们通过易弯曲的接头连接,核心包括至少12个保守的基序。但并非每个家族中都存在所有的基序[8],与家族之间相比,这些基序在家族内部的保守性更高。根据每个超家族成员的解旋酶核心结构域中存在的特征序列基序,可以将它们进一步分成几个家族:SF2超家族包含DEAD-box、DEAH-box和Ski2样RNA解旋酶家族。DEAD-box和DEAH-box蛋白家族是迄今为止人们鉴定出的最大的家族,包含所有真核生物和大多数原核生物中存在的解旋酶。研究者们发现在这两个家族中的大多数蛋白都具有RNA伴侣或RNP重塑剂功能,可以参与到RNA底物相互作用、NTP结合和水解以及解旋活性中。我们下面重点介绍DEAD/H-box这两个家族。
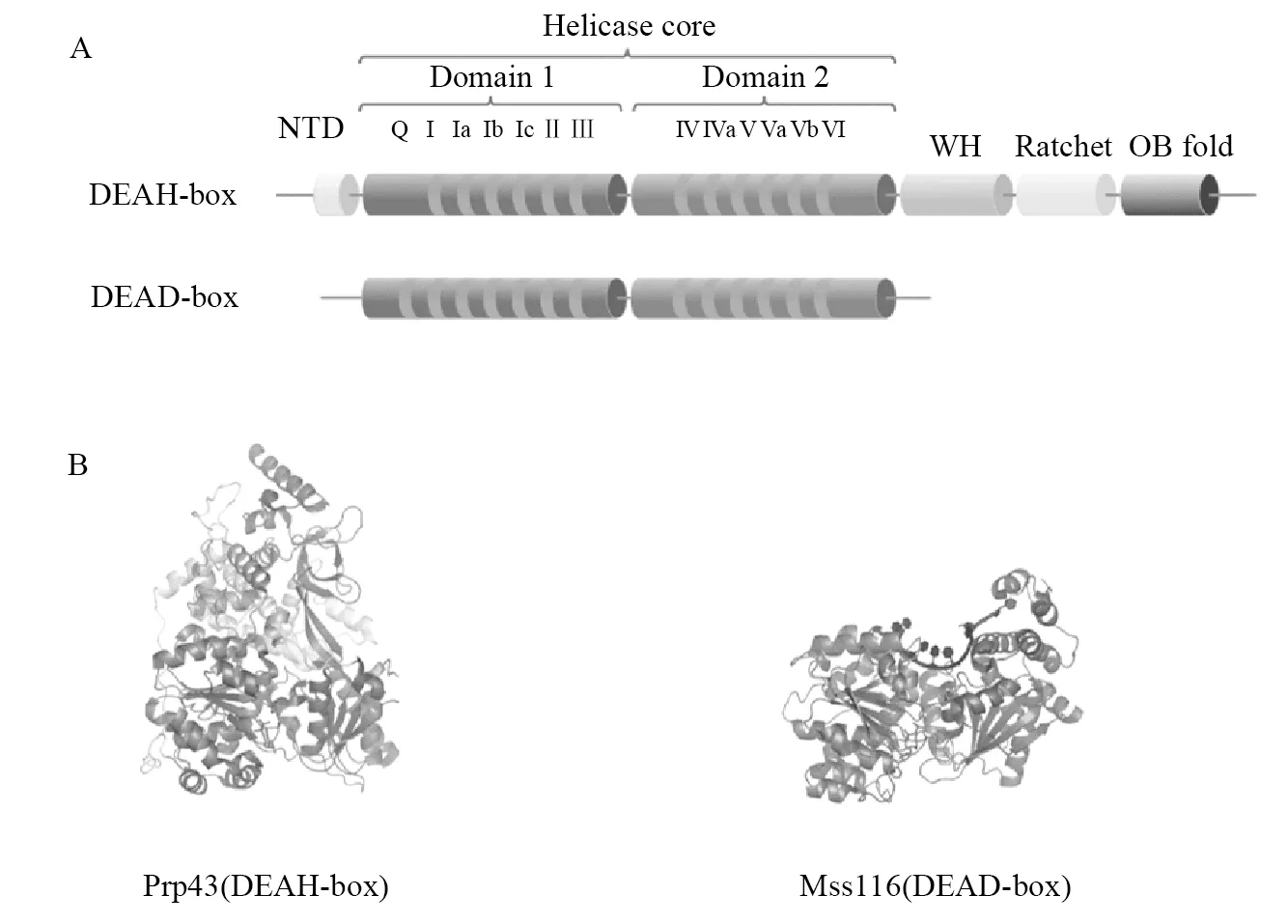
(A)解旋酶的保守序列分布 (B)DEAH-box(Prp43)和DEAD-box(Mss116)解旋酶的晶体结构
1.2 保守的RNA解旋酶基序的分子功能
在过去的20多年中,通过解析来自各类超家族的几种DNA和RNA解旋酶的晶体结构[10-11](图1B)发现,SF2解旋酶包含高度结构保守的解旋酶核心,存在于200~700个氨基酸区域内,其侧翼为氨基和羧基末端(C-和N-末端)延伸[8],保守的解旋酶基序则位于这两个解旋酶核心结构域中。SF2的解旋酶核心由两个相似的结构域组成,每个结构域通常由5个β链和5个α螺旋组成,其折叠类似于重组蛋白RecA的折叠[12-13]。C-和N-末端延伸/侧翼序列通常不保守,表现出大小和组成的高度可变性[14]。而DEAH-box蛋白却共享保守的C末端结构域,该结构域由翼状螺旋域(Winged helix,WH)、棘轮状域(ratchet)和寡糖结合域(oligosaccharide binding fold,OB fold)组成(图1A)。DEAD-box蛋白除了包含RecA样结构域的经典的5条β链和5条α螺旋外,还包含另外的β链和2条α螺旋,它们构成位于基序I上游的Q基序,折叠为D1结构域顶部的“帽”结构[15],该基序是DEAD-box蛋白家族独有的。
解旋酶核心的两个D1和D2结构域包含12个基序,其中结构域D1包括基序Q、I、Ia、Ib、Ic、II和III,而结构域D2中包含基序IV、IVa、V、Va和VI(表1)。其中,I(Walker A)、II(Walker B)和VI基序在SF1和SF2超家族的序列和结构中高度保守,而Q基序仅存在于RNA解旋酶DEAD-box家族中[16]。在活性的RNA解旋酶三级结构中,这些保守的基序可以形成RNA底物通道,可以与配体(包含NTP、二价离子和水分子等)接触,形成催化水解结合位点。

表1 RNA解旋酶保守基序的分子功能(改编自Katherine E Sloan and Markus T Bohnsack[17])
2 RNA解旋酶的活性机制
RNA解旋酶可以充当RNA和RNP的分子伴侣,通过破坏RNA碱基,除了使RNA具有结构化及其多样性外,SF2解旋酶还使用多种机制来促进RNA重排。本文只重点介绍DEAD-box和DEAH-box解旋酶的解旋活性机制,某些DEAD-box RNA解旋酶可以局部解开RNA,但不会沿RNA显著移位,而某些DEAH-box RNA解旋酶则沿RNA移位,但不会解旋。
2.1 通过DEAD-box解旋酶局部解旋
在没有NTP或RNA的情况下,DEAD-box RNA解旋酶的两个RecA样结构域(D1和D2)采用开放构象,从而限制了该酶的非特异性活性[27-28]。ATP最初与RNA解旋酶的D1结合,而RNA与解旋酶的D2结合,共同触发构象转换成为闭合状态。在闭合状态中,双链RNA、ATP和两个RecA样结构域D1和D2的保守基序之间形成了相互作用的复杂的三元复合物[29]。核心闭合步骤将两个结构域结合在一起并创建成ATP酶的活性位点[30]。过渡到闭合构象后,使结合的RNA显著扭曲,导致RNA的一条链保留在与酶结合的紧密复合物中,另一条链则从复合物中释放出来[11,20,31]。而RNA解旋酶的再循环则需要水解结合ATP和无机磷酸盐(Pi)的产生,促使解旋酶从紧密结合的RNA单链上解离出来[32-35]。
2.2 DEAH-box解旋酶分离RNA双链
DEAD-box蛋白通过RNA双链结合、解链和释放的简单循环而发挥作用,而DEAH-box解旋酶进行的RNA双链解链则通过移位方式起作用。通常认为DEAH-box RNA解旋酶是沿着RNA的单链3’至5’方向移位,前进方向可以破坏核酸结构而介导双链解旋,从而置换互补链[36-38]。此过程中,载有ATP的DEAH-box解旋酶与RNA结合,形成一种开放的构象。然后ATP水解诱导解旋酶的构象从开放状态转换为闭合构象,其中RNA解旋酶D1结构域仍锚定在RNA分子上,D2结构域则移近RNA方向。ATP水解并释放底物后,解旋酶再次形成开放构象,并且RNA解旋酶D2结构域与RNA在移动方向的下游更远处形成新的联系。相比之下,DEAD-box解旋酶的ATP水解是伴随着底物释放,而DEAH-box解旋酶是借助ATP向ADP的转换驱动着RNA双链的分离。
3 RNA解旋酶的细胞功能
RNA解旋酶作为RNA代谢中最大的酶类,参与到RNA代谢的各个方面。这里我们将对RNA解旋酶在复杂的细胞环境中,如何参与和调控mRNA前体剪接、核糖体生物发生、RNA的输出、mRNA翻译起始和RNA衰变的功能研究进行概述。
3.1 mRNA前体剪接加工(pre-mRNA splicing)
mRNA加工成熟过程包括转录的mRNA前体(pre-mRNA)在其5’和3’端进行修饰,并通过剪接过程去除内含子并连接外显子,从而形成成熟的mRNA,供核糖体翻译[39]。内含子序列通常位于真核蛋白编码基因序列中,包含一个5 ’剪接位点、一个分支点序列(包含保守腺苷酸A)和一个3 ’剪接位点,这些是剪接所必需的。剪接过程涉及RNA核苷酸之间发生的两次连续的酯交换反应。首先内含子分支点的保守A通过2’-OH对5’剪接位点的第一个核苷酸发动亲核攻击,形成套索-中间体;其次5’外显子的3’-OH攻击内含子3’剪接位点的最后一个核苷酸,将外显子连接在一起,释放出套索-内含子,导致成熟的mRNA形成。这些反应是由包含100多个相关蛋白因子和5个小核糖核蛋白(snRNPs U1、U2、U4、U5和U6)形成的剪接小体催化的,这些小核糖核蛋白snRNP是由一组特定的蛋白与相关的相同名称的小核RNA(snRNA U1、U2、U4、U5和U6)组成的。剪接反应包括外显子的连接、mRNA的释放、内含子的套索和剪接小体的解体,需要RNA-RNA和RNA-蛋白复合物的重新排列。这些构象的变化被认为需要来自NTP水解的能量。遗传和生化研究表明至少有8种RNA解旋酶参与上述过程[40],包括3个DEAD-box蛋白(Prp5、Sub2p和Prp28p)、1个Ski2样蛋白(Brr2p)和4个DEAH-box蛋白(Prp2p、Prp16p、Prp22p 和Prp43p)。这些解旋酶蛋白质与RNA二级结构重组和位移,促使剪接体组装、重新排列和分离控制,从而完成pre-mRNA的剪接。下面我们总结了目前关于解旋酶介导的啤酒酵母细胞剪接体重排的知识,以及其在pre-mRNA剪接中的作用。
pre-mRNA的剪接过程开始于U1 snRNP的5 ’剪接位点上的装配,该位点上的碱基与pre-mRNA配对不需要ATP,然后U2 snRNP以依赖ATP的方式与分支点序列(BPS)结合。这个装配步骤可能是由DEAD-box蛋白Prp5p和Sub2p解旋酶驱动的[41]:Prp5p被提出来改造U2 snRNP,从而通过ATP水解促进U2 snRNA与BPS结合。解旋酶Sub2p/UAP56(酵母菌/人类)被认为是将Mud2p/U2AF(酵母菌/人类)蛋白从富嘧啶区解离到靠近分支点,这与蛋白置换的作用一致[41-42]。
稳定的U2 snRNP绑定后,下一步三联体snRNP U4-U6-U5在5 ’剪接位点区域与初生的剪接小体结合,U1 snRNP被U6 snRNP取代。这一步涉及RNA-RNA的重排和RNA-蛋白质的相互作用,并且是在依赖ATP的方式下完成的。基于遗传数据,Prp28解旋酶需要解除U1 snRNP和pre-mRNA的5 ’剪接位点区域的配对而解旋,促进释放U1 snRNP和暴露5 ’剪接位点的碱基与U6 snRNP配对[43]。总的来说,这3种DEAD-box解旋酶蛋白的局部重排作用与DEAD-box蛋白的一般功能相一致,与下面描述的另外两类解旋酶介导的大量的重新排列现象形成对比。
接下来snRNP U4从剪接体中被移走。这需要Ski2家族蛋白Brr2p,它会破坏U4/U6双链[44]。然后DEAH盒蛋白Prp2p诱导第一次酯化反应所需的剪接体的结构重排。接下来的步骤需要其它DEAH-box蛋白包括Prp16p、Prp22p和Prp43p,它们分别参与到第二次反酯化反应、剪接mRNA的释放和剪接体的再循环利用。在这个过程中,Prp2p可能去除掉了与BPS结合的sf3ap和sf3bp蛋白以及其它一些蛋白,这有利于激活剪接体的催化活性[45]。套索的形成过程中,Prp16p在第二次酯交换反应之前或过程中促进了U2 snRNP重新折叠、U2/U6螺旋的不稳定、以及几种蛋白质的移位[46-47]。一旦发生了两个酯交换反应,Prp22p和Prp43p就会参与到剪接体的解体过程中,它们从剪接体中分别释放出催化成熟mRNA和套索-内含子。其中Prp22通过破坏U5 snRNP和mRNA之间的碱基对来释放mRNA[48]。最后Prp43介导大量的重新排列,使套索-内含子被释放,而U2、U5和U6 snRNPs得以循环再利用[49]。
3.2 核糖体的生物合成(ribosome biogenesis)
在真核生物中核糖体的生物发生开始于由RNA聚合酶I和RNA聚合酶III分别转录出35S / 45S(酵母/哺乳动物)和5S 核糖体RNA前体(pre-rRNA)。核糖体蛋白和非核糖体蛋白开始在新生的pre-rRNA上组装。这样的组装步骤会产生早期的90S核糖体复合物。这些复合物中的pre-rRNA将经历大量的加工过程,涉及小核仁核糖核蛋白(snoRNPs)的化学修饰以及一系列内切和外切核酸切割步骤,去除内部和外部转录间隔区序列(ITS和ETS)。成熟的90S核糖体复合物最终被拆分为60S核糖体亚基前体和40S核糖体亚基前体,分别包含有成熟的25S / 28S(酵母/哺乳动物),5S和5.8S rRNA以及18S rRNA。两种大小核糖体亚基(LSU和SSU)的成熟都遵循相同的路径,首先在核仁中,之后运输到核质中,最后是通过核孔转运至细胞质完成LSU和SSU的装配[50]。
核糖体的生物发生是一个精细且复杂的过程,涉及几个大片段的rRNA的剪切、修饰和折叠,在这个过程中涉及大量蛋白的装配和核糖核蛋白结构的变化[51]。酿酒酵母的DEAD-box蛋白的很大一部分在核糖体生物发生中起作用,几种DEAH-box解旋酶和Ski2样解旋酶也参与其中。尽管这些解旋酶在核糖体生物发生过程中的确切分子作用仍不清楚,但由于它们的ATP酶和解旋酶活性,它们可能参与了短snoRNA-rRNA或rRNA-rRNA双链的解除,或参与了RNA-蛋白质相互作用的重构[52]。
在酿酒酵母中,有19种解旋酶参与了核糖体的生物发生,其中17个是必需的。解旋酶蛋白的突变或敲除通常会影响到特定的pre-rRNA剪切加工,从而导致LSU和SSU的生成受损,有时两者都受损[53]。虽然很难区分到底是直接或间接的影响[51],但这些对研究解旋酶在核糖体生物发生中的功能提供了重要的见解。一些研究数据显示Ski2样解旋酶Mtr4p(Dob1p)是核外泌体复合物的辅因子,是5.8S rRNA的3’末端形成所必需的[54]。其它涉及核糖体生物发生的解旋酶都属于DEAD/DEAH-box家族。其中5个DEAD-box(Dbp4p、Dbp8p、Fal1p、Rrp3p和Rok1p)和2个DEAH-box(Dhr1p和Dhr2p)参与了合成SSU所需的早期切割,而合成LSU需要9个DEAD-box蛋白(Dbp2p、Dbp3p、Dbp6p、Dbp7p、Dbp9p、Dbp10p、Drs1p、Mak5p和Spb4p)[40]。值得注意的是DEAH-box Prp43p和DEAD-box Has1p都参与了核糖体大小亚基生物合成途径[40]。此外,Prp43p还参与了pre-mRNA剪接成熟的晚期步骤[55]。
小核仁RNA(snoRNAs)是小型结构化的RNA,可以与pre-rRNAs碱基配对,共同指导位点特异性的切割、甲基化和假尿苷化,并可指导pre-rRNA的折叠[16]。酵母中有75个snoRNAs,分别来自结构不同的box-C/D和H/ACA类,其中有3个必需的snoRNAs(U3、U14和snR30)可指导定点切割,72个非必需的snoRNAs参与到核苷酸修饰[16]。人们很容易推测RNA解旋酶可能参与调节snoRNAs与pre-rRNAs的结合和从pre-rRNAs上解离。一些解旋酶已被证明在snoRNAs的重排中起作用,snR30的解离需要Rok1p[56],而 U14 snoRNA的解离除了需要Has1p外还需要Dbp4p[57]。另一个例子是DEAD-box解旋酶Has1p,其不仅参与SSU和LSU的生物发生,而且参与了U3和U14 snoRNAs的解离,Has1p的缺失导致多个snoRNAs的积累。DEAH-box解旋酶Dhr1p和Dhr2p以及DEAD-box蛋白Dbp8p、Rrp3p和Rok1p是U3 snoRNA引导切割pre-rRNA所必需的[52]。这些表明去除一个给定的snoRNA可能需要多个解旋酶的直接或间接活性,一些解旋酶也被认为可能间接参与了核糖体中间体复合物的重排和释放[58]。
3.3 RNA 运输(RNA transport)
DEAD-box蛋白Dbp5p是从细胞核输出mRNA所必需的[59],而它主要存在于细胞质中[60-61],它可以将输出因子Mex67p/Mtr2p从细胞质中的核孔表面的mRNA上剔除掉[62-63]。并且也有研究人员发现在翻译终止过程中,Dbp5p起到参与终止密码子有效的识别和将eRF3募集到终止复合物中的作用[64]。
3.4 mRNA翻译起始(Translation initiation)
已经证明RNA解旋酶是mRNA翻译起始所必需的。其中之一是eIF4A是pre-mRNA帽子结构(5’-cap)依赖翻译起始因子[65-66]。推测eIF4A可以在真核mRNA的5’末端解开并重排RNA双链的二级结构,从而促进40S核糖体亚基的结合和扫描。另一种DEAD-box蛋白Ded1p参与翻译起始,其作为翻译的阻遏物[67-88]。而果蝇中的DEAD-box蛋白Vasa还可以通过与翻译起始因子2(dIF2)直接相互作用来激活mRNA翻译[69-70]。
3.5 RNA衰退(RNA decay)
外切体(exosome)是存在于真核细胞和古菌中由多于5种核酸外切酶构成的蛋白质复合体,也称为外切体复合物(exosome complex),外切体以一种外切酶方式从RNA的3’端对RNA进行水解。真核生物的外切体主要利用两种调节复合物的解旋酶:一种是细胞质Ski复合物的Ski2解旋酶以及另一种是核复合物TRAMP紧密相关的Ski2样解旋酶Mtr4[71]。两种解旋酶似乎都可以解开结构化的RNA,并将单链ssRNA导入外切体进行降解[72-73]。这些Ski2样解旋酶的定向3’至5’方向的解旋作用被认为可促进解旋和降解的耦合。RHAU解旋酶也可以与外切体短暂地相互作用,从而促进特定mRNA的降解,这可能是通过破坏RNA结构或蛋白质结合来实现的[74]。
4 小结
RNA解旋酶这个术语已经暗示出这类蛋白具有相当特殊的生化功能,越来越多的研究表明,RNA解旋酶参与到RNA分子代谢的所有环节。RNA解旋酶可以充当RNA和RNP的分子伴侣,通过RNA的局部解旋,或者蛋白质与RNA的结合/解离促进RNA结构化的重塑。如果想了解RNA代谢的分子基础,则需要了解RNA解旋酶的机制以及这些机制如何在细胞中表现出来,本文我们已经讨论了这两种不同的解旋机制。RNA代谢中的某些过程会使用大量RNA解旋酶,本文就RNA解旋酶在如何参与和调控前体mRNA剪接、核糖体生物发生、RNA的输出、翻译起始和RNA衰变的过程中的功能研究进展进行了概述。目前还不是很清楚RNA解旋酶的具体分子机制,但是已知的是RNA解旋酶的功能很广泛,例如有些RNA解旋酶还与精子发生、胚胎发生、细胞生长和增殖等相关[75]。越来越多的研究发现许多解旋酶通过调节各种基因的表达,在多种癌症中发挥重要作用[76]。相信在研究解旋酶的科研工作者们的共同努力下,解旋酶的分子机制将逐渐被阐明清晰,这也可能为肿瘤的靶向治疗提供新思路。
