基于生物信息学分析KCNQ1OT1在胃癌中的作用
2022-09-07张丽媛李家秋蔡锦威毕春华刘方花
张丽媛, 李家秋, 蔡锦威, 毕春华, 刘方花
(1)青岛滨海学院医学院 临床医学教研室,山东, 青岛 266555;2)潍坊医学院附属医院 肿瘤科,山东, 潍坊 261031)
胃癌(gastric cancer,GC)是起源于胃黏膜上皮的恶性肿瘤,根据国际癌症研究机构(International Agency for Research on Cancer,IARC)发布的2020年统计报告显示,胃癌发病率在全球范围内居于第5位,死亡率为第4位[1]。我国胃癌发病率和死亡率均处于第3位,防治胃癌的形势十分严峻。由于早期胃癌的症状常缺乏特异性且受各种条件的限制,胃癌早期诊断率较低,大多数病人首诊即晚期。加上目前晚期胃癌缺乏有效性的治疗手段,导致胃癌病人总体预后差,5年生存率很低[2]。因此,研究胃癌发生发展的分子机制,探索新的早期诊断和治疗胃癌的分子标记物,有助于提高胃癌的早期确诊率和有效控制其侵袭、转移,降低死亡率,对于胃癌的防治具有非常重要的科学价值和临床意义。
长非编码RNA(long non-coding RNA,lncRNA)是长度大于200个核苷酸的非编码RNA[3]。近年来的研究发现,lncRNA在剂量补偿效应、表观遗传调控、细胞周期调控和细胞分化调控等众多生命活动中发挥了重要作用,参与了机体的各种生理与病理过程[4,5],因此成为当前肿瘤学领域研究的热点。经研究发现,lncRNA KCNQ1OT1参与肿瘤的增殖、侵袭、转移和抗凋亡等过程,并在不同肿瘤的发生发展过程中发挥重要作用[6,7]。甚至有研究显示,它可调控糖酵解途径[8]。对于肿瘤中lncRNA KCNQ1OT1作用的研究有望为肿瘤诊疗提供新的靶点。目前,KCNQ1OT1 在胃癌中的研究文献报道较少,并且关于其表达和作用的结论缺乏一致性[9-10]。本研究通过癌症基因组图谱(The Cancer Genome Atlas, TCGA)等公共数据库综合分析和实验验证KCNQ1OT1在胃癌中的表达及作用机制,为深入研究该基因功能提供线索,为分子靶向治疗胃癌的临床研究提供新的靶点。
1 材料与方法
1.1 数据库的使用
1.1.1 GEPIA2数据库分析 通过GEPIA2数据库[11]Box Plot模块检索KCNQ1OT1在胃癌中的表达情况,匹配正常数据包含TCGA和GTEx数据,通过Stage Plot模块分析KCNQ1OT1在胃癌不同阶段的表达水平,通过Survival Analysis模块分析KCNQ1OT1在胃癌中总生存期(overall survival,OS)及无病生存期(disease-free survival,DFS)等预后数据,在Correlation Analysis模块通过Spearman相关系数分析前100个KCNQ1OT1相关基因,P<0.05为具有统计学意义。
1.1.2 TIMER2数据库分析 通过TIMER2数据库[12,13]Diff-Exp模块分析KCNQ1OT1在肿瘤中的表达水平,运用Survival模块比较不同表达KCNQ1OT1组的总生存期,通过Correlation模块运用Spearman相关系数分析KCNQ1OT1基因表达与谷氨酰胺酶1(glutaminase 1, GLS1)之间的相关性,通过Gene模块分析KCNQ1OT1与免疫细胞浸润之间的相关性。免疫细胞包括CD8+T细胞、CD4+T细胞、B细胞、单核细胞、巨噬细胞、树突状细胞、中性粒细胞、NK细胞和调节性T细胞,P<0.05为具有统计学意义。
1.1.3 UALCAN数据库分析 通过UALCAN数据库[14]分析KCNQ1OT1基因在胃癌及正常组织中的表达水平,比较KCNQ1OT1在胃癌不同分期、分级、病理类型、种族、年龄以及TP53突变状态的表达情况,分析KCNQ1OT1在胃癌中的总生存期,P<0.05为具有统计学意义。
1.1.4 StarBase数据库分析 通过StarBase数据库[15]分析GLS在胃癌中的表达,以及KCNQ1OT1的表达与PD-L1(CD274)、GLS表达之间的相关性,P<0.05为具有统计学意义。
1.1.5 cBioPortal数据库分析 通过cBioPortal数据库[16,17]分析KCNQ1OT1的表达与GLS表达之间的相关性,P<0.05为具有统计学意义。
1.1.6 Kaplan-Meier Plotter数据库分析 Kaplan-Meier Plotter数据库[18]包含基因表达综合(gene expression omnibus, GEO)和TCGA等数据,通过对该数据库的挖掘,评估KCNQ1OT1表达的预后价值,包括总生存期(OS)、无病生存期(DFS)、首次进展生存期(first progression survival, FP)和复发生存期(relapse survival, PPS),P<0.05为具有统计学意义。
1.1.7 lnCAR数据库分析 通过lnCAR数据库[19]探索KCNQ1OT1的共表达网络,对前200个基因网络进行可视化,并进行富集通路分析(kyoto encyclopedia of genes and genomes, KEGG)。
1.1.8 DAVID分析 通过GEPIA2和lnCAR数据库得到的KCNQ1OT1相关基因全部输入DAVID数据库[20]进行注释、可视化和综合分析,基因功能分析包括生物过程(biological process, BP),分子功能(molecular function, MF)和细胞成分(cellular component, CC),相关通路主要是KEGG途径。
1.2 细胞培养
细胞全部来源于中国科学院上海生命科学研究院细胞资源中心,使用DMEM或者1640培养基在含有5% CO2的 37 ℃ 细胞培养箱中培养,培养基中添加 10%胎牛血清以及 100 U/mL 青霉素和 100 μg/mL 链霉素,根据细胞生长状态及时更换培养液,取对数生长期的细胞用于实验。
1.3 siRNA干扰
将处于对数生长期的胃癌细胞SGC7901和BGC823接种于6孔板中(1×105/孔),贴壁过夜后,利用Lipofectamine RNAiMAX转染目标siRNA,转染24 h后更换正常培养基,48 h后收集细胞进行下一步实验。所用到的siRNA序列如下:NC-S:5′-UUCUCCGAACGUGUCACGUTT3′, AS:5′-ACGUGA CACGUUCGGAGAATT-3′; KCN1OT1-siRNA-1#-S:5′-GCAACAGCUAUUCUUCAUATT3′, AS:5′-UAUG AAGAAUAGCUGUUGCTT-3′; KCN1OT1-siRNA-2#:S: 5′-GCCACAUCUAACACCUAUATT-3′, AS:5′-UAUAGGUGUUAGAUGUGGCTT-3′。
1.4 平板克隆形成实验
取对数生长期的细胞,在6孔板中处理细胞后,重新消化种板,1 000个细胞/孔,每3 d换1次液,2周后用福尔马林固定,结晶紫染色后计算克隆个数。
1.5 台盼蓝染色细胞计数
取对数生长期细胞,在6孔板中处理细胞完成后,用0.4%台盼蓝染色,每d计数细胞数,以培养时间为横轴,细胞数为纵轴(对数),描绘细胞生长曲线图。
1.6 RNA提取及逆转录
取处理好的细胞,加入Trizol提取总RNA。RNA定量后,使用高容量cDNA逆转录试剂盒,取2 μg RNA进行逆转录。
1.7 实时定量PCR
使用实时定量PCR试剂盒,检测KCNQ1OT1和GLS1的RNA表达水平。其中,α-微管蛋白(α-tubulin)作为内参,引物序列如下:GLS1-F:5′-GCAACAGCGAGGGCAAAGA-3′,R:5′-TTCAACCTG GGATCAGACG-3′; KCNQ1OT1-F:5′-GATCTGCCCT GCTTACCTCTC-3′,R:5′-GTGAGTGACTGAAGGGGA TGA-3′; Tubulin-F:5′-GAAGCAGCAACCATGCGTG A-3′,R:5′-AAGGAATCATCTCCTCCCCCA-3′。
1.8 统计学分析
所有数据均以平均值±标准差表示。应用SPSS 20.0软件进行统计学分析,采用Student’st检验进行统计学显著性分析,P<0.05具有统计学意义。
2 结果
2.1 KCNQ1OT1在胃癌中高表达且与预后不良相关
为了排除单一数据库数据造成的结果偏倚,本研究利用TIMER和UALCAN两个数据库综合分析了KCNQ1OT1的表达,发现KCNQ1OT1在多种肿瘤类型中高表达,包括胃癌(Fig.1A, B)。而它对胃癌病人的预后的影响,通过GEPIA2、UALCAN和Kaplan Meier- plotter 3个数据库分析,发现高表达KCNQ1OT1的胃癌病人总生存期(OS)、无病生存期(DFS) 、进展生存期(FP)和复发生存期(PPS)均明显缩短(Fig.1C, D, E),提示高表达KCNQ1OT1的胃癌病人可能预后不良。因此,KCNQ1OT1在胃癌中可以作为一个诊断性和预后性的肿瘤标记物。
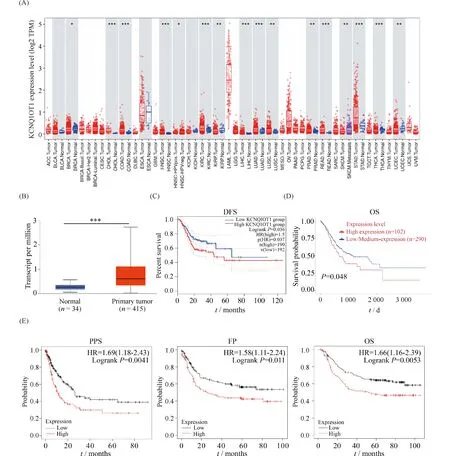
Fig.1 Expression levels and prognostic value of KCNQ1OT1 in gastric cancer (A) The expression level of KCNQ1OT1 in different tumors or specific tumor subtypes was analyzed through the TIMER2 database. (B) The expression level of KCNQ1OT1 between normal and gastric cancer tissues in the UALCAN database (***P<0.001). (C) The prognostic value of KCNQ1OT1 was analyzed by GEPIA2 database (DFS, disease-free survival). (D) The prognostic value of KCNQ1OT1 was analyzed by UALCAN database (OS, overall survival). (E) The prognostic value of KCNQ1OT1 was analyzed by Kaplan-Meier Plotter database. (FP, first progression survival, PPS, relapse survival)
2.2 KCNQ1OT1与胃癌各种临床因素密切相关
通过UALCAN数据库,进一步分析了KCNQ1OT1与胃癌各种临床因素的相关性。结果发现,KCNQ1OT1在胃癌的不同分期、分级、不同病理类型、不同人种和不同年龄段中普遍高表达(Fig.2A-2E),具有明显的统计学意义(P<0.05)。适用于大部分胃癌病人的诊断,其中在年轻的胃癌病人中(21~40岁)显著高于其他年龄段的胃癌病人(P<0.05)。更重要的是发现,KCNQ1OT1的表达与TP53的突变有一定相关性(P<0.05, Fig.2F)。这也部分揭示了KCNQ1OT1在胃癌中高表达的原因。
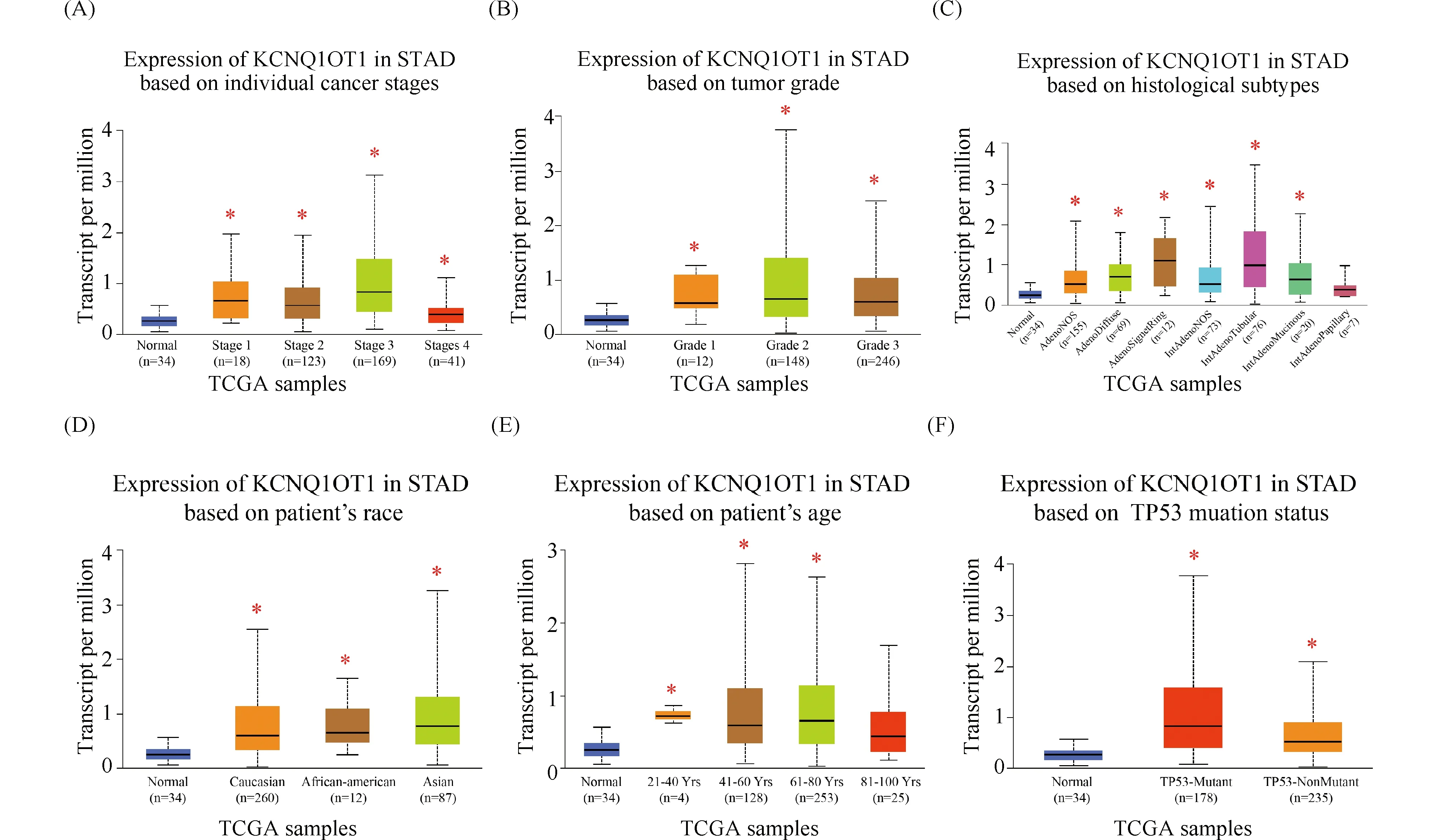
Fig.2 The correlation between the expression of KCNQ1OT1 and clinical factors in gastric cancer via UALCAN database (A) The expression of KCNQ1OT1 in different stages of gastric cancer (*P<0.05). (B) The expression of KCNQ1OT1 in different grades of gastric cancer (*P<0.05). (C) The expression of KCNQ1OT1 in different pathological types of gastric cancer (*P<0.05). (D) The expression of KCNQ1OT1 in different patient’s race (*P<0.05). (E) The expression of KCNQ1OT1 in different patient’s age (*P<0.05). (F) The expression of KCNQ1OT1 in different TP53 mutation status (*P<0.05)
2.3 KCNQ1OT1的表达与免疫细胞浸润密切相关
本研究利用TIMER2数据库分析了KCNQ1OT1的表达与免疫细胞浸润之间的关系。结果发现,KCNQ1OT1的表达分别与CD4+T细胞、调节性T细胞和内皮细胞的浸润呈正相关关系,而与CD8+T细胞、单核细胞、巨噬细胞、树突状细胞、嗜酸性粒细胞和γδT细胞的浸润呈负相关关系(Fig.3A)。而且发现,KCNQ1OT1表达与程序性死亡配体-1(programmed death ligand-1, PD-L1(CD274))的表达呈负相关关系(Fig.3B-3D), 提示高表达KCNQ1OT1的胃癌病人可能不适合免疫检查点治疗。
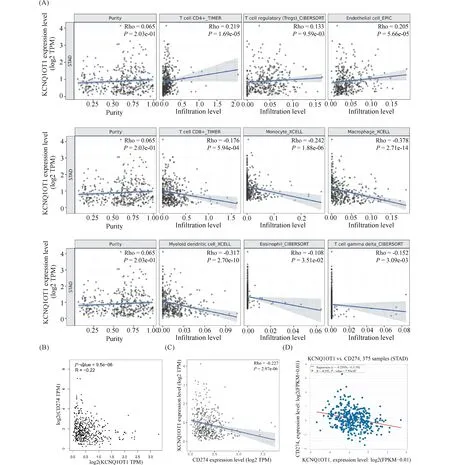
Fig.3 The correlation between the expression of KCNQ1OT1 and immune factors in gastric cancer (A) Correlation analysis between KCNQ1OT1 expression and various immune cell infiltration levels via TIMER2 database. (B-D) The expression of KCNQ1OT1 was negatively correlated with the expression of PD-L1 (CD274) via different databases. (B, GEPIA2, C, TIMER2, D, StarBase)
2.4 KCNQ1OT1促进胃癌肿瘤细胞的增殖能力
为了探索直接靶向KCNQ1OT1的可能性,本文研究了KCNQ1OT1在胃癌肿瘤细胞中的作用。首先检测了KCNQ1OT1在胃癌肿瘤细胞系中的表达情况。结果发现,KCNQ1OT1在胃癌肿瘤细胞系中普遍高表达(Fig.4A)。随后,应用特异性的siRNA敲低KCNQ1OT1的表达,发现胃癌肿瘤细胞的生长受到了明显的抑制(Fig.4B-4D)。说明KCNQ1OT1能促进胃癌肿瘤细胞的增殖能力,可作为1个新的分子靶点。
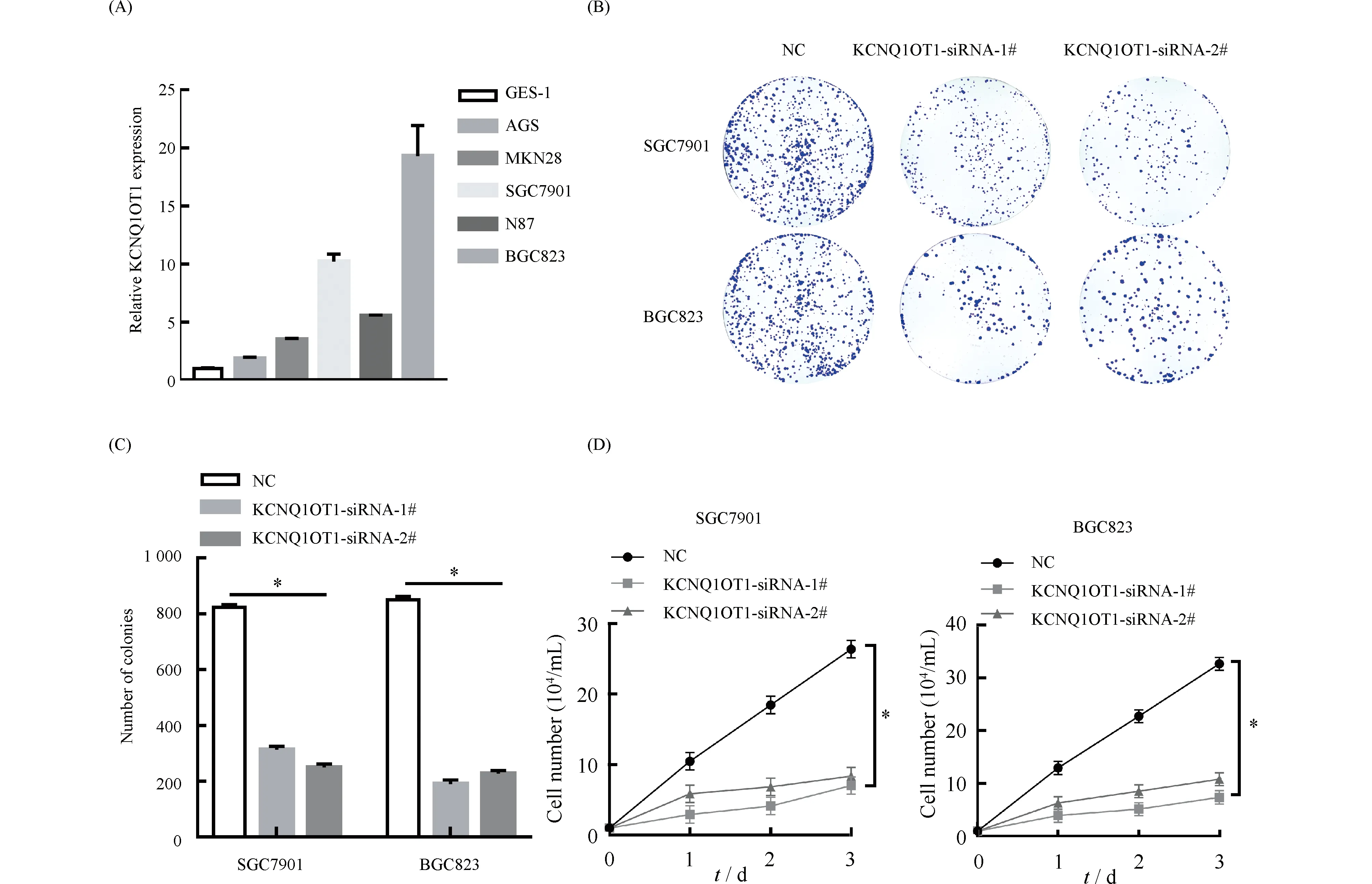
Fig.4 KCNQ1OT1 promotes the proliferation of gastric cancer cells (A) The relative expression of KCNQ1OT1 in gastric cancer cell lines by quantitative PCR assays. (B-D) Colony formation assays and growth curve were used to evaluate the proliferation of gastric cancer cells after knockdown of KCNQ1OT1 for 72 hours. Values are the mean ± SD of three determinations from separate experiments (*P<0.05)
2.5 KCNQ1OT1共表达网络富集通路分析
既然KCNQ1OT1在胃癌肿瘤细胞中发挥着重要的作用,本文想探索其发挥作用的具体机制。首先,在lnCAR数据库中分析了KCNQ1OT1共表达网络(前200个基因)(Fig.5A)。并且发现KCNQ1OT1跟多种通路活性密切相关,最明显的是代谢通路(Fig.5B)。本文合并GEPIA2(前100个基因)和lnCAR(前200个基因)2个数据库的相关基因,进行了基因功能分类(gene ontology, GO)和富集通路分析(KEGG)(Fig.5C-F)。有意思的是,结果显示,KCNQ1OT1可能参与到谷氨酰胺代谢通路当中。
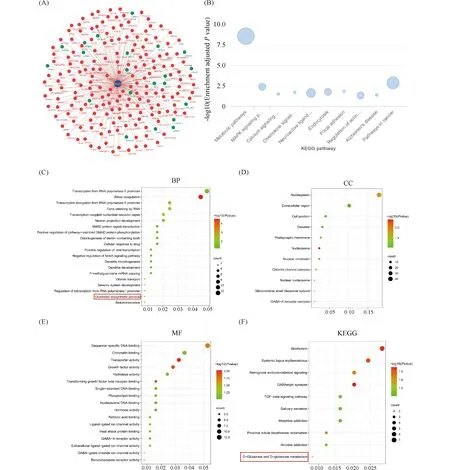
Fig.5 Analysis of KCNQ1OT1 coexpression gene network (A) Analysis of KCNQ1OT1 coexpression network via lnCAR database. (B) KEGG analysis of the KCNQ1OT1 gene network via lnCAR database. (C-F) GO and KEGG analysis of the KCNQ1OT1 gene network using lnCAR and GEPIA2 data (BP, biological process, CC, cellular component, MF, molecular function, KEGG, Kyoto encyclopedia of genes and genomes)
2.6 KCNQ1OT1通过GLS1调控胃癌肿瘤细胞的生长
谷氨酰胺酶1(GLS1)是调控谷氨酰胺代谢最重要的酶。利用TIMER2和UALCAN两个数据库,综合分析了GLS1的表达。发现GLS1在多种肿瘤类型中也高表达,包括胃癌(Fig.6A,B),而高表达GLS1的胃癌病人也提示预后不良,总生存期(OS)、进展生存期(FP)均明显缩短(Fig.6C)。进一步探索KCNQ1OT1是否是通过调控GLS1促进胃癌肿瘤细胞的增殖? TIMER2等数据库显示,KCNQ1OT1与GLS1 mRNA表达呈明显正相关关系(Fig.6D-6F)。敲低KCNQ1OT1的表达,GLS1 mRNA确实明显下调(Fig.6G),在敲低KCNQ1OT1的同时过表达GLS1会部分逆转对胃癌肿瘤细胞生长的抑制作用(Fig.6H)。上述结果说明,KCNQ1OT1能通过调控GLS1表达促进胃癌肿瘤细胞的增殖。
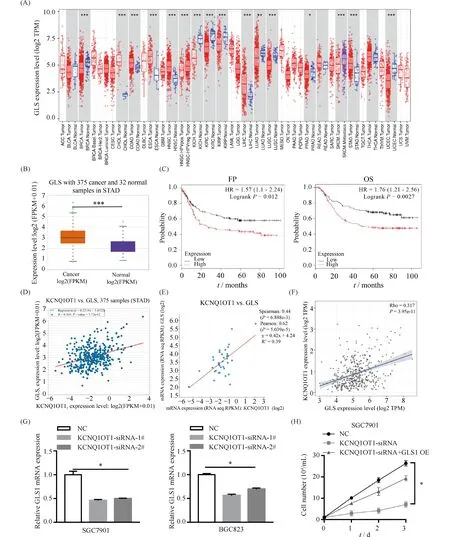
Fig.6 KCNQ1OT1 regulates the growth of gastric cancer cells through GLS1 (A) The expression level of GLS1 in different tumors or specific tumor subtypes was analyzed through the TIMER2 database. (B) The expression level of KCNQ1OT1 between normal and gastric cancer tissues in the StarBase database (***P<0.001). (C) The prognostic value of GLS1 was analyzed by Kaplan-Meier Plotter database (FP, first progression survival, OS, overall survival). (D-F) The expression of KCNQ1OT1 was positively correlated with the expression of GLS1 in gastric cancer via different databases (D, StarBase, E, cBio, F, TIMER2). (G) The relative expression of GLS1 mRNA was significantly downregulated after KCNQ1OT1 knockdown by quantitative PCR assays. (H) Overexpression of GLS1 can partially rescue the inhibitory effect on the growth of gastric cancer cells with KCNQ1OT1 knockdown. Values are the mean ± SD of three determinations from separate experiments (*P<0.05)
3 讨论
胃癌的发生发展是一个涉及多基因与多步骤的复杂过程[21]。我国的胃癌发病率明显高于世界水平,虽然已经在胃癌的诊疗方面取得了巨大进步,但是大部分病人的预后仍然较差,需要进一步研究胃癌的发病机制,发现新的分子靶点。多项研究[22-24]表明,长非编码RNA KCNQ1OT1在肝癌、肺癌、乳腺癌、胶质瘤、黑色素瘤等多种类型肿瘤的进程中存在表达异常,但是在胃癌中的研究文献报道较少,并且关于其表达和作用的结论缺乏一致性。Feng等[9]研究表明,KCNQ1OT1在胃癌中低表达,过表达后可以抑制胃癌肿瘤细胞的生长。而Zhong等[10]研究表明,KCNQ1OT1在胃癌中高表达,敲低后可以抑制胃癌肿瘤细胞的生长。造成这种相反结论的原因可能是他们研究的病例数太少,取材有一定的片面性,并且研究的细胞也是不同的。本研究通过TCGA不同公共数据库综合分析了KCNQ1OT1的表达。结果发现,KCNQ1OT1在胃癌中普遍高表达,并且通过实验证明敲低后可以抑制胃癌肿瘤细胞的生长。这些结论与Zhong等的研究结果一致。由于本文通过TCGA数据库分析了上千例胃癌病人KCNQ1OT1的表达,能够较好地克服少量临床病例造成的偏倚,更加真实的反映了KCNQ1OT1在胃癌中的表达水平。通过进一步对KCNQ1OT1与胃癌病人临床因素的相关性的生物信息学分析,结果发现,KCNQ1OT1在各种类型胃癌中普遍高表达,且KCNQ1OT1的表达与TP53的突变有一定相关性,这也解释了KCNQ1OT1在胃癌高表达的部分原因。此外,本文通过生物信息学分析还发现,KCNQ1OT1与胃癌病人预后不良相关。由此表明,KCNQ1OT1在对胃癌不仅可以作为一种新型的诊断性的肿瘤标记物,也可以作为一种预后性的肿瘤标记物。
随着免疫治疗的研究进展,特别是PD-L1/PD1抑制剂,胃癌也成为有望治愈的恶性肿瘤之一,但是需要筛选真正适合免疫治疗的病人。肿瘤微环境中主要的成分是免疫细胞,这些免疫细胞跟肿瘤形成、进展和转移密切相关[25,26]。本文的研究发现,KCNQ1OT1的表达与免疫细胞浸润也存在明显的相关性,而更重要的是KCNQ1OT1的表达与PD-L1(CD274)的表达呈负相关关系。目前,应用PD-L1抑制剂的条件是通过检测PD-L1的表达含量以及肿瘤突变负荷(TMB)等条件,但是仍然存在较多的缺点[27,28],造成符合条件的病人未取得理想的效果,而且漏掉了不符合这些条件反而能够取得较好疗效的病人。因此,需要更多的肿瘤标记物去预测是否使用PD-L1抑制剂。而本文的研究提示:高表达KCNQ1OT1的胃癌病人有可能不适合应用PD-L1抑制剂进行治疗,但是由于其表达与免疫细胞浸润存在明显的相关性,有可能适合其他类型的免疫治疗。当然这种假设需要进一步的临床实验去验证,从而帮助临床找到真正适合免疫治疗的胃癌病人。
谷氨酰胺代谢是恶性肿瘤存活的关键,特别是为肿瘤细胞的增殖及生物大分子合成方面具有十分重要的作用[29]。最新的研究显示,相对于肿瘤微环境喜欢利用葡萄糖代谢,肿瘤细胞更加喜欢利用谷氨酰胺作为代谢原料[30]。谷氨酰胺酶1(glutaminase 1,GLS1)是一种代谢酶,在谷氨酰胺分解中发挥重要作用,GLS1常在高度增殖的癌细胞中过表达以满足增强的谷氨酰胺代谢的需求[31]。目前,多项研究证实,GLS1是致癌过程中的重要靶点[32,33]。本研究通过生物信息学综合分析了KCNQ1OT1在胃癌中可能发挥的作用机制,发现KCNQ1OT1与肿瘤代谢有密切的相关性,尤其是对谷氨酰胺代谢的影响。本文通过生物信息学和实验证明,KCNQ1OT1可以通过调节GLS1介导的谷氨酰胺代谢,促进胃癌肿瘤细胞的生长。有研究证实,KCNQ1OT1在结直肠癌中可以调控糖酵解途径[34]。而本文是第一个报道KCNQ1OT1可调控谷氨酰胺代谢的研究。本研究进一步丰富了关于KCNQ1OT1调控肿瘤代谢的理论。
本研究尚存在较多的局限性。例如,通过生物信息学进行了分析,但是生物信息学本身由于算法等问题也可能存在一定的误差,需要临床数据进行验证。另外,本研究尚存在几个问题没有解决:首先,TP53基因突变为什么造成了KCNQ1OT1表达的差异?KCNQ1OT1为什么与免疫细胞浸润明显相关,而与PD-L1的表达却是负相关的?临床病例中KCNQ1OT1对免疫治疗的指导意义是怎样的?KCNQ1OT1调控GLS1表达的具体机制是什么?这些问题将在后续的工作中进行研究。总之,本研究通过生物信息学和分子实验揭示了KCNQ1OT1在胃癌组织中的表达及功能,提示KCNQ1OT1很可能通过调控GLS1介导的谷氨酰胺代谢来促进胃癌的发生和发展,并导致胃癌病人预后不良,为深入研究该基因功能提供了线索,也为分子靶向治疗胃癌的临床研究提供了新的靶点和思路。
