嵌合RNA的形成机制及生物学意义
2022-09-07秦付军
陈 晨, 秦付军, 2), 3), 4)*, 唐 悦*
(1)郑州大学基础医学院微生物学与免疫学系, 郑州 450001;2)郑州大学医学科学院分子病理中心, 郑州 450052;3)郑州大学医学科学院转化医学平台, 郑州 450052;4)郑州大学省部共建食管癌防治国家重点实验室, 郑州 450052)
科学家从慢性粒细胞白血病(chronic myelocytic leukemia, CML)患者细胞中发现了一条较短的22号染色体,并将其命名为费城染色体[1];随后在这种异常染色体上发现了由9号染色体的C-ABL基因与22号染色体的BCR基因发生易位而形成的融合基因BCR-ABL。这种染色体易位所导致的融合基因及其产物也是早期发现的嵌合RNA(或蛋白)的一种类型。BCR-ABL的发现及其靶向药物格列卫在临床的成功应用,促进了使用嵌合RNA作为生物标志物和药物靶标相关研究的快速发展[1]。
由于早期的嵌合RNA主要是在肿瘤中被发现,因而人们通常认为嵌合RNA都具有癌症特异性。随着RNA-Seq技术的广泛使用和二代测序技术成本的不断降低,越来越多的RNA-seq数据被上传到公共数据库中并被下载和分析,通过使用不同的嵌合RNA分析软件和检测方法,在不同癌症数据库中能快速发现并筛选出可以作为该癌症生物标志物的新型嵌合RNA[2-10]。近来,通过对人类正常组织的RNA测序数据库GTEx(genotype-Tissue Expression)分析,我们发现正常组织中的嵌合RNA数量不仅远超科学家们的预期,而且具有独特的作用模式,在正常细胞中也具有一定的生理功能[11-15]。
嵌合RNA是由来自于不同基因的外显子片段融合在一起组成的融合转录本[12, 16]。传统观点认为,染色体重排是生成嵌合RNA的唯一机制,然而,已有足够证据表明,嵌合RNA也会通过各种RNA的转录调控和剪接形成,且未发生染色体的改变[11, 12, 16-19]。
本文将综述嵌合RNA的形成机制及生物学意义。对这一新的研究领域的探索,将有助于从更深层面上挖掘人类基因组的生理功能和调控机制,便于更全面地了解嵌合RNA在分子与细胞水平和各种疾病中所发挥的功能和意义。
1 嵌合RNA形成机制
1.1 染色体重排
1.1.1 染色体重排的定义 染色体重排(chromosome rearrangement)是指染色体发生断裂,与其他染色体相连,构成新的染色体,包括插入、缺失和易位,可导致细胞遗传学上不同的基因位点并列在一起(Fig.1A)。过去由于技术限制,大多数基因融合只在血液学疾病和儿童恶性肿瘤中被发现,包括慢性粒细胞白血病中t(9;22)染色体易位导致的融合基因BCR-ABL;急性淋巴细胞白血病中t(12;21)染色体易位导致的融合基因TEL-AML1[20]等。这些融合基因的发现表明,嵌合RNA可通过DNA水平的染色体重排产生。
1.1.2 染色体重排形成的分子机制
(1)DNA复制障碍诱导染色体重排
DNA复制障碍能诱发染色体重排以及基因组拷贝数变异(copy number variation,CNV)。在癌症发展过程中,环境因素和致癌基因诱导的增殖会增加复制压力。当压力阻止DNA复制时,DNA复制子和停顿的DNA复制叉处于连接状态,且受到S期检测点的保护。当所受压力得到缓解时,停顿的DNA复制叉可重新进行有效复制。然而,当DNA聚合酶从DNA复制叉解离或者复制叉结构被破坏时,DNA复制则需要同源重组或者是微同源序列介导才能重新启动[21]。 Mizuno等[22]在研究中表明,同源重组启动DNA复制叉,同时DNA复制叉容易出错的特点可诱发癌症细胞中的染色体重排和基因扩增。
(2)DNA双链断裂诱导染色体重排
染色体重排过程的关键一步是诱导2个DNA双链断裂(DNA double-strand break, DSB)。诱发DNA双链断裂发生的可能机制有[23]:1)由于电离辐射、自由基氧化损伤或自发水解导致的随机位置的断裂;2)与拓扑异构酶抑制剂治疗相关的断裂;3)由未知的链断裂机制介导的直接或反向重复序列的断裂。在淋巴样细胞中,DNA双链断裂通常是生理性的在淋巴样抗原受体位点的V(D)J重组(H链可变区的D基因的任一段与J基因拼成DJ基因后再与V基因任一段相结合而形成新基因的过程)、或类别转换重组(class switch recombination, CSR)的结果;4)、重组活化基因(recombination activating gene, RAG)复合体在符合其正常V(D)J重组信号靶点的位点上引起序列特异性的DNA双链断裂;5)在单链到双链的过渡区域发生的结构特异性的DNA双链断裂;6)单链DNA位点的激活诱导的胞苷脱氨酶(cytidine deaminase, CyD)作用,可能发生在类别转换重组区域和体细胞高频突变区域(somatic hypermutation, SHM)的正常靶基因位点以外的病理位置。
1.2 反式剪接
1.2.1 反式剪接的定义 在反式剪接(trans-splicing)过程(Fig.1B)中,成熟的RNA通过2个独立的mRNA前体分子融合而成。前体mRNA分子通常来源于多个不同基因(基因间反式剪接)[24-26]或相同基因(基因内反式剪接)[27-29](Fig.2)。基因内反式剪接产物包括外显子复制或重排[12],以及由互补链转录的外显子构成的RNA[28, 30]。理论上,一些外显子重排会由于重新剪接而形成环状RNA。环状RNA不具有游离末端,因此它们会通过经由核糖核酸酶R处理与链式RNA区分[31]。目前,仍不清楚在不同谱系中是丢失还是获得了反式剪接[32]。
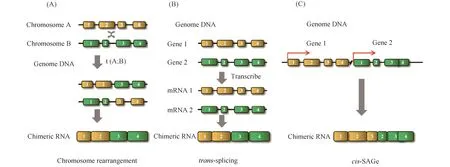
Fig.1 Three mechanisms for the production of chimeric RNAs are known Three known generating mechanisms for chimeric RNAs. Exons are depicted as blocks, and introns are indicated by lines. (A) Chromosomal rearrangement including translocation, deletion, and inversion. Shown here is a case of translocation. Gene fragments from different genomic loci are juxtaposed together. (B) RNA trans-splicing. Two separate pre-mRNA transcripts are spliced together. (C) cis-splicing between adjacent genes.The transcription machinery reads through two neighboring genes, and the exons from the two genes are spliced together

Fig.2 Classification of different splicing mechanisms Classification of different types of splicing. The two main classes are cis- and trans-splicing (TS). Canonical cis-splicing involves transcripts from one gene. Adjacent genes that transcribe in the same direction can also form chimeric RNAs, via the cis-SAGe mechanism. In primitive organisms, TS usually occurs between a short leader sequence (SL) and the transcript of another gene; however, an SL is not required in all of the cases—joined transcripts can derive from different genes, via intergenic TS, or from different exons of the same gene, via intragenic TS,which includes sense-antisense chimeras, as shown here. The latter also includes exon shuffling and exon repetition (not depicted in this figure)
以嵌合RNAJAZF1-JJAZ1为例,它是由7号染色体p15上的JAZF1基因的5′-端外显子和17号染色体q11上的JJAZ1/SUZ12基因的3′-端外显子组成。正常的子宫内膜基质细胞中存在该嵌合RNA,其组成与在子宫内膜间质肉瘤(endometrial stromal sarcoma, ESS)[33]中发现的JAZF1-JJAZ1融合是相同的。前期的细胞遗传学分析表明,人子宫内膜间质细胞中7号和17号染色体均正常,利用Southern印迹及FISH实验表明,人子宫内膜间质细胞中未发现染色体重排现象;通过体外反式剪接检测表明,人子宫内膜间质细胞中的JAZF1-JJAZ1嵌合体是RNA 反式剪接的结果[24]。以上结果说明,嵌合RNA可以通过RNA反式剪接机制形成。
1.2.2 反式剪接形成的分子机制
(1)低等生物中反式剪接的分子机制:短前导序列介导的反式剪接
在锥虫体中最早发现了一种古老的反式剪接类型,SL反式剪接(short leader sequence trans-splicing,SLTS)[34]。该类型后来在其他低等真核生物中也被发现,包括:甲藻[35]、眼虫门生物[36]、线虫类生物,例如秀丽隐杆线虫[37]等。在这些生物体中,SL反式剪接通过非编码的短前导序列(short leader sequence,SL)连接到转录物的5′端产生。在这个过程中,短前导序列mRNA的内含子中的剪切体结合位点会结合核小核糖核酸蛋白(small nuclear ribonucleic protein,snRNP)(U1 snRNP除外),其利用传统的剪接供体和受体位点进行后续进程。因此,转录本具有共同的前导序列。有趣的是,SL RNA可能直接从U1 snRNA进化而来,已有研究支持这一观点。在该研究中,U1 snRNA只通过添加1个剪接供体序列就被转化为SL RNA,而核苷酸序列几乎未见变化[38]。高等真核生物中SL反式剪接的发生极少,在原始脊索动物中观察到了SL反式剪接,但在植物、真菌和昆虫中未观察到[30, 37]。然而,人体内尚未发现SL反式剪接存在的证据,其中的反式剪接不涉及任何常见的短序列,而是2个独立的基因转录物相互作用的结果[30, 37, 39]。
(2)脊椎动物反式剪接可能的分子机制
脊椎动物反式剪接的分子机制仍不清晰。此前的研究提出了一些模型:
1)剪切体介导的反式剪接
与顺式剪接类似,反式剪接通常使用经典剪接位点来连接2个独立的原始转录物。因此,反式剪接可能使用与顺式剪接相同的基本组件。体外研究已经证明,剪接体机制能够产生反式剪接的嵌合体[40, 41]。有研究表明,RNA剪接体产生的反式剪接RNA能用来对疾病进行RNA治疗[42]。但是,是否所有反式剪接事件都是利用与常规剪接相同的机制,仍然有待研究。
2)短同源序列(short homologous sequence,SHS)介导的反式剪接
这种机制假定转录组件能从“第一”转录本中解离出来,并以某种方式移动到新的位置并继续转录。这主要涉及短同源序列,它可能将转录组件引导到另一个位点,甚至可能是附近的染色体上[43]。这种机制不能解释大部分利用经典剪接位点的嵌合RNA。此外,已知同源序列会诱导假阳性结果,因为它们的相似性在反转录或PCR过程中会驱动模板转换[31, 44]。
3)tRNA介导的反式剪接
两个单独的转录物可以通过普遍的tRNA序列连接,它们与伴侣基因相邻并与tRNA核酸内切酶共同作用[45]。
4)三维空间距离邻近可能诱导反式剪接
较近的三维空间距离可能是嵌合RNA形成的必要因素。据推测,嵌合RNA的母基因彼此(三维空间)靠近,可以共享相同的转录机制,或者至少在形成嵌合RNA的条件下瞬时共享。无论如何,在获得更多研究数据以前这仍然是一个有吸引力的假设。
1.3 顺式剪接
1.3.1 顺式剪接的定义 传统顺式剪接(cis-splicing)是指前体mRNA分子的外显子连接在一起形成成熟mRNA的过程(Fig.2),这些mRNA通常翻译为蛋白质[30]。前体mRNA分子的剪接,即内含子从前体mRNA上被切除后形成成熟mRNA的过程。但在某些情况下,能观察到嵌合RNA分子通常由2个不同基因编码的转录物片段组成。不同于常规的、正常剪接形成的RNA,嵌合体是通过基因间剪接机制产生的非传统的RNA融合物[12, 46]。
1.3.2 顺式剪接形成的分子机制
(1)5′端基因的聚A信号突变可产生嵌合RNA
一般认为,顺式剪接也存在不同的分子机制,且在一定程度上与上游基因的转录终止信号的变化有关。早期的研究指出,5′端基因的聚A信号突变可能导致嵌合RNA的产生[47]。但目前仍未获得更多的研究证实[48],而且也不能完全阐明大量顺式剪接嵌合体存在的原因。
(2)扭转应力促进相邻基因间顺式剪接嵌合RNA的形成
Meyer和Beslon在研究DNA螺旋的力学性质时提出,扭转应力是相距较近(仅仅几千个碱基)的2个基因连接的原因之一[49]。扭转应力可能会一方面促进上游基因的活跃转录,另一方面促进下游基因转录机制的启动。然而,此理论仅能解释相邻基因间顺式剪接嵌合RNA的形成原因,却在多数其他情况下并不适用。
(3)转录抗终止作用
早期,大鼠细胞中的终止信号旁路作用主要由于转录抗终止所导致[50],人类正常的卵巢细胞中也存在该作用[51]。此外,在低等生物体细菌和噬菌体中也观察到类似的作用现象,并且它似乎是一个高度调控的事件[52]。具有短同源序列的母基因可通过“转录滑动”模型形成嵌合体。在这个模型中,模板序列之间的高度相似性会干扰转录机制的进行,导致其迁移到附近的染色体区域,甚至迁移到另一个位点(三维结构中的邻近区域)[43]。然而,在这些情况下,很难辨别模板转换是一种生理过程,还是研究分析中逆转录过程产生的假象[31, 44, 53],后者可能导致产生假阳性结果。McManus等的研究也表明[28],所有成年果蝇中的嵌合体都已被证实为模板切换所造成的假象。
1.4 相邻基因顺式剪接
在对大量双末端RNA-Seq的数据研究中发现,有许多相邻基因外显子连接形成的嵌合RNA。通过对前列腺癌和非癌症样本的分析研究,本文作者从中筛选分离到300多个嵌合RNA,其中30%的嵌合RNA也被鉴定为由相邻基因外显子形成[54]。因此,将此类由相邻基因外显子经顺式剪接机制产生的RNA 定义为“相邻基因顺式剪接” (cis-splicing of adjacent genes,cis-SAGe)嵌合RNA(Fig.1c)。直观上看,cis-SAGe需要来自5′母基因的活跃转录,基因边界的转录通读,以及2个基因外显子之间的选择性剪接。转录因子CTCF结合到相邻基因间的绝缘子序列,形成基因边界,从而影响嵌合RNA的形成[19]。以SLC45A3-ELK4为例:SLC45A3-ELK4由于其可作为潜在的前列腺癌生物标志物已得到了大量的关注[55]。这个嵌合体的两个母基因位于染色体1q32,相隔25 kb。利用小干涉RNA特异性地敲低CTCF的表达,可引起该嵌合RNA表达的增高。FISH、CGH阵列、Southern印迹和qPCR的研究结果表明,参与融合的2个外显子之间并未发生任何DNA缺失而导致嵌合RNA的产生。体外反式剪接研究没有检测到SLC45A3-ELK4嵌合RNA,表明其不是RNA 反式剪接的结果[56]。相反,用特异嵌合RNA下游引物进行qRT-PCR实验,可从相邻母基因的基因间区域检测到RNA前体转录本。上述结果表明,这类嵌合RNA是cis-SAGe的产物[18]。
通过对形成cis-SAGe的母基因的序列及相关特征进行分析[54],结果显示,参与形成嵌合RNA的基因间距各不相同,但通常在8.5~30.6 kb范围内,显著地小于人类基因组内的平均基因间距(大约为50 kb)。最常见的顺式剪接嵌合RNA的剪接,通常发生于5′端基因的倒数第2个外显子和3′端基因的第2个外显子[54, 57](Fig.3)。这个结果并不令人意外,因为它包含了5′端基因的最后1个剪接供体位点和3′端基因的第1个剪接受体位点。目前,在嵌合连接位点周围尚未发现特定的序列模板,这可能是由于目前的研究仅限于一小部分真正的顺式剪接案例。随着越来越多的顺式剪接嵌合RNA被发掘,传统基因的定义也亟需不断的完善与补充。基因频繁地越过边界进行转录并与相邻的基因形成嵌合RNA,对定义一个基因的确切起始位置提出了挑战。
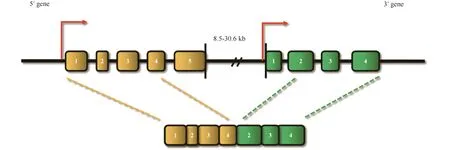
Fig.3 General characteristics of cis-SAGe chimeric RNAs Common characteristics of cis-SAGe chimeras: (1) parental genes are usually separated by a relatively small distance, 8.5~30.6 kb region, (2) 50 gene is actively transcribed, and (3) the fusion transcripts tend to have the second-to-last exon of the 50 gene joining to the second exon of the 30 gene
2 嵌合RNA的生物学意义
2.1 嵌合RNA广泛存在于人类多种正常组织和细胞中
基因融合及其产物(RNA和蛋白质)曾被认为是癌症的特有现象。近年来研究发现,嵌合RNA在人类多种正常组织和细胞中也广泛存在。此前,本文对来源于30种人类正常组织和细胞以及15种小鼠正常组织的300个RNA-Seq数据库进行了嵌合RNA的生物信息学分析[12]。在这些样本中,发现了9 778个嵌合RNA,大部分嵌合RNA只出现在一个组织(细胞)样本中,大约10%的嵌合RNA存在于一个以上的组织(细胞)样本中。通过分析发现,大多数嵌合RNA使用经典的剪接位点进行融合,且较大可能是相邻基因顺式剪接或RNA反式剪接的产物。通过一系列分析发现,51个嵌合RNA在多种不同组织(超过5种以上)中表达,例如嵌合RNACTBS-GNG5、CTNNBIP1-CLSTN1等[12]。近来,本课题组研究发现,下调或过表达嵌合RNACTNNBIP1-CLSTN1能够影响HEK-293T、HUVEC和LO2的细胞增殖和迁移。其中,下调CTNNBIP1-CLSTN1的表达能够诱导细胞凋亡,并引起细胞周期G2/M期阻滞。在转染siCTNNBIP1-CLSTN1的HEK-293T细胞中,过表达CTNNBIP1-CLSTN1,可拯救因下调CTNNBIP1-CLSTN1导致的细胞增殖减慢。而过表达母基因CTNNBIP1后,无明显拯救作用[17]。在对大脑新皮层发育的研究中发现,CTNNBIP1-CLSTN1在外放射状胶质细胞中富集,在人类进化的过程中与大脑皮层发育相关,通过对Wnt/β-catenin信号通路的精细调控来实现大脑皮层的发育模式[58]。
嵌合RNA可在肿瘤细胞和正常细胞中同时存在,其对生理功能的影响并不局限于肿瘤细胞。嵌合RNAJAZF1-JJAZ1可以在子宫内膜基质细胞和子宫内膜间质肉瘤中同时存在[33]。嵌合RNADUS4L-BCAP29曾被认为是前列腺癌和胃癌中存在的癌症特异嵌合RNA,并具有一定的致瘤作用[59]。本课题组进行深入研究后发现,DUS4L-BCAP29也存在于多种正常组织中,并与癌细胞中的表达水平相当,是相邻基因顺式剪接的产物。细胞功能研究表明,DUS4L-BCAP29可以促进非癌症细胞的生长和迁移能力,其促生长作用并非癌细胞所独有[60]。这些结果对使用嵌合RNA作为肿瘤生物标志物,尤其是用于检测癌症疾病的应用提出警示,在寻找真正的癌症特异性嵌合RNA时,需要考虑正常情况下基因间剪接产生嵌合RNA的可能性。
嵌合RNA的表达具有组织特异性和细胞阶段特异性。西班牙国家癌症研究中心的生物信息学研究者通过高通量RNA测序、质谱分析和功能注释等,在16个人类组织样本中发现了175个嵌合RNA转录本和12个嵌合蛋白质,这些嵌合RNA明显比非嵌合RNA呈现更高的组织特异性。嵌合RNA的组织特异性和细胞阶段特异性的特征,为识别未分化肿瘤的细胞来源提供了依据。本文作者对横纹肌肉瘤RH30细胞系的整个肌生成过程中的4个不同时间点细胞进行了转录组分析,最终发现18个嵌合RNA是在肌肉生成过程的一个特定时间点内细胞中特异性表达的[61]。此类嵌合RNA的挖掘为鉴定细胞来源提供了另一种独特的视角,并为研究其他肿瘤提供思路和可能。
2.2 嵌合RNA能作为癌症的特异性标志
嵌合RNA通过编码的嵌合蛋白质(Fig.4A)行使功能进而诱导肿瘤发生,也可以作为长非编码嵌合RNA调控癌症细胞的增殖和迁移(Fig.4B)[16, 18]。目前,BCR-ABL1融合基因已被广泛地应用于临床诊断和靶向治疗[62],它存在于95%的慢性粒细胞白血病中,其编码的P210蛋白能增强酪氨酸激酶的活性,从而改变细胞内多种蛋白质酪氨酸磷酸化水平,影响细胞内正常信号的传导,并抑制细胞凋亡的发生。格列卫是一种有效针对该嵌合蛋白质,且已在临床上被广泛应用的抗癌药物。格列卫具有特异的ABL酪氨酸激酶抑制作用,可有效地抑制BCR-ABL1激酶底物中酪氨酸残基的磷酸化,并使得该酶失活,从而发挥治疗作用[63, 64]。
嵌合RNA也可通过干扰母基因的表达而发挥致癌作用。约50%的前列腺癌患者过表达ETS转录因子家族的基因,其中90% ETS 表达嵌合RNATMPRSS2-ETS。TMPRSS2-ETS是由TMPRSS2的5′UTR区域与ETS转录因子家族的一个成员结合形成。此嵌合RNA可诱导一种雄激素应答调控元件与ETS结合,该元件在应对雄激素激活时可上调ETS转录因子的表达水平,从而导致肿瘤的发生[65](Fig.4C)。环状RNA由于其持久的化学稳定性成为受人关注的癌症治疗靶标。近年Pandolfi等研究人员,利用 RNase R 消化线性嵌合 RNA,设计反向引物,进行 RT-PCR、Sanger 测序等实验,鉴定出一类重要环状RNA分子——融合(嵌合)环状RNA[66]。融合环状 RNA由嵌合 RNA反向剪切形成,也是转录物组复杂性的重要体现[67, 68]。目前的融合环状RNA多源于急性早幼粒细胞白血病(acute promyelocytic leukemia)、急性骨髓性白血病(acute myeloid leukemia)等疾病的融合基因[67-69]。由嵌合转录本形成的融合环状RNA可以由“Fcirc”的算法特异性识别,但需要开展更多研究,以便理解融合环状RNA如何发挥作用[70]。

Fig.4 The central theory of chimeric RNAs Chimeric RNA-centric view. Chimeric RNAs can be generated by gene fusion, trans-splicing, and cis-splicing between adjacent genes (cis-SAGe). They can function as chimeric proteins, long non-coding chimeric RNAs (lnccRNA), or affect parental gene expression. Traditionally, they have been thought to be specific to cancer. However, increasing numbers are being found in normal physiology
2.3 嵌合RNA通过影响染色体重排从而诱导基因组畸变
参与染色体重排的母基因和参与基因间剪接的母基因存在部分基因重叠,这可能并不是巧合,嵌合RNA可能是RNA介导双链断裂后修复和重排的潜在模板。基因组的三维邻近区域双链断裂[71]后的错配修复可能会增加该区域发生易位的风险。嵌合RNA的形成使母基因在三维空间上彼此靠近,进而影响染色体重排。已有研究表明,RNA模板或相对应的cDNA(complementary DNA)可在无同源染色体的情况下,介导同源重组和双链断裂修复[72]的发生。有学者认为,反式剪接的嵌合RNA或逆转录的cDNA可以作为染色体重排的模板[73],这将为发生在DNA水平的基因融合提供另一种分子机制。在正常细胞和肿瘤细胞中,均出现类似JAZF1-JJAZ1和DUS4L-BCAP29的嵌合RNA,支持了这种可能性[60, 74]。
2.4 嵌合RNA可作为潜在的竞争性内源RNA
尽管部分嵌合RNA缺乏编码新蛋白质的功能,但与其他基因仍存在序列同源、组织特异和进化保守等表达特点。由于与母基因序列的相似性,嵌合RNA可作为竞争性内源RNA(competing endogenous RNA,ceRNA)或微小RNA(microRNA, miRNA)发挥作用。ceRNA能够与某些具有类似核酸序列的转录本竞争,作为海绵来为miRNA提供结合位点,进而调控目标基因表达[75]。miRNA的生理调控作用也已经在许多癌症和其他疾病中被发现和证实[76]。嵌合RNAPAX3-FOXO1和母基因PAX3因序列相似而具有共同的下游靶点,而miR-495则可以特异性地结合到人类PAX3基因的3′UTR,从而调控PAX3蛋白的表达。嵌合RNA的形成使人类细胞逃避了miR-495对PAX3基因表达的调控,从而使PAX3在人类干细胞和肌肉分化过程中的表达水平显著低于其他哺乳动物[77]。
2.5 嵌合RNA影响干细胞的分化
在基因组未改变的情况下,细胞获得新的生物学特性的连续过程,被称为干细胞分化。该过程主要是由转录因子(transcription factor,TF)通过驱动表观基因组的反复变化,最终调控细胞的成熟及命运[78]。在细胞分化阶段,基因组的染色质高级结构会发生显著的变化,进而影响基因间相互作用的频率。这些染色质相关的变化可能会破坏或引入嵌合RNA的表达。因此,很多嵌合RNA表现出明显的组织特异性[12, 61]。参与形成嵌合RNA的母基因也会由转录因子组成,在细胞分化的过程中转录因子会受到嵌合RNA的调控[61],且在癌症中特异表达或上调[16, 56, 79]。
选择性的基因内反式剪接RNA(trans-spliced RNA,tsRNA)可以调节胚胎干细胞的分化。Wu等[80]发现,有4种在分化和未分化的人胚胎干细胞中呈现出特异性差异表达的tsRNA,它们可通过与全能性相关因子NANOG和SUZ12的相互作用来抑制分化特异性基因的表达,从而影响细胞全能性的维持。
3 问题与展望
人类与低等生物(果蝇和线虫)之间存在许多不同,但基因数量却相近。显然,基因数量不是这些物种差异的根本原因。人类基因组包含约20 000个基因,而人体内蛋白质的种类却远多于理论数量。人类和小鼠的基因组的相似度高达95%,但人类转录组中的嵌合RNA数量却显著超过小鼠,并且比非嵌合RNA具有更显著的组织特异性表达的特征。上述内容中提及的几个案例表明,嵌合RNA在关键的细胞过程中发挥作用,这已经不是孤立的现象。嵌合RNA在人体中的存在形式,以及在不同组织中的生理功能将是未来需要研究的重要方向之一。
目前,GTEx项目已经收集了包括54个正常组织在内的948个非癌症捐献者的数据资料,为科学界研究人类基因组的表达调控及其与遗传变异的关系提供了资源[13, 15]。目前,它被广泛应用于研究基因表达数量或表达数量性状基因座(expression quantitative trait loci,eQTL),但它也是研究非癌组织中嵌合RNA表达的非常有用的资源。这些丰富的原始RNA-Seq数据能够用于验证之前发现的嵌合RNA和分离鉴定新的嵌合RNA。通过对正常机体中嵌合RNA的序列、表达、来源和分布等基本特征进行深入了解,挖掘嵌合RNA生成、调控和功能等机制,将丰富人们对RNA分子家族的认识,从而便于全面理解转录组的复杂性和蛋白质组多样性。
嵌合RNA的发现和研究,较大程度上依赖于生物信息分析算法和检测技术的发展[10]。目前,基因测序技术已经发展到了第3代,可以实现单分子测序和超长读长。基于3代测序结果的分析,已实现全长嵌合RNA电子克隆,也将提高嵌合RNA的阳性检测和验证效率。
相当一部分的嵌合RNA会以长非编码嵌合RNA(lnccRNA)形式存在,同时也有证据证实了融合环状RNA的存在[67, 68]。它们与经典的lncRNA、circRNA在表达特征、作用方式上会有何不同?这是今后需要解决的问题。
此外,嵌合RNA产生的详细机制尚不完全清楚,其发挥生理作用的相关分子机制有待探索。嵌合RNA未来将成为潜在的新型生物标记物和治疗靶标,并为疾病的临床治疗和致癌病理提供新的理论和研究方向,同时有助于理解嵌合RNA在不同物种进化过程中的功能和意义。
