GSIS超高维变量选择
2015-06-01马学俊
马学俊
(中国人民大学 应用统计科学研究中心, 北京 100872)
GSIS超高维变量选择
马学俊
(中国人民大学 应用统计科学研究中心, 北京 100872)
变量选择在超高维统计模型中非常重要。Fan 和Lv基于简单相关系数提出确保独立筛选法(SIS),但当自变量被分成组时,SIS就会失效。因为SIS只能对单个变量进行选择,不能对组变量进行选择。为此,基于边际组回归提出组确保独立筛选法(GSIS),该方法不仅对组变量有效,对单个变量也有效,或者两者的混合也同样有效。Monte Carlo模拟结果显示,GSIS的表现优于SIS。
组确保独立筛选法;确保独立筛选法;变量选择;边际组回归
一、引 言
伴随着人类社会的发展,收集数据的种类越来越多。通常存在一种样本量n相对于变量个数p特别小的数据,或者说变量的个数远大于样本量个数的数据,如遗传、基因芯片和磁共振成像等[1]。一般地,我们假设lnp=O(nα),其中α∈(0,1/2),即自变量个数是样本量个数的指数倍,也称为NP(Nonpolynomial)灾难,或超高维(Ultrahigh)灾难。通常假定只有很少的自变量对于因变量产生影响,这也是统计学中经常说的“稀疏性(Sparsity)”假设。这种假定具有一定的合理性,影响某一个事物的因素也许有很多个,但是起主要作用也许只有少数几个或很少的几个因素。稀疏性的假定是处理超高维(高维)问题的基本假定。
变量选择是统计学研究的重要问题。比较常见的方法有LASSO、 SCAD、MCP、 GLASSO、GSCAD和GMCP等,但是这些方法不能处理超高维的问题[2]。为了处理超高维问题。Fan和Lv基于简单相关系数提出确保独立筛选法(Sure Independence Screening),为了方便记为SIS[3]。确保(Sure)意味着该方法可以保证真实对于因变量有显著影响的自变量几乎都可以被选出。独立(Independence)的含义是指考虑每一个自变量与因变量的关系。Hall和Miller利用广义简单相关系数处理超高维问题[4]。Fan等利用边际似然对广义线性模型和分类模型进行了超高维的变量选择[5-6]。Fan等利用边际回归对可加模型和变系数模型进行了超高维的变量选择[7-8]。Liu基于条件相关系数对变系数模型进行了超高维的变量选择[9]。
实际应用中,存在某些变量组成一个整体,它们“同甘苦共患难”。在变量选择过程中,要么都保留,要么都删去。如我们在处理多分类变量时,经常使用虚拟变量。这些虚拟变量就是一组变量,变量选择时它们不能分开。此时SIS将不能胜任对于组变量的选择,因为简单相关系数针对的是两个变量而言,不是两组组变量。为此,本文提出组确保独立筛选法(Group Sure Independence Screening,简记为GSIS)。该方法不仅对组变量有效,对单个变量也有效,或者两者的混合也同样有效。
二、研究方法
(一)边际组回归
Y是因变量,Z=(Z1,Z2,…,Zp)′是p维的自变量向量。我们研究如下线性模型:
Y=β0+Zγ+ε
(1)
其中γ=(γ1,γ2,…,γp)′是p维未知参数,ε是随机误差项。
一般而言,超高维变量选择通过两个步骤实现。第一步是通过某一个“规则”初步选择变量;第二步是利用传统的方法对第一步选择出来的变量再进行变量选择。经过第一步计算,对于因变量有影响的自变量已初步选出,并且此时的自变量个数,传统的方法已可以快速计算。显然,第一步非常重要,第二步只是利用已有的方法进行变量选择。所以,本文重点阐述第一步的实施,第二步可以利用GLASSO、GSCAD和GMCP进行变量选择[2]。
设Z为已列标准化(即每个自变量已标准化),SIS是计算Y与每一个Zt(t=1,2,…,p)的相关系数,即:
w=Z′y
(2)
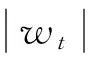
假设p个自变量可以分为J组,此时模型(1)可以写为:
(3)
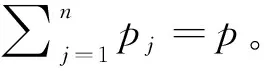
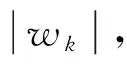
与SIS类似,GSIS考虑边际组回归,让第j组自变量Xj对Y进行回归,即最小化为:
(4)
如果Xj对Y有作用,那么边际回归模型的残差平方和就比较小;反之,如果Xj对Y没有作用,那么它们拟合的残差平方和比较大。SIS利用简单相关系数,而GSIS利用残差平方和。具体算法如下:
GSIS有效地利用了模型的残差信息,不仅对组变量有效,对单个变量也有效,或者两者的混合都有效。另外,GSIS可以推广到变系数模型、可加模型和分位数回归模型等统计模型。
(二)GSIS的延拓
对于变系数模型和可加模型,我们先用B样条逼近变系数部分或非参数部分,从而将非参数边际回归转为参数边际回归,然后对边际回归的残差平方和排序即可。以变系数模型为例,变系数模型的表达式一般是:
Y=a0(u)+a′(u)X+ε
(5)
其中a(u)={a1(u),a2(u),…,ap(u)}′是p维未知的函数向量,u∈[0,1]是指示变量(如时间等),ε是随机误差项,且E(ε|X,u)=0。
我们考虑边际非参数回归,即对第j个自变量Xj对Y进行回归,即最小化:
(6)
设B(u)={B1(u),B2(u),…,BK(u)}′是B样条基函数,其中K=T+r+1,r是样条函数的阶数,T为节点数,所以aj(u)≈η1jB1(u)+η2jB2(u)+…+ηKjBK(u),则式(6)可以转化为:
(7)
可以看出通过B样条逼近,将非参数边际回归式(6)转化为参数边际回归式(7)。也就是说,对于单个自变量Xj的选择转化为对于组自变量B1(u)Xj,B2(u)Xj,…,BK(u)Xj的选择,进而利用GSIS对自变量进行变量选择。可加模型与变系数模型类似,只需将式(7)修改为:
(8)
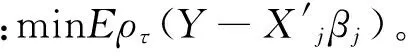
另外,对于分位数变系数模型和分位数可加模型,只需将式(7)、式(8)中的平方损失函数换成ρτ(v)=v(τ-I(v>0))损失函数即可。
三、Monte Carlo模拟
这里将进行Monte Carlo模拟,我们考虑如下模型:
模型(A):Y=2X1+X2+1.5X3+ε
其中X1=2Z1+3Z2+0.5Z3,X2=Z4+5Z5+3Z6,X3=1.2Z7+3Z8+Z9。
模型(B):Y=Z1+2Z2+3Z3+ε
其中Z={Z1,Z2,…,Zp}′~N(0,Σ),Σ是p×p正定矩阵,其元素ρij=0.5|i-j|,误差项ε~N(0,1)。我们重复模拟1 000次。样本量n=50,p=1 000,从而[n/log(n)]=13。我们考虑d=v[n/log(n)]=13v,其中v取1、2和3时相应的d记d1、d2和d3。
为了与SIS相比较,我们考虑如下指标:第一,bj为第j个非零自变量被正确选取的比例;第二,b为M⊂Mδ的比例,即全部非零自变量被正确选出的比例,其中M是真实非零自变量的下标集合。对于模型(A),GSIS和SIS的M分别是{1,2,3}、{1,2,3,4,5,6,7,8,9}。对于模型(B),GSIS和SIS的M都是{1,2,3}。SIS和GSIS对于模型(A)和模型(B)的结果见表1和表2。
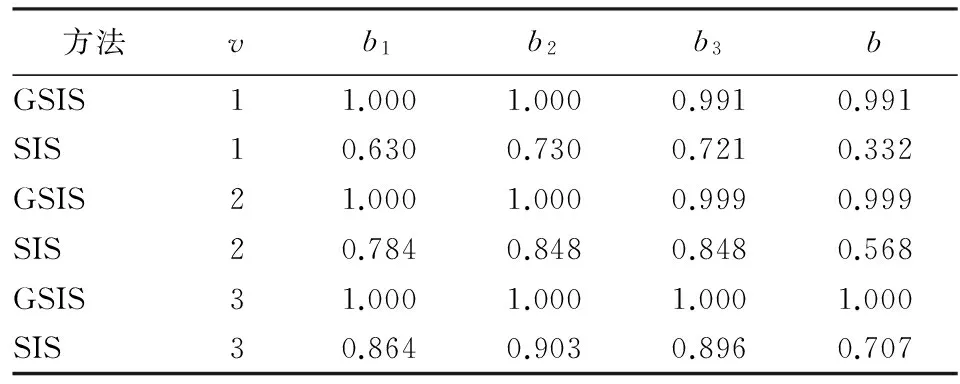
表1 模型(A)的SIS和GSIS结果
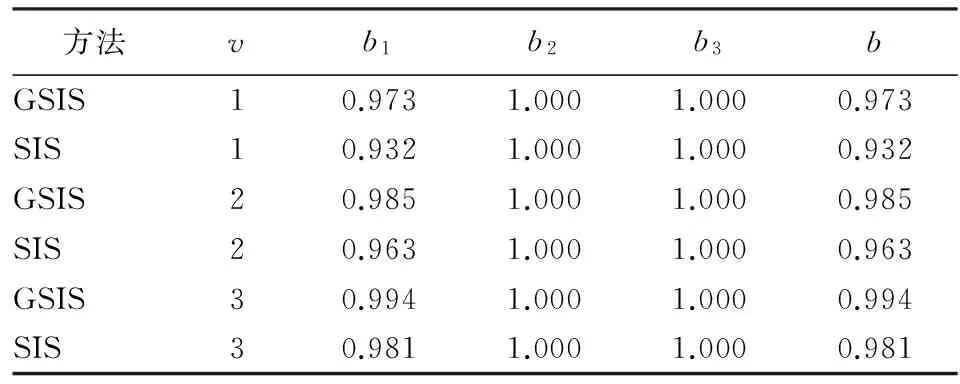
表2 模型(B)的SIS和GSIS结果
对于模型(A)而言,无论是单个自变量的选择,还是全部自变量的选择,GSIS的效果均优于SIS。如d=d1时,GSIS的b3=0.991,即1 000次模拟,只有9次不包含有X3,但SIS的b3=0.721,也就是说模拟1 000次,有279次没有选择X3。不同的d,GSIS的结果变化比较小,而SIS的结果变化比较大。如从d1变成3倍的d1,GSIS的b值只提高了0.009,而SIS从0.332 提高到0.707,其提高了2倍多。可见SIS对d比较敏感。对于实际问题,我们建议GSIS的d=[n/log(n)]。对于比较大的d,SIS对于个别组变量的识别效果还算可以,但是对全部自变量的识别能力欠佳。如d=d3时,SIS对于单个自变量(X1、X2或X3)的识别率均达到85%,但全部自变量的识别率只达到70%左右。相反,无论单个自变量,还是全部自变量,GSIS识别能力均可以达到99%以上。
对于模型(B)而言,SIS和GSIS效果都非常好,几乎都可以正确识别。对于X1和全部自变量的识别,GSIS 的效果仍略优于SIS。这一方面说明SIS和GSIS对于单个自变量的有效性,另一方面也说明了GSIS不仅仅可以胜任组自变量的选择,也可以胜任单个自变量的选择。综上所述,说明GSIS优于SIS。
四、实例分析
用心肌病数据研究基因对于G蛋白耦联受体(G protein-coupled receptor)Rol的影响。该数据的样本量是30,自变量是6 319个基因,因变量是Rol。为了消除量纲的影响,我们将自变量进行标准化处理。GSIS得到排列最靠前的2个基因是 Msa.2 134.0 和Msa.2 877.0。为了检验我们方法的合理性,我们总结了关于心肌病数据研究的几种结果(见表3)。Segal等得到Msa.2 877.0对于Rol的影响最大[10];Hall和Miller利用广义相关系数法排序得到前两个基因是Msa.2 877.0和Msa.1 166.0[4];Li等利用距离相关系数法排序得到前两个基因是Msa.2 134.0 和Msa.2 877.0。另外,Li等论证过:相对于Msa.2 877.0和Msa.1 166.0或Msa.2 877.0, Msa.2 134.0 和Msa.2 877.0对于Rol的影响更加显著[11]。从表3可以得到我们的方法支持Li等的结论,这也印证了GSIS方法的合理性。
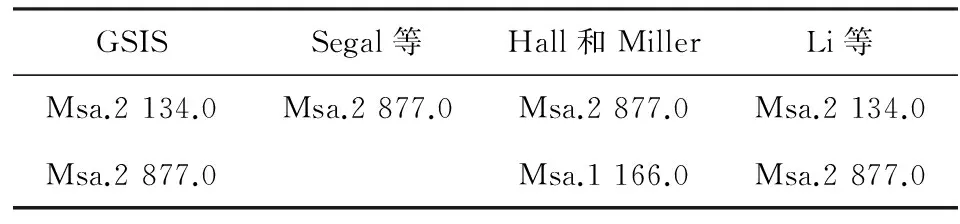
表3 心肌病数据研究的几种结果
五、结 论
本文基于边际组回归,针对组自变量提出GSIS。该方法有效利用模型拟合的残差信息,即如果自变量对于因变量有影响,那么它们边际回归模型的残差平方会比较小,相反,残差平方和会比较大。GSIS方法可以推广到变系数模型、可加模型以及分位数模型的超高维变量选择。根据Monte Carlo模拟的结果,我们可以得到SIS不能胜任组自变量的选择,而GSIS不仅可以胜任组自变量的选择,也可以胜任非组自变量的选择。对于单个自变量选择效果,GSIS仍会略优于SIS。
[1] 马超. 基于多基因组合选择模型的结肠癌特征基因选取[J]. 统计与信息论坛, 2012,27(6).
[2] Huang J, Breheny P, Ma S. A Selective Review of Group Selection in High Dimensional Models[J]. Statistical Science, 2012,27(4).
[3] Fan J, Lv J. Sure Independence Screening for Ultrahigh Dimensional Feature Space[J]. Journal of the Royal Statistical Society, Ser. B, 2008,70(5).
[4] Hall P, Miller H. Using Generalized Correlation to Effect Variable Selection in Very High Dimensional Problems[J]. Journal of Computational and Graphical Statistics, 2009,18(3).
[5] Fan J, Samworth R, Wu Y. Ultrahigh Dimensional Feature Selection: Beyond the Linear Model[J]. Journal of Machine Learning Research, 2009(10).
[6] Fan J, Song R. Sure Independence Screening in Generalized Linear Models With NP-Dimensionality[J]. The Annals of Statistics,2010, 38(6).
[7] Fan J, Feng Y, Song R. Nonparametric Independence Screening in Sparse Ultra-high-dimensional Additive Models[J]. Journal of the American Statistical Association, 2011,106(494).
[8] Fan J, Ma Y, Dai W. Nonparametric Independence Screening in Sparse Ultra-high-dimensional Varying Coefficient Models[J]. Journal of the American Statistical Association, 2014,109(507).
[9] Liu J, Li R, Wu S. Feature Selection for Varying Coefficient Models with Ultrahigh-dimensional Covariates[J]. Journal of the American Statistical Association, 2014, 109(505).
[10]Segal M, Dahlquist D, Conklin B. Regression Approach for Microarray Data Analysis[J]. Journal of Computational Biology,2003,10(6).
[11]Li R, Wei Z, Zhu L. Feature Screening Via Distance Correlation Learning[J]. Journal of the American Statistical Association,2012,107(499).
(责任编辑:李 勤)
Group Sure Independence Screening for Ultrahigh Dimensional Variable Selection
MA Xue-jun
(Center for Applied Statistics, Renmin University of China, Beijing 100872, China)
Variable selection plays an important role in high dimensional models. Fan and Lv showed sure independent screening(SIS) based on simple correlation. But when independent variable can be naturally grouped, SIS does not work. Because SIS is designed for individual variable selection, but not group selection. In this paper, we propose group sure independent screening(GSIS) based on marginal group regression . The method is designed for either variable selection or group selection, also for both. Monte Carlo simulations indicate that GSIS has superior performance in group and individual variable selection relative to SIS.
GSIS; SIS; variable selection; marginal group regression
2014-11-18;修复日期:2015-03-20
中国人民大学2014年度拔尖创新人才培育资助计划项目《变系数模型的变量选择》
马学俊,男,安徽颍上人,博士生,研究方向:应用数理统计。
F224.0∶O212
A
1007-3116(2015)08-0016-04
