CLPTM1L与miR-494靶向关系的双荧光素酶报告实验验证
2015-04-18张圣洁张学研臧文巧
张 仁,张圣洁,张学研,李 彤,臧文巧
郑州大学基础医学院微生物与免疫学教研室 郑州 450001
CLPTM1L与miR-494靶向关系的双荧光素酶报告实验验证
张 仁,张圣洁,张学研,李 彤,臧文巧#
郑州大学基础医学院微生物与免疫学教研室 郑州 450001
#通信作者,女,1979年8月生,博士,副教授,研究方向:食管癌病因学,E-mail: zangwenqiao@sina.com
miR-494; CLPTM1L; 双荧光素酶报告系统
目的:确定miR-494与CLPTM1L的靶向关系。方法:采用生物信息学方法预测miR-494与CLPTM1L基因的结合位点。采用PCR技术扩增CLPTM1L基因3’UTR片段,并克隆至pmirGLO载体,构建野生型及突变型重组双荧光素酶报告质粒。将培养的293T细胞分为4组,分别共转染miR-494或阴性对照和pmirGLO-CLPTM1L-W 3’UTR或pmirGLO-CLPTM1L-M 3’UTR,检测4组细胞中荧光素酶活性。结果:酶切和测序证实成功构建了野生型及突变型重组双荧光素酶报告质粒pmirGLO-CLPTM1L-W 3’UTR和pmirGLO-CLPTM1L-M 3’UTR;与共转染miR-494 mimics和突变型CLPTM1L 3’UTR质粒的293T细胞中荧光素酶活性(1.056 4±0.163 4)、共转染阴性对照序列和突变质粒的293T细胞中荧光素酶活性(0.961 3±0.177 9)或野生型质粒的293T细胞中荧光素酶活性(0.983 4±0.001 2)相比,共转染miR-494 mimics和野生型CLPTM1L 3’UTR质粒的293T细胞中荧光素酶活性(0.651 6±0.136 4)明显降低(F=4.476,P=0.040),其他3组间比较差异无统计学意义(P>0.05)。结论:CLPTM1L与miR-494存在靶向关系。
微小RNA(microRNA,miRNA)是一组内源性的、高度保守的非编码RNA,通过与mRNA 3’非编码区(3’UTR)结合,进行转录或转录后调控,从而调控靶基因的表达[1-2],进而参与一系列生物学过程,如脂肪代谢、细胞分化、凋亡等[3-4]。作者通过生物信息学方法分析了唇腭裂跨膜1样蛋白(cleft lip and palate transmembrane 1 like,CLPTM1L)基因与miR-494的靶向关系,并进一步应用双荧光素酶报告实验来验证miR-494对CLPTM1L的靶向调控作用。
1 材料与方法
1.1 主要材料 人肾上皮细胞系293T细胞株购自ATCC公司,T4 DNA 连接酶、限制性内切酶XbaⅠ和XhoⅠ、Taq DNA 聚合酶、dNTP 购自TaKaRa 公司,DH5α感受态大肠杆菌购自Solarbio公司;PCR产物回收试剂盒、质粒提取试剂盒、总RNA提取试剂盒购自Invitrogen公司,质粒pmirGLO、荧光素酶检测系统、DNA提取试剂盒购自Promega公司;根据CLPTM1L基因3’UTR序列(NM_030782)设计合成引物,上下游引物分别引入限制性内切酶XbaⅠ和XhoⅠ识别位点,PCR 引物由上海生工生物工程技术服务有限公司合成,序列见表1,由Invitrogen公司完成测序; miR-494 mimics 及阴性对照购自上海吉玛公司。
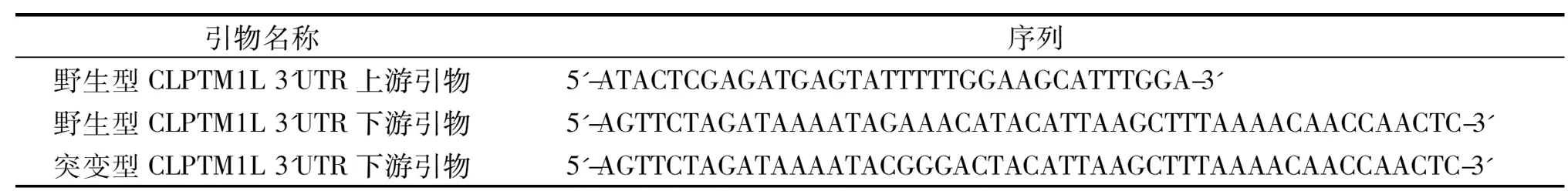
表1 PCR引物序列
1.2 miR-494靶基因的生物信息学预测 采用TargetScan 6.0、miRBase和miRanda软件在线搜索可能与CLPTM1L基因3’UTR作用的miRNA。
1.3 CLPTM1L基因野生型和突变型的3’UTR片段的扩增及纯化 以健康人外周血DNA 为模板,以野生型CLPTM1L 3’UTR的上、下游引物进行野生型3’UTR片段扩增,以野生型CLPTM1L 3’UTR上游引物和突变型CLPTM1L 3’UTR下游引物进行突变型3’UTR片段扩增。总体系均为30 μL。扩增条件:95 ℃预变性3 min;95 ℃ 35 s,55 ℃ 35 s,72 ℃ 40 s,共35个循环;循环结束后72 ℃延伸3 min。取扩增产物,用15 g/L的琼脂糖凝胶进行电泳检测,切胶纯化回收。
1.4 野生型及突变型CLPTM1L基因T载体的构建 将PCR扩增的CLPTM1L基因野生型及突变型胶回收产物与pGEM-T载体连接,在T4连接酶的作用下,置于4 ℃过夜连接,转化入Top10感受态细菌,进行重组子的筛选与鉴定。
1.5 CLPTM1L基因野生型及突变型双荧光素酶报告载体的构建 参照质粒提取试剂盒说明书分别提取野生型和突变型重组pGEM-T质粒,经限制性内切酶XbaⅠ和XhoⅠ双酶切,酶切产物经再次纯化回收,用T4 DNA 连接酶与荧光素酶报告载体pmirGLO连接,转化DH5α 感受态大肠杆菌,提取质粒,XbaⅠ和XhoⅠ双酶切初步鉴定,测序。重组质粒分别命名为pmirGLO-CLPTM1L-W 3’UTR和pmirGLO-CLPTM1L-M 3’UTR。
1.6 细胞培养及转染 于37 ℃、体积分数5%CO2、饱和湿度的培养箱中,用含体积分数10%胎牛血清的DMEM高糖培养基培养293T细胞,细胞贴满培养瓶瓶底约75%时可传代培养。实验用对数生长期细胞。采用胰蛋白酶消化293T细胞,PBS洗涤后,再加1.4 mL PBS调整细胞密度至2×107mL-1,分4组(A组:共转染阴性对照序列和野生型CLPTM1L 3’UTR;B组:共转染miR-494 mimics和野生型CLPTM1L 3’UTR;C组:共转染阴性对照序列和突变型CLPTM1L 3’UTR;D组:共转染miR-494 mimics和突变型CLPTM1L 3’UTR)进行电转,每组250 μL细胞悬液中分别加入30 μL miRNA-494 mimics或阴性对照序列和相应的重组质粒(pmirGLO-CLPTM1L-W 3’UTR/pmirGLO-CLPTM1L-M 3’UTR)6 μL,混匀后分别置于4个BTX电转杯中(4 mm),ECM2001电转仪电转,之后逐滴加入有血清的培养液中混匀,接种于24孔板中,每组设置3个复孔,置于37 ℃、体积分数5%CO2培养箱中培养。
1.7 各组细胞中荧光素酶活性的变化 收集转染30 h后的293T细胞,按照双荧光素酶报告基因检测试剂盒说明,使用荧光发光检测仪测定细胞中萤火虫荧光素信号及海肾荧光素信号。
1.8 统计学处理 采用SPSS 17.0进行分析,采用单因素方差分析和LSD-t检验比较4组细胞中荧光素酶活性的差异,检验水准α=0.05。
2 结果
2.1 生物信息学预测结果 见图1。通过在线分析软件分析,CLPTM1L基因3’UTR 465-471序列与miR-494序列间存在特定结合区域,CLPTM1L是miR-494的靶基因。

图1 miR-494在CLPTM1L 3’UTR 上的预测结合位点
2.2 野生型及突变型CLPTM1L基因T载体的构建结果 见图2。PCR结果显示,扩增野生型CLPTM1L的3’UTR 序列(446 bp)及突变型CLPTM1L 3’UTR 序列(452 bp),与理论片段大小一致( 图2左)。重组T载体通过XbaⅠ和XhoⅠ双酶切初步鉴定,分别得到野生型3’UTR片段及其突变型3’UTR片段( 图2右),与预期相符。

图2 野生型及突变型CLPTM1L基因T载体的构建结果
左:PCR 扩增CLPTM1L基因3’UTR 序列(M: DNA Marker; 1: 野生型;2:突变型);右:T载体重组质粒XbaⅠ、XhoⅠ双酶切(M:DNA Marker;1:野生型重组质粒双酶切片段;2:突变型重组质粒双酶切片段)。
2.3 重组野生型及突变型CLPTM1L基因pmirGLO载体的构建结果 见图3。将重组pmirGLO载体转化感受态大肠杆菌DH5α,随机挑取野生型/突变型菌落进行菌液PCR鉴定,结果显示与理论扩增的PCR片段大小一致(图3左)。测序结果证实插入的片段与预期完全一致(图3右)。

图3 重组野生型及突变型CLPTM1L基因pmirGLO载体的构建及部分测序结果
A:PCR 扩增CLPTM1L基因3’UTR 序列(M:DNA Marker;1:野生型CLPTM1L 3’UTR;2:突变型CLPTM1L 3’UTR);右:pmirGLO-CLPTM1L-W 3’UTR和pmirGLO-CLPTM1L-M 3’UTR部分测序图。
2.4 各组细胞中荧光素酶活性的变化 D组(1.056 4±0.163 4)、C组(0.961 3±0.177 9)或野生型质粒(0.983 4±0.001 2)的293T细胞中荧光素酶活性相比,共转染miR-494 mimics和野生型CLPTM1L 3’UTR的293T细胞中荧光素酶活性(0.651 6±0.136 4)明显降低(F=4.476P=0.040),其他3组间比较差异无统计学意义(P>0.05)。
3 讨论
在miRNA的研究中,经常用荧光素酶报告技术来验证目的基因是否为miRNA的靶基因。荧光素酶报告实验的原理是以荧光素为底物来检测萤火虫荧光素酶活性的一种报告系统[5]。荧光素和荧光素酶这一生物发光体系可以极其灵敏、高效地检测基因的表达。在以往实验[6]中通常使用pGL3报告载体,还需加入对照载体pRL-TK,实验时需要同时转染2个载体,步骤较为复杂,无形中增加了成本和劳动量,而且结果不稳定,批次内和批次间差异较大。该实验使用的pmirGLO报告载体[7]同时含有萤火虫荧光素酶基因和海肾荧光素酶基因,可以在单管中进行双荧光素酶报告基因测试,快速、灵敏、简便,还可减少内在因素的变化对实验准确性的影响,如不同实验组中细胞的数目和活力的差别、细胞转染和裂解效率的不同等[8]。
近年来研究[9]发现,miRNA的异常表达与肿瘤发生密切相关,其通过多种靶基因、信号通路对肿瘤发生发展进行调控。目前已证实miR-494的异常表达与多种肿瘤的发生有关[10],如胃癌[11]、胃肠道间质瘤、胆管癌、淋巴瘤[12-13]、肺癌[14-15]等。作者利用生物信息学技术预测CLPTM1L可能是miR-494的靶基因,通过构建野生型和突变型CLPTM1L基因双荧光素酶报告载体,经过双荧光素酶报告实验证实了miR-494与CLPTM1L基因具有靶向关系,且荧光素酶报告实验证实miR-494可以特异性作用于CLPTM1L基因的3’UTR位置的465-471序列,对其上游Luc基因表达进行负调控。这一结果为进一步研究miR-494在肿瘤中的作用提供了部分参考。
[1]Jaskiewicz L,Filipowicz W.Role of Dicer in posttranscriptional RNA silencing[J].Curr Top Microbiol Immunol,2008,320:77
[2]Lai EC.Micro RNAs are complementary to 3’UTR sequence motifs that mediate negative post-transcriptional regulation[J].Nat Genet,2002,30(4):363
[3]Brennecke J,Hipfner DR,Stark A,et al.Bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila[J].Cell,2003,113(1):25
[4]Ma L,Teruya-Feldstein J,Weinberg RA.Tumour invasion and metastasis initiated by microRNA-10b in breast cancer[J].Nature,2007,449(7163):682
[5]洪欣,尹昭云,裴雪涛,等.bax转录调控pGL3’-Basic荧光素酶报告基因载体的构建[J].中国应用生理学杂志,2004,20(4):317
[6]张衡,贾雅丽,岳文,等.细胞角蛋白19启动子调控的双报告载体的构建及其在肝干/祖细胞分化研究中的应用[J].生物化学与生物物理进展,2010,7(7):728
[7]谭靖,蒋先镇,何乐业,等.构建p53双荧光报告载体及其功能验证[J].中南大学学报:医学版,2013(11):1135
[8]高晓宁,林季,李永辉,等.FLT3基因3’-非翻译区-荧光素酶报告载体的构建及其活性鉴定[J].中国实验血液学杂志,2010,18(3):694
[9]杨生生.MicroRNA-494抑制肝癌细胞增殖与迁移能力的研究[D].上海:第二军医大学,2013.
[10]林燕明,张庆,唐志,等.miR-494慢病毒表达载体的构建[J].现代肿瘤医学,2013,21(5):930
[11]He W,Li Y,Chen X,et al.miR-494 acts as an anti-oncogene in gastric carcinoma by targeting c-myc[J].J Gastroenterol Hepatol,2014,29(7):1427
[12]Jones K,Nourse JP,Keane C,et al.Plasma microRNA are disease response biomarkers in classical Hodgkin lymphoma[J].Clin Cancer Res,2014,20(1):253
[13]Liu C,Iqbal J,Teruya-Feldstein J,et al.MicroRNA expression profiling identifies molecular signatures associated with anaplastic large cell lymphoma[J].Blood,2013,122(12):2083
[14]Romano G,Acunzo M,Garofalo M,et al.MiR-494 is regulated by ERK1/2 and modulates TRAIL-induced apoptosis in non-small-cell lung cancer through BIM down-regulation[J].Proc Natl Acad Sci U S A,2012,109(41):16570
[15]Ohdaira H,Sekiguchi M,Miyata K,et al.MicroRNA-494 suppresses cell proliferation and induces senescence in A549 lung cancer cells[J].Cell Prolif,2012,45(1):32
(2014-09-02 收稿 责任编辑赵秋民)
Identification of miR-494 target gene-CLPTM1L by luciferase reporter assay
ZHANGRen,ZHANGShengjie,ZHANGXueyan,LITong,ZANGWenqiao
DepartmentofImmunologyandMicrobiology,CollegeofBasicMedicalSciences,ZhengzhouUniversity,Zhengzhou450001
miR-494;CLPTM1L;luciferase reporter assay
Aim: Analysis and identification of miR-494 potential target gene CLPTM1L. Methods: Bioinformatics analysis suggested CLPTM1L was a target of miR-494. PCR was performed to obtain wild-type and mutant CLPTM1L 3’UTR fragments, which were cloned into pmirGLO and constructed the recombinant plasmids pmirGLO-CLPTM1L-W and pmirGLO-CLPTM1L-M, respectively. 293T cells were cultured. Two recombinant plasmids were co-transfected into 293T cells with the miR-494 mimics or scrambled oligonucleotide(negative control), and the relative luciferase activity was determined by luciferase reporter assay. Results: With identification of restriction enzyme digestion and sequencing, the sequences of CLPTM1L-W 3’UTR and CLPTM1L-M 3’UTR were cloned into pmirGLO successfully.Compared with the group of miR-494 mimics co-transfected with pmirGLO-CLPTM1L-M(1.056 4±0.163 4) and the groups of miR-NC co-transfected with pmirGLO-CLPTM1L-W(0.983 4±0.001 2) or pmirGLO-CLPTM1L-M(0.961 3±0.177 9), the relative luciferase activity in group of miR-494 mimics co-transfected with pmirGLO-CLPTM1L-W showed a significant decrease(0.651 6±0.136 4;F=4.476,P=0.040). No difference was found among the aforementioned three groups(P>0.05).Conclusion: CLPTM1L is a target gene of miR-494.
10.13705/j.issn.1671-6825.2015.03.033
Q786
