MALDI-TOF质谱在院内感染致病菌检测中的应用*
2015-03-04屠博文史伟峰韩晓冬徐晓怡
屠博文,史伟峰,韩晓冬,杜 强,徐晓怡,郝 超△
(1.江苏省常州市疾病预防控制中心 213000;2.江苏省常州市第一人民医院检验科 213000;3.南京大学医学院,南京210093)
院内感染具有较高的发病率和较强的传染性,逐渐成为一个全球性的问题,微生物耐药的不断增强威胁着全球人类健康安全。抗菌药物滥用现象不断加剧,细菌产生的耐药性日趋泛滥。产碳青霉烯酶是革兰阴性菌对碳青霉烯类抗菌药物耐药的主要原因[1]。随着新德里β内酰胺类B 型碳青霉烯类耐药菌在印度新德里的一家医院被首次检出,并逐渐在全球范围蔓延[2-4],耐药菌感染成为院内感染的主要致病因素。
耐药菌的早期诊断和疾病预防过程中,快速高效的微生物鉴定成为挽救患者和控制疾病蔓延的关键。传统的微生物鉴定方法通常采用血平板培养和生化反应鉴定结合的方法,目前该方法已经成为医院菌株鉴定的金标准技术。然而,随着院内感染状况的不断加剧,为了争取更多的治疗时间,致病菌的快速鉴定已经成为临床和疾控工作迫在眉睫的技术要求。MALDI-TOF质谱近年来开始在微生物鉴定中应用[5-6],该方法只在微生物实验室中被运用,MALDI-TOF质谱技术能够以稳定分子量的生物大分子为指纹图谱,通过建立大量的目标菌株图谱数据库来实现快速鉴定菌株的目的,具有高效、快捷和准确的特点。此外,蛋白质指纹图谱还能用于分析菌株的亲缘关系,但近年来菌株同源性的MALDI-TOF 质谱分析鲜有文献报道[7]。本文首先将两种快速鉴定院内感染致病菌的方法进行比较,并通过不同来源的同种耐药菌进行MALDI-TOF质谱图谱对比,分析不同来源致病菌的MALDI-TOF 质谱同源性,以此辅助评估耐药致病菌的传播风险,为建立临床突发院内感染的快速检测方法提供理论研究经验。
1 材料与方法
1.1 材料
1.1.1 样品收集及分离纯化 院内感染病例及污水样品取自某市第一及第二人民医院,待检样品经过营养肉汤复苏及血平板分离培养纯化获得待测菌株,制备成麦氏浊度5.0 的菌悬液,其余由MicroBank 细菌保存磁珠(Pro-Lab 公司)冻存于-70 ℃冰箱中。大肠杆菌标准菌株ATCC8739由法国梅里埃公司代购自美国菌株保藏中心。
1.1.2 培养基 营养肉汤干粉购自青岛海博生物技术有限公司,营养琼脂及血平板培养基平板购自科玛嘉生物技术有限公司。
1.1.3 仪器与试剂 主要仪器:MALDI-TOF 生物质谱仪(VITEK MS,梅里埃)、VITEK-2生化分析仪(VITEK-2Compact 30,梅里埃)、全自动革兰染色仪(PREVI Color Gram,梅里埃)、ISOLA 全自动平板涂布仪(PREVI ISOLA 29500,梅里埃)、恒温培养箱(GNP9050,精宏)、-80 ℃超低温冰箱(DW-86L338,海尔)。主要试剂:甲酸(formic acid,FA)、乙腈(acetonitrile,ACN)、乙醇和三氟乙酸(trifluoracetic acid,TFA),为色谱纯,购自Sigma公司;α-氰-4-羟基肉桂酸(α-Cyano-4-hydroxy-cinnamic acid,CHCA)粉末购自法国梅里埃公司;CHCA 饱和基质液是由超纯水、乙醇和乙腈按体积比1∶1∶1 配制而成。
1.2 实验方法
1.2.1 菌株培养与VITEK-2生化分析鉴定 将样品菌株通过ISOLA 全自动平板划线仪接种至普通大豆营养琼脂平板,37 ℃培养24h。选取单一菌落通过全自动革兰染色仪进行革兰染色,选用合适的GN 或GP芯片进行鉴定,每个样品设置2个平行组。
1.2.2 MALDI-TOF 质谱鉴定 参数调整:激光强度62 Hz,每孔采样率100 次,相对分子质量2 000~20 000,有效检测3 000~13 000,峰值调整模式为线性。质控校准:选取大肠杆菌ATCC8739作为质控菌,靶板点样48个样品进行分析后检出率达99.90%以上即为质控合格。本实验采用同一待测样品连续点样的方式进行分析,随机选取未知样品(编号:CZ14000267)和质控菌株ATCC8739 大肠杆菌,分别进行点样。在不同靶板上点样质控菌株(2组平行)和未知样品菌株(10组平行),收集每次检出的结果及特征峰图谱叠加进行比对。样品制备:吸取1.0μL的样品菌悬液(麦氏浊度5.0),滴加于MALDI-TOF 质谱细菌靶板对应孔,随后滴加1μL 甲酸乙醇溶液(甲酸∶乙醇∶水=1∶1∶1),充分混合菌斑后常温静置20min。接着滴加1.0μL 的CHCA 饱和基质液常温静置10min。待孔位结晶完整置于MALDI-TOF生物质谱仪进行分析。以ATCC8739 大肠杆菌为鉴定质控菌为阳性对照孔。质谱分析:采用RUO 系统软件对采集的样品图谱进行比对分析,与Myla指纹数据库中的对应标准菌株的超级图谱和参考图谱进行多重比对。以参考检出率表示为鉴定结果。超过75.00%的则为检出,75.00~79.90%代表可信性较低,80.00~89.90%代表有部分可信度,90.00~94.90%代表有较高的可信度,95.00~99.90%代表有极高的可信度;低于75%则代表未检出。
1.2.3 VITEK-2及MALDI-TOF 质谱方法比较 将院内感染患者样品分离获得的待测菌株,同时进行VITEK-2 和MALDI-TOF 质谱鉴定。以大肠杆菌ATCC8739为内参分别以检测时间、检出数量、检出率进行差异分析。
1.2.4 致病菌MALDI-TOF 质谱同源性分析 选取样品检出率最高的平行组图谱进行聚类分析。通过VITEK MS SARAMIS软件对图谱进行共性峰计算及峰值统计,共性峰分析数量设置为100~200,以树形图中距离远近表示同源性的差异。
2 结 果
2.1 VITEK-2及MALDI-TOF质谱分析结果 VITEK-2检出27株肺炎克雷伯氏菌和28株大肠埃希氏菌,有4株菌未检出。其中肺炎克雷伯氏菌的鉴定率为85.00%~99.00%,大肠埃希氏菌的鉴定率则为87.00%~99.00%。从培养单菌落获得菌悬液至检出共耗时43h(平板培养24h,生化鉴定19 h)。MALDI-TOF质谱鉴定检出28株肺炎克雷伯氏菌,鉴定率 为91.00%~99.90%;31 株 大 肠 埃 希 氏 菌,鉴 定 率 为94.60%~99.90%。从单菌落靶板制备至检出耗时33min(前处理及质控菌株校准需25min,所有菌株鉴定仅需8min),见表1、2。环境样品中VITEK-2和MALDI-TOF还检出了2株解鸟苷酸拉乌尔菌,2株粘质沙雷氏菌,2株产气肠杆菌,2株蜡样芽孢杆菌和1株凝结芽孢杆菌。VITEK-2无法鉴别区分蜡样芽孢杆菌与苏云金芽孢杆菌,且所有样品的鉴定率低于MALDI-TOF质谱的结果,样品菌株B07和B08经过伴胞晶体染色为阴性,则鉴定为蜡样芽孢杆菌。该结果与质谱分析相同,见表3。
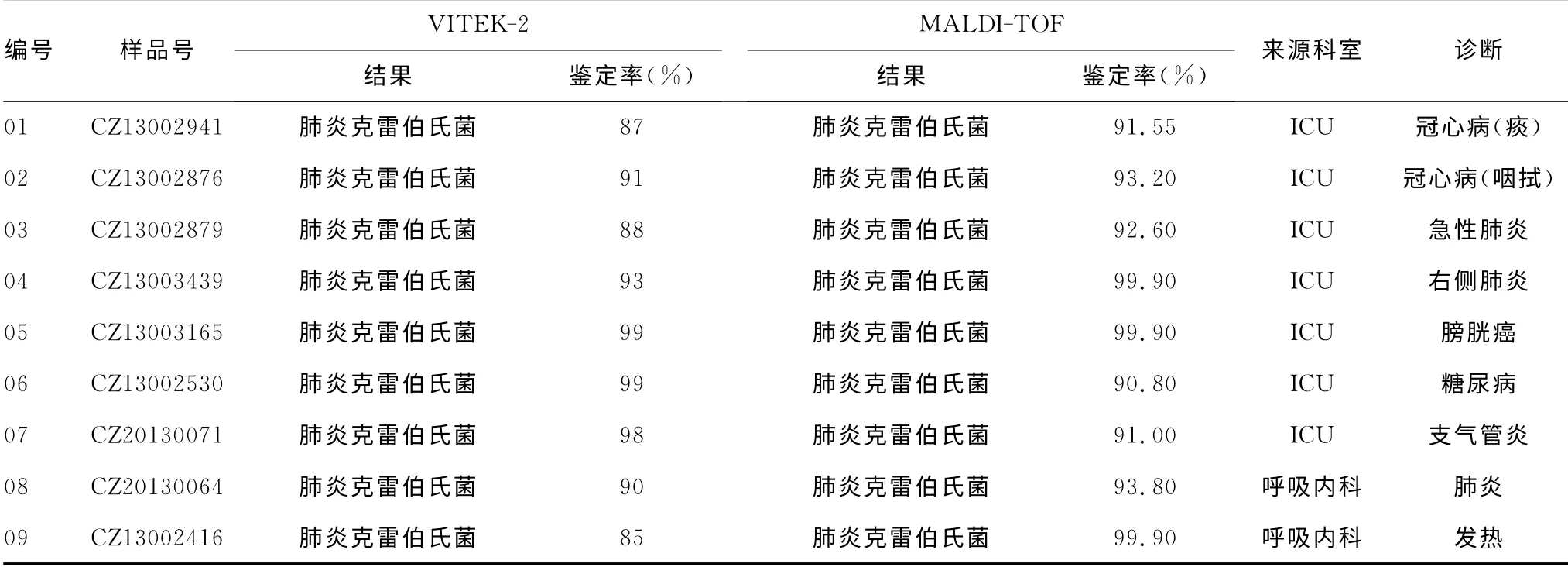
表1 肺炎克雷伯氏菌的VITEK-2和MALDI-TOF质谱检测结果

续表1 肺炎克雷伯氏菌的VITEK-2和MALDI-TOF质谱检测结果
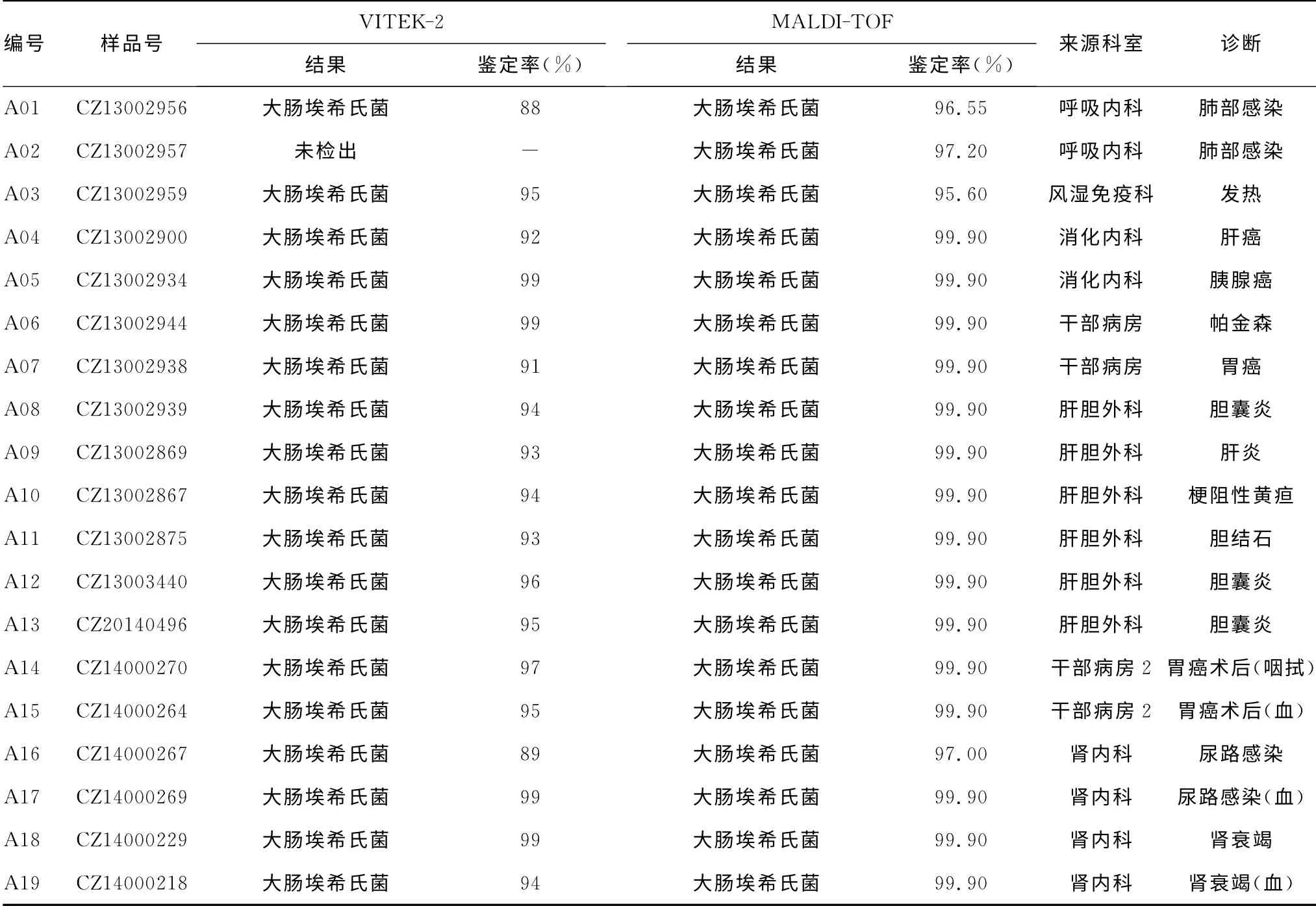
表2 大肠埃希氏菌的VITEK-2和MALDI-TOF质谱检测结果
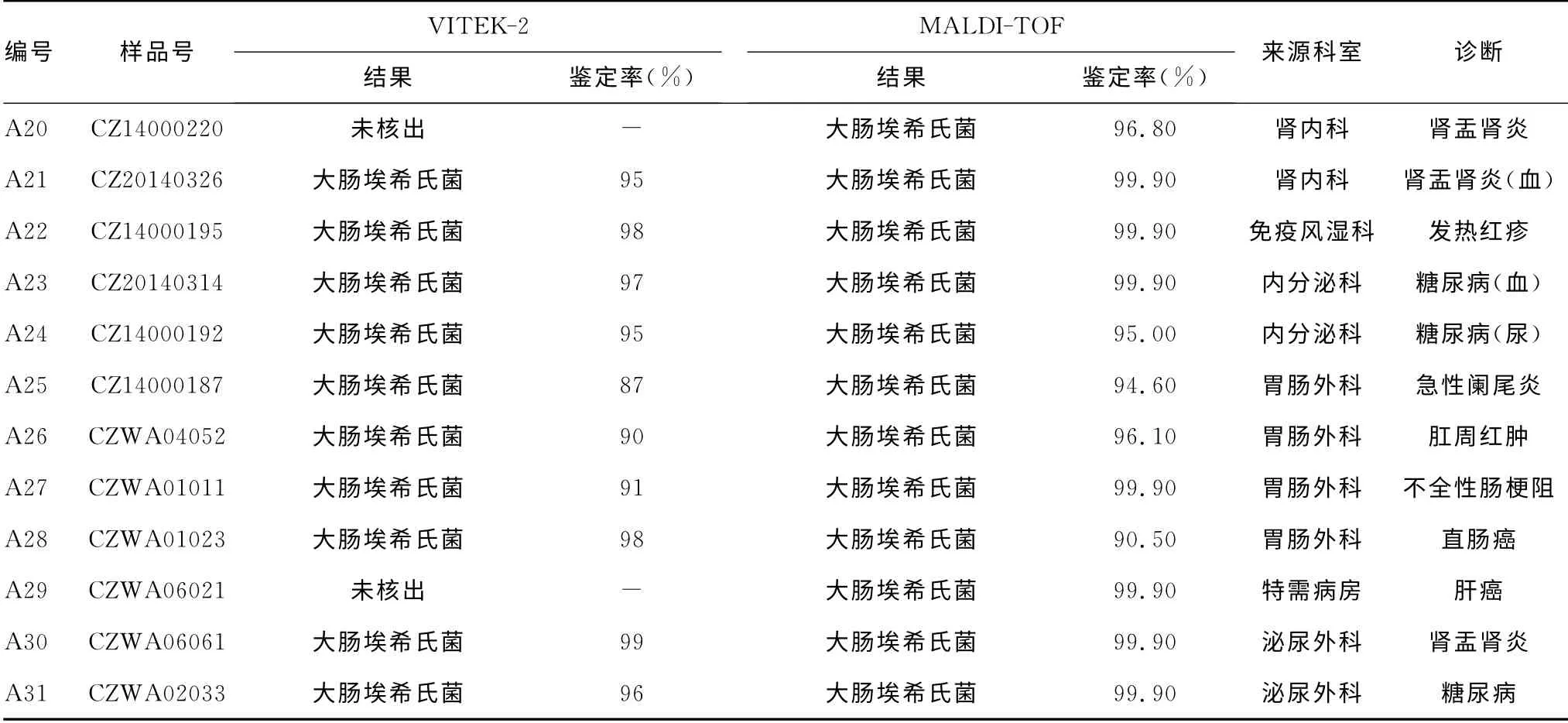
续表2 大肠埃希氏菌的VITEK-2和MALDI-TOF质谱检测结果

表3 其他菌株的VITEK-2和MALDI-TOF质谱检测结果

图1 2次质控菌株ATCC8739鉴定的特征峰图谱叠加图像
2.2 MALDI-TOF微生物鉴定的重复性与兼容性分析 数据统计共获得14对主要的特征峰,图中黑色箭头表示的是其中明显的6个特征峰。与数据库中多个大肠埃希氏菌超级图谱(Myla数据库超级图谱编号为:superspectrumEscherichiacoli38/0DSM10235 1/4/30/23/39/0DSM1576 1/31/10/18/20)比对,14个特征峰完全吻合,见图1。未知样品为大肠埃希氏菌,重复检测获得的MALDI-TOF图谱特征峰重叠,峰型完整平滑,噪音峰少,信噪比高。较高峰值的特征峰几乎没有差异,且未见缺失及其他杂峰。由此可见,MALDI-TOF 微生物鉴定方法的重复性极好,检测结果可信度很高,见图2。本鉴定方法通过检出峰与数据库中大量的超级图谱特征峰匹配率的分析,通过鉴定率百分比来确定检出结果的可信度,具有很高的兼容性。
2.3 院内感染致病菌同源性分析 通过图谱特征峰及共性峰的统计归纳,经过计算获取不同来源的致病菌样品其亲缘关系,以此反映院内感染致病菌传播的潜在方向及风险。图3和4所示为不同来源的肺炎克雷伯氏菌图谱亲缘性分析结果。结合表1数据,大部分肺炎克雷伯氏菌的样品同源性很高,样品1、2和25、26为同一患者不同样品检出菌,结果显示其峰形一致,而大部分来自ICU、呼吸内科等同科室院感样品菌株,其同源性都非常接近。而大肠埃希氏菌分析结果也有类似现象,其中1和2、14和15、16和17、18和19、20和21及23和24菌株来源于同一个患者,其图谱相同,属同源菌株。大部分亲缘性较近的菌株都来源于同一个科室。院内感染的发生和传播都以科室为中心呈现多点源传播的模式。

图2 10个平行组的未知菌株在不同初始电压下的叠加图谱
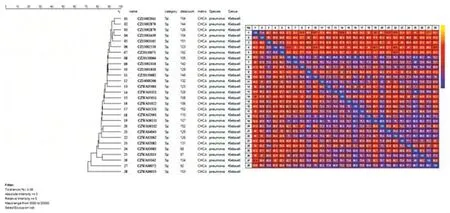
图3 肺炎克雷伯氏菌检出特征峰的同源性分析
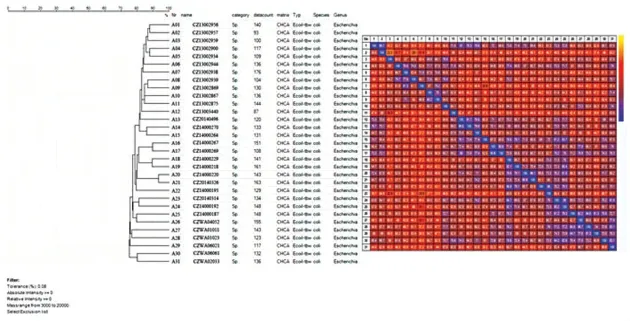
图4 大肠埃希氏菌检出特征峰的同源性分析
3 讨 论
传统的微生物鉴定方法通常以生化分析、菌落形态等表型鉴定,以及PCR 和测序等分子鉴定为手段[8-9],同步的还需进行致病菌毒力实验等辅助分析方法进行鉴定[10]。MALDITOF主要以微生物内的核糖体蛋白、DNA 结合蛋白及冷休克蛋白等作为检测目标蛋白[11],他们是管家基因编码的,相对稳定。由此MALDI-TOF质谱能检出一些生化反应不明显,抗原丢失及需要特殊染色辅助的致病菌。本文中VITEK-2未检出的3株大肠埃希氏菌,后续通过抗原修复和增菌,恢复了部分特征生化反应,经过再次分析仍只获得87~90%的鉴定率,且检测耗时3d,而MALDI-TOF质谱则展现出高效快速的特征在1h内全部检出待测菌。本实验通过与基于生化鉴定方法为基础的VITEK-2同步鉴定对比,证明MALDI-TOF微生物鉴定系统无论在鉴定时间、准确率及鉴定率都具有显著的优势。
岛津公司田中耕一在1988年首次利用MALDI-TOF 质谱鉴定生物大分子[12]。而同年Karas和Hillenkamp也成功利用MALDI-TOF技术建立了10 000 的分子鉴定技术[13]。过去的15年,通用化的MALDI-TOF质谱才逐步成为一种研究生物大分子的有效分析工具[14]。MALDI-TOF质谱的运行基本原理是样品在紫外吸收基质辅助下的光致电离散射。粒子散射经过飞行时间质谱分析获得待测物质信息[15]。MALDITOF运用于微生物鉴定则是基于基质处理后的细胞质蛋白质结晶的激光电离解析而建立的质谱分析技术,无论蛋白表达、细胞甚至组织学研究领域,MALDI-TOF已经成为一种新型的检测生物大分子的研究方法[16-18]。然而,大部分的MALDITOF质谱分析都是基于实验室应用而言,很少直接运用于临床研究[9,16-19]。本文基于RUO 分析系统及Myla微生物图谱数据库,尝试建立临床微生物快速分析的方法。为快速诊断急性和危重患者的院内感染及其他感染提供了理论基础。
此外,通过共性峰的同源分析,样品的亲缘关系还可以由质谱鉴定的方式展现出来,质谱分析获得的肽指纹谱能够通过峰值分析和计算获得样品的亲缘关系[7]。蛋白溯源法主要通过一些保守而稳定高表达的核糖体蛋白及DNA 结合蛋白为标签进行的,在快速检出致病菌的同时,还能通过聚类分析得出致病菌株的亲缘关系,为及时有效的判断院内感染提供了帮助。但是,每一种微生物都具有各自特征性的表达蛋白,完善特征蛋白的质谱特征峰的数据可能使得蛋白溯源的准确性大大提高。完善的微生物鉴定质谱系统的建立对于耐药监控和高致病性致病菌的检测工作至关重要,MALDI-TOF质谱在临床诊断运用有利于耐药监管工作及积极地预防突发公共卫生事件的发生。
[1] Nordmann P,Naas T,Poirel L.Global spread of Carbapenemaseproducing Enterobacteriaceae[J].Emerg Infect Dis,2011,17(10):1791-1798.
[2] Bonomo RA.New Delhi metallo-beta-lactamase and multidrug resistance:aglobal SOS[J].Clin Infect Dis,2011,52(5):485-487.
[3] Moellering RC.NDM-1-a cause for worldwide concern[J].N Eng J med,2010,363(30):2377-2379.
[4] Rolain JM,Parola P,Cornaglia G.New Delhi metallo-beta-lactamase(NDM-1):towards a new pandemia[J].Clin Microbiol Infect,2010,16(20):1699-1701.
[5] Salah Z,Gülay G,Stina B,et al.Identification of clinical Pasteurella isolates by MALDI-TOF-a comparison with VITEK 2and conventional microbiological methods[J].Diag Microbiol Infec Dis,2013,77(1):96-98.
[6] Haag AM,Taylor SN,Johnston KH,et al.Rapid identification and speciation of Haemophilus bacteria by matrixassisted laser desorption/ionization time-of-flight mass spectrometry[J].J Mass Spect,1998,33(8):750-756.
[7] Prasanna D,Mark A.Mass Spectrometry in the Clinical Microbiology Laboratory,Part II:MALDI-TOF MS[J].Clin Microbiol Newsl,2012,34:18.
[8] Hauser D,Popoff MR,Kiredjian M,et al.Polymerase chain reaction assay for diagnosis of potentially toxinogenic Coryne bacterium diphtheriae strains:correlation with ADP ribosylation activity assay[J].J Clin Microbiol,1993,31(10):2720-2723.
[9] Schuhegger R,Lindermayer M,Kugler R,et al.Detection of toxigenic Corynebacterium diphtheria and Corynebacterium ulcerans strains by a novel real-time PCR[J].J Clin Microbiol,2008,46(8):2822-2823.
[10] Engler KH,Glushkevich T,Mazurova IK,et al.A modified Elek test for detection of toxigenic corynebacteria in the diagnostic laboratory[J].J Clin Microbiol,1997,35(2):495-498.
[11] Liu P,Li P,Jiang X,et al.Complete genome sequence of Klebsiellapneumoniaesubsp.pneumoniae HS11286,a multidrug resistant strain isolated from human sputum[J].J Bacteriol,2012,194(7):1841-1842.
[12] Tanaka K,Waki H,Ido Y,et al.Protein and polymer analyses up to m/z 100,000by laser ionization time-of-flight mass spectrometry[J].Rapid Commun Mass Spectrom,1988,2(8):151-153.
[13] Karas M,Hillenkamp F.Laser desorption ionization of proteins with molecular masses exceeding 10,000daltons[J].Anal Chem,1988,60(30):2299-2301.
[14] Stults JT.Matrix-assisted laser desorption/ionization mass spectrometry[J].Curr Opin Structu Biol,1995,5(7):691-698.
[15] Zenobi R,Knochenmuss R.Ion formation in Maldi mass spectrometry[J].Mass Spectr Rev,1998,17(4):337-366.
[16] Van Veen SQ,Claas EC,Kuijper EJ.High-throughput identification of bacteria and yeast by matrix-assisted laser desorption ionization time of flight mass spectrometry in conventional medical microbiology laboratories[J].J Clin Microbiol,2010,48(3):900-907.
[17] Seng P,Drancourt M,Gouriet F,et al.Ongoing revolution in bacteriology:Routine identification of bacteria by matrix-asisted laser desorption ionization time-of-flight mass spectrometry[J].Clin Infect Dis,2009,49(4):543-551.
[18] Sauer S,Kliem M.Mass spectrometry tools for the classification and identification of bacteria[J].Nature Rev Microbiol,2010,8(1):74-82.
[19] Mellmann A,Cloud J,Maier T,et al.Evaluation of matrix-assisted laser desorption ionization-time-of-flight mass spectrometry in comparison to 16SrRNA gene sequencing for species identification of nonfermenting bacteria[J].J Clin Microbiol,2008,46(6):1946-1954.
