不同产区艾叶ISSR遗传多样性分析
2023-07-06余孟娟赵博宇董诚明
余孟娟,赵博宇,董诚明,李 询,邢 冰,李 曼
不同产区艾叶ISSR遗传多样性分析
余孟娟,赵博宇,董诚明*,李 询,邢 冰,李 曼
河南中医药大学药学院,郑州 河南 450046
对不同产区艾叶进行遗传多样性分析,为艾叶规范化种植及新品种选育工作奠定基础。采用ISSR分子标记技术对36个不同产区的艾叶样品进行扩增,然后利用POPGENEv1.32软件进行遗传学分析,得出它们的遗传特征系数,用Ntsyspc2.1对艾叶ISSR分子标记结果进行UPGMA聚类分析。筛选出16条引物,共扩增出条带402条,其中多态性条带398条,多态性百分比为99%;在对栽培种和野生种之间遗传距离分析后发现,两者之间的遗传一致度为0.989 1,遗传距离为0.011 0;对按纬度划分的4个区域艾资源进行遗传结构与遗传变异分析,4个区域间基因多样性指数(diversity index,t)为0.192 7、遗传多样性指数(genetic diversity index,s)为0.164 2、遗传变异指数(genetic variation index,st)为0.147 9、基因流(gene flow,m)为2.881 8,NTSYSpc软件对4个区域进行系统聚类分析,可将4个区位划分为2大类,且4区位相邻2个之间均具有一定的遗传相似性。艾叶具有极其丰富的遗传多样性,栽培种和野生种之间亲缘关系接近、遗传背景差异较小。艾叶其遗传特性随着纬度的变化呈现出一种递进式遗传扩散,其中河南南部居群多样性最为丰富,其次为河南中西部和河南中部,北部居群遗传多样性最低。
艾叶;ISSR分子标记;遗传多样性;栽培;野生;不同纬度
艾叶为菊科植物艾LevletVant的干燥叶,性温,味苦、辛,有温经止血、散寒止痛之功效。在临床上可分为外用和内服2类,其中外用以火灸著名,而内服用途甚广。我国艾分布范围较广,从东北到华南均有生长[1]。国内外对艾叶成分含量、药理药效及化学成分的提取均有比较深入的研究[2-3],但利用分子生物学手段对不同产地艾叶种质资源遗传多样性的探索却较少[4-5]。遗传多样性的本质是遗传物质变异,探索遗传多样性是研究植物进化和亲缘关系的基础[6],故将现代分子生物技术应用于艾规范化种植及辅助育种进而发掘优良资源或改良资源显得尤为重要[7]。王惠君等[5]在2015年首次将分子标记方法应用到艾叶的种质资源多样性研究领域,但其却未能收集到北艾的种质资源,分析其遗传多样性,在《国药提要》《中国药学大词典》以及国外的《泰西本草名疏》中均以北艾作为艾叶的正品基原,本实验所用样品包括北艾,填补了北艾种质资源多样性研究的空白。
简单重复序列间扩增(inter-simple sequence repeat,ISSR)是基于微卫星系列发展来的分子标记技术,具有快速高效、引物特异性强、多态性好等优点,目前被广泛应用于品种鉴定、物种分类、居群遗传学研究等领域,是研究植物遗传多样性常用的分子生物学方法[8-10]。因此本研究拟采用ISSR分子标记技术对我国传统产区的36份艾叶从分子水平分析其遗传多样性,旨在从DNA水平上探明艾叶资源遗传多样性现状;野生艾叶和栽培艾叶遗传多样性差距以及纬度与艾叶遗传多样性的关系,为今后艾的种质资源利用和品种选育等奠定基础[11-12]],因而具有非常重要的现实意义。
1 材料和仪器
1.1 材料
查阅《中国植物志》《中药志》《河南植物志》《河南志》等相关资料和文献,了解艾叶生境、分布、栽培等信息,以北纬38°20′40″到北纬33°03′6″划分采样区域,南起湖北蕲春,北至河北安国,基本覆盖了全国艾叶主要产地。经河南中医药大学董诚明教授鉴定为艾Levl.et Vant.,取样部位均为其幼嫩叶片,总计36份样品(表1),野生种和栽培种各有18种。所有材料在采样前均经过了去杂处理,以确保种质纯度。采集后封装于有干燥剂的自封袋内,放置入冰盒,并及时送至实验室用锡箔纸包裹浸入液氮,迅速冷冻后放入于−80 ℃冰箱保存备用。
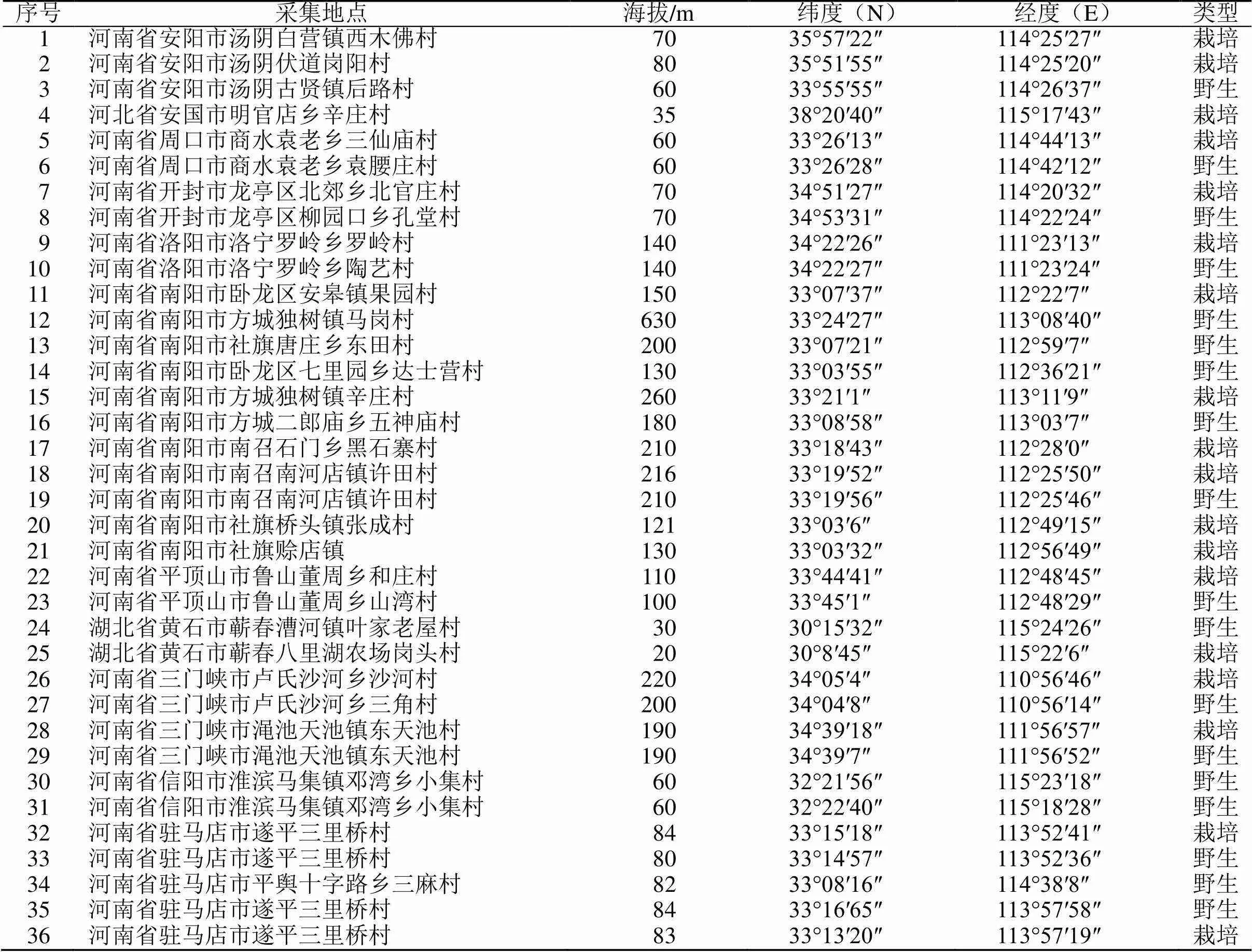
表1 样品生长环境信息
1.2 试剂与仪器
植物组织基因组DNA抽提试剂盒(生工生物公司,编号B518261)、SanPrep柱式DNA胶回收试剂盒(生工生物公司,编号B518131)、Taq Plus DNA 聚合酶(BBI,编号B600090)、灭菌水。
SW-CJ-1D型洁净工作台(江苏苏洁净化设备厂)、PCR-96型PCR反应扩增仪(BBI)、G508009型高速微量离心机(100~14 800 r/min,生工生物公司)、DYY-6C型电泳仪(北京六一公司)、FR980型凝胶成像系统(上海复日科技有限公司)。
2 方法
2.1 DNA提取与检测
采用试剂盒(生工生物,批号B518261)法提取样品叶片基因组DNA。
2.2 ISSR引物筛选及PCR反应体系确定
以提取的艾基因组DNA为模板,从哥伦比亚大学(UBC)公布的100条引物[13]中筛选出16条引物,均能扩增出清晰完整、多态性和稳定性均较好的条带。通过一系列实验优化,最终确定本实验的ISSR-PCR反应体系为(50 μL):10×PCR Buffer 5 µL,引物F 2 µL,引物R 2 µL,Dntp 2 µL,Taq Plus DNA Polymerase 0.5 µL,最后用灭菌水加至50 µL。扩増反应程序:95 ℃预变性5 min;95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸50 s,进行35个循环;72 ℃延伸8 min;4 ℃保存。
2.3 ISSR-PCR扩增及检测
将需要使用的反应体系取出,对所有样品基因组DNA扩增,将扩增后的产物用2%的琼脂糖凝胶在150 V 100 mA的电泳条件下电泳20 min,置于全自动凝胶成像分析系统上成像,调节至最佳光圈及亮度后拍照保存。
2.4 数据处理与分析
将实验中所做出来的琼脂糖凝胶胶板拍照,对胶板中的电泳条数进行判读。统计原则以琼脂糖凝胶电泳上的每1个条带当成1个分子标记,其所代表的意义为1个基因位点。对于不同产地的艾叶DNA样品在琼脂糖凝胶电泳统计时按照有条带为1,无条带为0;相同相对分子质量中,以较为清晰的记为1不清晰的记为0,模糊条带记为0的原则,在EXCEL中记录成0、1矩阵形式。用Ntsyspc2.1对艾叶ISSR分子标记统计好的0、1矩阵数据做UPGMA聚类分析。用POPGENEv1.32软件对艾叶ISSR分子标记统计好的0、1矩阵数据进行遗传学分析,得出它们的遗传特征系数。
3 结果与分析
3.1 引物扩增结果分析
以16条引物对36个不同产地的艾叶资源进行ISSR-PCR扩增(表2),共扩增出402条清晰的条带,其中多态性条带有398条,多态性百分比为99%。16条引物扩增出的有效条带数范围为17~31条,均值为24.875条。引物UBC840扩增出的条带最多,为33条,引物UBC822扩增条带最少,为17条(图1~3)。ISSR扩增结果表明艾种质资源具有较为丰富的遗传多样性。
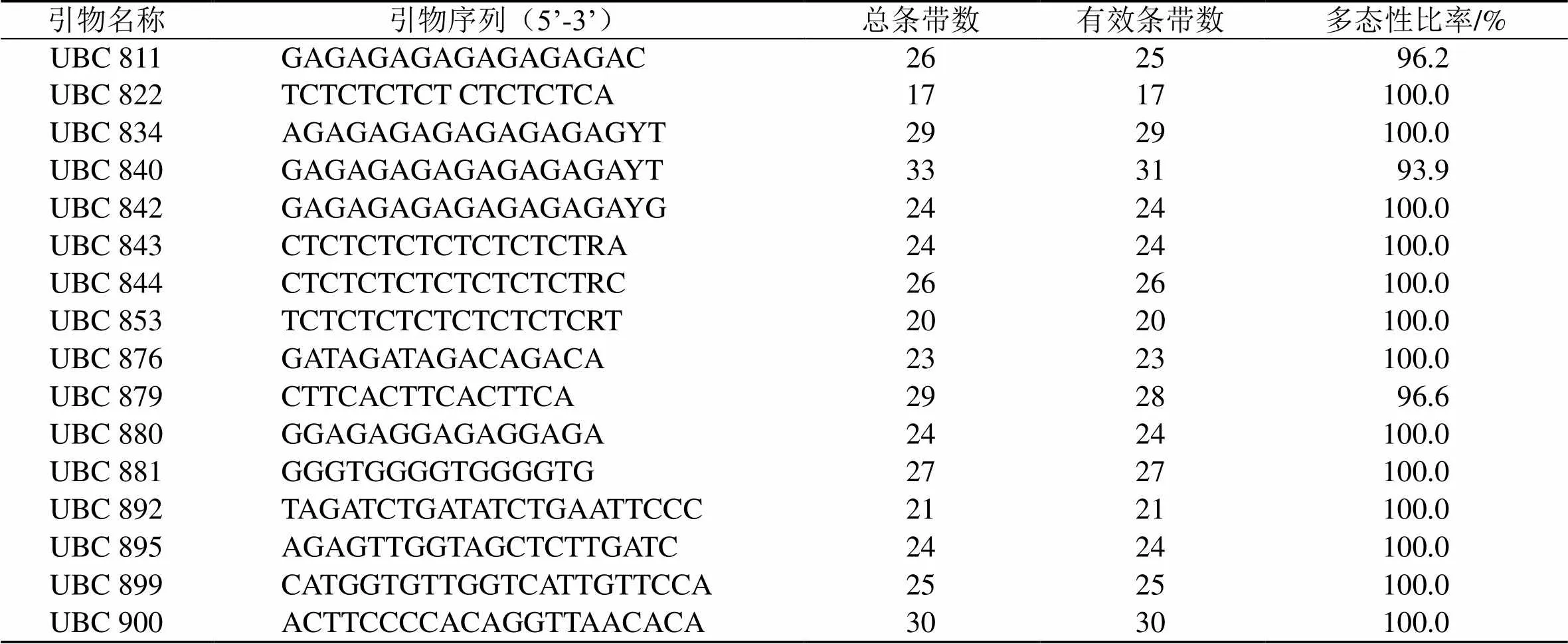
表2 ISSR引物扩增结果

图1 引物892琼脂糖凝胶扩增结果

图2 引物822琼脂糖凝胶扩增结果

图3 引物834琼脂糖凝胶扩增结果
3.2 不同产区艾野生种质与栽培种质遗传多样性分析
对ISSR引物电泳结果进行统计后,通过POPGENEv1.32软件对艾种质资源栽培种与野生种的遗传背景进行分析,从2个资源的遗传多样性指数来看,遗传多样性指数栽培艾要略高于野生品,但遗传多样性差距并不显著。等位基因数(number of alleles,a)、有效等位基因数(effective number of allele,e)、Nei’s 氏基因多样性指数(Nei’s gene diversity index,h)、Shannon’s 信息指数(Shannon's information index,)等指标结果见表3。对栽培种和野生种之间遗传距离分析后发现,两者之间的遗传一致度接近于1.000 0,为0.989 1,遗传距离较小,为0.011 0,表明两者亲缘关系接近,说明栽培种来自于野生种的驯化,且驯化时间并不很长,因此遗传背景差异较小[14]。对人工栽培艾与野生艾种质资源进行遗传结构与遗传变异分析可以发现,有2.77%的遗传分化存在于2个亚群间,在采样过程中能够观察到同一采集点的每株艾的表型均略有不同,生物的表观多样性主要由基因决定,基因差异明显则表观形态多样性会更加丰富,由于艾的人工驯化时间短,栽培品种并非严格意义上遗传背景高度纯合的品种,而是多为一些混合品种,故种内遗传分化程度高则使得在同一样点单株表观形态特性呈现多样性。栽培种与野生种之间基因流(gene flow,m)为17.520 8,由于艾的繁殖更多是通过无性繁殖途径实现,此处的基因流更多是反映野生种与栽培种两者之间遗传多样性趋同的一种关系,即两者遗传背景差距并不大[15-17]。
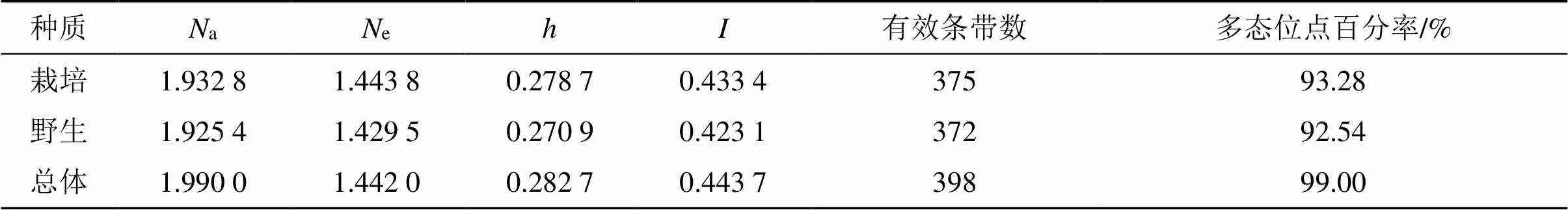
表3 人工栽培艾与野生艾种质资源遗传多样性
3.3 不同产区(不同纬度)艾遗传多样性分析
按照地理纬度,将本次采集的纬度差异相对较小的产地划分为4个大的区域(表4),从这些样本中共扩增出396条多态性较为丰富的条带,多态性位点占比98.51%,该扩增结果表明不同纬度间艾具有较好的遗传多样性。通过POPGENEv1.32软件对按表4方法划分的不同区域艾种质资源进行分析,结果如表5所示,从遗传多样性指数来看,4个区域的艾叶之间具有显著的遗传多样性。对4个区域资源进行遗传结构与遗传变异分析后发现,4个区域间艾种群基因多样性指数(gene diversity index,t)为0.192 7、遗传多样性指数(genetic diversity index,s)为0.164 2、遗传变异指数(genetic variation index,st)为0.147 9、m为2.881 8。由此表明在地理区划上4个居群间的艾品种具有一定的基因交流。在地理区划上4个居群间的艾叶具有一定的基因交流。对4个区域间遗传距离分析后发现(表6),它们之间的遗传一致度接近于1,遗传距离较小,接近于0,表明这4个居群间亲缘关系极近。再次证明了4个居群具有一定的联系。利用PopGen对4个区域进行聚类,由聚类图(图4)可知,中西部ZXB与南部NB亲缘关系更为接近,被聚成第1类。中部ZB次之,与第1类单独聚成第2类。北部BB遗传分化最强,单独与第2类聚为一类。按地理划分的聚类结果显示出不同区域间由南向北依次具有进阶相似性,即不同产区艾在纬度从南向北相互两两具有联系,这表明各地区艾的亲缘关系受自身生长的纬度和相邻地域间的距离影响。

表4 不同产区不同纬度区划

表5 不同产区区划艾种质资源遗传变异结果
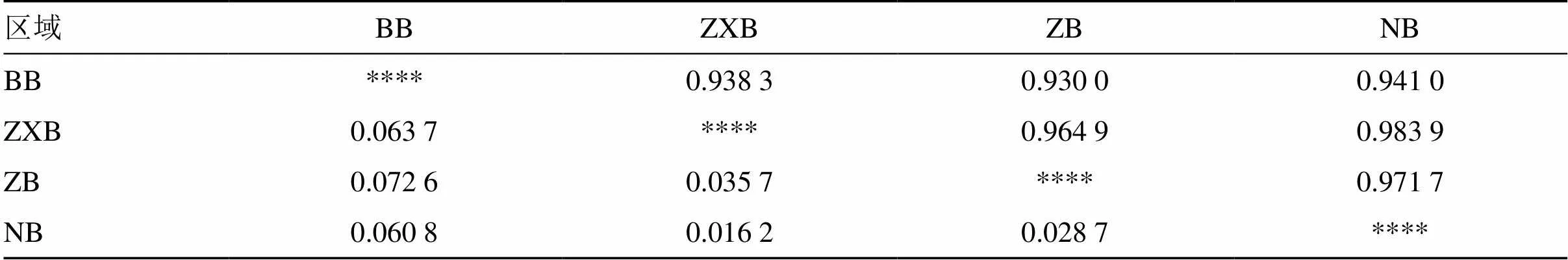
表6 不同纬度区域遗传距离相似度
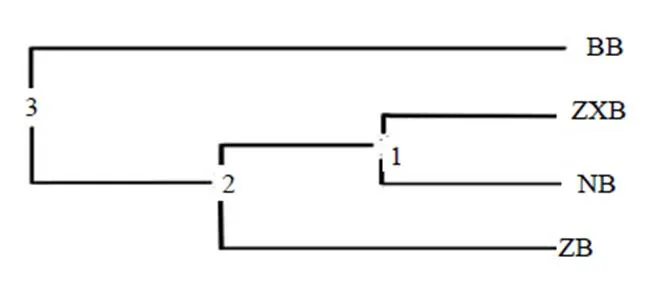
图4 不同纬度区域聚类结果
4 讨论
以筛选出的16条引物对36个不同产地的艾叶资源进行扩增,多态性百分比为99%。有效条带数范围为17~31条,均值为24.875条。扩增结果表明艾种质资源具有较为丰富的遗传多样性。在采集样品的过程中也能够看出田间艾草临株之间的差异性,表明即便在同一地点生长的不同植株间也存在一定的差异[18-19]。植物遗传多样性的成因较为复杂,植物在不同环境下与不同异栽培物之间存在协同共存,通过虫媒等繁殖途径,与其他异栽培物进行接触,导致其出现了较高的遗传多样性。
从栽培种与野生种2个类型来看,其两种生存状态下的艾均具有丰富的遗传多样性。两者之间的遗传一致度接近于1,遗传距离也很小,趋近于0,这表明人类对于艾的驯化历史相对较短,人工栽培种和野生种之间尚未分化完全。由于艾的繁殖更多是通过无性繁殖途径实现,因此栽培种与野生种之间存在的m为17.520 8时更多是反映野生种与栽培种两者之间遗传多样性趋同的一种关系。即两者遗传背景差距并不大,亲缘关系接近,有利于植物间育种。本研究在将纬度差异相对较小的采集地划分为的4个大的区域时,发现其遗传多样性整体呈现出一种递进式的遗传扩散。由聚类结果表明,相邻区域间艾叶两两联系,说明艾叶遗传距离与纬度有关,从侧面也能反映出不同种质艾叶间基因交流程度较大。其中,南部居群多样性最为丰富,中西部和中部分别居于第2和第3位置,北部居群遗传多样性在4个居群中最低。根据艾叶4个居群的遗传分析可以推断出,艾的分布是由南部向中部、北部依次扩散的。性状表型是基因型和环境间互做的结果,河南中西部属于伏牛山脉分布区域,其气候多样性强,生态类型复杂多变,由此为不同产区艾的生存和变异提供了物质基础,而北部居群所在区域受北部大陆季风影响较强,气候类型较为单一,这也是其遗传类型较少的主要原因[20-21]。
综上可得,艾叶的遗传多样性与地理距离具有一定的相关性,种质资源遗传多样性是育种的重要内容之一,种质资源遗传多样性越丰富越有利于新品种选育,种质亲缘关系越近越有利于植物间育种。本研究通过分子标记技术为艾叶规范化种植及新品种选育工作奠定了一定的基础。
利益冲突 所有作者均声明不存在利益冲突
[1] 宋川. 艾叶生药及化学成分的研究 [D]. 昆明: 云南中医学院, 2013.
[2]田璐. 艾叶化学成分分析及其抗炎功效研究 [D]. 广州: 暨南大学, 2017.
[3]于晓. 艾叶化学成分及一测多评分析方法的研究 [D]. 济南: 山东中医药大学, 2016.
[4]焦倩. 艾叶和野艾叶指纹图谱及多成分含量测定研究 [D]. 石家庄: 河北医科大学, 2018.
[5]王惠君, 卢诚, 黎明, 等. 传统中药艾种质资源的ISSR遗传多样性分析 [J]. 贵州农业科学, 2015, 43(8): 1-5.
[6]李冰敏. 分子标记在园林植物育种中的应用 [J]. 现代园艺, 2019(15): 148-149.
[7]Nasim N, Sandeep I S, Sahoo A,. Population genetic structure and diversity analysis in economically important(Forssk.) Kuntze accessions employing ISSR and SSR markers [J]., 2020, 143: 111894.
[8]李璐, 董诚明, 朱畇昊, 等. 基于ISSR的连翘遗传多样性研究 [J]. 中草药, 2019, 50(15): 3673-3680.
[9]Shahabzadeh Z, Mohammadi R, Darvishzadeh R,. Genetic structure and diversity analysis of tall fescue populations by EST-SSR and ISSR markers [J]., 2020, 47(1): 655-669.
[10]姜武, 吴志刚, 陶正明, 等. 基于ISSR和SRAP标记的栀子种质遗传多样性研究 [J]. 中草药, 2019, 50(2): 510-516.
[11]Sudha G S, Ramesh P, Sekhar A C,. Genetic diversity analysis of selected Onion (L.) germplasm using specific RAPD and ISSR polymorphism markers [J]., 2019, 17: 110-118.
[12]俞文灏, 吴保锋, 刘勇波. 生境破碎化对动植物遗传多样性的影响研究进展 [J]. 应用与环境生物学报, 2019, 25(3): 743-749.
[13]周延清. DNA分子标记技术在植物研究中的应用 [M]. 北京: 化学工业出版社, 2005.
[14]梁文汇, 刘凯, 黄开顺, 等. 肉桂家系遗传背景的ISSR分析 [J]. 广西林业科学, 2016, 45(1): 35-39.
[15]赵琬玥, 冷秋思, 屈燕, 等. 全缘叶绿绒蒿() 的ISSR遗传多样性分析 [J]. 分子植物育种, 2019, 17(20): 6891-6899.
[16]邢婀娜, 徐诗涛, 任明迅. 海南特有毛花马铃苣苔小尺度局域种群的年龄结构与基因流 [J]. 热带生物学报, 2018, 9(1): 37-46.
[17]刁毅, 周丽娟. 地木耳遗传多样性的ISSR分析 [J]. 北方园艺, 2018(14): 143-147.
[18]宋凤. 基于SCoT分子标记的甘草属植物自然居群的遗传多样性与基因渐渗研究 [D]. 石河子: 石河子大学, 2017.
[19]赵博宇, 董诚明. 河南艾不同种质资源形态特征比较 [J]. 中医学报, 2019, 34(5): 1024-1029.
[20]彭国清, 唐绍清. 喀斯特耐阴灌木淡黄金花茶的精细空间遗传结构与基因流 [J]. 生态学报, 2017, 37(21): 7313-7323.
[21]吴蕊. 紫牡丹引种驯化及远缘杂交后代早期鉴定研究 [D]. 北京: 中国农业科学院, 2011.
ISSR analysis on genetic diversity offrom different habitats
YU Meng-juan, ZHAO Bo-yu, DONG Cheng-ming, LI Xun, XING Bing, LI Man
College of Pharmacy, Henan University of Traditional Chinese Medicine, Zhengzhou 450046, China
The genetic diversity analysis ofin different producing areas will lay the foundation for the standardized planting and selection of new varieties ofISSR molecular marker technology was used to amplify artemisia sylvestris samples from 36 different production areas, and then genetic analysis was performed using POPGENE v1.32 software to obtain their genetic characteristic coefficients. Ntsyspc2.1 was used to perform ISSR molecular marker results UPGMA cluster analysis.16 primers were selected, and a total of 402 bands were amplified, including 398 polymorphic bands, and the percentage of polymorphism was 99%.. After analyzing the genetic distance between the wild species and the wild species, it was found that the genetic identity between the two is 0.989 1 and the genetic distance is 0.011 0. The diversity index (t) is 0.192 7, the genetic diversity index (s) is 0.164 2, the genetic variation index (st) is 0.147 9, and the gene flow (m) is 2.881 8. NTSYSpc software performs a systematic cluster analysis on 4 regions. The 4 loci are divided into 2 categories, and there are certain genetic similarities between the two adjacent 4 loci.Artemisia oleifera is extremely rich in genetic diversity, the genetic relationship between cultivated and wild species is close, and the genetic background is small. The genetic characteristics ofexhibit a progressive genetic spread with changes in latitude. Among them, southern Henan has the most abundant population diversity, followed by central and western Henan and central Henan, and northern population has the lowest genetic diversity.
Artemisiae Argyi FoliumISSR molecular marker; genetic diversity; cultivated germplasm; wild germplasm; different latitudes
R286.12
A
0253 - 2670(2023)13 - 4306 - 06
10.7501/j.issn.0253-2670.2023.13.023
2023-02-06
国家自然科学基金项目(81603232)
余孟娟(1997—),女,硕士研究生,从事中药资源保护与开发研究。E-mail: 1600213809@qq.com
董诚明(1963—),E-mail: dcm663@sina.com
[责任编辑 时圣明]
