庆大霉素诱导粪肠球菌耐药及其对氯霉素协同敏感相关特性研究
2023-05-30孙瑶徐雯雅周翠吴庆陈栎江徐春泉叶建中周铁丽
孙瑶 徐雯雅 周翠 吴庆 陈栎江 徐春泉 叶建中 周铁丽
摘要:目的 通過研究庆大霉素诱导粪肠球菌耐药突变株对氯霉素协同敏感(collateral sensitivity, CS)的相关特性,为基于协同敏感治疗策略的临床应用提供依据。方法 采用庆大霉素通过梯度浓度平板对3株庆大霉素敏感的粪肠球菌临床分离株和1株粪肠球菌标准菌株ATCC29212进行体外实验诱导耐药(experimental evolution);通过微量肉汤稀释法测定庆大霉素诱导后耐药突变株对常见抗菌药物的最低抑菌浓度(minimum inhibitory concentrations, MICs),分析抗菌药物间交叉敏感性;PCR检测诱导耐药菌株氨基糖苷类修饰酶编码基因携带情况;通过琼脂平板稀释法测定庆大霉素诱导耐药(evolved gentamicin-resistant)菌株和亲本菌株对其协同敏感药物(氯霉素)的防耐药突变浓度(mutant prevention concentration, MPC),最后,测定氯霉素对庆大霉素诱导耐药菌株和亲本菌株的时间杀菌曲线。结果 结果显示4株粪肠球菌亲本菌株经过庆大霉素诱导后,对庆大霉素MICs升高了16~
64倍,并对阿米卡星表现出交叉耐药;诱导耐药菌株及其亲本菌株均未检出aac(6')-Ie-aph(2")-Ia。此外,研究发现诱导后菌株对氯霉素表现出协同敏感。氯霉素对庆大霉素诱导耐药粪肠球菌及其亲本菌株的MPC均大于1024 μg/mL,突变选择窗较宽;时间杀菌曲线显示当氯霉素浓度为亲本菌株的1/2×MIC或1/4×MIC时,庆大霉素诱导耐药菌株的生长被有效抑制,而亲本菌株则不能被抑制。结论 基于协同敏感性制定抗菌药物治疗策略可能有助于有效清除耐药病原菌并延长抗菌药物的使用寿命。
关键词:粪肠球菌;协同敏感;庆大霉素;氯霉素;诱导耐药
中图分类号:R978.1 文献标志码:A
Study on the characteristics of collateral sensitivity to chloramphenicol
of evolved gentamicin-resistant Enterococcus faecalis
Sun Yao, Xu Wen-ya, Zhou Cui, Wu Qing, Chen Li-jiang, Xu Chun-quan, Ye Jian-zhong, and Zhou Tie-li
(Laboratory Medicine Center, the First Affiliated Hospital of Wenzhou Medical University, Wenzhou 325000)
Abstract Objective To study the characteristics of collateral sensitivity to chloramphenicol of evolved gentamicin-resistant Enterococcus faecalis, and to provide foundation for clinical application of treatment strategies based on collateral sensitivity (CS). Methods Three clinically isolated antimicrobial susceptible Enterococcus faecalis and E. faecalis standard strain ATCC29212 were used to adaptation laboratory evolution in vitro with gentamicin gradient concentration plates. The minimum inhibitory concentrations (MICs) of evolved gentamicin -resistant strains were determined by the microbroth dilution method, and the cross susceptibilities between antimicrobials were analyzed. The prevalence of aminoglycoside modifying enzyme encoding gene was detected by PCR in the evolved resistant strains. The agar plate dilution method was performed to determine the mutant prevention concentration (MPC) of evolved gentamicin-resistant strains and parental strains to the collateral sensitivity drug (chloramphenicol). Finally, the time-kill curve of chloramphenicol on the strains before and after evolution of gentamicin was determined. Results The results showed that the MICs of four strains of E. faecalis were increased 16~64 fold after evolution to gentamicin, and they showed cross-resistance to Amikacin. aac(6')-Ie-aph(2")-Ia had not been detected in all evolved resistant strains and their parental strains. Interestingly, the evolved resistant strains exhibited collateral sensitivity to chloramphenicol. The MPC of chloramphenicol in E. faecalis before and after evolution to gentamicin was higer than 1,024 μg/mL, indicating a wide mutant selection window (MSW). The time-kill curve showed that when the concentration of chloramphenicol was 1/2×MIC or 1/4×MIC of the ancestral strain, the evolved resistant strains were effectively inhibited, while the parental strains could not be effectively inhibited. Conclusion Antimicrobials therapy strategies based on collateral sensitivity possibly help to effectively eliminate drug-resistant pathogens and prolong the lifespan of antimicrobial drugs.
Key words Enterococcus faecalis; Collateral sensitivity; Gentamicin; Chloramphenicol; Evolved resistance
抗菌药物在人类医疗、畜牧业以及农业上的广泛使用甚至滥用,使得细菌几乎对所有抗菌药物均出现耐药并迅速播散[1]。新型抗菌药物的研发难度大、周期长[2],人們迫切需要寻找新的对策来对抗或者延缓耐药性的产生。近年来研究不断强调基于进化的策略(evolution-based strategies)在优化和延长现有药物疗效方面的前景,包括药物循环使用、联合用药、抗菌药物管理等[3]。近期多项研究表明协同敏感(collateral sensitivity,CS)可能成为一种有效的手段延缓或者逆转抗菌药物的耐药性[4]。
交叉敏感性(cross susceptibility)包括交叉耐药(cross resistance)及协同敏感,指耐药突变导致细菌耐药的同时对另外一种抗菌药物敏感性降低或者升高。近年来,研究人员发现[5]在细菌及癌症中均有多种药物显示出协同敏感现象。不断有研究发现[6]基于协同敏感现象制定交替或者联合用药策略能够一定程度上延缓细菌耐药的发生。然而,协同敏感发生机制及其发生规律尚不明确。部分研究显示[7]协同敏感谱表现出高度异质性和非重复的特性,使得利用这一机制制定用药策略复杂化。我们前期研究[8]采用5类8种抗菌药物对粪肠球菌进行体外诱导耐药,结果显示交叉耐药及协同敏感现象广泛存在。
目前,协同敏感相关研究主要集中在最低抑菌浓度(minimum inhibitory concentration,MIC)水平,仅少数研究[9]开始着眼于阐释防耐药突变浓度(mutant prevention concentration,MPC)水平对细菌的作用。MPC指防止特定大小菌群中一步耐药突变菌株选择性富集的最低抗菌药物浓度。MIC至MPC之间的浓度范围称为突变选择窗(mutant selection window,MSW),当抗菌药物浓度落在MSW中时能够选择性富集一步耐药突变株[10]。有研究表明[13]交叉敏感性的发生能够引起MSW的改变。协同敏感菌株可能能够缩小MSW,从而防止耐药突变株被选择性富集并有效清除耐药细菌。
目前,国内外对于协同敏感现象及其临床用药策略的研究仍较少,并且主要集中于大肠埃希菌、金黄色葡萄球菌等[9],罕有肠球菌相关研究。肠球菌作为一种临床常见条件致病菌,可引起人类尿路感染、血流感染等多种感染性疾病,是最重要的医院获得性感染病原菌之一。因此,本部分研究内容针对庆大霉素诱导耐药引起氯霉素敏感性升高的粪肠球菌菌株进行MPC测定和时间杀菌实验,了解协同敏感菌株在各抗菌药物浓度水平下的清除效率,为临床制定有效清除感染耐药病原菌的合理方案提供理论依据。
1 材料与方法
1.1 菌株来源
实验菌株为3株对庆大霉素敏感的粪肠球菌临床分离株(菌株编号分别为FC254、FC446和FC490,分别分离自创面、胆汁及脓液标本)、24株庆大霉素高水平耐药的粪肠球菌临床分离株和1株粪肠球菌标准菌株ATCC 29212,临床菌株为分离自温州医科大学附属第一医院的非重复标本,由法国梅里埃MALDI-TOF MS质谱仪和VITEK?2 Compact型全自动微生物分析仪进行菌种鉴定和抗菌药物敏感性初步测定。标准菌株粪肠球菌ATCC29212购自卫生部临检中心。
1.2 线性梯度平板法体外诱导耐药
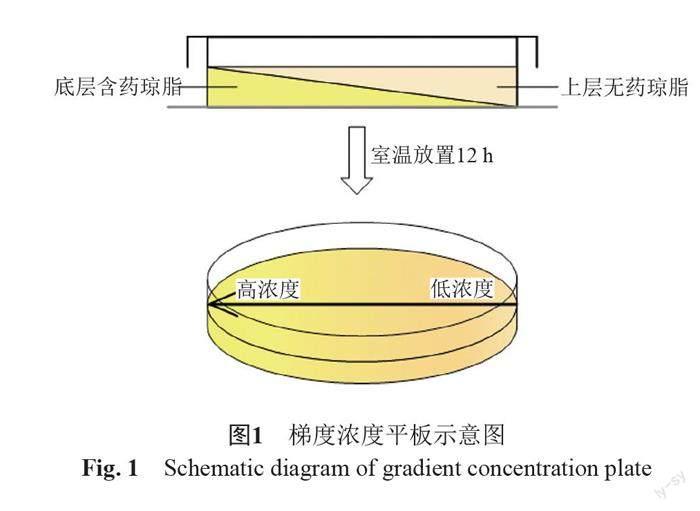
线性梯度平板的制备参考文献[8]。将加入抗菌药物的15 mL LB琼脂培养基(例如终浓度为1×MIC)倾注于90 mm培养皿中,将平皿倾斜放置;琼脂凝固后使培养皿水平放置,加入另外不含药的15 mL LB琼脂培养基,放置扩散12 h,所得梯度平板浓度即0~1×MIC,如图1。
将过夜培养的4株待测菌株以无菌生理盐水调成1.5×108 CFU/mL菌悬液;取100 μL上述菌液均匀涂布于琼脂平板表面(起始梯度平板浓度:0~2×MIC),每4 d传代1次;4 d后挑取高浓度端生长菌落保种,新鲜梯度平板上;当包括最高浓度处全板均有细菌生长后,提高最高抗菌药物浓度为原浓度5倍进一步诱导;传代10代,共40 d,同时将亲本菌株在不含抗菌药物LB琼脂平板上传代,作为空白平行对照。每株待测菌株分别平行诱导3系(lineage)。分别取第3次、第6次和第9次传代菌株进行药物敏感性的监测;庆大霉素体外诱导耐药菌株在不含抗菌药物平板上连续传代10代,观察其药物敏感性改变是否稳定存在。
1.3 抗菌药物敏感性试验
采用微量肉汤稀释法测定诱导耐药菌株及其亲本菌株对包括庆大霉素在内的13种抗菌药物的抗菌药物敏感性,即庆大霉素、阿米卡星、青霉素、氨苄西林、亚胺培南、环丙沙星、左氧氟沙星、加替沙星、万古霉素、氯霉素、红霉素、四环素和呋喃妥因;确认庆大霉素高水平耐药粪肠球菌临床分离株对庆大霉素的MICs值。抗菌药物购自温州市康泰生物科技有限公司。操作规范及折点判读参照美国临床和实验室标准协会(Clinical and Laboratory Standards Institute,CLSI)标准2021版。采用粪肠球菌质控菌株ATCC 29212进行质控。
1.4 氨基糖苷类修饰酶编码基因PCR检测
采用Biospin细菌基因组DNA提取试剂盒提取庆大霉素诱导耐药菌株及其亲本菌株、24株庆大霉素高水平耐药粪肠球菌临床分离株的基因组DNA。PCR扩增氨基糖苷类修饰酶aminoglycoside modifying enzymes, AMEs)编码基因aac(6')-Ie-aph(2")-Ia。引物序列为F:CAGAGCCTTGGGAAGATGAAG;R:CCTCGTGTAATTCATGTTCTGGC,产物大小为
348 bp。PCR产物经1%琼脂糖凝胶电泳,Gel-Red染色后凝胶成像观察结果。
1.5 防耐药突变浓度的测定
根据参考文献[11]中琼脂平板稀释法检测庆大霉素诱导耐药粪肠球菌和亲本菌株对氯霉素的防耐药突变浓度。以诱导耐药粪肠球菌对氯霉素的MIC为基准,采用倍比稀释方法进行含抗菌药物的平板配制,药物浓度自 MIC到1024 μg/mL;将待测菌株37℃培养18~24 h,挑取单个纯菌落于20 mL MH 肉汤培养基中震荡培养18~24 h,3000 r/min离心
10 min 后将沉淀重悬并加入到200 mL MH肉汤培养基中,200 r/min震荡培养6 h,再次以3000 r/min离心10 min,取沉淀调节菌液浓度为3.0×1010 CFU/mL;吸取100 μL上述菌液均匀涂布于每个含有倍比稀释抗菌药物的胰酶大豆琼脂平板上,每个浓度接种4个平板,并以菌落计数方法计数确定每个平板细菌总接种量大于1.0×1010 CFU,将平板置于37℃孵育;于 24、48和72 h时分别观察菌落生长情况,将72 h无菌落生长的最低药物浓度称为暂定MPC(MPCpr);以MPCpr为基准,按抗菌药物浓度的10%依次递减配制各浓度含药平板,重复上述MPCpr测定时操作,将72 h无菌落生长的最低药物浓度称为MPC。选择指数(Selection index, SI)=MPC/MIC。
1.6 时间-杀菌实验
将新鲜培养的待测纯菌株以无菌生理盐水调节菌悬液浓度并加入MH肉汤培养基中,使初始接种细菌浓度约为105~106 CFU/mL;向上述体系中加入抗菌药物,使抗菌药物终浓度为1/4×MIC、1/2×MIC、1×MIC、4×MIC 和16×MIC,并设置不含抗菌药物的空白生长对照,置37℃培养;分别于0、2、4、6、8、10、12 和24 h对培养液进行菌落计数;以每毫升细菌数(CFU/mL)为纵坐标,以时间(h)为横坐标,取3次重复实验结果绘制各诱导耐药菌株的时间-杀菌曲线。
2 结果
2.1 体外诱导粪肠球菌对庆大霉素药物敏感性情况分析
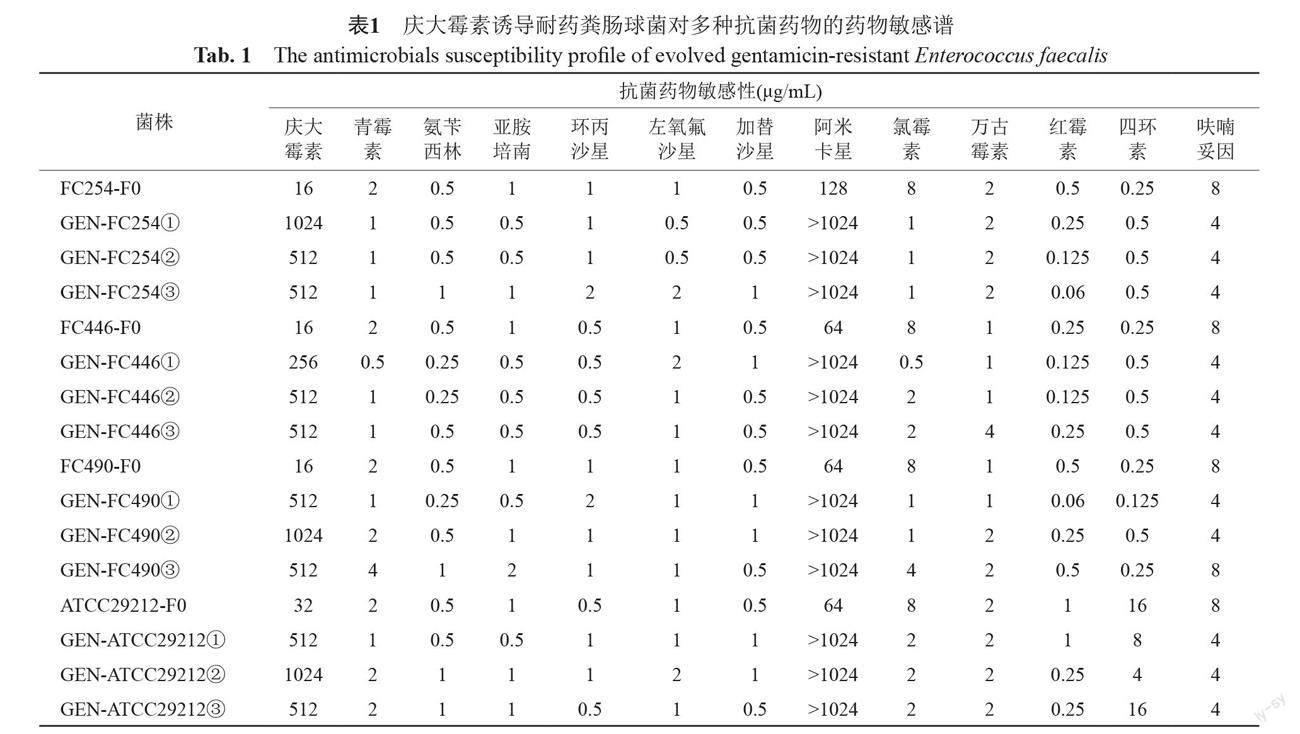
4株亲本菌株以庆大霉素进行诱导,每株菌设置3组平行,共获得12系诱导耐药菌株(GEN-FC254①、GEN-FC254②、GEN-FC254③、GEN-FC446①、GEN-FC446②、GEN-FC446③、GEN-FC490①、GEN-FC490②、GEN-FC490③、GEN-ATCC29212①、GEN-ATCC29212②和GEN-ATCC29212③)。相比诱导前亲本菌株,12系诱导耐药突变株对庆大霉素MICs提高了16~64倍;不含抗菌药物平行传代获得的菌株MICs未见明显改变。3组平行重复试验诱导获得的耐药株间相比,MICs升高倍数无明显差异。12系诱导耐药菌株在不含抗菌药物的平板上连续传代10代后耐药性均呈稳定存在。
2.2 诱导耐药菌株对其他各种药物交叉敏感性情况分析
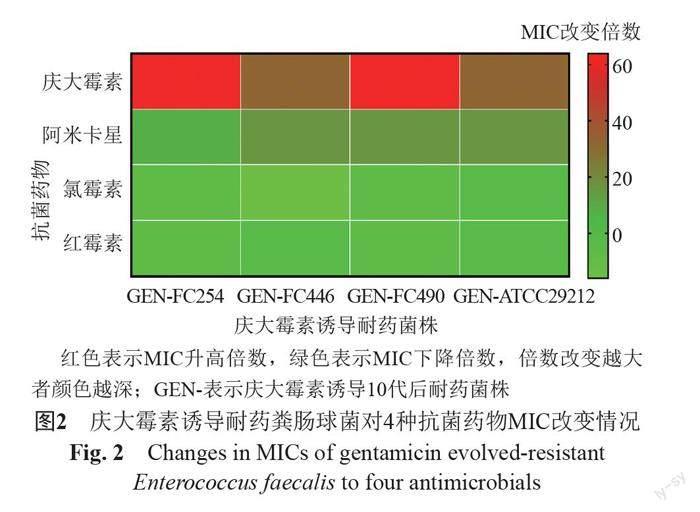
药敏结果显示庆大霉素诱导耐药菌株对3种抗菌药物敏感性发生明显改变,即12系诱导耐药菌株均对阿米卡星MICs升高大于8倍(诱导前4株粪肠球菌对阿米卡星MIC为64~128 μg/mL,庆大霉素诱导菌株均>1024 μg/mL),对氯霉素和红霉素表现为不同程度的协同敏感性(MIC下降倍数≥4倍),其中以氯霉素较为显著,MIC下降4~16倍,见图2。3组平行诱导获得的耐药株间的药物敏感谱无明显差异,临床菌株和标准菌株相比,诱导耐药菌株药物敏感谱同样无明显差异,具体见表1。提示经同种抗菌药物诱导耐药后,细菌对抗菌药物敏感性的改变可能具有一定的规律性和可预测性。
2.3 氨基糖苷类修饰酶基因携带情况
PCR检测结果显示24株慶大霉素高水平耐药粪肠球菌临床分离株(MICs≥512 μg/mL)均携带aac(6')-Ie-aph(2")-Ia,而12株庆大霉素诱导耐药菌株及其4株亲本菌株均不携带该基因。
2.4 防耐药突变浓度的测定
氯霉素对4株粪肠球菌亲本菌株和12株庆大霉素诱导耐药粪肠球菌的MPC均大于1024 μg/mL,远高于MIC值,选择指数均SI>512,防耐药突变选择窗MSW较宽。
2.5 时间杀菌实验分析
时间杀菌实验结果显示抗菌药物浓度为亲本菌株氯霉素MICs的1/2×MIC或1/4×MIC时,诱导耐药菌株可以被有效抑制,而亲本菌株则不能被有效抑制生长(图3),1/4×MIC抗菌药物浓度时,诱导耐药菌株仅在12 h后生长曲线开始升高;药物浓度为1×MIC时,诱导耐药菌株生长被抑制,而亲本菌株仍能缓慢生长;当抗菌药物水平为亲本菌株氯霉素MICs的4倍及16倍时,诱导耐药前后的菌株均被完全抑制生长,两者时间杀菌曲线无明显区别,见图3。
3 讨论
数十年来,多重耐药病原菌的快速出现和流行给公共卫生安全造成严重威胁。基于协同敏感现象制定抗菌药物使用策略能够有效减低耐药的发生率甚至逆转已经存在的耐药性[8]。在本研究中通过庆大霉素对粪肠球菌进行体外诱导耐药实验,研究结果显示交叉耐药及协同敏感广泛存在,相同作用机制的抗菌药物之间存在交叉耐药,而协同敏感发生于作用机制不同的抗菌药物之间。然而,有研究显示交叉耐药不仅能发生于同种类抗菌药物间,也可以发生于不同种类药物之间。例如,Lázár等[13]研究发现拓扑异构酶基因gyrA突变的喹诺酮类耐药的大肠埃希菌,能够引起氨苄西林、头孢西丁及妥布霉素等药物的耐药性升高,以及对呋喃妥因和多西环素敏感性的升高。协同敏感普遍存在,但在不同种细菌甚至同种细菌中也存在异质性,可能与细菌不同的基因背景、适合度代价以及可移动元件等因素有关[9]。研究显示大肠埃希菌经氨基糖苷类药物诱导耐药后,对四环素和氯霉素发生协同敏感,可能与质子泵动力势(proton-motive force,PMF)有关[14]。本研究中粪肠球菌对氯霉素协同敏感的机制尚待进一步阐明。
臨床病原菌对氨基糖苷类耐药可以由多种耐药机制引起,其中以氨基糖苷类修饰酶灭活抗菌药物分子最为常见,即乙酰基转移酶(aminoglycoside acetyltransferases,AACs)、腺苷酸/核苷酸转移酶(adenylyltransferases or nucleotidyltransferases,ANTs)和磷酸转移酶(phosphotransferases,APHs),其中以双功能酶aac(6')-Ie-aph(2")-Ia最为常见[15]。本研究中临床分离庆大霉素高水平耐药粪肠球菌中均检出aac(6')-Ie-aph(2")-Ia基因,诱导耐药菌株及其亲本菌株均未携带该基因,提示诱导耐药菌株可能是由其他耐药机制引起对庆大霉素耐药,如30S核糖体蛋白编码基因突变[3]、外排泵表达量升高[14]等。研究提示与可移动基因元件携带AMEs引起临床菌株对庆大霉素耐药不同,长期用药引起的庆大霉素耐药演变可能由其他多种机制介导,并引起对其他抗菌药物协同敏感,为临床选用发生协同敏感的药物进行交替治疗提供指导。
临床上治疗感染性疾病时常采用高于MIC的抗菌药物浓度来清除致病菌,然而治疗过程中当抗菌药物浓度介于MIC之上而低于MPC,即落在MSW内时,耐药突变株菌株则能够被选择性富集[16],因此,临床用药时应使感染部位抗菌药物浓度保持在MPC之上。有研究发现体外诱导耐药菌株存在协同敏感现象,协同敏感菌株在MIC降低的同时MPC也相应减低[17],较低的抗菌药物浓度则达到防耐药突变浓度,从而在防止耐药突变株不被筛选富集的同时保证临床用药的安全性。然而,本研究中,庆大霉素诱导耐药菌株相比亲本菌株,氯霉素MIC降低到1/4~1/16,但诱导前后菌株MPC均达到1024 μg/mL以上,远高于氯霉素的最高血药浓度(Cmax=26 μg/mL)及安全用药范围[18],防耐药突变选择窗MSW较宽,本研究中氯霉素MIC降低没有能够显著减小其MSW,提示该理论可能并不适用于采用氯霉素治疗粪肠球菌感染时耐药突变株的清除。相同药物对不同菌种的选择指数SI不同,如氯霉素对大肠埃希菌的选择指数SI为6.3,而对金黄色葡萄球菌的SI则为5.7[19]。本研究中氯霉素对粪肠球菌的选择指数SI达512以上。因此,系统、全面地测定临床常见抗菌药物对不同病原菌的协同敏感及MPC值是临床应用的前提。
有研究报道诱导耐药菌株对其他抗菌药物产生协同敏感现象时,在野生菌株MIC的16倍抗菌药物浓度作用下,协同敏感菌株相比野生菌株能够更快速地被有效清除[17]。本研究中时间杀伤实验结果显示在高于亲本菌株MIC的抗菌药物水平下,即4×MIC和16×MIC时,庆大霉素诱导耐药前后菌株生长被抑制程度相似,24 h后菌落数仍保持在与接种菌量相当水平,这可能与氯霉素仅具有抑菌作用而无杀菌作用有关。而当抗菌药物浓度低于MIC,即为1/4×MIC和1/2×MIC时,庆大霉素诱导耐药菌株被有效抑制,而亲本菌株则不能被有效抑制。该研究结果提示我们协同敏感现象的发生使得耐药菌株能够在较低的抗菌药物浓度作用下被清除,尤其对存在较严重基础疾病患者使用具有较强副作用二线药物情况下,将能够允许安全用药的前提下有效清除耐药菌株。
综上所述,本研究显示粪肠球菌经庆大霉素体外诱导耐药能够快速获得耐药性,并对氯霉素协同敏感。低水平氯霉素能够有效抑制协同敏感菌株的生长,这为基于协同敏感的交替用药策略的应用奠定基础,协同敏感的发生发展规律及其发生机制仍有待进一步的阐释。
参 考 文 献
Holmes A H, Moore L S, Sundsfjord A, et al. Understanding the mechanisms and drivers of antimicrobial resistance[J]. Lancet, 2016, 387(10014): 176-187.
Watkins R R, Bonomo R A. Overview: Global and local impact of antibiotic resistance[J]. Infect Dis Clin North Am, 2016, 30(2): 313-322.
Maltas J, Wood K B. Pervasive and diverse collateral sensitivity profiles inform optimal strategies to limit antibiotic resistance[J]. PLoS Biol, 2019, 17(10): e3000515.
Imamovic L, Ellabaan M M H, Dantas Machado A M, et al. Drug-driven phenotypic convergence supports rational treatment strategies of chronic infections[J]. Cell, 2018, 172(1-2): 121-134 e114.
Zhao B, Sedlak J C, Srinivas R, et al. Exploiting temporal collateral sensitivity in tumor clonal evolution[J]. Cell, 2016, 165(1): 234-246.
Yoshida M, Reyes S G, Tsuda S, et al. Time-programmable drug dosing allows the manipulation, suppression and reversal of antibiotic drug resistance in vitro[J]. Nat Commun, 2017, 8: 15589.
Nichol D, Rutter J, Bryant C, et al. Antibiotic collateral sensitivity is contingent on the repeatability of evolution[J]. Nat Commun, 2019, 10(1): 334.
Sun Y, Lu H, Zhang X, et al. Phenotype and genotype alteration during adaptive evolution of Enterococcus faecalis to antimicrobials[J]. Infect Genet Evol, 2018, 62: 80-85.
Podnecky N L, Fredheim E G A, Kloos J, et al. Conserved collateral antibiotic susceptibility networks in diverse clinical strains of Escherichia coli[J]. Nat Commun, 2018, 9(1): 3673.
Lozano-Huntelman N A, Singh N, Valencia A, et al. Evolution of antibiotic cross-resistance and collateral sensitivity in Staphylococcus epidermidis using the mutant prevention concentration and the mutant selection window[J]. Evol Appl, 2020, 13(4): 808-823.
Daoud Z, Sokhn E S, Azar E, et al. Mutant prevention concentrations of ciprofloxacin against urinary isolates of Escherichia coli and Klebsiella pneumoniae[J]. J Infect Dev Ctries, 2014, 8(2): 154-159.
Szybalski W, Bryson V. Genetic studies on microbial cross resistance to toxic agents. I. Cross resistance of Escherichia coli to fifteen antibiotics[J]. J Bacteriol, 1952, 64(4): 489-499.
Lazar V, Nagy I, Spohn R, et al. Genome-wide analysis captures the determinants of the antibiotic cross-resistance interaction network[J]. Nat Commun, 2014, 5: 4352.
Furusawa C, Horinouchi T, Maeda T. Toward prediction and control of antibiotic-resistance evolution[J]. Curr Opin Biotechnol, 2018, 54: 45-49.
Costa L B D, Corá L F, Correa F E L, et al. High prevalence of the aac(6')-Ie-aph(2'')-Ia gene in hospital isolates of Enterococcus faecalis co-resistant to gentamicin and penicillin[J]. Microb Drug Resist, 2019, 25(9): 1275-1281.
Gianvecchio C, Lozano N A, Henderson C, et al. Variation in mutant prevention concentrations[J]. Front Microbiol, 2019, 10: 42.
Imamovic L, Sommer M O. Use of collateral sensitivity networks to design drug cycling protocols that avoid resistance development[J]. Sci Transl Med, 2013, 5(204): 204ra132.
Eliakim-Raz N, Lador A, Leibovici-Weissman Y, et al. Efficacy and safety of chloramphenicol: Joining the revival of old antibiotics? Systematic review and meta-analysis of randomized controlled trials[J]. J Antimicrob Chemother, 2015, 70(4): 979-996.
Drlica K. The mutant selection window and antimicrobial resistance[J]. J Antimicrob Chemother, 2003, 52(1): 11-17.
收稿日期:2021-09-24
基金项目:国家自然科学基金青年科学基金项目(No. 81802069)
作者简介:孙瑶,女,生于1990年,主管技师,主要研究方向为临床微生物耐药机制研究,E-mail: sunyaowzmu@163.com
通訊作者,E-mail: wyztli@163. com
