3种致病菌多重real-time PCR检测方法的建立及其在散装即食肉制品中的应用
2022-02-16高晓月李贺楠董雨馨李宇轩刘虹宇郭文萍
范 维,高晓月,李贺楠,董雨馨,李宇轩,刘虹宇,郭文萍
(中国肉类食品综合研究中心,北京食品科学研究院,北京 100068)
随着人们膳食习惯的改变,食品生产加工模式和销售方式也发生了变化,即食食品由于方便、快捷等优点已成为广大消费者推崇的快销食品[1-2]。此类食品主要是指不需要额外加工处理即可食用,该类食品或是未经烹煮或已经煮熟、烫热或冰冻,无需再经加热处理(包括翻热)[3]。即食食品较为常见的销售形式为散装销售,散装即食食品因略去繁琐的包装,买卖随意方便,降低了食品的成本,倍受消费者和生产者的青睐。但是由于散装即食食品的制作、销售过程中,销售者与食品的接触以及食品与空气的接触,导致微生物性“二次污染”的可能性加大,增加了散装即食食品的致病菌污染风险,特别是熟肉制品、凉拌菜等[4-5]。目前,针对散装即食食品国家卫生健康委员会已经拟订了《散装即食食品中致病菌限量》标准草案,该草案中对散装即食食品中需检测的致病菌以及各致病菌的限量提出了明确要求,其中热处理散装即食食品要求检测的致病菌有沙门氏菌(Salmonella,SAL)、金黄色葡萄球菌(Staphylococcus aureus,SA)和蜡样芽孢杆菌(Bacillus cereus,BC)3种。对于这3种致病菌的检测,传统的微生物检测和鉴定方法一般要经过增菌培养、分离纯化、生化鉴定和血清学验证等多个步骤,检测周期长,操作繁琐[6-7];免疫学检测技术存在灵敏度偏低、检测通量小等缺点[8];生物传感器技术[9]、质谱技术[10-11]成本较高,需要专业人员操作,不易在基层普及推广。因此,建立一种高通量、准确、快速的致病菌检测方法显得尤为重要。
多重实时聚合酶链式反应(real-time polymerase chain reaction,real-time PCR)技术因其在检测通量、时效性、灵敏度以及成本方面的优势得到迅速发展[12-13]。Elisa等[14]利用三重real-time PCR方法实现了肉制品中SAL、大肠埃希氏菌O157:H7和单核细胞性李斯特菌3种食源性致病菌的同时检测。Qin Hong等[15]利用多重real-time PCR技术实现对牛奶中单核细胞性李斯特菌、坂崎克罗诺杆菌、SA的检测。李涛等[16]利用多重realtime PCR技术实现对海产品中霍乱弧菌、副溶血性弧菌和单核细胞性李斯特菌的快速检测。但目前对于采用多重TaqMan探针real-time PCR技术同时定量检测肉制品中SAL、SA和BC的研究较少。因此,本研究旨在建立一种高通量、快速、准确、灵敏的SAL、SA和BC三重realtime PCR检测方法,以应用于市场上散装即食肉制品的检验,为监管部门进行散装即食食品病原微生物风险监测提供技术支持。
1 材料与方法
1.1 材料与试剂
1.1.1 样品与菌种
实验采用散装即食肉制品指熟的纯肉制品,例如酱牛肉、酱肘子、烤羊腿肉、熏鸭肉、熏鸡胸肉等,购自超市、门市店或农贸市场。
菌株共有27 株,包括SAL 6 株、SA 3 株、BC 3 株、非目标菌株15 株,保存于-80 ℃冰箱。其中SA(ATCC 25923)、鸭沙门氏菌(ATCC 9270)、BC(ATCC 8187)、大肠埃希氏菌(ATCC 25922)、伊氏李斯特氏菌(ATCC 19119)、产气荚膜梭菌(ATCC 13124)、志贺菌(ATCCA 9207)、铜绿假单胞菌(ATCC 27853)、产气肠杆菌(ATCC 13048)、英诺克李斯特氏菌(ATCC 33090)、副溶血性弧菌(ATCC 17802) 美国模式培养物集存库;鼠伤寒沙门氏菌(CGMCC 1.1190)、SA(CGMCC 1.0089)、表皮葡萄球菌(CGMCC 1.2429)、弗氏柠檬酸杆菌(CGMCC 1.1732) 中国微生物菌种保藏中心;甲型副伤寒沙门氏菌(CICC 24167)、肠炎沙门氏菌(CICC 21482)、婴儿沙门氏菌(CICC 21481)、乙型副伤寒沙门氏菌(CICC 10437)、BC(CICC 10041)、SA(CICC 10384)、阪崎肠杆菌(CICC 21544)、荧光假单胞菌(CICC 21620) 中国工业微生物菌种保藏中心;BC(CMCC 63303)、单核细胞性李斯特菌(CMCC 54002)、枯草芽孢杆菌(CMCC 63501)、普通变形杆菌(CMCC 49027) 中国医学微生物菌种保藏中心。
1.1.2 试剂
营养琼脂(nutrient agar,NA)、脑心浸液肉汤(brain heart lnfusion,BHI)、胰蛋白胨大豆肉汤(trypticase soy broth,TSB)、缓冲蛋白胨水(buffered peptone water,BPW)、李氏增菌肉汤(listeria enrichment broth base,LB)、7.5%氯化钠肉汤(7.5%盐)、胰酪胨大豆多黏菌素肉汤(trypticase soy polymyxin broth base,TSPBB) 北京陆桥技术有限责任公司;细菌基因组DNA提取试剂盒 广州迪澳生物科技有限公司;2×PCR Premix ExTaqTM大连宝生物科技有限公司;引物、探针合成 上海英潍捷基科技有限公司。
1.2 仪器与设备
FTC3000荧光定量PCR 加拿大枫岭科技有限公司;DHP-9272恒温培养箱 上海一恒科学仪器有限公司;TD4低温高速离心机 鑫港仪器有限公司;SterilGARD III Advance 生物安全柜 美国贝克公司。
1.3 方法
1.3.1 引物与TaqMan探针设计与合成
根据GenBank中登录的SALinvA基因、SASa442基因和BCCereolysin AB基因的保守序列,应用Priemer Express和Primer Premier 5.0软件进行引物和探针设计,再通过GenBank中的BLAST以及Snapgene软件验证引物和探针的可行性,具体序列见表1。
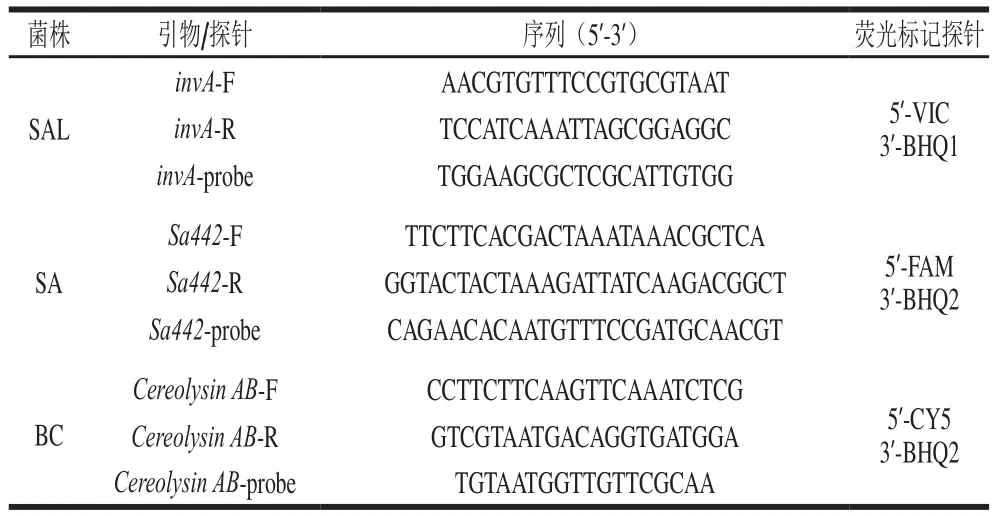
表1 实验所用引物及TaqMan探针序列Table 1 Primers and TaqMan probes used in this study
1.3.2 细菌的培养及模板DNA提取
考察不同增菌液对3种目标菌同时增菌的效果。将3种目标菌分别接种于BHI肉汤中,36 ℃培养24 h,各取1 mL进行10 倍递增稀释(10-1~10-8),并选择适当稀释浓度进行倾注平板计数。将10-8稀释水平的3种菌悬液进行等体积混合后,分别用5 种常见的前增菌液(TSB、BPW、LB、7.5%盐和TSPBB)于36 ℃条件下,对混合菌悬液进行5 h前增菌。通过对比5 种增菌液中3种目标菌的比生长速率衡量其增菌效果。菌种比生长速率按下式计算。DNA的提取采用细菌基因组DNA提取试剂盒,按照说明书进行,将DNA模板冻存于-20 ℃备用。
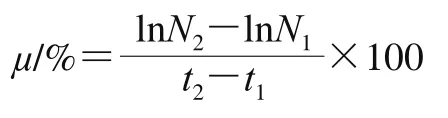
式中:μ为菌种比生长速率/%;N1和N2分别为t2/h、t1/h对应的菌落总数/(CFU/mL)。
1.3.3 多重real-time PCR体系及扩增条件
反应体系为50 μL:25 μL 2×PCR Premix ExTaqTM,3种探针(10 μmol/L)各1.0 μL,3种上、下游引物(10 μmol/L)各1.0 μL,DNA模板3.0 μL,补水至50 μL。扩增条件:95 ℃预变性3 min,94 ℃变性5 s,60 ℃退火延伸30 s,40 个循环,在每个循环延伸阶段收集荧光信号。阳性结果判定:Ct值不大于40且扩增曲线呈S型。
1.3.4 特异性实验
提取12 株目标菌株和15 株非目标菌株的DNA,并以此为模板,分别进行SAL、SA、BC三重real-time PCR实验,以检测本方法的特异性。
1.3.5 标准曲线及敏感性实验
将经活菌计数的SAL 2416、SA 25923、BC 8187菌液,分别进行10 倍梯度稀释至10-7,各取1 mL进行DNA提取。将提取后的3 株菌的DNA,依据同级别浓度梯度分别进行等体积混合,以混合后的DNA为模板进行realtime PCR扩增,以浓度的对数为横坐标,Ct值为纵坐标制作标准曲线。
1.3.6 重复性实验
参考张静等[17]的方法,分别在2 批次菌株培养液梯度稀释样本中选取3个梯度(103、104、105CFU/mL),用2 批次试剂进行多重real-time PCR检测,每一梯度重复检测5 次,每次2个平行,计算Ct值的组间和组内变异系数以评估其重复性和稳定性。
1.3.7 多重real-time PCR方法检出限
考察不同肉基质样品的检出限。对比样品不经过增菌和经过5 h增菌后进行多重real-time PCR检测,2 种前处理方法检出限的差异,具体做法如下:取经验证不含3种目标致病菌的5 种即食肉制品(猪、牛、羊、鸡、鸭)样品各40 份(每份25 g),每种源性样品平均分成4 组,向其中加入225 mL增菌液,均质1 min。之后将活化好的菌悬液加入250 mL体系中,使体系中3种目标菌(SAL 2416、SA 25923、BC 8187)的终浓度分别达到100、101、102、103CFU/mL,充分混匀。每组中取5 份样品直接进行多重real-time PCR检测;另5 份样品于37 ℃恒温培养箱增菌5 h后,进行多重real-time PCR检测。
1.3.8 实际样品检测
从不同超市、门市店及农贸市场购买即食肉制品样品100 份,包括猪肉制品(20 份)、牛肉制品(20 份)、羊肉制品(20 份)、鸡肉制品(20 份)、鸭肉制品(20 份),为了保证一定的检出率,对其中40 份样品进行目标菌人为污染,具体样品信息见表2。取各样品25 g添加225 mL增菌液制备250 mL体系,充分混匀后,于37 ℃恒温培养箱增菌5 h。之后取1 mL增菌液进行DNA提取及多重real-time PCR检测。同时采用GB 4789.4—2016《食品微生物学检验 沙门氏菌检验》[18]、GB 4789.10—2016《食品微生物学检验 金黄色葡萄球菌检验》[19]及GB 4789.14—2014《食品微生物学检验 蜡样芽孢杆菌检验》[20]对样品中的SAL、SA和BC分别进行检测,对比2 种方法的检测结果。
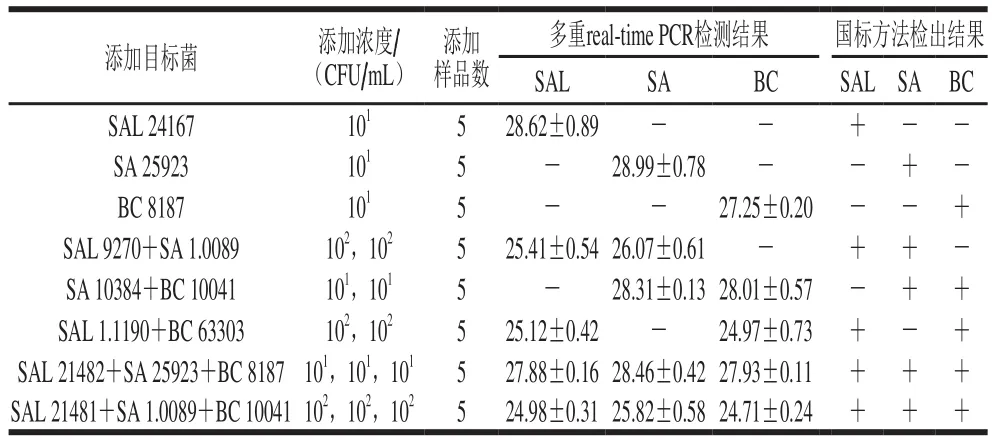
表2 40 份人为污染肉制品样品信息及检测结果Table 2 Results of real-time PCR and national standard method for 40 artificially contaminated meat samples
1.4 数据处理
2 结果与分析
2.1 引物、TaqMan探针可行性及特异性验证结果
从NCBI获取SAL、SA和BC的基因组序列,使用Snapgene软件打开基因组序列,并将设计的引物、探针导入Snapgene软件中,对其可行性进行分析,结果见图1。由图1A~C可知,所设计的3 组引物、探针分别可以在各自对应的目标菌基因组序列中搜索到,说明3 组引物探针可以用于SAL、SA和BC的real-time PCR检测。此外,通过对27 株目标菌和非目标菌进行多重realtime PCR检测发现,只有相对应的目标菌株出现“S型”阳性扩增曲线,其他非目标菌株均未出现。说明针对这3种致病菌建立的多重real-time PCR方法具有较好的特异性,与其他食源性致病菌存在交叉反应的概率较小,结果见图1D。
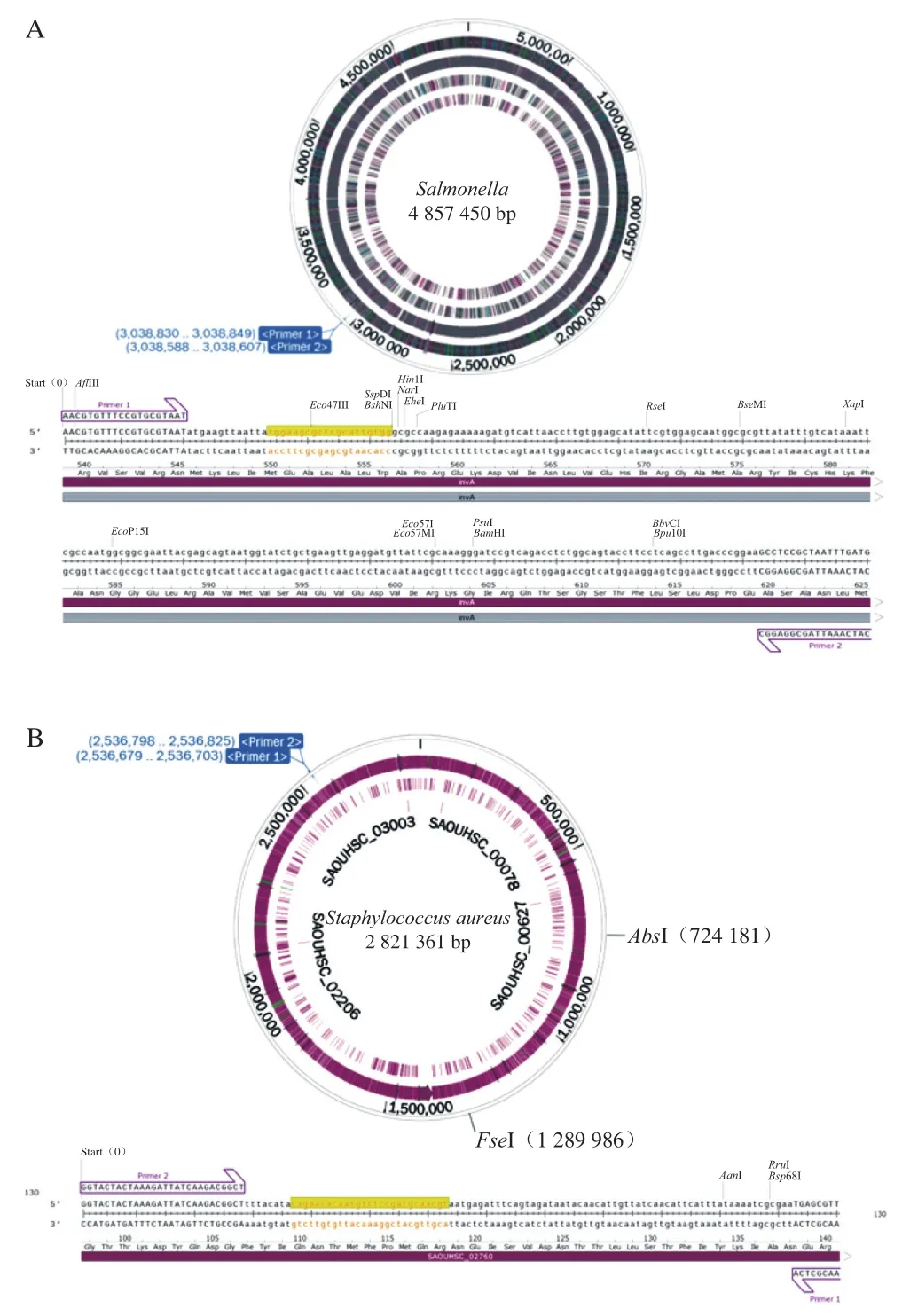
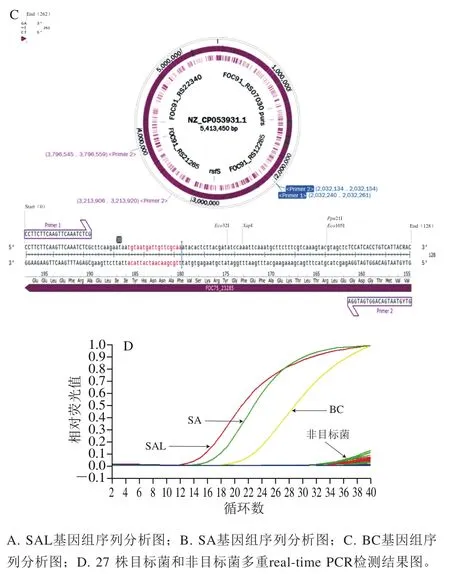
图1 引物、TaqMan探针可行性及特异性分析Fig. 1 Feasibility and specificity analysis of primers and TaqMan probes
2.2 增菌液的确定
为了选出一种增菌液可以使3种目标菌均达到较好的增菌效果,本研究通过对比3种目标菌在不同增菌液中的比生长速率,确定最佳增菌液,结果见图2。BPW、LB、7.5%盐和TSPBB分别为GB 4789.4—2016《食品微生物学检验 沙门氏菌检验》[18]、GB 4798.30—2016《食品微生物学检验 单细胞增生李斯特氏菌检验》[21]、GB 4789.10—2016《食品微生物学检验 金黄色葡萄球菌检验》、GB 4789.14—2014《食品微生物学检验 蜡样芽孢杆菌检验》[20]中规定的SAL、单核细胞性李斯特菌、SA和BC的增菌液。BC在LB和7.5%盐增菌液中,经过5 h培养后并没有生长,比生长速率为0;SAL和BC在TSB、BPW、TSPBB增菌液中的增菌效果相似(P>0.05);而SA在TSB和7.5%盐增菌液中增菌效果较好(P<0.05)。综上,选取TSB作为3种目标菌的增菌液。
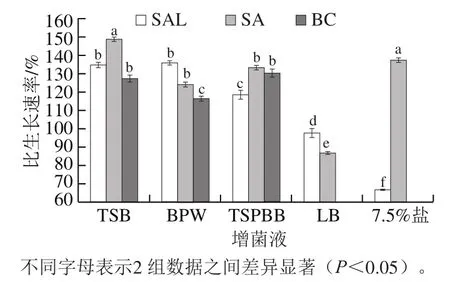
图2 不同增菌液增菌效果Fig. 2 Efficiencies of different enrichment solutions
2.3 多重real-time PCR方法敏感性实验结果
以Ct值为纵坐标(y),菌液浓度的对数为横坐标(x)绘制标准曲线,结果如图3所示。由图3B~D可知,SAL 2416的标准曲线在3.8×102~3.8×108CFU/mL浓度范围内具有良好的线性相关性(y=-3.300 0x+43.587,R2=0.998 3),SA 25923的标准曲线在4.9×102~4.9×108CFU/mL浓度范围内具有良好的线性相关性(y=-3.631 8x+44.592,R2=0.995 7),BC 8187的标准曲线在5.7×102~5.7×108CFU/mL浓度范围内具有良好的线性相关性(y=-3.516 1x+43.073,R2=0.998 7)。由此可知,多重real-time PCR法定量下限分别为3.8×102、4.9×102CFU/mL和5.7×102CFU/mL。而当3种目标菌的浓度低于该检出限时,仍有扩增(Ct=35~40),但不呈线性关系,且重复性和准确性差。
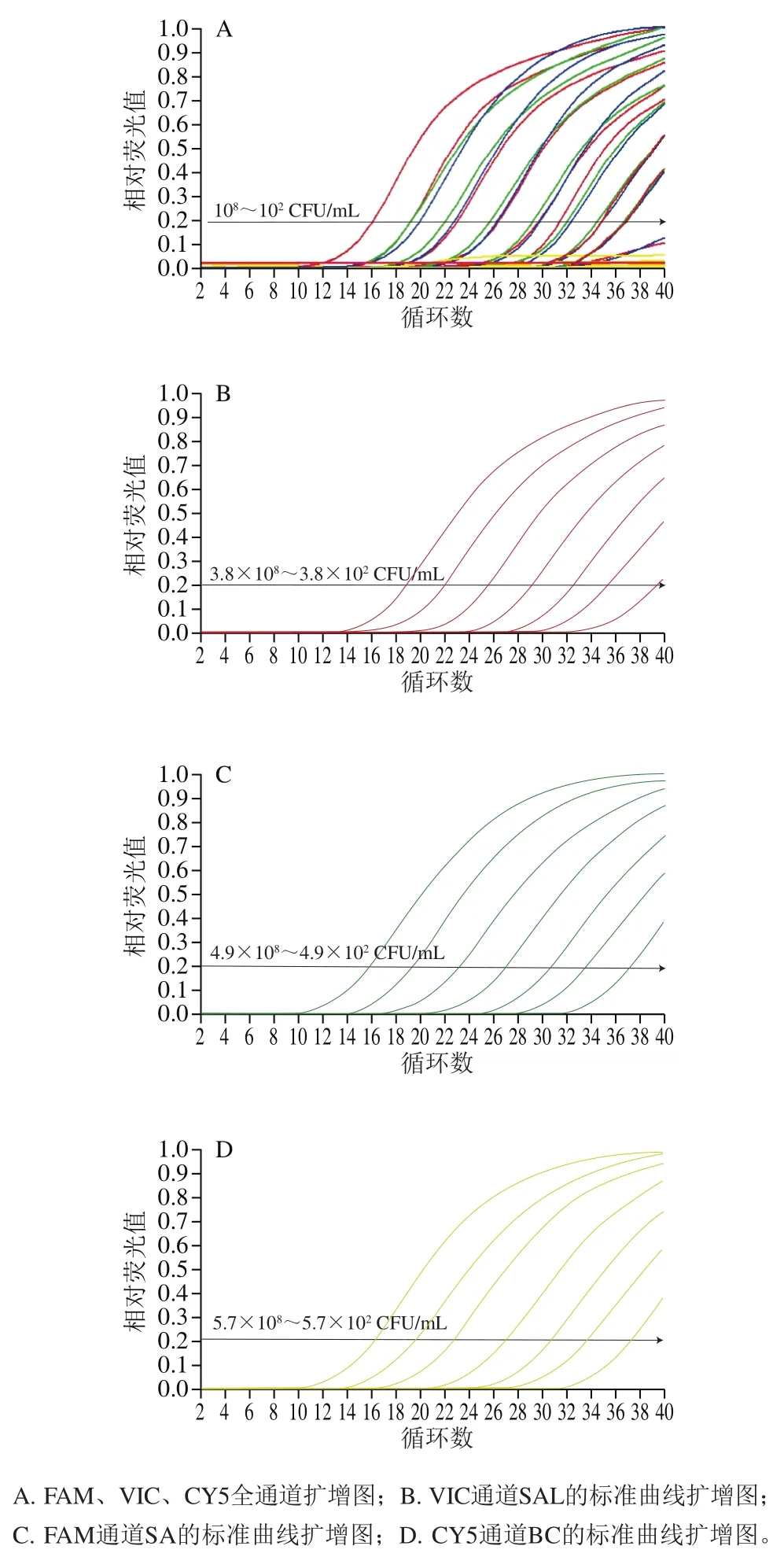
图3 3种目标菌扩增曲线及标准曲线Fig. 3 Amplification curves and standard curves for three target bacteria
2.4 重复性实验结果
如表3所示,SAL、SA、BC的批内和批间变异系数均小于2%,表明本方法具有良好的重复性。
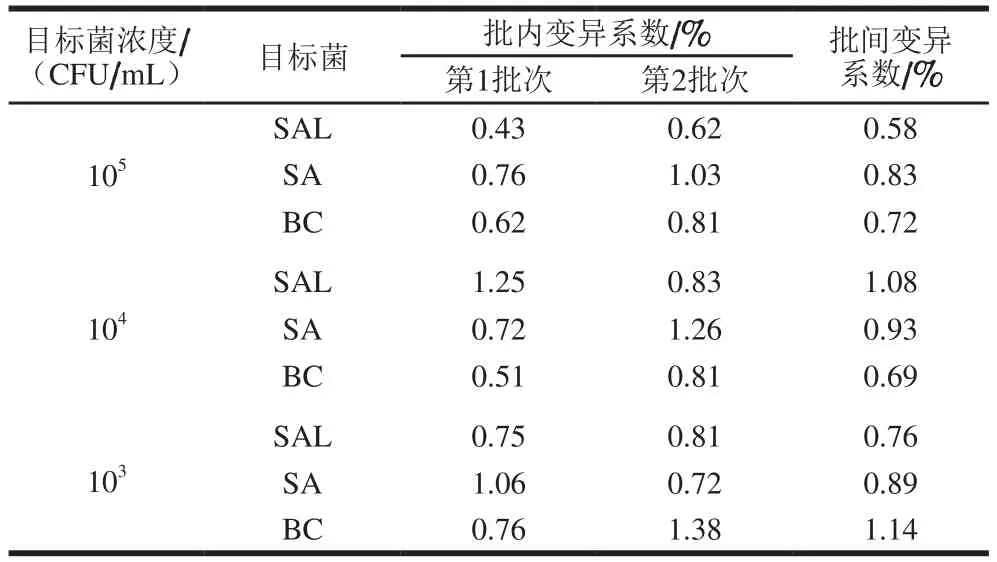
表3 多重real-time PCR方法的重复性检验结果Table 3 Reproducibility of multiple real-time PCR
2.5 多重real-time PCR方法检出限
以猪、牛、羊、鸡和鸭5 种纯肉制品作为基质,对比样品不经过增菌和经过5 h增菌,这2 种前处理方法多重real-time PCR检出限的差异。从结果可知,不同纯肉基质对目标菌的检出限影响较小,这与Fereidoun[22]、Yang Youjun[23]和Suo Biao[24]等的研究结果一致,且SN/T 1059.7—2010《进出口食品中沙门氏菌检测方法 实时荧光PCR法》[25]中对于不同食品基质,SAL的检出限相同。此外,通过对不同加标浓度样品检测证明,2 种前处理方法多重real-time PCR的检出限不同:未增菌组,5 种基质中SAL、SA、BC的检出限分别为3.8×102、4.9×102CFU/mL和5.7×102CFU/mL;增菌组,检出限分别为3.8、4.9 CFU/mL和5.7 CFU/mL。该检出限结果较Paola[26]、李新福[27]等的研究低,说明本方法更为灵敏;与李慧芳[28]和Elisa等[14]的研究结果相似,但增菌时间(5 h)较短(12 h或18 h),可能是优化了前增菌液的原因。
2.6 实际样品检测结果
采用本方法与国标方法同时对100 个实际样品(包括40 个人为污染样品和60 个自然样品)进行检测,其中除了人为添加的40 个样品检出目标菌外,还有1个自然样品检出SAL,2个自然样品检出SA。总体上,2 种方法的检测结果一致(表2、4),说明所建方法结果可靠,可以用于即食肉制品中3种致病菌的检测。
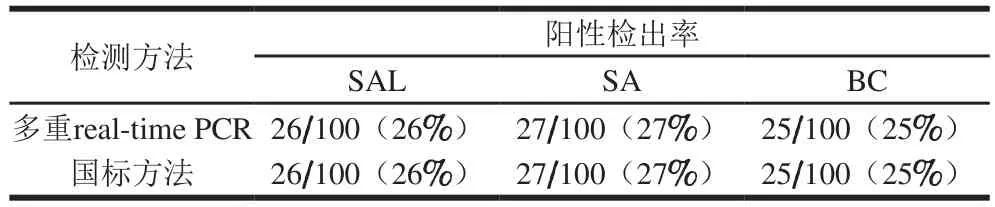
表4 不同方法检测即食肉制品中SAL、SA、BC的阳性检测结果Table 4 Positive detection rates of SAL, SA and BC in ready-to-eat meat products determined by different methods
3 结 论
本研究建立即食肉制品中SAL、SA和BC的三重TaqMan探针real-time PCR检测方法,实现在一个反应管中同时扩增3种目标菌,达到了缩短检测时间、提高检测效率的目的。TaqMan探针法由于只能结合特定的扩增子,因此较SYBR-Green染料法具有更好的特异性,无需再借助熔解曲线分析鉴定PCR的单峰[29-30]。通过对12 株目标菌和15 株非目标菌进行特异性验证,发现本方法具有良好的特异性,15 株非目标菌均无典型扩增曲线,且对6 种(SAL 9270、1.1190、24167、21482、21481、10437)同属但不同血清型的SAL均可检出。此外,该方法还具有较高的灵敏度和重复性,即食肉制品样品中的检出限可达100CFU/mL水平,满足各标准中对3种目标菌限量值的要求,且定量检测批内和批间的变异系数均小于2%。对100 份即食肉制品实际样品进行检测均与国家标准检测的结果符合,且该方法从前增菌到多重realtime PCR检测完成仅需7 h,远低于国标方法(检测周期为2~6 d)。综上,本研究建立的三重TaqMan探针realtime PCR检测方法可以作为一种可靠地检测手段,为监管部门进行散装即食肉制品中病原微生物风险监测提供技术支持。
