人源RNA解旋酶DDX1的表达、究
2021-10-23高宝才段树燕苏晓琴李继喜
高宝才,段树燕,苏晓琴,纪 锐,李继喜
(1. 复旦大学 生命科学学院,上海 200438; 2. 复旦大学 遗传工程国家重点实验室,上海 200438)
RNA解旋酶几乎参与RNA代谢的所有主要步骤,包括pre-mRNA剪切、mRNA转运、RNA编辑和衰减、核糖体合成[1-2].DEAD -box蛋白(DDXs)属于RNA解旋酶超家族II,也是最大的解旋酶家族[3].DDX蛋白包含有9个保守基序,分别命名为Q、Ⅰ、Ⅰa、Ⅰb、Ⅱ、Ⅲ、Ⅳ、Ⅴ和Ⅵ.其中基序Ⅱ也称为Walker B基序,是以DEAD(Asp-Glu-Ala-Asp)氨基酸序列为特征,并在抗病毒免疫应答和肿瘤发生发展中起着核心作用[4].DDX家族蛋白在发挥解旋酶功能时,ATP与RNA之间存在协同作用[5].目前已知有60余人源DDX蛋白,其中DDX1除了具有共有的解旋酶结构域外,其基序Ⅰ和Ⅰa之间包含一个SPRY结构域,该结构域可介导蛋白质相互作用以及蛋白-RNA相互作用[6].
DDX1最初是在神经母细胞瘤和视网膜母细胞系中发现,并在肿瘤组织中过度表达,因此DDX1被作为乳腺癌的潜在生物标志物和卵巢癌的预测标志物[7].DDX1还参与多种细胞功能,例如mRNA/miRNA加工[8-9],与hnRNP K相互作用[10],参与NF-κB介导的转录[11-12],参与富含AU的元素介导的衰变,以及募集到含有RNA-DNA结构的DNA损伤位点[13-15].此外,DDX1在病毒复制,尤其是在HIV-Rev的复制中发挥重要的作用[16];也可与DDX21、DHX36以及接头蛋白TRIF组成一个巨大复合物,可以感知和识别流感病毒A、呼肠孤病毒以及长短链poly(I: C)信号,激活NF-κB和I型干扰素的表达[17].目前DDX家族中只有少数几个成员,如DDX58(也叫RIG-I)[18]、MDA5[19]和DHX36的结构得以解析[20].我们实验室最近也获得了DDX21识别和解旋底物RNA的多个状态的结构信息,阐明了DDX21识别和结合流感病毒NS1蛋白的作用机理.但目前有关DDX1如何识别、结合和解旋RNA的结构基础尚未报道.蛋白质结构的解析和分析对于研究其功能机制具有极为重要的意义,解析DDX1蛋白的晶体结构及空间构象,将有助于我们进一步理解其发挥解旋酶功能的机制以及在各种生理病理过程中发挥功能的分子机理.
1 材料和方法
1.1 材料和主要试剂
1.1.1 材料
各种限制性内切酶、T4 DNA连接酶等均购自TaKaRa公司.E.coliDH5α和E.coliBL21(DE3)plysS感受态菌株购自北京全式金生物技术有限公司.pSMT3表达载体为本实验室库存,其N端带有6×His-Sumo标签.剪切Sumo标签的Ulp1酶由本实验室自行纯化保存.
1.1.2 主要试剂
所用试剂均为分析纯,大肠杆菌培养基,卡那霉素、氯霉素等抗生素均购自生工生物工程(上海)股份有限公司,Ni-NTA亲和层析柱、凝胶过滤层析柱(Superdex75 10/300 GL,Superdex75 16/600 GL,Superdex 200 10/300 GL,Superdex200 16/600 GL)均购自GE有限公司.
1.2 方法
1.2.1 人源DDX1的克隆、表达及纯化
以人源DDX1基因(NM_004939.2)为模板,设计两端引物,采用BamHⅠ和XhoⅠ进行双酶切(见表1,酶切位点已用下划线标出),从人cDNA文库中克隆全长及不同截短体DDX1到pSMT3载体上[21].经菌落PCR验证,并送公司测序以确定各种截短体质粒构建是否正确,相关信息见表2.

表1 引物信息
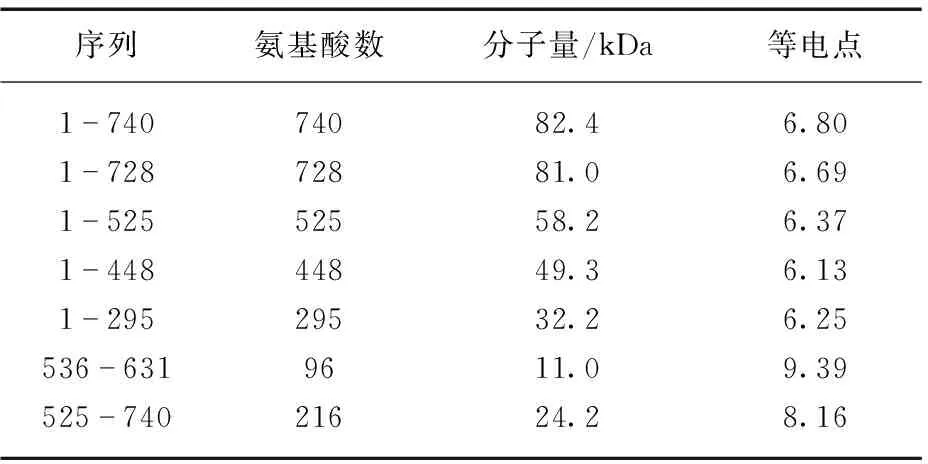
表2 人源DDX1蛋
之后将构建好的不同表达质粒通过热激法转入表达菌株E.coliBL21(DE3) plysS中.将转化后的菌液加入到卡那霉素终浓度为50 μg/mL、氯霉素抗性终浓度为34 μg/mL的新鲜LB培养基(100 mL)中,于37 ℃摇床中振荡培养过夜.第二天按1∶50的体积比将菌液接种到1 L加有卡那霉素、氯霉素的LB培养基中;37 ℃振荡培养至OD600为0.6~0.8时,加入IPTG至终浓度为0.5 mmol/L,18 ℃ 诱导表达18~20 h.经7 000 r/min离心20 min收集菌体,弃去上清,用Buffer(50 mmol/L Tris,500 mmol/L NaCl,10 mmol/L Imidazole,5% glycerol,pH 8.0)进行重悬菌体(按每克30 mL的量),然后使用低温超高压细胞破碎机进行菌体破碎.
在4 ℃条件下,18 000 r/min离心1 h,收集破菌液上清.上清蛋白样品用Ni-NTA亲和层析柱进行梯度洗脱纯化(洗脱Buffer: 50 mmol/L Tris,500 mmol/L NaCl,250 mmol/L Imidazole,5% glycerol,pH 8.0).洗脱样品加Ulp1酶(1∶1 000),于4 ℃过夜酶切去除N端的Sumo标签[22],再进行二次Ni-NAT亲和层析.流出液(目的蛋白)通过SDS -PAGE检测,根据蛋白纯度进行凝胶过滤层析(Buffer: 20 mmol/L Tris,100 mmol/L NaCl,2 mmol/L DTT,pH 8.0).最后将获得的高纯度蛋白收取浓缩,液氮速冻保存于-80 ℃待用.
1.2.2 动态光散射分析
动态光散射(Dynamic Light Scattering, DLS)分析参考文献[22],测定仪器为动态激光光散射仪(DynaPro NanoStar),配套软件为DYNAMICS.测定时,目的蛋白浓度为1 mg/mL,每组数据采集10次,每个样品独立测量5次(测量时蛋白Buffer: 20 mmol/L Tris,100 mmol/L NaCl,2 mmol/L DTT,pH 8.0).
1.2.3 X射线小角散射分析
X射线小角散射(Small-Angle X-ray Scattering, SAXS)分辨率为1~25 nm,能够直接测定溶液中蛋白质分子的轮廓和分布状态,在蛋白质-蛋白质相互作用及蛋白质-核酸相互作用研究中发挥重要作用.SAXS数据在上海同步辐射光源BL19U2线站进行收集.样品在检测前,需在低温下进行17 000 r/min离心10 min,以去除沉淀和不溶物质.样品浓度可以设置浓度梯度,每个样品上样量为70 μL.数据处理软件为ATSAS (https:∥www.embl-hamburg.de/biosaxs/atsas-online/).
1.2.4 DDX1蛋白的生物信息学研究
多氨基酸序列比对: 将需要比对的目的氨基酸序列提交至网站(https:∥www.genome.jp/tools-bin/ clustalw),获得比对结果;然后将结果以及相关蛋白的PDB结构文件提交至ESPript 3.0网站(http:∥espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi),即可获得基于三维结构的比对结果[23].
IUPred2有序度分析: 可以通过IUPred2(http:∥iupred2a.elte.hu)对目的蛋白进行有序度分析,发现目的蛋白中潜在的无序区.将人源DDX1的氨基酸序列提交,即可获得预测的有序度分析情况.
同源建模分析: SWISS -MODEL是一种全自动蛋白质结构同源性建模服务器,可通过ExPASy Web服务器或从DeepView程序(Swiss Pdb-Viewer)访问.将目的蛋白的氨基酸序列输入,待系统进行识别;等成功识别完成,进行模板寻找或者是直接的模型构建.
2 结果与分析
2.1 人源DDX1蛋白的表达及纯化
本文所表达的目的蛋白N-端均带有6×His-Sumo标签.DDX1全长蛋白为740aa,等电点为6.69.DDX1主要由N-端的解旋酶ATP结合区(Helicase ATP binding,氨基酸2-428)和C-端解旋酶区(Helicase C-terminal domain,氨基酸493-681)组成(图1(a,b),见第430页).在共有基序I和Ia之间包含一个B30.2/SPRY结构域(氨基酸70-247),介导蛋白质相互作用以及蛋白-RNA相互作用[6].目前也有文献报道DDX1的C-末端区域(氨基酸525-740)与poly(A) RNA及HNRNPK蛋白相互作用[6].经大肠杆菌表达,通过Ni-NTA柱纯化,我们发现除DDX1(536-631)及DDX1(525-740)外,全长DDX1和其余截短体均可以获得纯度较高,性质稳定的蛋白(图1(c),见第430页);进一步经过凝胶过滤层析(Superdex200 16/600 GL)方法,我们可以获得单一稳定蛋白(图2(a,b),见第431页).从分子筛图可以看出,DDX1全长蛋白主要呈现出一个单体峰,峰尖位置在72 mL,对应分子量大小为82 kDa,表明DDX1主要以单体形式存在.其余截短体DDX1(1-728)、DDX1(1-448)和DDX1(1-295)均表现出单一峰,出峰位置与这些蛋白分子量一致(图2(a)).DDX1(1-525)截短体在分子筛上出现2个峰,76 mL峰对应于单体,而45 mL峰表明该截短体有高聚倾向.此外,截短体DDX1(536-631)和DDX1(525-740)蛋白性质极其不稳定,很容易发生沉淀,故而未能进行分子筛检测.
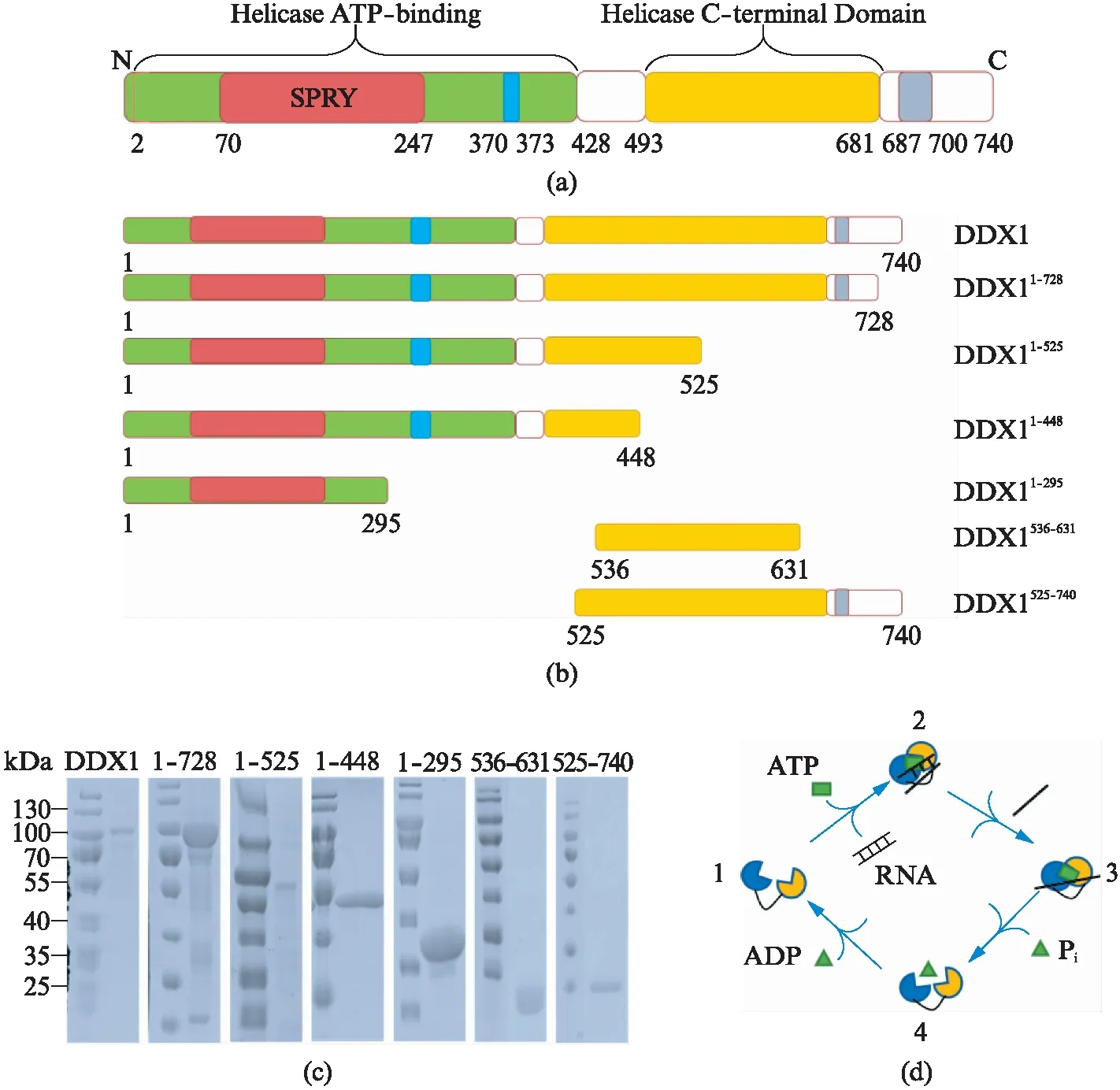
图1 人源DDX1蛋白结构域及表达纯化结果Fig.1 Domain schematic and the in vitro purification results of human DDX1(a) 人源DDX1结构域示意图.绿色区域代表Helicase ATP-binding结构域,蓝色区域代表DEAD -box,灰色区域代表无规则序列区域,SPRY结构域用红色表示.(b) DDX1不同截短体结构域组成信息.(c) DDX1全长及不同截短体蛋白体外表达纯化结果.(d) DEAD -box蛋白解旋RNA机制模型.蓝色和橙色分布代表DDX家族核心功能域D1以及D2,在没有配体情况下,蛋白采用开放的构象;当结合ATP以及RNA时,蛋白处于闭合状态,形成ATP酶活位点以及RNA结合位点.在这种状态下,RNA发生形变.解离完成,ATP水解为ADP以及游离的磷离子.
2.2 DLS及SAXS结果分析
我们将所有纯化的DDX1蛋白用DLS进行了分析,从图2(c~e)(见第431页)结果可以看到,DDX1, DDX1(1-728)、DDX1(1-525)、DDX1(1-488)以及DDX1(1-295)的粒径均小于6 nm,相对Pd值也低于20%,所占的质量除了DDX1(1-525)外,其余均为100%.因此,这5个蛋白在溶液中均一度良好(图2(c~e)).同时,DDX1(536-631)和DDX1(525-740)两个截短体的分子半径均大于8 nm,相对应为高聚状态;而且二者的Pd值均大于30%,表明其分子半径分布波动较大,进一步佐证了这些截短体的聚合状态不均一.结合DLS与分子筛分析(图2(a~e)),DDX1的全长蛋白及包含N-末端的截短体比较稳定、性质均一;而包含C-末端的截短体蛋白容易高聚,性质不均.这可能是DDX1的C-末端具有柔性区域,适应于其他相互作用蛋白/核酸的结合.

图2 人源DDX1蛋白分子筛及DLS结果Fig.2 The gel-filtration profiles and DLS results of DDX1 and its truncations(a~b)DDX1全长及不同截短体蛋白的分子筛(Superdex200 16/600 GL,上面)和SDS-PAGE(下面)结果.其中L代表上样前样品.M代表蛋白标准品.(c~e)DDX1全长以及截短体蛋白的动态光散射实验结果.其中(c)图表示不同蛋白在溶液中的分子粒径大小;(d)图表示蛋白所占的质量百分比;(e)图表示不同蛋白的Pd值.
进一步用X射线小角散射(SAXS)方法分析DDX1的溶液轮廓,结果显示DDX1全长蛋白呈现出较为均一的分布情况(图3),其完整性良好且呈单分散状态.根据P(R)值计算DDX1蛋白的最大尺寸(Dmax)和回旋半径(Rg)分别为9.953 nm和3.752 nm(图3).

图3 人源DDX1全长蛋白SAXS数据分析Fig.3 The SAXS results of human DDX1 protein(a) 通过SAXS获得的强度曲线.(b) P(R)曲线函数.DDX1蛋白最大尺寸(Dmax)和回旋半径(Rg)分别为9.953 nm和3.752 nm.
2.3 DDX1与同源家族解旋酶的多序列比对
在同源家族多重氨基酸序列比对中,选择与人源DDX1蛋白相似度较高且有结构报道的嗜热脂肪芽孢杆菌(Geobacillusstearothermophilus10)来源CshA蛋白(PDB_ID: 5IVL,相似度29%,Positive: 48%)为DDX1蛋白结构模板,分别与人源DDX21(NC_000010.11),DDX3X(NC_000023.11,相似度29%,Positives: 46%),IF4A3(NC_000017.11,相似度28%,Positives: 46%)以及DDX56(NC_000007.14,相似度27%,Positives: 41%)进行多重序列比对.通过比对结果可知,DDX1蛋白具有典型的DEAD -box家族特征,包含一个典型DEAD氨基酸序列,且在多个位点上表现出相同性和一致性.此外,DEAD -box家族在进化上具有一定的保守性.除去典型的DEAD序列,其他如74-LVVAPTRELA-83,182-SAT-184以及324-YVHRUGRTGRA-334等序列片段也呈现出高度保守性(见图4).
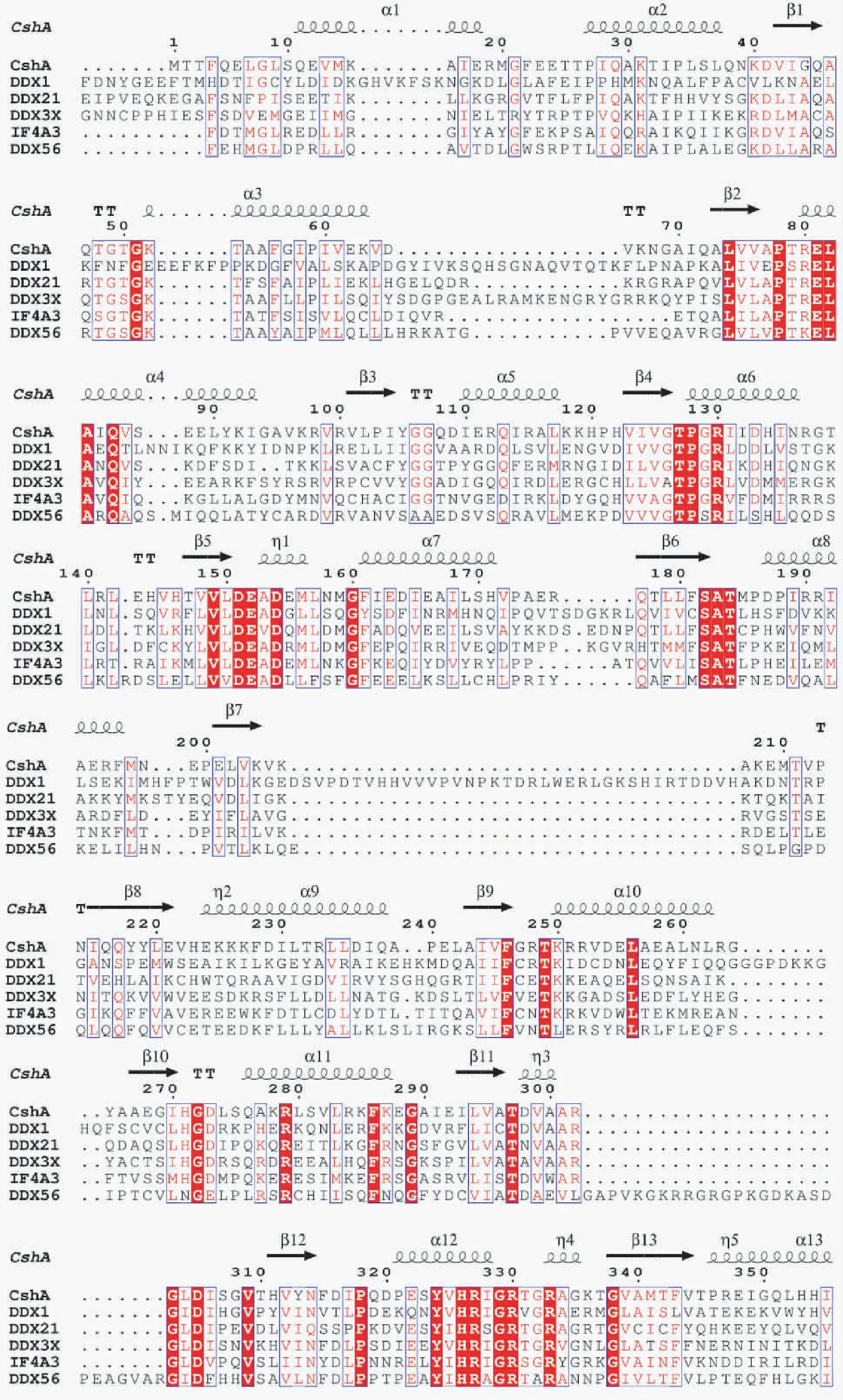
图4 DDX1蛋白与DEAD -box家族蛋白的多重氨基酸序列比对Fig.4 Multiple sequence alignment of DDX1 with its homologous proteins所比对的蛋白序列分别来自嗜热脂肪芽孢杆菌CshA蛋白(PDB_ID: 5IVL),人源DDX21,DDX3X,IF4A3以及DDX56.相同和高度保守的氨基酸残基分别用红色和白色表示.
通过Blastp在线程序对不同物种来源的DDX1进行分析,发现DDX1在高等哺乳动物中具有高度的保守性;人源DDX1与苏门答腊猩猩源蛋白具有99.86%的同源性,与小鼠源蛋白具有98%的同源性,与牛源蛋白具有97.28%的同源性.
2.4 人源DDX1蛋白的有序度分析
IUPred2是目前较为广泛使用的预测蛋白质有序度的服务器[24].将人源DDX1蛋白提交网站进行计算.当IUPred2以及ANCHOR2的评分结果高于0.5时,代表其处于无序状态的可能性较大;分值越高,无序程度越高.从图5可知,人源DDX1蛋白整体得分情况较低,多数氨基酸位点得分在0.5以下.在IUPred2的评分结果中,氨基酸序列为75-80,277-280,441-445,466-481,554-558的片段,得分处于0.5以上;而ANCHOR2评分均低于0.5,这表明DDX1蛋白整体有序度较高,不存在较长片段的无规则序列卷曲.DDX1蛋白无序度最高的区域处于两个主要功能结构域之间,表明这些柔性区可能不利于蛋白质晶体生长,这也与我们实验室最近报道的DDX21结构类似[20].

图5 人源DDX1蛋白的IUPred2预测结果Fig.5 IUPred2 prediction result of human DDX1图中红线表示通过IUPred2对不同氨基酸的评分结果.横坐标表示氨基酸位点,纵坐标表示分值;其中分值大于0.5的区域代表无序或者柔性区.
2.5 人源DDX1蛋白的同源建模
我们将获得的全长DDX1蛋白及多个截短体蛋白进行了1 000多个晶体条件筛选,尝试了在不同温度下生长不同浓度的蛋白.遗憾的是,目前尚未能获得足够用于X射线衍射的蛋白晶体.由于DDX1与已报道的嗜热脂肪芽孢杆菌CshA蛋白结构具有29%的相似性[25],我们采用SWISS -MODEL[26]进行同源建模,以期获得DDX1的可能结构信息.提交人源DDX1蛋白后,我们获得了以嗜热脂肪芽孢杆菌CshA蛋白(PDB_ID: 5IVL)为模板的三维结构模型.模型与模板之间的序列相似度在34.88%,且整体序列覆盖率在90%,GMQE评分为0.34,整体可信度较高.通过PyMOL软件(https:∥pymol.org/2/)显示(图6),DDX1氨基酸序列为248-644,其余区域并没有建模成功.所获得的结构模型表明,DDX1由两个结构区域组成.其中氨基酸序列248-430为区域1,即N端解旋酶ATP结合区域,包括7个α螺旋,7个β折叠;氨基酸序列481-644为区域2,主要包括C端解旋酶区域,含6个α螺旋,7个β折叠(图6).两个结构域均呈现出典型的α/β/α亚结构域特征,即β折叠被α螺旋所包裹.两大核心区域之间有一段较长的无规则序列卷曲(431-481),与IUPred2分析结果中无序区域(441-445,466-481)一致.

图6 人源DDX1蛋白三维结构模型Fig.6 The structural model of human DDX1DDX1模型结构由2个结构域组成,包括15个α螺旋和14个β折叠.右图为旋转180°的视野图.紫色代表β折叠,天蓝色代表α螺旋,黄色代表无规则序列卷曲.
3 讨 论
DDX1作为RNA解旋酶DEAD -box家族中重要的一员,其在RNA代谢方面发挥着极其重要的作用[11].此外,DDX1与病毒复制及核酸代谢相关[27-28].有研究表明,DDX1可被劫持作为细胞辅因子参与冠状病毒[28]、丙型肝炎病毒[29-30]以及人类多瘤病毒的复制[31].DDX1也在HIV病毒的复制转录中发挥功能[32],可结合HIV的Rev蛋白和RRE RNA,并具有RRE-RNA依赖的催化活性[33-34].本研究通过对不同功能区的截短体进行表达纯化以及DLS分析,发现涉及N端解旋酶ATP结合区域的截短体其稳定性普遍较好,蛋白聚合状态均一且多为单体存在.其中截短体1-295性质最稳定,与前人报道的DDX1蛋白以及SPRY结构域蛋白相似[35].DDX21的C端截短体性质不同,蛋白稳定性差,容易发生沉淀,不利于后期晶体生长(图2).多重序列比对结果表明,DDX1具有保守的DEAD序列,其中74-LVVAPTRELA-83,182-SAT-184及324-YVHRUGRTGRA-334等片段也呈现出高度保守性,这表明DEAD -box家族在进化上具有一定的保守性,保守位点可能对其功能发挥产生作用(图4).IUPred2分析显示氨基酸序列75-80,277-280,441-445,466-481,554-558片段的无序度较高(图5);SWISS -MODEL建模则表明两大核心区域呈现出α/β/α折叠状态.这种“sandwich conformation”结构与已报道的DDX1蛋白SPRY结构域表现出一致性[35].两大核心区域之间有一段较长的无规则序列卷曲(431-481),结合IUPred2分析推测氨基酸序列(441-445,466-481)在DDX1的正确折叠中起到负面作用,不利于晶体的生成.这也提示今后的研究中,可以尝试将这部分序列进行相应的处理,或许可以获得稳定性更高,聚合状态更加均一的DDX1蛋白.DDX21与ssRNA的复合物结构(PDB_ID: 6L5N)呈现出典型的结合模型[36],即经典的DEAD -box RNA解旋酶在结合ssRNA时,其解旋酶核心的两个RecA与功能区域(D1,D2)进行结合,形成ATPase酶活功能结构域和核酸结合的裂缝.并且通过进一步分析发现,在DDX21发挥解旋酶作用时,ATP和RNA协同发挥结合作用,这与之前报道的DDX1蛋白的功能研究结论相类似[34],推测ssRNA以及ATP/ADP对DDX1的功能发挥起到重要作用,也可能对于其结晶起到一定作用,值得进一步尝试.
