严重急性呼吸综合征冠状病毒2型4种结构蛋白特性分析
2020-10-10
(吉林大学动物科学学院,吉林长春 130062)
2019新型冠状病毒病(coronavirus disease 2019,COVID-19)是由严重急性呼吸综合征冠状病毒2型(severe acute respiratory syndrome coronavirus type 2,SARS-CoV-2)引起的一种主要通过近距离空气、飞沫传播和密切接触传播的呼吸道传染病[1],目前在世界范围内广泛传播,并已造成世界范围的大流行。截至北京时间2020年5月15日2时31分,全球累计确诊人数已达4 386 761人,累计死亡人数已达297 043人[2],并且仍在增加。该病毒属于基因组最大的一类RNA病毒,容易发生变异,这给疾病的诊断、预防和治疗都带来了较大困难。
人感染SARS-CoV-2后的常见体征有发烧、乏力,部分出现干咳,少数有流鼻涕、鼻塞以及胃肠道症状[3],严重的可导致肺炎、严重急性呼吸综合征、肾衰竭,甚至死亡[1]。报道[1,4]显示:该病毒在人群中传染性明显增加,致死率为2%~4%,虽然比普通流感高,但远比严重急性呼吸综合征冠状病毒(severe acute respiratory syndrome coronavirus,SARS-CoV)、埃博拉病毒和H7N9流感病毒低;该病的中位潜伏期只有3 d,长的可达24 d以上,且在家庭和社区有明显的聚集发病现象。
与SARS-CoV相同,SARS-CoV-2含有单股正链RNA基因组,具有5'帽子(cap)和3' poly(A)尾巴的结构,长度在29.5~30.3 kb之间[5-6]。基因组由11个开放阅读框(open reading frame,ORF)组成,分别编码ORF1ab,刺突蛋白(spike protein,S),ORF3a,囊膜蛋白(envelope,E),膜蛋白(membrane protein,M),ORF6,ORF7a,ORF7b,ORF8,核衣壳蛋白(nucleocapsid protein,N)和ORF10[6-8]。序列同源性分析发现,SARS-CoV-2与SARS-CoV的核酸序列约有80%的相似性[9],而与蝙蝠源冠状病毒RaTG13(GenBank号 MN996532)的全基因组同源性达93.7%~96.2%,其ORF1ab、N和S蛋白的氨基酸同源性分别为98.5%、99.05%和97.41%[10]。
研究表明,冠状病毒的刺突蛋白(S),囊膜蛋白(E)、膜蛋白(M)、核衣壳蛋白(N)对病毒感染有重要作用。它们的主要功能是入侵宿主细胞,维持病毒粒子形态,帮助病毒粒子装配、释放,其中S、M、E 3种蛋白位于病毒囊膜表面[11],而N蛋白则位于病毒囊膜内侧。
以往的SARS-CoV研究发现:在感染过程中,S蛋白与宿主表面受体血管紧张素转化酶2(angiotensin I converting enzyme 2,ACE2)结合[12],并经过网格蛋白和小窝蛋白非依赖型内吞途径进入细胞[13],然后将病毒基因组释放到宿主细胞质中。而在病毒组装过程中,M蛋白的C端和核衣壳相互作用形成一个核心结构,S蛋白则与病毒囊膜合为一体,然后再与M、E蛋白相互作用,从而完成病毒粒子装配,然后成熟的病毒粒子穿膜释放到胞外[14-15]。因此,M和E蛋白可能与S蛋白在病毒包膜形成过程中起共同作用[15-17]。也有研究[17-21]推测,E蛋白与病毒传染性有关。
目前研究[22]显示,SARS-CoV-2的S蛋白结构与SRAS-CoV非常相似,同样可与ACE2结合,并且靶向SARS-CoV S蛋白的中和抗体也可与SARS-CoV-2相结合[22]。因此,S蛋白受体结合域也是相关抗体、疫苗研发的重要靶点。
N蛋白最重要的功能是识别病毒基因组RNA的特征序列,与病毒基因组RNA结合形成螺旋状的核衣壳,并与其他结构蛋白相互作用,完成病毒粒子装配[9,11,15]。此外,N蛋白也可能与病毒RNA合成[23]以及病毒建立感染有关[24-25]。
众所周知,蛋白质的功能与其结构密切相关。本研究借助生物信息学蛋白分析工具,分析SARSCoV-2的4个结构蛋白的分子生物学特征,并预测4种蛋白的部分结构域,以期为更好地了解该病毒,并为SARS-CoV-2的相关分子生物学研究和针对其结构蛋白的疫苗开发等提供参考,也为后期的COVID-19防治奠定基础。
1 材料和方法
选取NCBI上公布的SARS-CoV-2的4种结构蛋白序列进行特性分析,其中S蛋白为YP_009724390,M蛋白为YP_009724393,E蛋白为YP_009724392,N蛋白为YP_009724397。
1.1 氨基酸组成分析
应用DNAstar软件的子程序Protean,分析蛋白的氨基酸组成。
1.2 信号肽预测
应用SignalP-5.0[26]在线服务器(http://www.cbs.dtu.dk/services/SignalP-5.0/),预测4种蛋白各自的信号肽及其剪切位点。
1.3 亚细胞定位预测
用在线服务器PSORT Prediction(http://psort1.hgc.jp/form.html),对4种蛋白进行亚细胞定位。
1.4 跨膜结构预测
使 用TMHHM 2.0(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0),预测4种蛋白的跨膜结构。
1.5 BUSCA综合分析
使用BUSCA在线服务器(http://busca.biocomp.unibo.it/),综合分析、预测4种蛋白的信号肽、跨膜区和亚细胞定位。
1.6 B细胞抗原表位预测
应用ABCpred[27]在线服务器(https://webs.iiitd.edu.in/raghava/abcpred/ABC_submission.html),直接预测S蛋白的B细胞抗原表位;应用DNAstar软件的子程序Protean,采用Kyte-Doolittle[28]方法,预测4种蛋白的亲水性;用Karplus-Schultz[29]方法,预测4种蛋白的可变性;用Emini[30]方法,预测4种蛋白的表面可及性;用Jameson-Wolf[31]方法,预测抗原指数。
2 结果与分析
2.1 氨基酸组成分析
应用DNAstar软件的子程序Protean,对蛋白的氨基酸组成进行分析发现,E蛋白的核酸序列含有228 nt(nucleotide),编码75个氨基酸。该蛋白相对分子质量为8 365.06 Da,等电点为8.33。在构成E蛋白的氨基酸中,亮氨酸(Leu)占比最高,达到18.67%;而天冬氨酸(Asp)、谷氨酰胺(Gln)、甲硫氨酸(Met)和色氨酸(Trp)占比最低,仅为1.33%。此外,E蛋白不含组氨酸、色氨酸和谷氨酰胺。
M蛋白的核酸序列含有669 nt,编码222个氨基酸。该蛋白相对分子质量为25 146.68 Da,等电点为9.48。在构成M蛋白的氨基酸中,Leu占比最高,达到 15.77%;而半胱氨酸(Cys)、Met和Gln占比最低,仅为1.8%。
S蛋白的核酸序列为3 822 nt,编码1 273个氨基酸。该蛋白相对分子质量为141 178.84 Da,等电点为6.47。在构成S蛋白的氨基酸中,Leu占比最高,达到8.48%;而Trp占比最低,仅为0.94%。
N蛋白的核酸序列有1 260 nt,编码419个氨基酸。N蛋白相对分子质量为45 625.69 Da,等电点为10.10。在构成N蛋白的氨基酸中,甘氨酸(Gly)占比最高,达到8.48%;而组氨酸(His)占比最低,仅为0.95%。此外,N蛋白不含Cys。
2.2 信号肽预测
经SignalP 5.0预测,N、M和E 3种蛋白N端存在信号肽的可能性极小。而S蛋白在N端存在信号肽,且有96.89%的可能性属于常规分泌(Sec/secretory)通路,由I型信号肽酶(SPase I,SPI)进行切除。信号肽剪切位点有53.53%的可能性在15~16 aa(SQC-VN)处(图1)。
2.3 亚细胞定位
用PSORT Prediction预测,发现N蛋白存在于细胞核内的可能性最大(表1),而S蛋白最可能存在于质膜,可能性为46.0%,其次是微体(表2)。
E、M蛋白最可能定位于质膜和内质网膜上,但M蛋白也有46.0%的可能性存在于高尔基体(表3~4)。
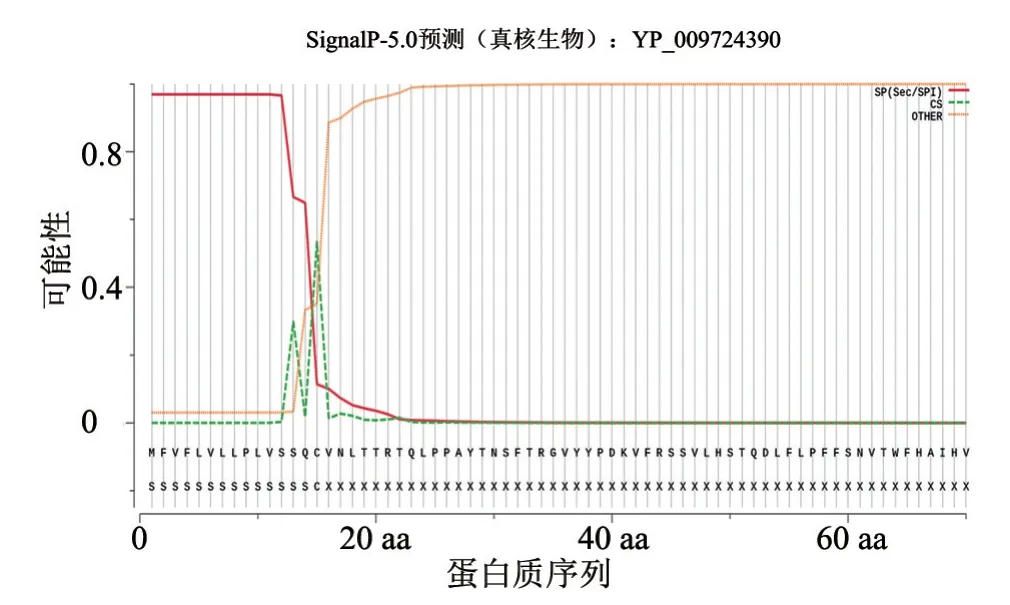
图1 S蛋白信号肽预测结果

表1 N蛋白亚细胞定位预测结果
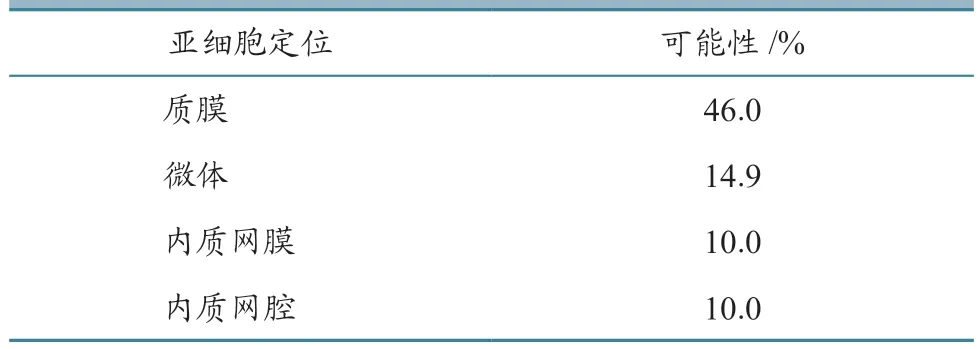
表2 S蛋白亚细胞定位预测结果
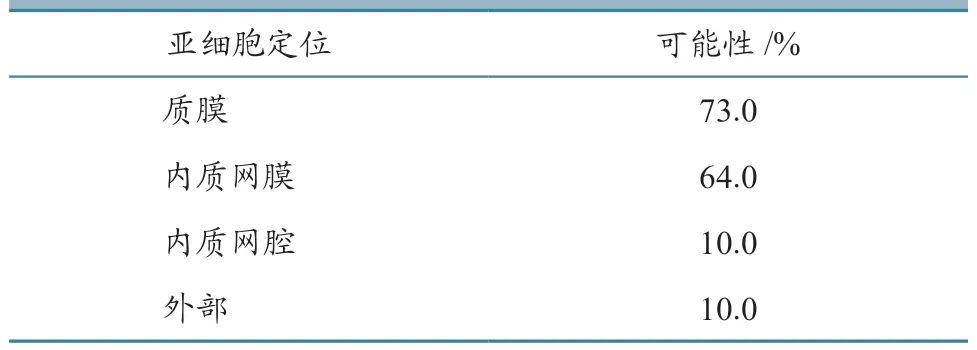
表3 E蛋白亚细胞定位预测结果
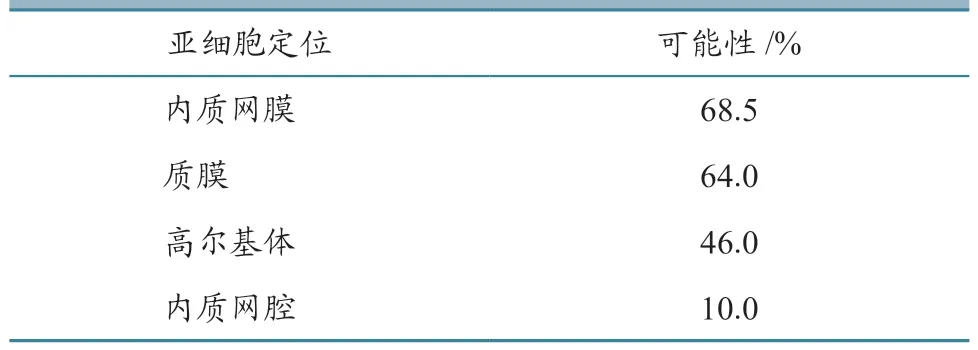
表4 M蛋白亚细胞定位预测结果
2.4 跨膜结构预测
使用TMHHM 2.0预测4种蛋白的跨膜结构发现:E蛋白1~11 aa在膜内,35~75 aa在膜外,12~34 aa为跨膜区(图2-A);M蛋白40~50、101~222 aa在膜内,1~19、74~77 aa在膜外,20~39、51~73、78~100 aa为跨膜区(图2-B);S蛋白1~1 213 aa在膜外,1 237~1 273 aa在膜内,1 214~1 236 aa为跨膜区(图2-C);N蛋白则全部处于膜外,无跨膜区(图2-D)。
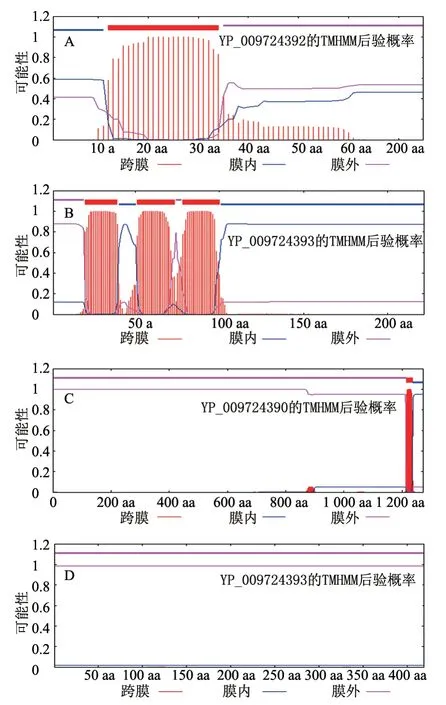
图2 S、E、M、N蛋白的跨膜结构预测结果
2.5 BUSCA预测
使用BUSCA在线服务器,对4种蛋白进行综合分析预测,发现结果与前面的预测基本相符,表明E、M、S蛋白均定位于质膜上,N蛋白定位于胞质内,但E、M蛋白也有可能定位于内质网膜。E蛋白9~42 aa为α螺旋跨膜区,M蛋白14~36、42~68、75~101 aa为α螺旋跨膜区,S蛋白1~21 aa为信号肽区段,1 051~1 062、1 213~1 234 aa为α螺旋跨膜区。
2.6 B细胞抗原表位预测
根据之前的分析,E、M蛋白多为α螺旋跨膜,N蛋白无跨膜结构域,因此选择S蛋白预测其抗原表位。由于抗原决定簇区域通常具有较好的亲水性、表面可及性和柔性,因此本研究使用DNAstar子程序Protean,对S蛋白的亲水性、柔性、表面可及性和抗原指数进行分析,选择这4种方法预测结果中共有的区段作为潜在的B细胞抗原表位,结果发现S蛋白的抗原位点较可能在20~29、75~81、95~98、111~116、148~152、181~186、458~466、575~581、676~685、773~779、809~815、1071~1 076、1 141~1 145、1 147~1 156、1 158~1 165、1182~1 187、1 204~1 208、1 258~1 262 aa处(图3)。

图3 Protean预测S蛋白抗原表位结果
而ABCpred则给出较多抗原表位预测结果,且表位主要集中在S蛋白序列的中间区域。将Protean和ABCpred预测的结果进行对比,筛选出共有抗原表位在25~29、75~81、112~116、148~152、773~779 aa。
3 讨论
关于SRAS-CoV-2的生物学和流行病学等特性仍存在许多未知。目前,对SARS-CoV-2结构蛋白的研究也大多集中在S蛋白的受体结合域(RBD)[32]、抗原表位[33-34]和疫苗设计[32]等方面,而对E、M、N蛋白结构的研究却较少,因此本研究对S、E、M、N蛋白的结构域进行多种分析。而鉴于它们的二级结构特征,本研究使用不同的方法[27,29-31,35]预测S蛋白B细胞抗原位点,并给出共有抗原表位。
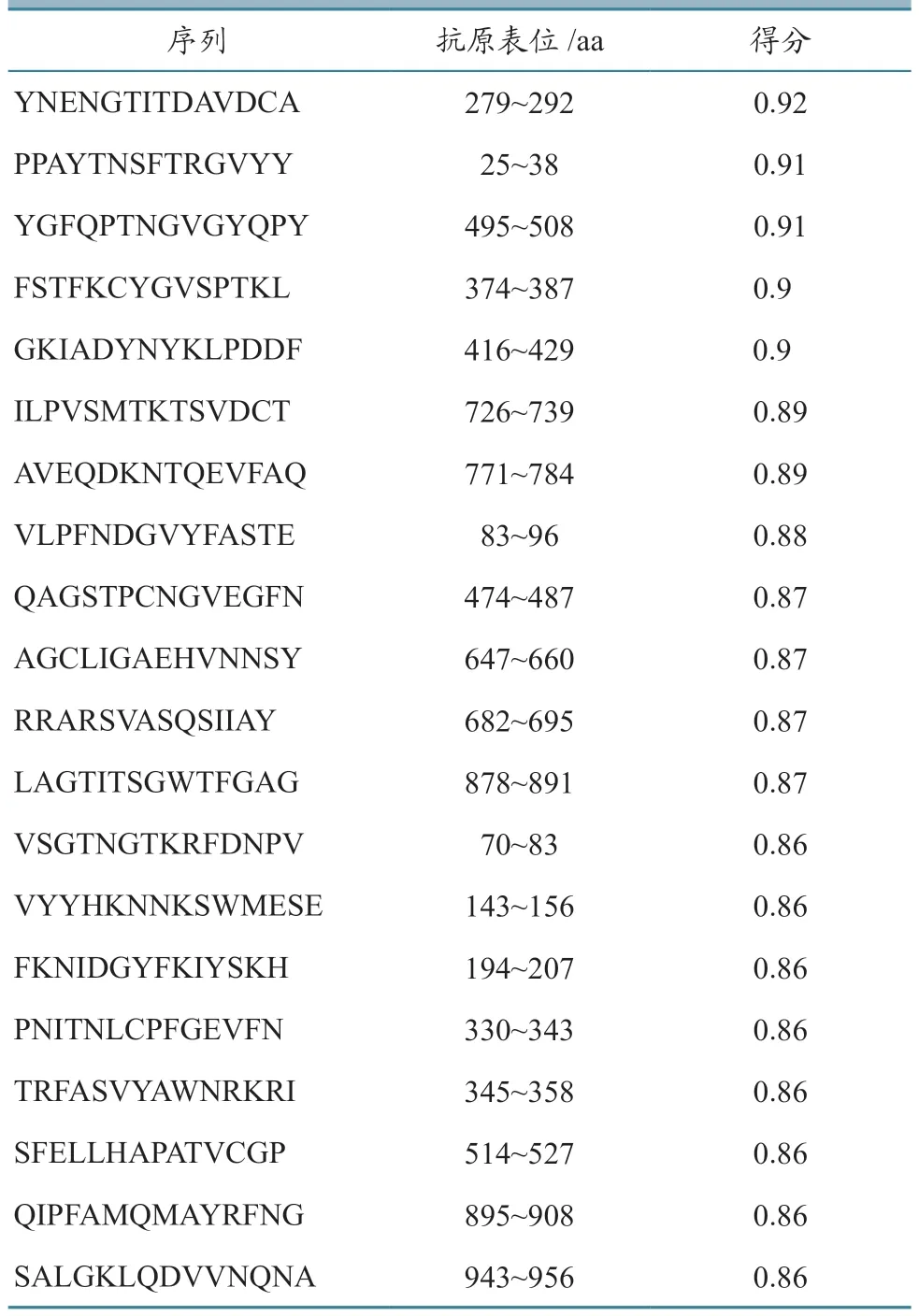
表5 ABCpred预测S蛋白抗原表位得分高于0.85的结果
本研究对不同的结构域预测使用了不同的方法。BUSCA得到的复合结果与之前使用SignalP、PSORT prediction和TMHHM得到的结果基本一致,均提示S蛋白N端存在信号肽,S、E、M蛋白存在跨膜结构域,N蛋白无跨膜结构域。这与报道的冠状病毒4种结构蛋白各自的分布及功能相符[9,11-12,24,36]。而在抗原表位预测中,基于S蛋白序列特征,预测出抗原表位25~29、75~81、112~116、148~152、773~779 aa更可能是潜在的抗体靶标。这一结果与之前报道的潜在抗原表位有所一致[34]。
本研究通过对SARS-CoV-2的4种结构蛋白结构特征进行分析,进一步了解了4种蛋白的结构域特征,也为下一步的分子生物学和实验动物学研究提示了方向。在后期工作中,将在目前研究基础上,分析该病毒结构蛋白特征,并寻找可能的药物靶点,合成抗体肽,进行更多分子生物学和医学学科的交叉研究,希望能为解析该病毒的感染和致病机制以及为相关药物设计、临床治疗提供参考。同时本研究采用的思路和方法对于研究其他病毒的结构蛋白,以及针对其蛋白设计相应的抗体、药物等也会有一定的参考价值。
