乙型肝炎病毒B和C基因型S蛋白特异性CTL表位保守性分析
2019-04-28陈庆新李文姝陈卓艳何佳莉汪文寰林茂叶菊秀
陈庆新,李文姝,陈卓艳,何佳莉,汪文寰,林茂,叶菊秀
(温州医科大学,浙江 温州 325035,1.微生物学与免疫学教研室;2.眼视光学院)
乙型肝炎病毒(hepatitis B virus,HBV)在中国高度流行,根据HBV基因序列,可将世界范围内的HBV分为A-H 8个基因型,各基因型间核苷酸序列同源性差异超过8%,我国流行的HBV基因型主要为B和C型[1]。HBV感染过程中的免疫耐受和免疫逃逸是HBV慢性感染发生的重要致病机制[2],而HBV特异性的细胞毒性T细胞(cytotoxic T lymphocyte,CTL)介导的细胞免疫应答,是最终清除HBV的关键,因此对国内流行基因型的CTL表位进行对比分析,将有助于了解HBV免疫逃逸特点,从而找到稳定特异性CTL靶点,为HBV感染后的治疗奠定基础。
本研究主要对HBV在国内流行的B和C基因型S区基因编码蛋白HLA-A*0201限制性CTL表位进行了生物信息学筛选,CTL表位长度选择为9个氨基酸,结合已鉴定和报道的表位及GenBank中已有的B和C基因型进行比较,寻找稳定的B和C基因型S区基因编码蛋白HLA-A*0201限制性CTL表位,探索可以用于HBV细胞免疫治疗的表位。
1 材料和方法
1.1 试剂与载体 pIRES载体、pGEM-T-HBV全基因组(HBV C基因型)重组载体由本实验室保存,Proteinase K购自德国Merck公司。IPTG、Ampicilin、X-Gal、进口CaCl2、胰蛋白酶(1∶250)、UNIQ-5柱离心式胶回收试剂盒、Taq plus DNA Ploymerase及dNTP Mix等购自上海生工生物工程有限公司。T4 DNA连接酶购自大连TAKARA公司。LB液体培养基、LB固体培养基、CaCl2溶液购自上海生工生物工程有限公司。
1.2 HBsAg基因的克隆和载体构建 根据HBV全基因组,设计一对引物,上游引物:5’-AGCTGCTAGCTCC GGAACAGTAAACCCTG-3’,含Nhe I酶切位点;下游引物:5’-GCCGATTAGGGTTCAAATGTA-3’,含Mlu I酶切位点。以pGEM-T-HBV全基因载体为模板,PCR扩增可得到长度为789 bp的HBsAg目的基因,将PCR产物用Nhe I和Mlu I双酶切后进行纯化,与同样酶切纯化后的pIRES混合,加入T4 DNA ligase置16 ℃恒温仪连接过夜,转化大肠杆菌DH5α。PCR和酶切鉴定为阳性的重组质粒送上海基康公司DNA测序。
1.3 候选CTL表位预测 选择HBV B基因型(D00330)和C基因型(AB033556)的S区基因序列,采用BIMAS、NetMHC和SYFPEITHI[3]预测软件分别预测S区基因编码蛋白HLA-A*0201限制性CTL表位。SYFPEITHI预测得分大于21作为CTL表位选择条件,NetMHC预测结果中选择affinity binding level<50 nmol/L的CTL表位,BIMAS预测得分大于25作为CTL标为选择依据。
1.4 候选CTL表位分析 将3种预测软件筛选出的高分CTL表位进行分析,在BIMAS、NetMHC和SYFPEITHI中的至少2个预测软件中都具有较高的预测得分选定为候选CTL表位;通过BLAST检索HBV蛋白库,分析候选CTL表位在大量HBV B和C基因型中保守性;将候选表位是否在其他文献中已经被鉴定过进行标注。
2 结果
2.1 HBsAg基因的克隆和鉴定 PCR产物核酸电泳可见大约0.8 bp的片段,与预期789 bp相符(见图1);重组载体pIRES-HBsAg酶切后可以得到6 100 bp的载体片段和800 bp的目的片段(见图2),测序分析显示与HBV S基因序列一致,说明HBsAg基因已经克隆并成功构建pIRES-HBsAg真核表达载体。
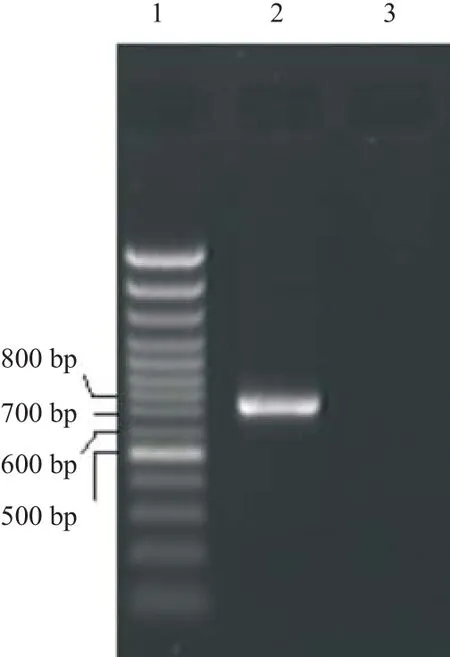
图1 HBV S基因的PCR产物核酸电泳图
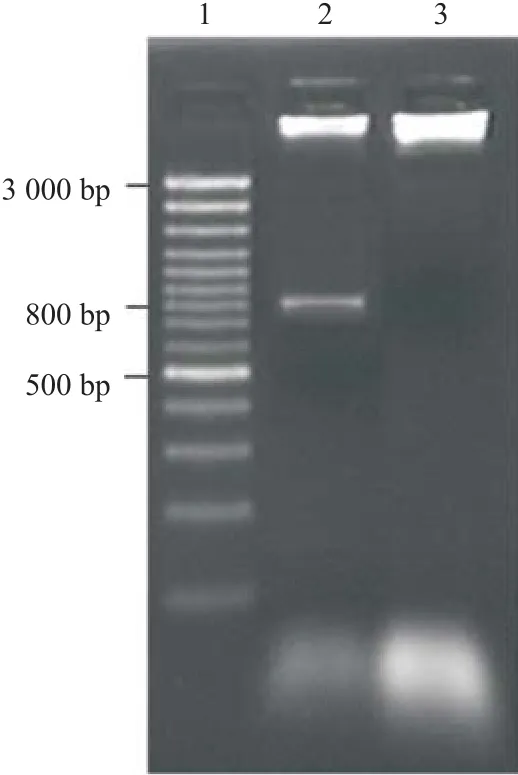
图2 pIRES-HBsAg重组质粒的酶切鉴定结果
2.2 预测CTL表位得分情况 根据HLA-A*0201限制性和9肽等条件,通过BIMAS、NetMHC和SYFPEITHI等方法对选择序列分别进行分析后得到如下结果。SYFPEITH分析共得到18个评分大于21的CTL表位(见表1)。NetMHC对CTL表位的亲和性分析后,根据评分和亲和力共得到了14个亲和性高的CTL表位(见表2)。BIMAS对S蛋白HLA-A*0201限制性CTL表位分子的解离半衰期进行评分,共预测得到20个可能存在的表位分子(见表3)。通过对表位在3种方法中的得分情况进行综合分析对比后,最后共选择17个表位进入后续的保守性分析。
2.3 候选CTL表位保守性分析 对预测表位进行分数比较、B和C基因型的保守性比较、是否鉴定等进行综合分析后发现,预测表位在B和C基因型的保守性分析发现除了S205-213(SLDSWWTSL)、S261-269(ILLLCLIFL)、S257-265(FLFILLLCL)、S270-278(VLLDYQGML)和S250-258(CLRRFIIFL)等保守率能够达到80%以上,其余表位保守性较低。已经鉴定过的表位发现,这些表位在B和C基因型中的保守性也较低,其中S215-223表位在C基因型中保守性为0%,在B基因型中则是70.2%;而S131-139和S194-202是在C基因型中保守性较高,B基因型中保守性较低,同样在未鉴定表位(S255-263和S276-284)中也存在相同现象(见表4)。

表1 SYFPEITHI预测S区编码蛋白HLA-A*0201限制性CTL表位
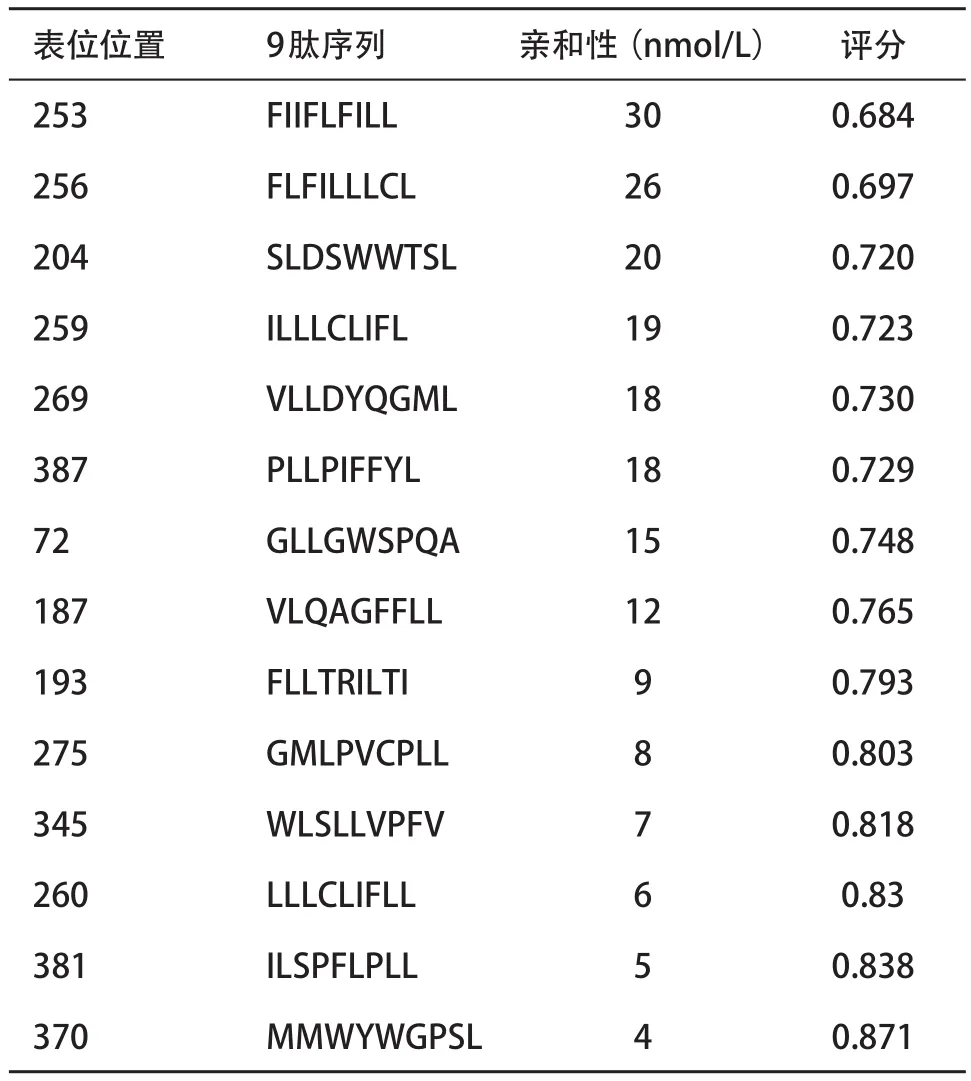
表2 NetMHCI预测S区基因编码蛋白HLA-A*0201限制性CTL表位结果

表3 BIMAS预测S区基因编码蛋白HLA-A*0201限制性CTL表位结果
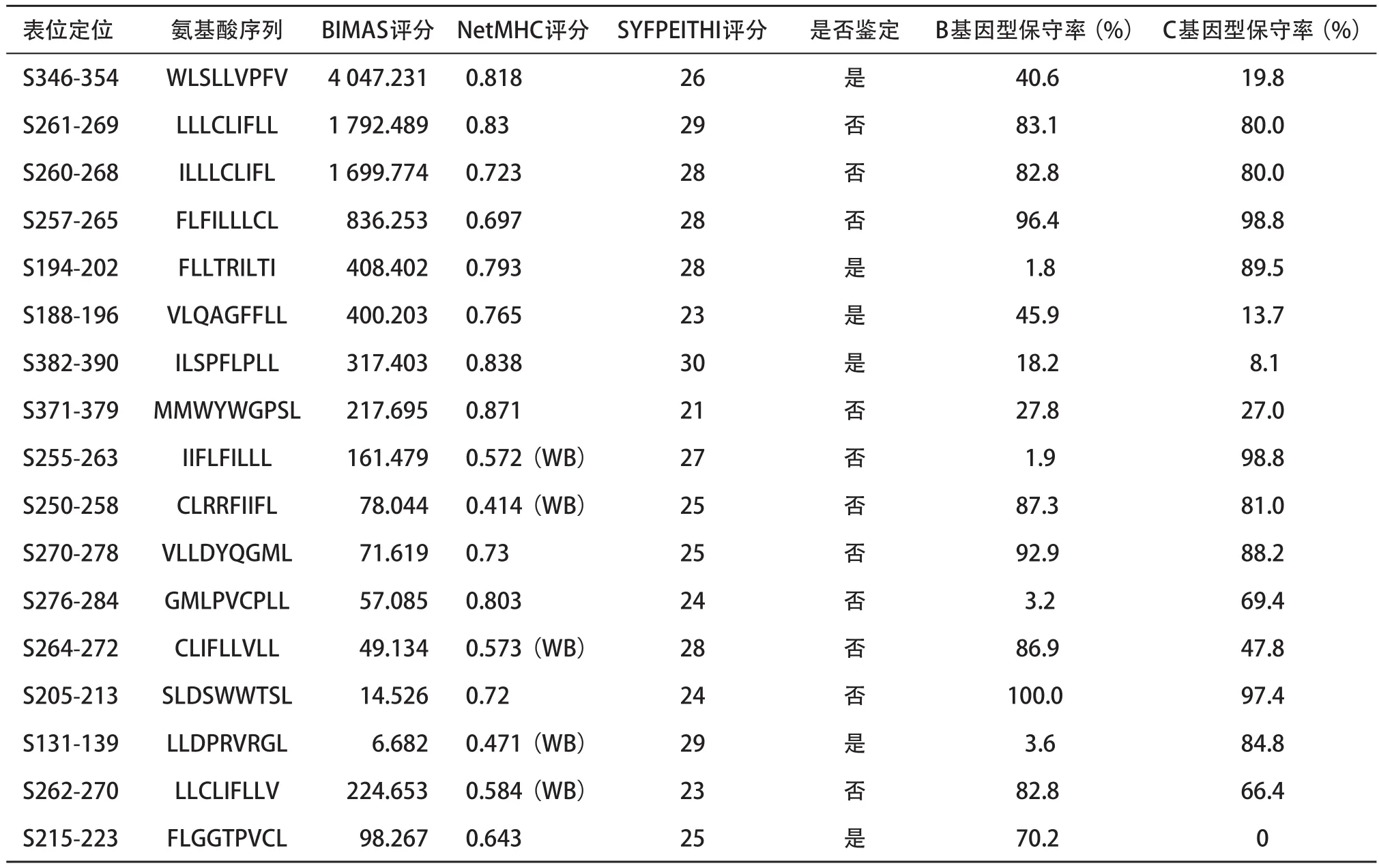
表4 候选HLA-A*0201限制性S区基因编码蛋白CTL表位的综合分析
3 讨论
HBV感染会导致慢性肝炎、肝硬化、肝癌等严重后果,而国内感染者众多,清除慢性感染者体内HBV是一项艰巨而复杂的任务[4]。HBV特异性细胞免疫应答的建立是最终清除HBV感染的根本途径,因此对HBV CTL表位的选择就显得至关重要[5]。在研究HBV表位的时候,应选择与国内流行的B和C基因型一致的表位,否则可能会影响其在国内的临床应用[6-8]。本研究结果显示蛋白库中HBV B和C基因型的S区基因编码蛋白特异性CTL表位与已鉴定CTL表位存在较大差异,这些差异会影响细胞免疫到对HBV的识别,所以选择保守性高的CTL表位对国内HBV感染后的免疫治疗具有重要意义。
本研究成功克隆HBV S基因并构建真核表达载体,通过HBV B和C基因型的S区基因编码蛋白的生物信息学分析,共筛选到17个CTL表位肽,其中已经鉴定过的有6个表位,其余表位均是未鉴定表位,保守性分析显示S区基因编码蛋白在B和C基因型中保守性不高,未鉴定的表位中有6个表位在HBV B和C基因型中保守率均超过80%,已鉴定表位在B和C基因型中保守性相对较低。这些结果显示S区基因是B和C基因型间差异的重要区域,由于S区基因的变异,导致CTL表位的改变。HBV CTL表位保守性分析提示通过S区基因编码蛋白表位的变异是HBV逃避机体的免疫应答,进而形成持续性感染和免疫耐受的重要机制。
研究发现不同HBV基因型感染人体后会导致不同的临床结局,C基因型比B基因型更能引起患者的肝脏纤维化和肝癌的发生[4,9-10]。通过对S区基因编码蛋白表位的保守性分析发现,S215-223表位在B基因型中保守率较高,而在C基因型中保守率为0,S131-1139、S194-202、S255-263和S276-284等表位在C基因型中保守率高,而在B基因型中保守率极低,说明这些表位具有型特异性,其是否与B型和C型致病性差异有关,需要通过进一步研究证实。另外,也可以将这种型特异性表位用于HBV基因型的分型鉴定[11]。
本研究克隆HBV S基因,构建真核表达载体,并对国内流行广泛的乙型肝炎病毒B和C基因型中S区基因编码蛋白的存在的可能表位进行了生物信息学筛选,分析了被筛选表位在B和C基因型中的保守性,共得到6个特异性CTL表位肽,它们在B和C基因型间均具有较高的保守性,为HBV表位在国内的进一步研究和应用提供数据支持。
