前列腺癌不良预后相关差异甲基化基因筛选
2020-08-14李猷柳东辉张国勇
李猷,柳东辉,张国勇
·论著·
前列腺癌不良预后相关差异甲基化基因筛选
李猷,柳东辉,张国勇
110016 沈阳,辽宁省金秋医院泌尿外科
识别与前列腺癌不良预后相关的差异甲基化基因,为寻找治疗靶点提供数据支持。
利用 GEO 数据库的 4 个前列腺癌基因芯片数据集 GSE46602、GSE69223、GSE6919 和 GSE32269进行差异基因的筛选,并与 TCGA 数据相比对。通过 David 数据库对其进行功能富集分析。采用 String 数据库构建了基因编码蛋白之间 PPI 网络,随后利用 Cytoscape 软件进行分析并实现可视化。通过 TCGA 甲基化数据,考量基因的甲基化水平,并利用临床数据观察其差异表达对预后的影响。
GEO 数据库筛选得到差异基因 600 个,与 TCGA 数据比对后,得到差异基因 301 个。激活了癌症、pI3K-Akt 和 cGMP-PKG 信号通路。构建 PPI 网络,分析出 10 个网络关键节点,进一步做差异甲基化分析,发现过表达基因2、2、1 和6 存在启动子区低甲基化情况,低表达基因1 启动子区高甲基化。其中基因2、1 和6 的过表达与前列腺癌的不良预后相关。
选取不同平台的前列腺癌数据,通过生物信息学分析,筛选出与不良预后相关的差异甲基化基因,为前列腺癌治疗提供新的分子靶点。
前列腺癌; 甲基化; 生物信息学分析
前列腺癌(prostate adenocarcinoma,PRAD)是男性泌尿系统最常见的肿瘤,全球范围内男性癌症第二大死因,尤其以欧洲老年男性最为常见[1]。据估算,2018 年美国前列腺癌新发 164690 例,死亡 29430 例[2]。虽然在我国发病和死亡仍以呼吸和消化道肿瘤为主[3],但随着生活方式的西化,全球范围内可以观察到,发展中国家前列腺癌的发病率和死亡率在逐渐增加[4]。近年来的表观遗传学研究表明,DNA 甲基化现象是各种恶性肿瘤发生发展的重要生物学机制[5]。甲基化的变异包括过表达基因的低甲基化和低表达基因的高甲基化,其存在于基因启动子和外显子区域的CpG岛内,异常甲基化能通过改变抑癌基因的表达水平来使之沉默,进而使基因调节失控,引发肿瘤[6]。本研究利用多平台数据筛选前列腺癌患者组织中差异表达的基因,并考察其启动子区甲基化水平,以期为临床靶点选择提供参考依据。
1 材料与方法
1.1 数据来源
下载 TCGA(The Cancer Genome Atlas,TCGA)数据库(https://www.cancer.gov/)的前列腺癌转录组数据(肿瘤组 498 例/正常组 52 例)和甲基化数据(肿瘤组 502 例/正常组 50 例)。选用GEO 基因表达数据库(https://www.ncbi.nlm.nih.gov/geo/)中两类前列腺癌转录组数据,一类为原发前列腺癌:GSE46602(肿瘤组 36 例/正常组 14 例)、GSE69223(肿瘤组 15 例/正常组 15 例);另一类是转移的前列腺癌:GSE6919(肿瘤组 25 例/原发组 65 例)、GSE32269(肿瘤组 29 例/原发组22 例)。
1.2 方法
1.2.1 前列腺癌差异基因筛选 差异基因筛选和甲基化分析采用 limma、WGCNA、data.table、GEOquery 和 Biobase 等 R 包,令前列腺癌组织与对照组织的数据相比对,筛选校正后< 0.05 并且上调表达基因log2FC > 1,下调表达基因log2FC < –1。
1.2.2 GO 分析及 KEGG 分析 选用 David 6.8 数据库(https://david.ncifcrf.gov/)对差异表达基因进行 GO分析和 KEGG分析。筛选< 0.05 的 GO 分析和 KEGG 分析。
1.2.3 蛋白质互作网络构建 将鉴别出的差异基因输入 String 数据库(https://string-db.org)构建蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络,随后利用 Cytoscape3.6.1 软件进行网络分析并实现 PPI 网络图可视化。
1.2.4 关键节点甲基化情况和预后生存分析 利用 TCGA 数据库中前列腺癌甲基化数据Infinium Human Methylation450 验证所筛基因启动子区域甲基化情况。利用 TCGA 临床数据,对关键节点使用 Kaplan-Meier 法做生存分析,使用log rank test 做比较,< 0.05 有统计学差异。
2 结果
2.1 差异基因筛选
选取 TCGA 和 GEO 两个平台的前列腺癌数据,既包含原位肿瘤的数据又选取了远端转移数据,具体筛选流程详见图 1。首先分析 TCGA 中 PRAD 数据,经鉴定得到差异表达基因 1595 个,其中上调表达 488 个,下调表达 1107 个。
为选取更具特征性的差异基因,分析了原发前列腺癌 GSE46602 数据,得到差异表达基因1393 个,其中上调表达 502 个,下调表达 891 个;GSE69223 数据,得到差异表达基因 1759 个,其中上调表达 592 个,下调表达 1167 个,两个结果取交集得到差异基因401 个。分析转移前列腺癌 GSE6919 数据,得到差异表达基因 1015 个,其中上调表达 469 个,下调表达 546 个;GSE32269 数据,得到差异表达基因 948 个,其中上调表达 604 个,下调表达 344 个,两个结果取交集得到差异基因 222 个。GEO 数据库原发和转移肿瘤差异基因结果分析发现只有 23 个基因存在重叠,数据见图 2A。
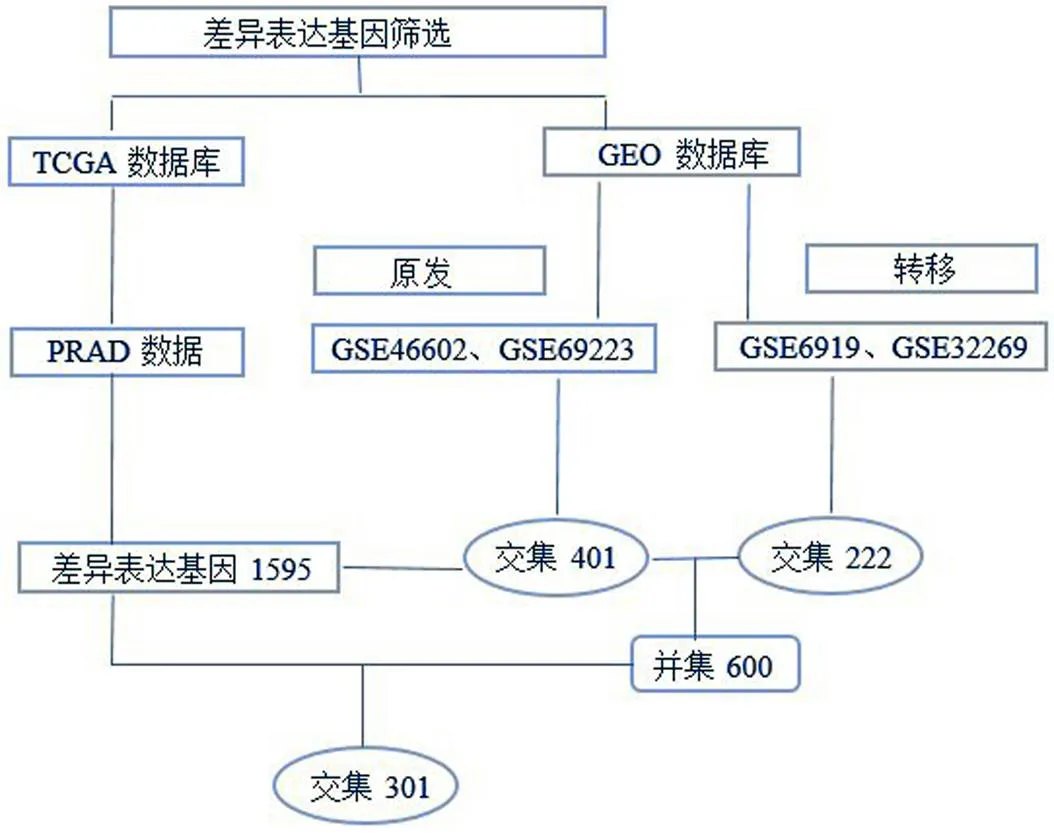
图 1 差异基因筛选流程图
Figure 1 Differentially expressed genes screening flow chart
对 TCGA和 GEO 分析结果取交集,最终得到差异表达基因 301 个,上调表达 102 个,下调表达 199 个,见图 2B。
2.2 GO 分析及 KEGG 分析
对 GEO 平台的原发和转移数据分别做 KEGG 分析,比对结果显示,两类数据均激活pI3K-Akt 信号通路和细胞-基质黏附信号通路。此外,转移肿瘤激活了癌症、cAMP、卵母细胞减数分裂、细胞外基质-受体相互作用和细胞周期信号通路;原发肿瘤激活了 AMP、Hippo、细胞色素 P450 对外源物质的代谢等通路。
对最终筛选的 301 个差异基因做 GO 分析和 KEGG 分析。GO 分析结果显示,生物过程(biological process,BP)主要围绕聚合酶 II 启动子转录的负性调节、细胞增殖的调控和细胞黏附及分化;细胞组分(cellular component,CC)主要表现在细胞质、质膜、胞浆和外泌体等结构;分子功能(molecular function,MF)主要体现在蛋白质间相互结合作用。KEGG 分析显示:激活了 pI3K-Akt 和 cGMP-PKG 两条重要信号通路同时还富集在癌症、microRNA 和 Hippo 信号通路中,具体见表 1。
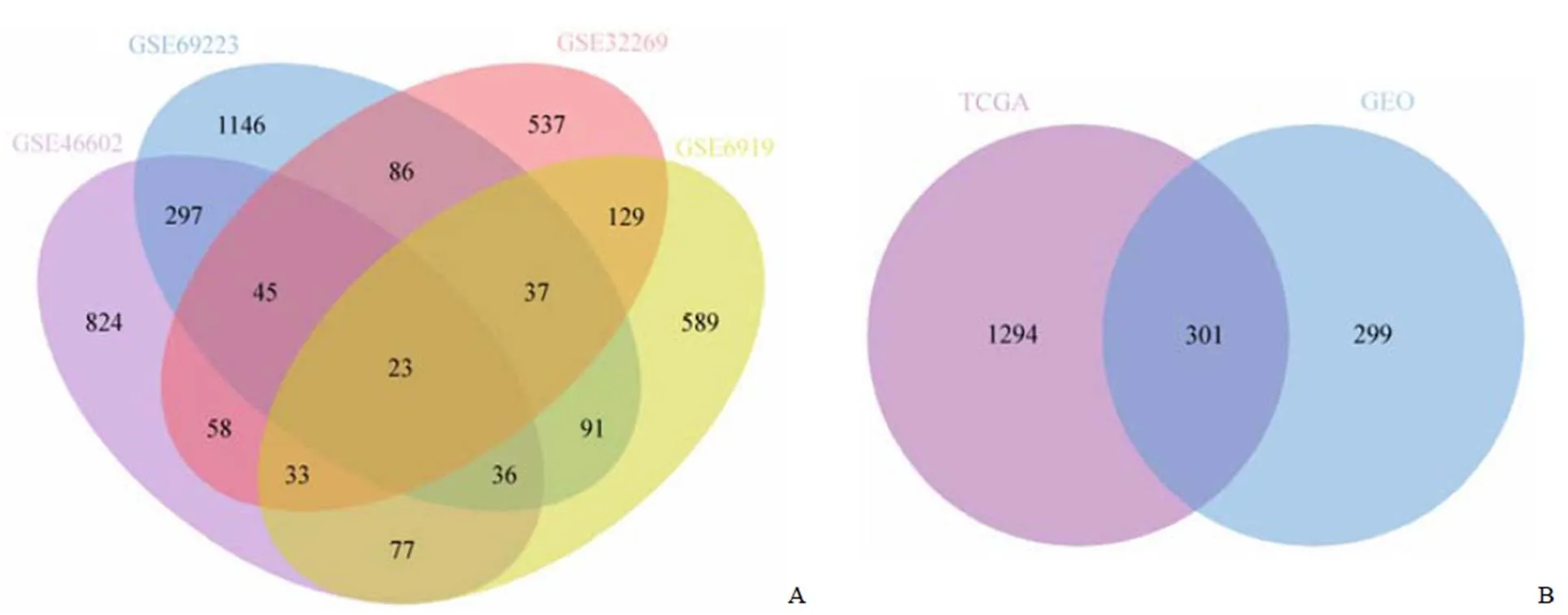
图 2 差异基因筛选韦恩图
Figure 2 Venn diagram of screening differentially expressed genes

表 1 差异基因的 GO 分析和 KEGG 分析
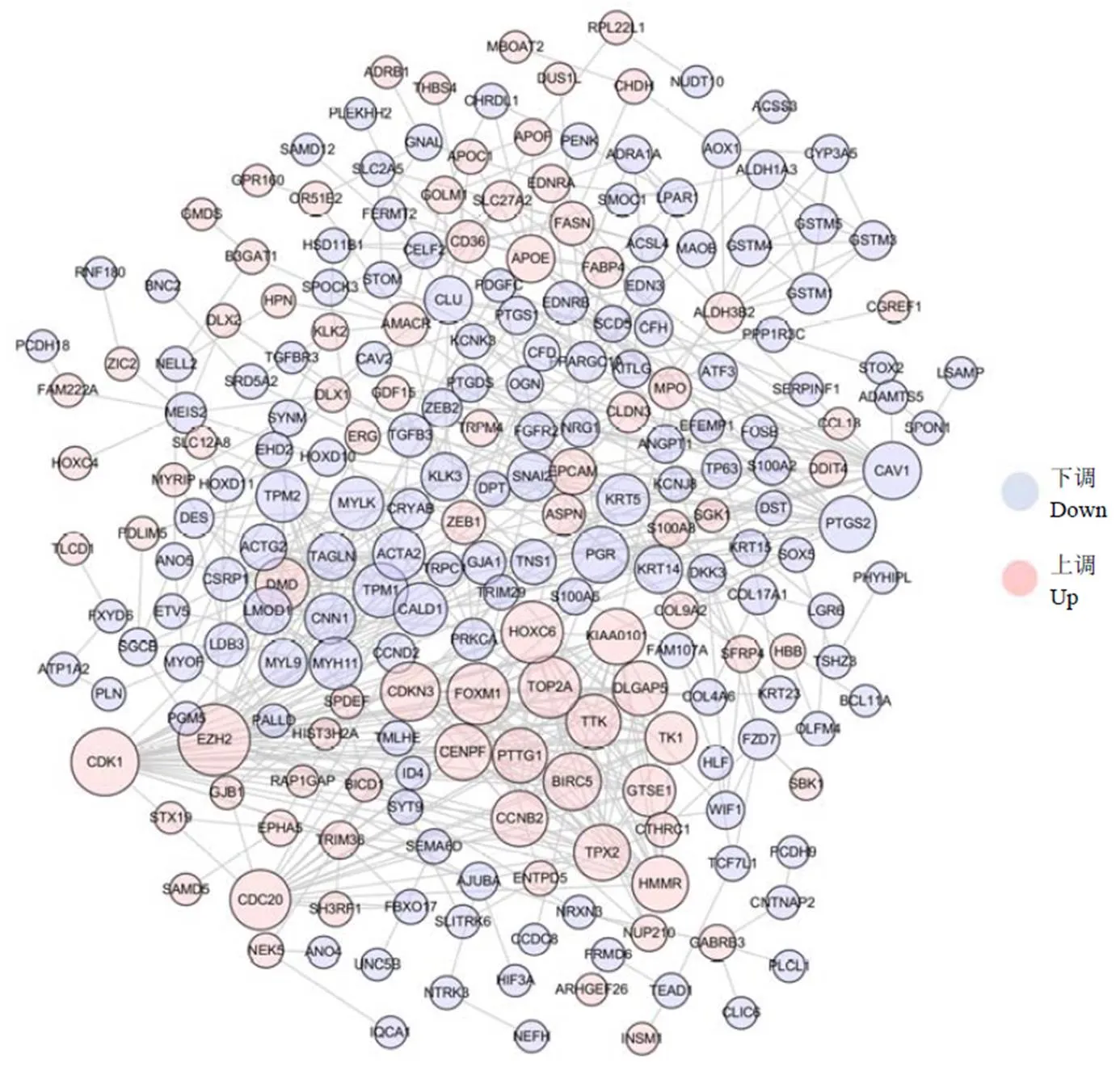
图 3 差异基因蛋白质互作网络
Figure 3 PPI network of differentially expressed genes
2.3 PPI 网络构建
将筛选出富集在生物过程中的基因导入到 String 数据库,分析得到 PPI 网络同时进行可视化分析,详见图 3。依据实验验证、共表达、连接度、结合分数等条件筛选 10 个核心蛋白作为网络关键节点,分别为 CDK1、EZH2、FOXM1、CDC20、TOP2A、CDKN3、GTSE1、HOXC6、BIRC5 和 CAV1。
2.4 关键节点基因甲基化情况
利用 TCGA 前列腺癌甲基化数据,验证关键节点蛋白的编码基因启动子区甲基化水平。筛选发现:过表达基因2、2、1 和6 存在启动子区低甲基化情况,低表达基因1 启动子区高甲基化,差异具有显著性,< 0.01,详见图 4。其他 5 个基因1、1、20、3 和5 甲基化水平与正常组相比较无统计学差异。
2.5 预后生存分析
利用 TCGA 临床数据评估上一步中筛选出的 5 个差异甲基化基因,考察其表达水平与生存时间的关系。K-M 法生存曲线显示,前列腺癌患者组织中2、1 和6 高表达组患者总生存率低于低表达组,差异具有显著性,< 0.05,详见图 5。
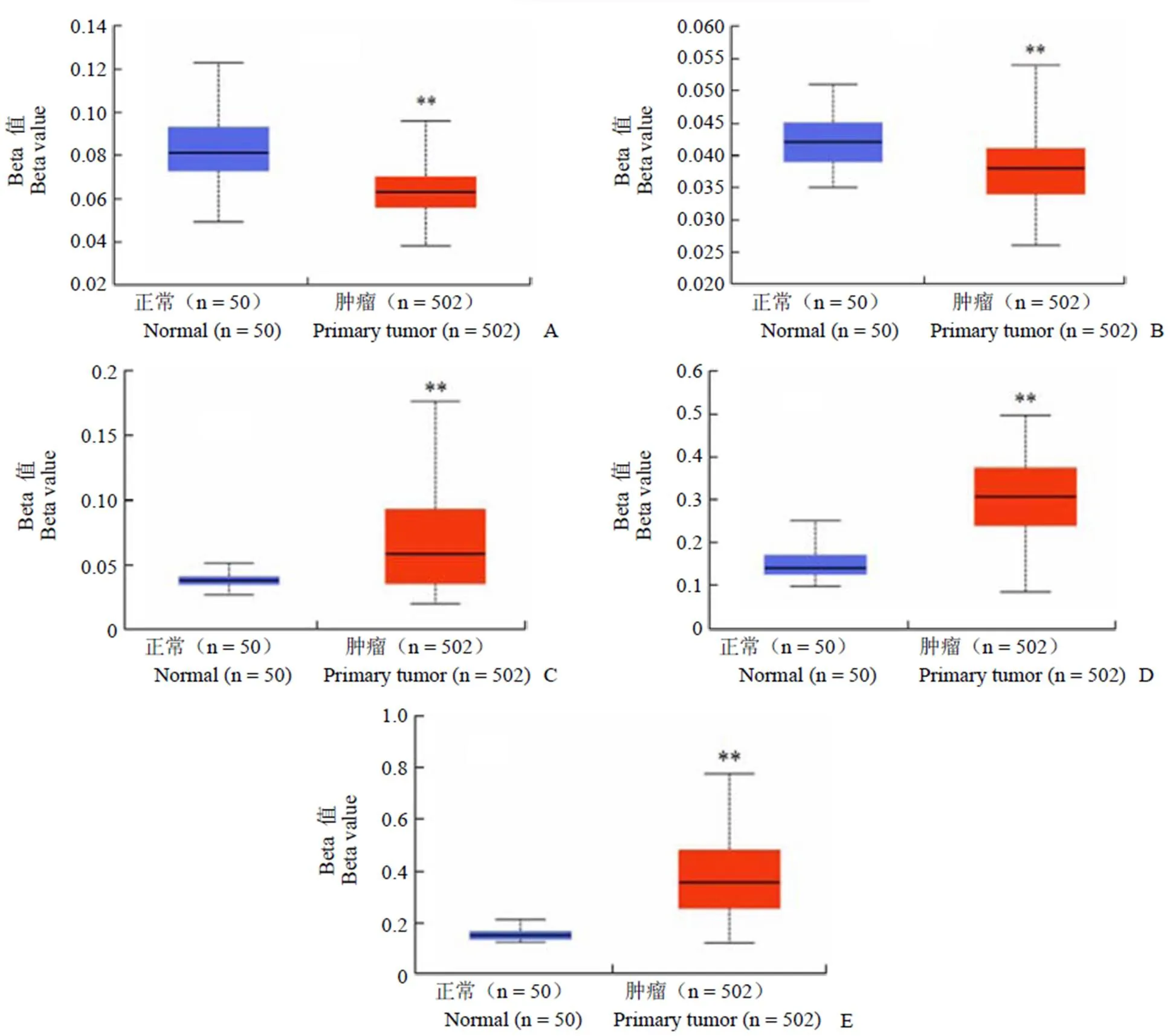
图 4 关键基因甲基化水平(A:EZH2,P = 6.84E-08;B:TOP2A,P = 2.49E-05;C:GTSE1,P = 1.62E-12;D:HOXC6,P < 1.0E-12;E:CAV1,P < 1.0E-12;**P < 0.01)
Figure 4 Methylation of key genes (A:2,= 6.84E-08; B:2,= 2.49E-05; C:1,= 1.62E-12; D:6,< 1.0E-12; E:1,< 1.0E-12;**< 0.01)
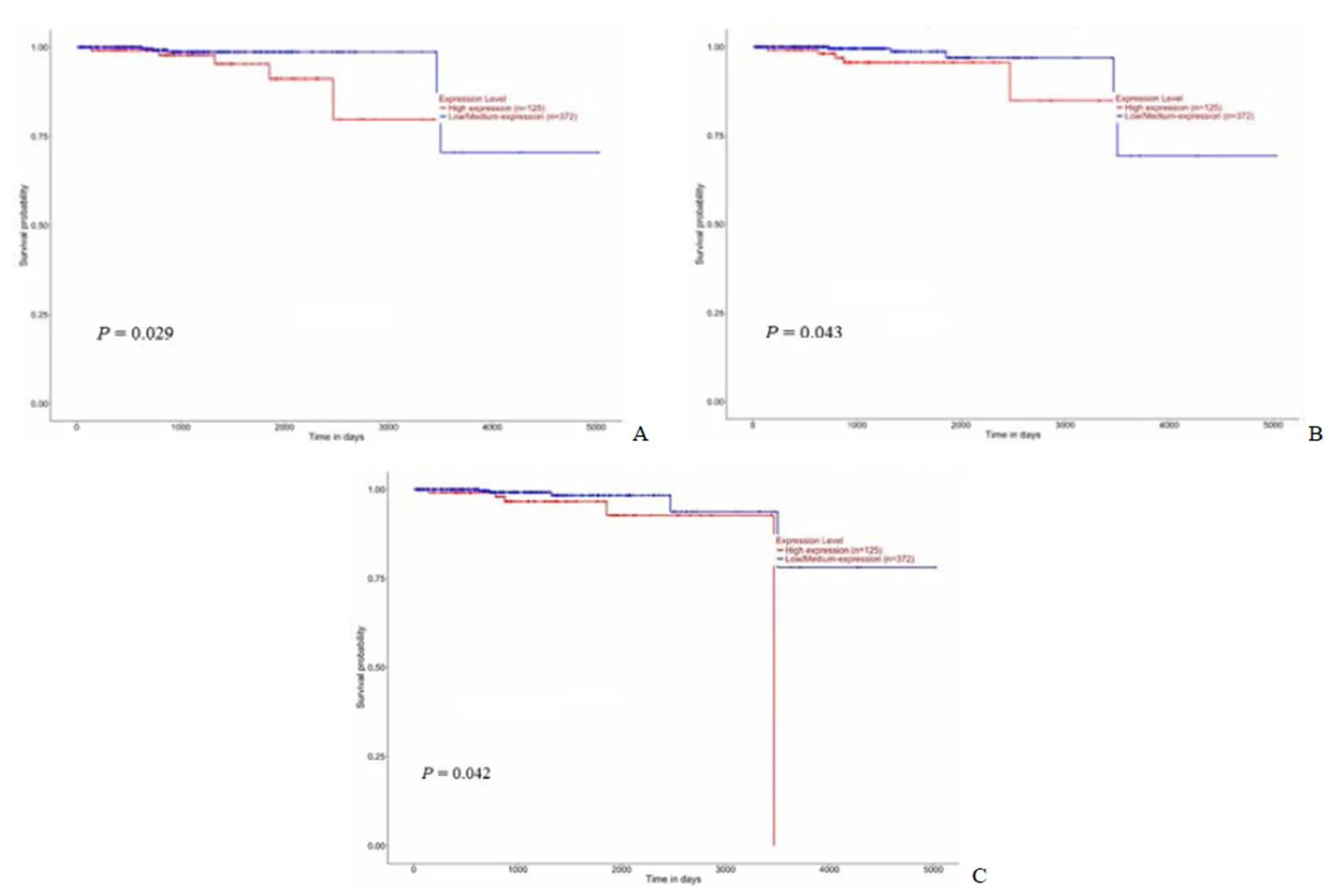
图 5 生存分析曲线(A:EZH2;B:GTSE1;C:HOXC6)
Figure 5 Survival analysis curve (A:2; B:1; C:6)
3 讨论
前列腺癌在欧美国家的病死率由于早期诊断和治疗而下降,但在发展国家却持续上升[7-9]。在中国平均每年新增 47000 例,不同于西方较为完善的主动监测体系和患者的重视程度[10],68% 的国人初次确诊时已经是晚期[11]。本研究通过不同平台数据的比较分析,筛选出差异甲基化的基因,为指导临床治疗提供依据。
研究选取了两类前列腺癌数据:一类是原发前列腺癌 GSE46602 和 GSE69223 数据,另一类是转移的前列腺癌 GSE6919 和 GSE32269 数据。分析发现前者差异基因有 401 个,后者有 222 个。虽然均是前列腺患者组织的差异表达基因,但是韦恩分析发现原发和转移的共有基因仅有 23 个。说明癌症转移激活了不同的基因和信号通路。Peng 等[12]研究表明,前列腺癌易发生骨转移,骨髓富含窦状血管,有利于癌细胞的迁移,文中 GSE32269 芯片即源于骨转移数据。本文研究发现,转移肿瘤激活了细胞外基质-受体相互作用信号通路,这可能与癌细胞通过分泌到细胞外基质的外泌体来调节前列腺癌细胞转移到骨组织初期的休眠活动[13]。外泌体是小囊泡结构,能携带蛋白质、mRNA、内源性 RNA 和 DNA,它从前列腺癌细胞释放到细胞外基质中[14],然后被骨髓基质细胞吸收,改变了原有的基因表达和信号通路,以促进癌细胞转移[15]。此外,转移的前列腺癌还激活了细胞周期信号通路,这可能会打破 G0/G1 细胞周期的阻滞,进入 G2/M 期,从而激活癌细胞的播散和促进癌症发展[16]。
DNA 甲基化作为一种已知的表观遗传模式在不同类别的癌症中均发生改变,目前的技术可以从患者的血液和尿液中分离纯化出 DNA 甲基化,并且安全无创,使其作为潜在的诊断或者预后判别的生物标记物[17]。本研究中筛选出的 10 个PPI 网络核心节点的编码基因中,有 5 个存在启动子区异常甲基化现象,过表达基因2、2、1 和6 存在启动子区低甲基化情况,低表达基因1 启动子区高甲基化。DNA 甲基化的异常改变包括促肿瘤发生的基因去甲基化,抑制肿瘤发展的基因甲基化[17]。后续的分析验证了2、1 和6 的高表达均与不良预后相关,而这 3 个基因均存在低甲基化现象。
Liu 等[18]研究发现,EZH2 通过与其启动子区域结合直接抑制多个下游靶点,包括 ADRB2、CDH1 和 miRNAs 等肿瘤抑制因子,从而发挥其致癌作用,并通过实验证实了阿司咪唑可以降低EZH2 蛋白水平,对晚期前列腺癌有效。Wu 等[19]研究发现,EZH2 介导了放射抗性机制,EZH2 表达的增加与放疗后前列腺癌复发有关,抑制 EZH2 的水平,会提高细胞对放射的敏感性。对 GTSE1 研究发现,其在肝癌、乳腺癌、胃癌、肺癌等多种癌症中表达增加,但在前列腺癌中还未引起研究者的关注。Wu 等[20]研究发现,肝癌组织中高表达 GTSE1,其与上皮向间质转化调控的迁移和侵袭密切相关,低表达GTSE1 可以增加化疗药物的敏感性。GTSE1 通过激活 AKT 途径来促进乳腺癌细胞增殖,它是 p53 的靶基因和负调控因子,通过将 p53 从细胞核转移到细胞质进行降解从而改变细胞周期,并下调 p53 表达[21]。Subhash 等[22]研究确定了 GTSE1 在顺铂诱导的细胞凋亡中起抑制作用,是胃癌细胞耐药的生物标志物。有研究显示,HOXC6 在前列腺癌组织中上调表达,并与不良预后密切相关[23-24],与本研究结论相符合。HOXC6 属于同源异型基因家族,是细胞分化和发育的重要调节因子,但其在前列腺癌中发挥作用的机制尚不十分清楚,推测其可能原因是 HOXC6 在前列腺癌中下游靶基因1、1 和存在下调表达,掩盖了其致癌效应[24]。综上所述,利用多平台数据筛选出与 3 个前列腺癌不良预后相关的差异甲基化表达基因:2、1 和6,为前列腺癌临床治疗和生物标记物的筛选提供了新的分子靶点。
[1] Baciarello G, Gizzi M, Fizazi K. Advancing therapies in metastatic castration-resistant prostate cancer. Expert Opin Pharmacother, 2018, 19(16):1797-1804.
[2] Siegel RL, Miller KD, Jemal A. Cancer statistics, 2018. CA Cancer J Clin, 2018, 68(1):7-30.
[3] Chen W. Cancer statistics: updated cancer burden in China. Chin J Cancer Res, 2015, 27(1):1.
[4] Ye D, Zhang W, Ma L, et al. Adjuvant hormone therapy after radical prostatectomy in high-risk localized and locally advanced prostate cancer: First multicenter, observational study in China. Chin J Cancer Res, 2019, 31(3):511-520.
[5] Tong Y, Song Y, Deng S. Combined analysis and validation for DNA methylation and gene expression profiles associated with prostate cancer. Cancer Cell Int, 2019, 19:50.
[6] Wang Q, Wang G, Liu C, et al. Prognostic value of CpG island methylator phenotype among hepatocellular carcinoma patients: A systematic review and meta-analysis. Int J Surg, 2018, 54(Pt A):92-99.
[7] Zhou CK, Littman AJ, Levine PH, et al. Male pattern baldness in relation to prostate cancer risks: an analysis in the VITamins and lifestyle (VITAL) cohort study. Prostate, 2015, 75(4):415-423.
[8] Zhou CK, Check DP, Lortet-Tieulent J, et al. Prostate cancer incidence in 43 populations worldwide: an analysis of time trends overall and by age group. Int J Cancer, 2016, 138(6):1388-1400.
[9] Zhou CK, Pfeiffer RM, Cleary SD, et al. Relationship between male pattern baldness and the risk of aggressive prostate cancer: an analysis of the Prostate, Lung, Colorectal, and Ovarian Cancer Screening Trial. J Clin Oncol, 2015, 33(5):419-425.
[10] Lancee M, Tikkinen K, de Reijke TM, et al. Guideline of guidelines: primary monotherapies for localised or locally advanced prostate cancer. BJU Int, 2018, 122(4):535-548.
[11] He D, Sun Z, Guo J, et al. A multicenter observational study of the real-world use of docetaxel for metastatic castration-resistant prostate cancer in China. Asia Pac J Clin Oncol, 2019, 15(3):144-150.
[12] Peng S, Yi Z, Liu M. Ailanthone: a new potential drug for castration-resistant prostate cancer. Chin J Cancer, 2017, 36(1):25.
[13] Widner DB, Park SH, Eber MR, et al. Interactions between disseminated tumor cells and bone marrow stromal cells regulate tumor dormancy. Curr Osteoporos Rep, 2018, 16(5):596-602.
[14] Zhang X. Interactions between cancer cells and bone microenvironment promote bone metastasis in prostate cancer. Cancer Commun (Lond), 2019, 39(1):76.
[15] Kobayashi A, Okuda H, Xing F, et al. Bone morphogenetic protein 7 in dormancy and metastasis of prostate cancer stem-like cells in bone. J Exp Med, 2011, 208(13):2641-2655.
[16] Sosa MS, Bragado P, Aguirre-Ghiso JA. Mechanisms of disseminated cancer cell dormancy: an awakening field. Nat Rev Cancer, 2014, 14(9):611-622.
[17] Kirby MK, Ramaker RC, Roberts BS, et al. Genome-wide DNA methylation measurements in prostate tissues uncovers novel prostate cancer diagnostic biomarkers and transcription factor binding patterns. BMC Cancer, 2017, 17(1):273.
[18] Liu Q, Wang G, Li Q, et al. Polycomb group proteins EZH2 and EED directly regulate androgen receptor in advanced prostate cancer. Int J Cancer, 2019, 145(2):415-426.
[19] Wu X, Scott H, Carlsson SV, et al. Increased EZH2 expression in prostate cancer is associated with metastatic recurrence following external beam radiotherapy. Prostate, 2019, 79(10):1079-1089.
[20] Wu X, Wang H, Lian Y, et al. GTSE1 promotes cell migration and invasion by regulating EMT in hepatocellular carcinoma and is associated with poor prognosis. Sci Rep, 2017, 7(1):5129.
[21] Lin F, Xie YJ, Zhang XK, et al. GTSE1 is involved in breast cancer progression in p53 mutation-dependent manner. J Exp Clin Cancer Res, 2019, 38(1):152.
[22] Subhash VV, Tan SH, Tan WL, et al. GTSE1 expression represses apoptotic signaling and confers cisplatin resistance in gastric cancer cells. BMC Cancer, 2015, 15: 550.
[23] Zhou J, Yang X, Song P, et al. HOXC6 in the prognosis of prostate cancer. Artif Cells Nanomed Biotechnol, 2019, 47(1):2715-2720.
[24] Vinarskaja A, Yamanaka M, Ingenwerth M, et al. DNA Methylation and the HOXC6 Paradox in Prostate Cancer. Cancers (Basel), 2011, 3(4):3714-3725.
Screening of methylation genes with poor prognosis in prostate cancer
LI You, LIU Dong-hui, ZHANG Guo-yong
Department of Urology, Jinqiu Hospital of Liaoning Province, Shenyang 110016, China
The aim of this study was to identify differentially methylated genes associated with poor prognosis of prostate cancer and provide data support for finding therapeutic targets.
In this experiment, four prostate cancer gene chip data sets of GSE46602, GSE69223, GSE6919 and GSE32269 from GEO database were used to screen differential genes, and compared with TCGA data. The functional enrichment analysis was carried out by David database. The PPI network between gene-encoded proteins was constructed by using String database, and then analyzed and visualized by Cytoscape software. Through the methylation data of TCGA, the methylation level of the gene was investigated, and the clinical data were used to observe the effect of differential expression on the prognosis.
600 differential genes were screened by GEO database. After overlapping analysis with TCGA data, 301 differential genes were obtained. These genes activate pI3K-Akt and cGMP-PKG signaling pathways. After building the PPI network, 10 key nodes
of the network were analyzed. Further differential methylation analysis of these core genes showed that the over-expressed genes2,2,1 and6 had low methylation in the promoter region, while the low-expressed gene1 had hypermethylation in the promoter region. Over-expression of2,1 and6 genes is associated with poor prognosis of prostate cancer.
Prostate cancer data from different platforms are selected, and the differential methylation state of genes related to poor prognosis is analyzed. The finding may provide potential molecular targets for the treatment of prostate cancer.
Prostate cancer; Methylation; Bioinformatics analysis
LI You, Email: liyoutgyx@126.com
李猷,Email:liyoutgyx@126.com
10.3969/j.issn.1673-713X.2020.04.013
2020-02-11
