肿瘤基因突变检测的标准化研究
2020-08-14张文新黄传峰孙楠高飞胡泽斌孙晶黄杰曲守方
张文新,黄传峰,孙楠,高飞,胡泽斌,孙晶,黄杰,曲守方
·论著·
肿瘤基因突变检测的标准化研究
张文新*,黄传峰*,孙楠,高飞,胡泽斌,孙晶,黄杰,曲守方
100050 北京,中国食品药品检定研究院非传染病诊断试剂室(张文新、孙楠、高飞、胡泽斌、孙晶、黄杰、曲守方);理化检测室(黄传峰)
探索制备和等肿瘤基因突变参考品的方法,用于评价肿瘤基因突变检测试剂盒(二代测序法)的性能。
收集和等基因突变的组织切片和细胞系,提取样本的 DNA 和 RNA。采用数字 PCR 方法对样本对应的基因突变进行验证和定量,确定阳性参考品及其突变比例;然后将阳性参考品和野生型样本混合,稀释至突变率为 5% 和含 100 个融合 RNA 拷贝数的样本,经数字 PCR 定量后,作为检测限参考品。采用肿瘤基因突变检测试剂盒(二代测序法)进行准确性和检测限项目检测。
数字 PCR 的结果表明成功制备了阳性参考品和检测限参考品,并确定其突变比例;制备的参考品能被肿瘤基因突变检测试剂盒(二代测序法)正确检出。
建立了制备肿瘤基因突变参考品的方法,制备的参考品可以用于评价肿瘤基因突变检测试剂盒(二代测序法)的性能。
靶向药物; 分子靶向治疗; 基因突变; 下一代测序; 标准物质研究
肿瘤驱动基因3等是细胞分化、增殖、凋亡、迁移等过程中关键的基因,这些基因突变会持续激活相关信号通路,导致肿瘤的发生和发展[1-2]。随着这些基因被不断发现,针对特定分子靶点的药物研究进展迅速,包括表皮生长因子酪氨酸激酶抑制剂(EGFR-TKI)和棘皮动物微管相关样蛋白-4-间变性淋巴瘤激酶(EML4-ALK)融合基因抑制剂(TKI)等[3]。检测这些相关基因是否发生突变或者融合,确定肿瘤患者的药物敏感性,可以为临床医生选择个体化靶向药物治疗提供参考。
传统的肿瘤突变检测方法包括免疫组织化学、荧光原位杂交、荧光聚合酶链反应法和 sanger 测序法等[4-6]。这些肿瘤突变检测方法的局限性在于每次只能检测一种基因,而且根据检测基因数量的增多需要更多的活检组织。随着精准医疗和分子诊断技术的发展,下一代测序技术(next generation sequencing,NGS)在肿瘤基因检测方面得到广泛的应用,可以在同一份活检组织中一次检测多个基因突变,包括点突变、插入、缺失、DNA 拷贝数改变及重排(融合)等多种突变形式[7-9]。本研究拟探索建立一种制备肿瘤基因突变参考品的方法,制备的参考品能够用于评价基于二代测序技术平台的肿瘤基因突变检测试剂盒的性能。
1 材料与方法
1.1 材料
1.1.1 细胞株购自美国菌种保藏中心(ATCC)。选取石蜡包埋组织样本,包括不同肿瘤基因的突变类型。
1.1.2 试剂 AllPrep DNA/RNA FFPE Kit 购自美国 Qiagen 公司;DNA/RNA 共提取试剂盒购自北京天根生物技术有限责任公司;Qubit®dsDNA HS Assay Kit、Qubit®RNA HS Assay Kit、QuantStudio™ 3D Digital PCR Master Mix v2 和TaqMan®Gene Expression Assay 购自美国 Life Technologies 公司;One-Step RT ddPCR Advanced Kit for Probes 购自美国 Bio-Rad 公司;人、、、3、、1 基因突变检测试剂盒(半导体测序法)购自天津诺禾致源生物信息科技有限公司。
1.2 方法
1.2.1 样本 DNA 和 RNA 的提取 选择石蜡包埋组织样本(FFPE)蜡卷片放入 1.5 ml 离心管中,进行脱蜡处理后,按照 AllPrep DNA/RNA FFPE Kit 说明书提取样本的 DNA 和 RNA。将野生型细胞株、正常人白细胞和带有肿瘤基因突变的细胞株,按照 DNA/RNA 共提取试剂盒说明书进行DNA和RNA 提取。使用 Qubit®3.0 进行样本DNA/RNA 定量。
1.2.2 阳性参考品突变比例确定 按照 3D 数字 PCR 检测流程,配制反应体系:含 10 ng DNA 样本 6.75 μl,2× QuantStudio™ 3D Digital PCR Master Mix 0.75 μl,20× Taqman ASSAY(primer/probe mix)0.75 μl,加双蒸水补足至 15 μl。取 14.5 μl 配置好的体系加入到加样孔,反应液均匀涂抹到芯片上。将芯片放置在平板 PCR 仪上,按照如下程序进行 PCR 反应:96 ℃ 10 min;56 ℃ 2 min,98 ℃ 30 s,39 个循环;56 ℃ 2 min,10 ℃保存。将芯片放入芯片读取仪进行读取。使用 QuantStudio 3D Analysis Suite 软件分析结果。将突变阳性的样本 DNA 作为待测样本,检测肿瘤基因的原始突变比例,将其作为阳性参考品。根据原始突变比例,将阳性参考品 DNA 和野生型 DNA 进行混合,制备 5% 突变比例的检测限参考品,然后经 3D 数字 PCR 验证。
按照伯乐数字 PCR 检测流程 One-Step RT ddPCR Advanced Kit for Probes 试剂盒配制反应体系:含 30 ng RNA 样本 11 μl,Super Mix 5 μl,Reverse transcriptase 2 μl,300 mmol/L DTT 1 μl,20 × Taqman assay(probe mix)1 μl。转移 20 μl 反应体系至 DG8 cartridge,放入微滴生成仪生成微滴,转移到 96 孔板,用 PX1 热封仪进行封膜。封好的 96 孔板放入 PCR 仪中进行扩增,反应程序如下:45 ℃ 60 min;95 ℃ 10 min;95 ℃ 30 s,60 ℃ 1 min,40 个循环;98 ℃ 10 min。将 96 孔板放入 QX200 Droplet Reader 仪器中,使用 QuantaSoft 软件进行分析。将融合突变阳性的样本 RNA 作为待测样本,检测后获得融合 RNA 拷贝数,将其作为阳性参考品。将阳性参考品 RNA 和野生型 RNA 混合,制备每 30 ng RNA 含 100 个融合 RNA 拷贝数的检测限参考品,然后经伯乐数字 PCR 验证。
1.2.3 文库的构建和定量 将制备的阳性参考品和检测限参考品,按照人、、、3、、1 基因突变检测试剂盒(半导体测序法)的说明书构建文库,步骤包括:RNA 样本的反转录,目的片段的 PCR 扩增,引物的消化,加 P1 接头和特异性接头以及文库的纯化。
然后采用荧光定量 PCR 仪将文库进行定量。根据文库浓度,分别将 DNA 文库和 RNA 文库稀释至 100 pmol/L 和 50 pmol/L,然后将所有的DNA 文库和 RNA 文库等体积混合,再将 4.8 μl 混合后的 DNA 文库和 2.4 μl 混合后的 RNA 文库混合后,进行模板制备。
1.2.4 模板的制备和富集 文库混合后进行乳液 PCR 扩增。将配制好的乳液 PCR 反应液和模板反应油注入到模板制备过滤器,装入 Ion OneTouchTM2仪器上选择程序进行反应,步骤包括:通过反应器形成油包水型,PCR 扩增反应,离心回收制备好的模板。然后用 Ion OneTouchTMES 的磁珠技术进行模板富集。
1.2.5 测序 将质控模板加入到富集的模板溶液中,退火,将混合液加入到测序芯片,加测序酶。将芯片上机进行测序。用癌症基因数据处理软件进行数据分析。
2 结果
2.1 阳性参考品和检测限参考品验证结果
肿瘤组织来源的 DNA 样本 PD1 在 3D 数字 PCR 的结果为基因 18 外显子突变阳性,且突变比例为10.1%,见图 1A。将其稀释至理论突变比例为 5% 的 DNA 样本 LD1,结果表明也为突变阳性,而且突变比例为5.9%,见图 1B。3D 数字 PCR 的验证结果表明,成功制备了 DNA 样本的阳性参考品 PD1 和检测限参考品 LD1。
细胞株来源的 RNA 样本 PR1 在伯乐数字 PCR 的结果为 EML4-ALK 融合突变阳性,且融合突变浓度为 768 拷贝/μl,见图 2A。将其稀释至理论融合突变浓度为 5 拷贝/μl 的样本 LR1,结果表明也为融合突变阳性,且融合突变浓度为 4.7 拷贝/μl,即 20 μl 反应体系中含有 94 个拷贝的融合 RNA,见图 2B。伯乐数字 PCR 的验证结果表明成功建立了 RNA 样本的阳性参考品 PR1 和检测限参考品 LR1。
2.2 测序结果
使用人、、、3、、1 基因突变检测试剂盒(半导体测序法)进行检测,利用癌症基因数据处理软件进行数据分析,得到了样本的变异信息。阳性参考品和检测限参考品的肿瘤基因突变类型的结果均与数字 PCR 的结果一致。测序结果显示,阳性参考品 PD1 在 7 号染色体的 55241708 位置处发生了鸟嘌呤 G 突变为胞嘧啶 C,支持突变的 reads 数为 474,经过与人类基因组(hg19)比对,结果是基因 18 外显子的 G719A 突变;阳性参考品 PR1 比对到参考序列上的 reads 数是 1520542,支持突变的 reads 数是 303249,经过与参考序列比对,结果是 EML4-ALK E13A20 融合形式;检测限参考品的测序结果也符合阳性参考品的突变类型(表 1 和表 2)。
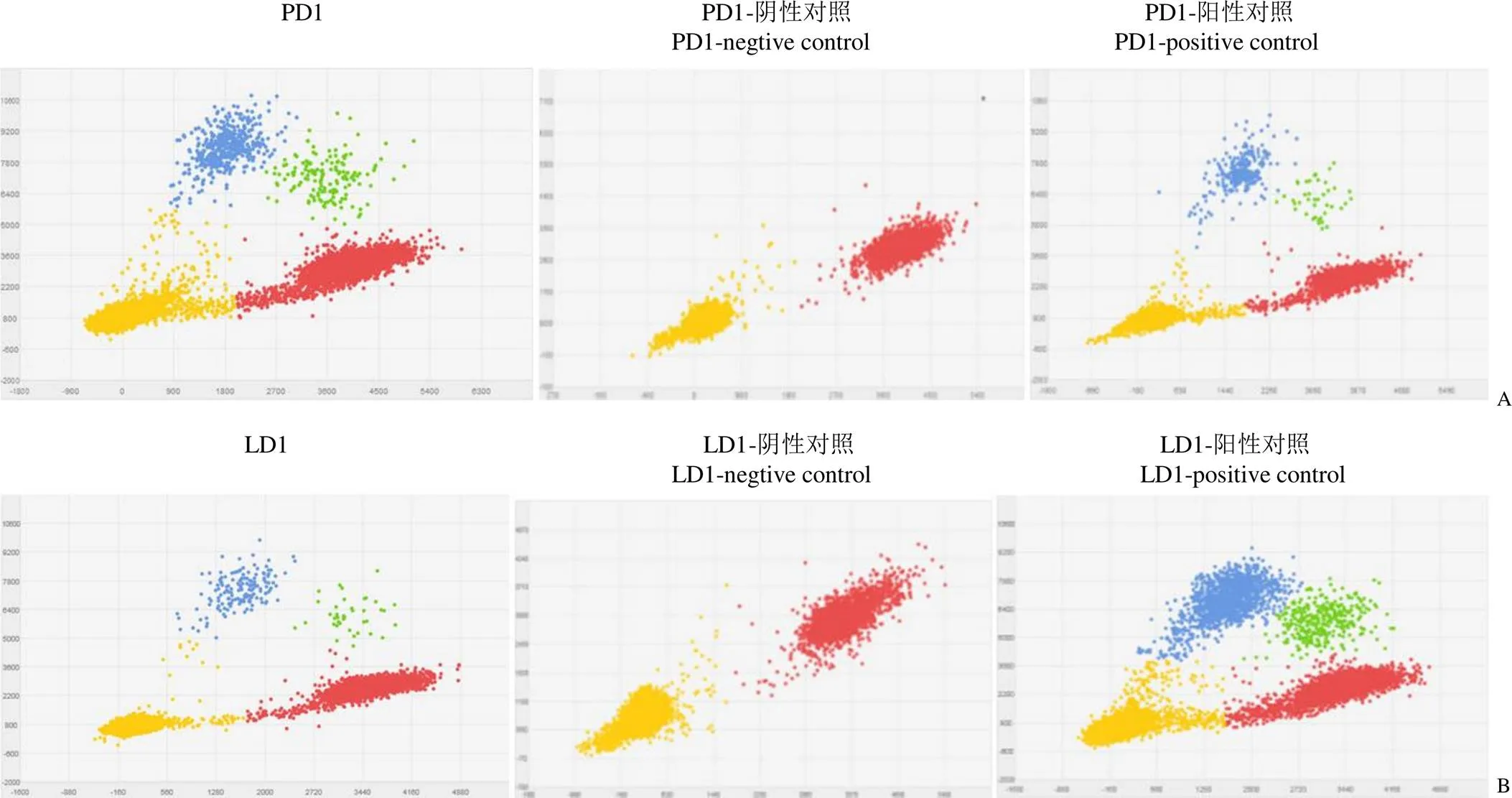
图 1 DNA 阳性参考品和检测限参考品的 3D 数字 PCR 结果(A:阳性参考品 PD1;B:检测限参考品 LD1)
Figure 1 The results of DNA positive and limit reference material by QuantStudio 3D digital PCR (A:PD1 positive reference;B:LD1 limit detection reference)
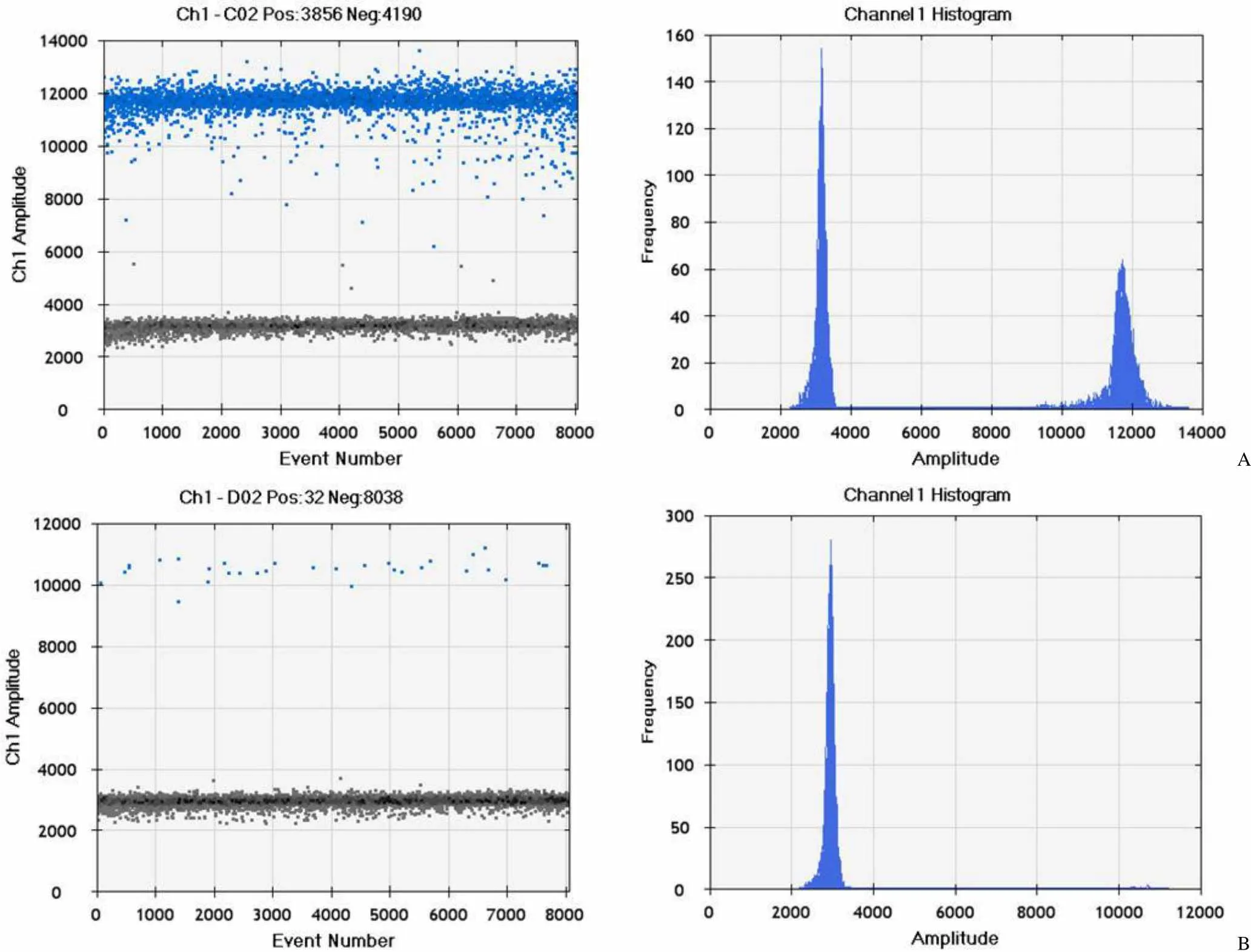
A B
Figure 2 The results of RNA positive and limit reference material by Bio-Rad droplet digital PCR (A: PR1 positive reference;B: LR1 limit detection reference)
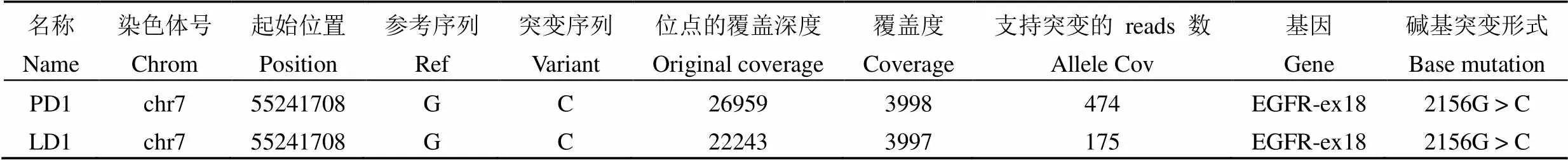
表 1 DNA 样本的阳性参考品和检测限参考品的测序结果

表 2 RNA 样本的阳性参考品和检测限参考品的测序结果
3 讨论
随着精准医学的发展,针对新的驱动基因的靶向治疗药物迅速发展。美国食品药品监督管理局(FDA)已经批准了表皮生长因子受体酪氨酸激酶抑制剂——吉非替尼和奥西替尼等和间变性淋巴瘤激酶酪氨酸激酶抑制剂——克唑替尼等[10-11]。这些靶向治疗药物均对携带某些特定基因变异的人群有效,因此其分子肿瘤伴随诊断具有十分重要意义。
是最常见的驱动基因,在非小细胞肺癌(non-small cell lung cancer,NSCLC)患者中高度表达。2004 年,Lynch 等[12]首次提出,TKI 对不同 NSCLC 的治疗效果的差异是由于 EGFR 外显子的突变状态不同。EGFR 19 外显子缺失突变(以 delE746-A750 为主要代表)及 21 外显子 L858R 点突变的非小细胞肺癌患者对 EGFR-TKI 治疗敏感。EML4-ALK 基因融合是新发现的致癌驱动基因[13],已从肺癌患者中发现了 10 多种不同的融合基因型,比例占到肺癌人群的 3% ~ 7%。美国国立综合癌症网络(NCCN)非小细胞肺癌(NSCLC)临床实践指南(2014 版)提出,对于腺癌、大细胞癌以及未分类的 NSCLC,常规进行表皮生长因子受体(EGFR)基因突变和 EMIA-ALK 融合基因检查,若 EGFR 突变阳性则选择 EGFR TKI 治疗如厄洛替尼,EML4-ALK 融合基因阳性者,可给予 ALK 小分子抑制剂克唑替尼治疗。因此提高肿瘤基因突变检测的敏感性和准确性,有助于改善 NSCLC 患者治疗。
与传统的 FISH 及荧光 PCR 法相比,高通量测序具有较高的灵敏度和准确度。国内已经开展研发对肺癌、乳腺癌、结直肠癌等恶性肿瘤的基于 NGS 技术的体外诊断试剂盒。体外诊断试剂产品质量的提高有赖于国家标准物质水平的提高和完善[14-15]。为了配合新的检测技术,应该建立基于二代测序技术检测的肿瘤基因突变国家参考品。建立肿瘤基因突变参考品的关键在于确定样本的肿瘤基因突变比例。数字 PCR 技术具有极高的敏感性,是一种核酸分子绝对定量技术。相比于传统的荧光定量 PCR 技术,数字 PCR 技术能够直接读出 DNA 和 RNA 分子的拷贝数。从检测突变频率的角度考虑,数字 PCR 技术能够检测到万分之一的基因突变。本研究探索了建立肿瘤基因突变参考品的方法,选用 3D 数字 PCR 和伯乐数字 PCR 对发生突变或者融合突变的组织样本和细胞株进行验证,确定肿瘤基因的突变比例,制备了阳性参考品和检测限参考品。研究结果表明肿瘤基因突变参考品的建立方法是可靠的,制备的参考品评价了基于 NGS 平台的肿瘤基因突变检测试剂盒的性能,完成了该类试剂盒的标准化研究工作,也为国家参考品的建立提供了技术指导。
[1] Zhu QG, Zhang SM, Ding XX, et al. Driver genes in non-small cell lung cancer: characteristics, detection methods, and targeted therapies. Oncotarget, 2017, 8(34):57680-57692.
[2] Su N, Zhang XL, Huang HY, et al. Driver gene status and first line treatment of advanced lung adenocarcinoma: a single centric real-world study. J Pract Med, 2019, 35(4):537-540. (in Chinese)
苏宁, 张贤兰, 黄惠怡, 等. 晚期肺腺癌驱动基因分布及其一线治疗现状——单中心真实世界研究. 实用医学杂志, 2019, 35(4):537- 540.
[3] Li WQ, Bai RL, Cui JW. Targeted therapies for driver gene mutation-positive lung cancer patients with brain metastasis. Chin
J Lung Cancer, 2019, 22(11):719-726. (in Chinese)
李汶倩, 白日兰, 崔久嵬. 驱动基因阳性肺癌脑转移患者的靶向治疗进展. 中国肺癌杂志, 2019, 22(11):719-726.
[4] Fang F, Qiao XB, Yang L, et al. Diagnosis of lung biopsy employing the 2015 WHO criteria and detection of related oncogenic driver mutations. Chin J Pathol, 2019, 48(4):270-275. (in Chinese)
方芳, 乔旭柏, 杨丽, 等. 用 2015 WHO肺肿瘤分类诊断肺穿刺活检标本及相关肿瘤驱动基因的突变检测. 中华病理学杂志, 2019, 48(4):270-275.
[5] Su F, Zheng K, Fu YY, et al. Influence of different therapies on EGFR mutants by circulating cell-free DNA of lung adenocarcinoma and prognosis. Chin J Lung Cancer, 2018, 21(5):389-396. (in Chinese)
苏飞, 郑可, 付祎云, 等. 不同治疗对肺癌患者血液循环游离 DNA 检测 EGFR 基因突变的影响及预后分析. 中国肺癌杂志, 2018, 21(5):389-396.
[6] Zhang TT, Li JL. Driven gene in patients with lung squamous cell carcinoma: analysis of clinicopathologic characteristics and prognosis. Chin J Lung Cancer, 2016, 19(10):648-652. (in Chinese)
张童童, 李峻岭. 存在驱动基因突变肺鳞癌患者的临床病理特征及预后分析. 中国肺癌杂志, 2016, 19(10):648-652.
[7] Chen LF, Chen XY, Lin J, et al. The characteristics of lung adenocarcinoma driver gene variants detected by high-throughput sequencing and quantitative fluorescence PCR. Chin J Pathol, 2019, 48(10):772-778. (in Chinese)
陈灵锋, 陈小岩, 林洁, 等. 应用高通量测序技术与荧光PCR 技术检测肺腺癌驱动基因变异分析. 中华病理学杂志, 2019, 48(10): 772-778.
[8] Liu XY, Wu XY, Shao Q, et al. Analysis of driving gene mutations in non-small cell lung cancer using UMI-based next-generation sequencing. J Mol Diag Ther, 2019, 11(6):468-473. (in Chinese)
刘小云, 吴小延, 邵琼, 等. 基于分子标签二代测序技术的非小细胞肺癌驱动基因变异分析. 分子诊断与治疗杂志, 2019, 11(6):468- 473.
[9] Paweletz CP, Sacher AG, Raymond CK, et al. Bias-corrected targeted next-generation sequencing for rapid, multiplexed detection of actionable alterations in cell-free DNA from advanced lung cancer patients. Clin Cancer Res, 2016, 22(4):915-922.
[10] Yang WJ, Yang GW. Choice of first-line treatment strategy for advanced non-small cell lung cance. Chin Clin Oncol, 2019, 24(8): 762-766. (in Chinese)
杨雯靖, 杨国旺. 晚期非小细胞肺癌一线治疗策略的选择. 临床肿瘤学杂志, 2019, 24(8):762-766.
[11] Xu AR, Ma W. The targeted therapy progress of advanced non-small cell lung cancer with ALK fusion gene positive. J Mod Oncol, 2019, 27(3):529-533. (in Chinese)
徐爱茹, 马为. ALK 融合基因阳性的晚期非小细胞肺癌靶向治疗进展. 现代肿瘤医学, 2019, 27(3):529-533.
[12] Lynch TJ, Bell DW, Sordella R, et al. Activating mutations in the epidermal growth factor receptor underlying responsiveness of non-small-cell lung can-cer to gef tinib. N Engl J Med, 2004, 350(21): 2129-2139.
[13] Gao QQ, Jiang XL, Huang C. Clinical advanced in early-stage ALK-positive non-small cell lung cancer patients. Chin J Lung Cancer, 2017, 20(2):124-129. (in Chinese)
高琼琼, 蒋湘俐, 黄纯. 早期非小细胞肺癌 ALK 阳性患者的临床研究进展. 中国肺癌杂志, 2017, 20(2):124-129.
[14] Lu S, Ji WX, Yu YF. Updated interpretation of clinical practice guidelines for NCCN non-small cell lung cancer (2014). Chin
J Postgrad Med, 2015, 38(5):318-320. (in Chinese)
陆舜, 纪文翔, 虞永峰. 2014 版《NCCN 非小细胞肺癌临床实践指南》更新解读. 中国医师进修杂志, 2015, 38(5):318-320.
[15] Qu SF, Yu T, Guo LP, et al. The establishment of control materials for variant 1 and variant 3 of human EMLA-ALK fusion gene. Chin
J Pharm Anal, 2015, 35(3):500-505. (in Chinese)
曲守方, 于婷, 郭李平, 等. EML4-ALK 融合基因变体 1 和变体 3 质控品的建立. 药物分析杂志, 2015, 35(3):500-505.
[16] Yang Z, Huang J, Yu T, et al. Status and countermeasures of national standard substances for in vitro diagnostic reagent in China. Chin J Biol, 2015, 28(7):765-771. (in Chinese)
杨振, 黄杰, 于婷, 等. 我国体外诊断试剂国家标准物质现状及对策分析. 中国生物制品学杂志, 2015, 28(7):765-771.
·学术活动预告·
中国医药生物技术协会移植技术分会(以下简称“移植技术分会”)于 2019 年 10 月 27 日在武汉成立,华中科技大学同济医学院附属医院器官移植研究所陈刚教授为第一届主任委员,现有 72 名委员。移植技术分会是一个为临床医生、科研人员和生产企业提供多方交流与合作,注重产学研结合、促进新技术临床转化的平台。各位委员已经开始规划通过高质量临床研究,促进移植相关检测及治疗技术的最优化和标准化,推动新技术和新产品应用转化,制订或完善行业规范和标准,以更好地为患者服务。
在我国抗击新冠疫情取得阶段性成果之际,为了加强技术与临床的交流,推动移植技术的发展和完善临床诊疗方案,由中国医药生物技术协会移植技术分会主办,国药集团东风总医院承办的“第一届中国移植技术高峰论坛暨 2020 年中国医药生物技术协会移植技术分会工作会议”将于 2020 年 9 月 25 – 26 日在湖北省十堰市召开。本次会议的主题是“移植延续生命技术成就梦想”,会议将围绕器官移植临床与技术前沿,邀请国内外知名专家参与大会学术交流。
会议详情及报名请登录中国医药生物技术协会网站https://www.bagevent.com/event/6628908 查阅。
Study on the standardization of tumor gene mutation detection
ZHANG Wen-xin, HUANG Chuan-feng,SUNNan,GAO Fei, HU Ze-bin, SUN Jing, HUANG Jie, QU Shou-fang
Division of Diagnostic for Non-infectious Disease (ZHANG Wen-xin, SUN Nan, GAO Fei, HU Ze-bin, SUN Jing, HUANG Jie, QU Shou-fang), Division of Physical and Chemical Testing (HUANG Chuan-feng), National Institutes for Food and Drug Control, Beijing 100050, China
To explore the method for preparing the reference material for human tumor gene such asandfusion gene in order to evaluate the performance of tumor gene mutation detection kit (next-generation sequencing, NGS).
Different tissue sections and cell lines including these gene mutations were collected and DNA and RNA of samples were extracted. The genetic mutation corresponding to the sample was verified and quantified by digital PCR method to determine the positive control material and its mutation frequency. Then positive reference material of DNA or RNA and wild type samples were mixed to dilute them to a sample with a mutation rate of 5% and a number of 100 fusion RNA copies after being quantified by digital PCR as a test limit reference. The accuracy and detection limit was carried out by using tumor gene mutation detection kit (NGS).
The results of digital PCR method showed that positive and limit reference material were successfully established, and their mutation frequency were determined. The reference materialcould be correctly detected by kit (NGS).
The preparation method ofreference material is establishedand it could be used for evaluating the performance of tumor gene mutation detection kit (NGS).
Targeted drug; Molecular targeted therapy; Gene mutation; Next generation sequencing; Research on reference material
QU Shou-fang, Email: qushoufang@126.com; HUANG Jie, Email: jhuang5522@126.com
10.3969/j.issn.1673-713X.2020.04.010
曲守方,Email:qushoufang@126.com;黄杰,Email:jhuang5522@126.com
2020-02-12
*同为第一作者
