利用生物信息学分析鉴定鼻咽癌转移的信号通路及核心基因
2019-12-23宫伟李涛
宫伟,李涛
论著
利用生物信息学分析鉴定鼻咽癌转移的信号通路及核心基因
宫伟,李涛
利用基因芯片技术和生物信息学分析方法,筛选出鼻咽癌转移相关的核心基因和相关信号通路,为寻找鼻咽癌转移早期诊断和靶向治疗潜在标志物提供依据。
GSE103611 的表达芯片从 Gene Expression Omnibus(GEO)数据库中下载,该数据库中包含 48 个样本,包括 24 个原发鼻咽癌样本和 24 个放疗后转移的鼻咽癌样本。整理微阵列数据集获得差异表达基因(DEGs)与本课题组前期构建的相对于 CNE-2 侵袭转移能力更强的 CNE-2SI 细胞对比芯片差异基因进行比对获得共同差异基因。利用基因本体论(GO)和京都百科全书基因和基因组数据库(KEGG)对共同差异基因进行富集并利用 DAVIDE 在线进行分析。共同差异基因的蛋白质互作(PPI)网络由 STRING 数据库构建。Hub 基因分析通过 Cytoscape 软件中的 cytoHubba 插件制作。关键基因的生存分析通过 Kaplan Meier-plotter 数据库分析获得。
GSE103611 数据集中共鉴定出差异基因共 3301 个,其中上调 506 个,2795 个基因被下调。本课题组的芯片中差异基因共 2691 个,其中上调 1349 个,1342 个基因被下调。两个芯片共同上调基因 47 个,下调基因 135 个,共计 182 个。GO 分析表明共同差异基因的生物学功能主要集中在基本的生物学过程和细胞黏附;主要的细胞成分包括细胞膜和细胞质;分子功能包括 ATP 结合等(< 0.05)。KEGG 通路分析显示这些共同差异基因主要参与脂类代谢、MAPK 信号通路和细胞黏附信号通路(< 0.05)。CytoHubba 插件分析从 PPI 网络中找到前 20 个具有高度关联性的核心基因。生存分析发现 CXCL10、CUL7、PLCB2 可能与在鼻咽癌不良预后相关(< 0.05)。
利用生物信息学分析筛查鼻咽癌转移相关 DEGs 和通路可以帮助了解鼻咽癌转移发生的分子机制,对鼻咽癌转移的早期诊断具有临床意义。
鼻咽肿瘤; GEO 数据库; 肿瘤转移
复发和转移是鼻咽癌患者预后差的主要原因[1]。肿瘤的转移历经上皮间质转化(epithelial-mesenchymal transition,EMT)、定向侵袭、侵入血液循环或淋巴循环、渗出脉管并形成微转移灶和定植的过程[2]。在 EMT 过程中,一些细胞可获得有助于肿瘤细胞转移的肿瘤干细胞样特性[3]。本课题组前期通过无血清悬浮培养 CNE-2 细胞获得鼻咽癌肿瘤干细胞亚群,并血清诱导其分化成 CNE-2SI 细胞[4]。CNE-2SI 与 CNE-2 相比细胞侵袭能力明显增强。探究其发生机制,用基因芯片技术检测两者的基因表达,并利用生物信息学的手段对两种细胞差异表达基因分析。GEO 数据库中 GSE103611 是 24 个原发与 24 个放疗后转移的临床鼻咽癌组织芯片。通过比对两芯片,筛选出与鼻咽癌转移发生、发展相关的核心基因并进行综合分析。为将来临床上提高鼻咽癌患者诊断率以及针对新的靶点开发防治鼻咽癌转移的更高效的治疗药物奠定理论基础。
1 材料与方法
1.1 材料
1.1.1 实验材料 鼻咽癌分化细胞株 CNE-2SI 由本实验室构建。DMEM 培养基、胎牛血清和 0.25% Trypsin-EDTA 购自美国 Gibco 公司;Transwell 小室和基质胶购自美国 Corning 公司。
1.1.2 数据来源 鼻咽癌及鼻咽癌转移芯片表达谱下载自美国国立生物技术信息中心 NCBI-GEO DataSet 数据库(https://www.ncbi.nlm.nih.gov),编号为 GSE103611。其中包括 24 个原位鼻咽癌样本及 24 个放疗后转移的鼻咽癌样本。直接下载其矩阵文件,通过 R 软件处理数据将其去除重复的基因,保留每个基因最大表达量结果。选取|log2fc| > 1,< 0.05 的差异基因进行后续分析。CNE-2SI 与 CNE-2 之间对比基因表达芯片制作及差异基因分析由博奥生物有限公司完成。
1.2 方法
1.2.1 细胞迁移实验 将 CNE-2 和 CNE-2SI 细胞数调整为 5 × 105个/ml,加 200 µl 细胞悬液于已经水化的侵袭小室上室。培养 6 h 后用棉签擦去上层胶和未迁移细胞,4% 多聚甲醛固定 15 min 后用0.1% 结晶紫染色 15 min,PBS 清洗。倒置显微镜随机选取 10 个视野,拍照后计数细胞数。采用 SPSS 19.0 对数据进行统计学分析。数据符合正态分布,两组比较用检验,< 0.05 具有统计学意义。
1.2.2 筛选共同差异基因及 GO 和 KEGG 途径富集 将 GSE103611 上调差异基因和下调差异基因与本课题组芯片相应差异基因进行比对,筛选出共同上调、下调差异基因用于进一步分析。对共同差异基因使用 DAVIDE 数据库(https://david. ncifcrf.gov/)进行 GO 功能注释及 KEGG 途径分析。在本研究中,我们将差异基因共同分析,< 0.05 被认为具有统计学意义。
1.2.3 蛋白质互作网络(PPI) String 数据库(http://string-db.org)常用于描述经实验验证和预测的蛋白质之间相互作用关系,用来全面整体分析蛋白质之间的生物信息学分析;Cytoscape 软件(http://www.cytoscape.org/)是一种可视化软件,可以将生物分子之间的相互作用关系更加形象直观地描述出来。每个节点是一个基因、蛋白质或分子,节点间的连接表示这些生物分子之间的相互作用。而其插件“cytoHubba”可以通过计算每个节点与其他节点的关联强度,进一步筛选出共同差异基因中的核心基因。

图 1 CNE-2 与CNE-2SI 的迁移能力及差异基因热图[A:Transwell 实验比较CNE-2 与CNE-2SI 的迁移能力;B:穿过基质膜的CNE-2 与CNE-2SI 细胞数比较;C:CNE-2 与CNE-2SI 的差异基因热图(|log2fc| > 1 且P < 0.05),红色代表基因表达上调,绿色代表基因表达下调,黑色表示没有显著差异变化(2-1、2-2、2-3:CNE-2 细胞的差异基因(重复检测 3 次);2S-1、2S-2、2S-3:悬浮培养 CNE-2 细胞获得的干细胞亚群的差异基因;2S-24h、2S-48h、2S-72h:血清诱导干细胞亚群分化的不同时间点的差异基因(每时间点重复检测3次);2S-7d-1、2S-7d-2、2S-7d-3:CNE-2SI 细胞的差异基因(重复检测 3 次)]
Figure 1 Migration capability and differential gene heat map of CNE-2 and CNE-2SI [A: Tranwell cell invasion experiment; B: The number of CNE-2 and CNE-2SI cells across the matigel basement membrane; C: Differential gene heat map of CNE-2 and CNE-2SI (|log2fc| > 1 and< 0.05). Red indicates up-regulation of gene expression, green indicates relative down-regulation of differential gene expression, and black indicates no significant change in gene expression (2-1 to 2-3: differential genes in CNE-2 cells; 2S-1 to 2S-3: differential genes in stem cell subpopulations obtained from suspension culture of CNE-2 cells (repeated detection for 3 times); 2S-24h, 2S-48h and 2S-72h: differential genes at different time points of serum induced stem cell subpopulation differentiation (repeat the test 3 times at each time point); 2S-7d-1 to 2S-7d-3: differential genes in CNE-2SI cells]
1.2.4 Hub 基因的生存分析 cytoHubba 插件筛选出的前 20 个 Hub 基因对头颈部肿瘤预后的影响用 Kaplan Meier-plotter 数据库(http://kmplot. com/analysis/)分析获得。
2 结果
2.1 CNE-2SI 相较于 CNE-2 有更强的迁移能力
Tranwell 实验研究结果显示,CNE-2 细胞与 CNE-2SI 细胞均由上室穿过聚碳酸酯膜到下室。CNE-2 穿过基质膜细胞数为(25 ± 4.04),CNE-2SI 穿过基质膜细胞数为(163 ± 14.2)。两独立样本检验分析显示 CNE-2SI 的侵袭能力明显强于 CNE-2(= 9.33,< 0.01),如图 1 所示。
2.2 芯片数据信息共同差异基因
GSE103611 数据集中共鉴定出差异基因3301 个,其中上调 506 个,2795 个基因被下调。本课题组的芯片中差异基因共 2691 个,其中上调 1349 个,1342 个基因被下调。两个芯片共同上调基因 47 个,下调基因 135 个,共计 182 个。如图 2 所示。
2.3 共同差异基因的 GO 功能富集及 KEGG 通路分析
使用 DAVIDE 在线分析数据库分析两芯片 182 个共同差异基因进行生物学功能注释及通路富集。其中 GO 功能注释包括生物过程、细胞组成及分子生物学功能。将获得的数据导入在线画图软件 imageGP(http://www.ehbio.com/ImageGP/index. php/Home/Index/index.html),结果如图 3 所示。分析显示共同差异基因的生物学功能主要集中在基本的生物学过程和细胞黏附(表 1),主要的细胞成分包括细胞膜和细胞质,分子功能包括 ATP 结合等(< 0.05)。KEGG 通路分析显示这些共同差异基因主要参与脂类代谢、MAPK 信号通路和细胞黏附信号通路(表 2)(< 0.05)。
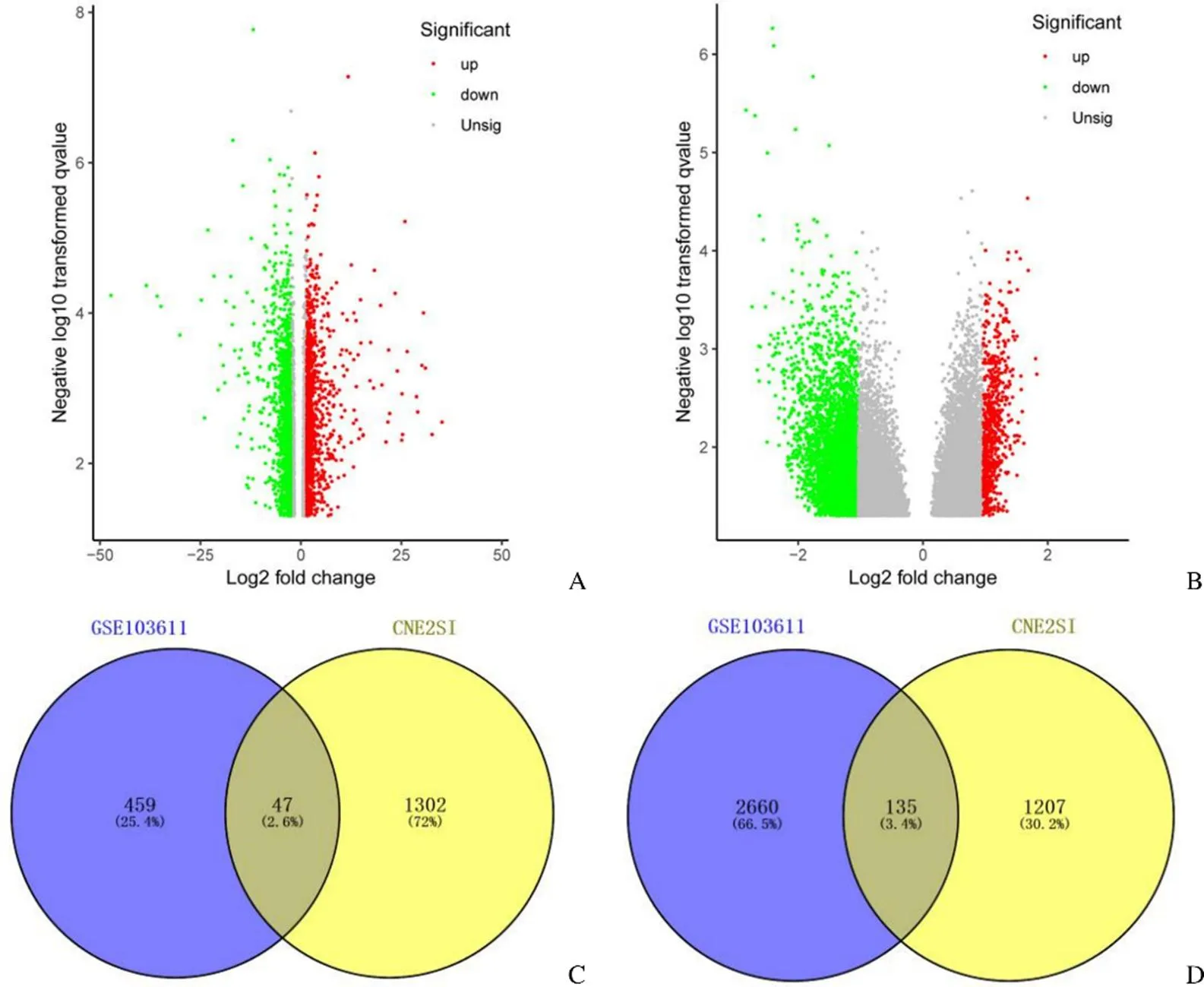
图 2 筛选鼻咽癌转移相关差异基因(A:CNE-2SI 与CNE-2 差异基因火山图;B:GSE103611 差异基因火山图,x 轴代表对数转换后的P值,y 轴代表基因表达的平均差异。注:两个火山图显示了所有DEG;灰点表示样本之间不存在差异表达的基因,绿点和红点分别表示样本中下调和上调的基因。|log2fc| > 1 和P < 0.05 被设定为截止标准;C:GSE103611 和CNE-2SI 数据集上调DEG 的交叉点;D:GSE103611 和CNE-2SI 数据集下调DEG 的交叉点。相交的DEG 被定义为共同的DEG)
Figure 2 Identification of significant differentially expressed genes (DEGs) in nasopharyngeal carcinoma (A: Volcano plot showing the DEGs identified from our microarray; B: Volcano plot showing the DEGs identified from GSE103611. x axis represents log transformedvalue, and y axis indicates the mean expression differences of genes. Note: The two volcano plots showed all of the DEGs; the grey dots represent genes that are not differentially expressed and the green dots and red dots represent the down-regulated and up-regulated genes in nasopharyngeal carcinoma samples, respectively. |log2fc| >1 and< 0.05 were set as the cut-off criteria; C: The intersection of up-regulated DEGs of CNE-2SI and GSE103611 datasets; D: The intersection of down-regulated DEGs of CNE-2SI and GSE103611 datasets. The intersected DEGs were defined as the common DEGs)
2.4 PPI 网络筛选关键核心基因及其预后分析
为了进一步了解差异基因间的相互作用关系,用 String 软件对共同差异基因进行网络互作分析。并将结果导入到 Cytoscape 软件中将其可视化(图 4)。并用 cytoHubba 插件计算每个节点与其他节点的关联强度,进一步筛选出共同差异基因中的核心基因(IRF6、OAS1、CXCL10、CUL7、HERC6、KBTBD7、HACE1、PJA1、HERC3、ISG15、MYO6、PLCB2、MGLL、LGALS3、EGFR、CDH1、ITGA6、LAMA3、LAMB3、COL17A1)。
2.5 核心基因的预后分析
在对 20 个核心基因在头颈部鳞癌中进行预后分析发现,CXCL10(= 0.014)、CUL7(= 0.048)、PLCB2(= 0.0003)的低表达与头颈部鳞癌的不良预后相关,如图 5 所示。

图 3 GO 功能富集以及KEGG 通路富集[A:生物过程(前20);B:细胞成分;C:分子功能;D:KEGG 途径;图中的圆圈颜色改变指示P值的变化,圆圈的大小表示功能中差异基因富集的数量]
Figure 3 GO function enrichment and KEGG pathway enrichment [A: Biological process (top 20); B: Cellular component; C: Molecular function; D: KEGG pathway; the circle color change in the figure indicates the change ofvalue, and the circle size change indicates the richness of DEGs in function number of sets]

表 1 GO:0007155 细胞黏附 term 中的共同差异基因

表 2 差异基因 KEGG 通路富集

图 4 Cytoscape 软件对蛋白质相互作用可视化(方块代表差异基因,线条代表基因之间的相互作用,黄色至红色代表关联强度前20 的基因,颜色越深关联强度越强)
Figure 4 Cytoscape visualizes protein interactions (squares represent differentially expressed genes, lines represent interactions between genes, and colors from yellow to red represent the top 20 genes with stronger correlation, with darker colors showing stronger correlation)

ABC
Figure 5 Effect of Hub gene on survival of patients with head and neck carcinoma (A: CXCL10,= 0.014; B: CUL7,= 0.048;C: PLCB2,= 0.0003)
3 讨论
鼻咽癌是我国华南地区常见的肿瘤之一,缺乏对复发及转移的有效控制是患者存活率下降的主要原因。放疗目前是鼻咽癌治疗的主要手段,但是研究发现放疗可引起癌细胞的局部复发和远处转移[5]。电离辐射(IR)以不同的方式促进癌细胞的存活和侵袭,包括促进上皮间质转化(EMT)、促进肿瘤干细胞(CSCs)和改变肿瘤微环境
(TME)[6]。因此,本文通过将课题组芯片与临床样本芯片进行对比,利用生物信息学方法与基因芯片技术的有机结合,可以宏观、整体地观察鼻咽癌转移的相关基因。为快速寻找鼻咽癌靶点及相关调控通路提供可能。
本研究通过 Transwell 实验发现 CNE-2SI 细胞的迁移能力明显高于 CNE-2。比对实验室两者表达芯片结果,筛选 |log2fc| > 1 且< 0.05 的差异表达基因共 2691 个,其中上调 1349 个,1342 个基因被下调。比对 GEO 数据库中 GSE103611芯片,两个芯片共同上调基因 47 个,下调基因 135 个,共计 182 个。将共同差异基因进行 GO 功能富集及 KEGG 通路富集。GO 分析表明共同差异基因的生物学功能主要集中在细胞黏附。细胞表面黏附于细胞外基质是组织完整性的基础[7],不仅包含细胞与细胞外基质的黏附也包含细胞与细胞之间的黏附。黏附结构的破坏是细胞迁移的重要步骤之一[8]。细胞黏附力下降和运动功能的增强便于肿瘤细胞逃离原发部位,发生转移[9]。本研究得到的结果与已报道的鼻咽癌转移密切相关[10-11]。KEGG 通路分析显示这些共同差异基因主要参与脂类代谢、MAPK 信号通路和细胞黏附信号通路。文献[12]报道脂类代谢的异常与鼻咽癌增殖转移相关。MAPK 信号通路参与细胞增殖、分化和迁移等多种功能。之前的研究发现 MAPK 信号通路的过度激活可诱导 EMT 和鼻咽癌细胞的侵袭[13]。
本文还构建了共同差异基因的 PPI 网络,并筛选了前 20 的核心基因:IRF6、OAS1、CXCL10、CUL7、HERC6、KBTBD7、HACE1、PJA1、HERC3、ISG15、MYO6、PLCB2、MGLL、LGALS3、EGFR、CDH1、ITGA6、LAMA3、LAMB3、COL17A1。其中 CXCL10(= 0.014)、CUL7(= 0.048)、PLCB2(= 0.0003)的低表达与头颈部鳞癌的不良预后相关。CXCL10 基因编码一种细胞因子,在 CXCL9、-10、-11/CXCR3 轴中调控免疫细胞迁移、分化和活化,发挥肿瘤抑制功能[14]。因此 CXCL10 的低表达对免疫细胞的刺激减弱,很可能间接促进肿瘤的转移。CUL7 基因编码的蛋白是 E3 泛素蛋白连接酶复合物的组成部分,研究发现在多种肿瘤中 CUL7 的高表达有促瘤作用[15]。PLCB2基因编码的蛋白是一种磷酸二酯酶,在多种肿瘤的侵袭和转移中的作用[16]。
综上所述,利用生物信息学的方法将鼻咽癌细胞系芯片与 GEO 数据库临床样本芯片相比对,寻找出与细胞转移相关的差异表达基因及可能涉及的调控通路。其部分基因经文献检索已报道。这些发现增加了对鼻咽癌远处转移的发病机制及分子机制的理解,对鼻咽癌的早期诊断和预防具有重要的临床意义。因此证明生物信息学分析有望为日后肿瘤发病机制研究以及诊治方法提供新的策略。
[1] Hui EP, Leung SF, Au JS, et al. Lung metastasis alone in nasopharyngeal carcinoma: a relatively favorable prognostic group. A Study by the Hong Kong Nasopharyngeal Carcinoma Study Group. Cancer, 2004, 101(2):300-306.
[2] Cheng GZ, Zhang W, Wang LH. Regulation of cancer cell survival, migration, and invasion by Twist: AKT2 comes to interplay. Cancer Res, 2008, 68(4):957-960.
[3] Li H, Batth IS, Qu X, et al. IGF-IR signaling in epithelial to mesenchymal transition and targeting IGF-IR therapy: overview and new insights. Mol Cancer, 2017, 16(1):6.
[4] Ji TX, Zhang X, Li XY, et al. Isolation and characterization of cancer stem cell-like sphere-forming cell subpopulation from the human nasopharyngeal cell line CNE-2. Chin J Clin Oncol, 2013, 40(13): 754-757. (in Chinese)
冀天星, 张鑫, 李祥勇, 等. 鼻咽癌肿瘤干细胞样细胞亚群的分离及初步鉴定. 中国肿瘤临床, 2013, 40(13):754-757.
[5] Lee AW, Ma BB, Ng WT, et al. Management of nasopharyngeal carcinoma: current practice and future perspective. J Clin Oncol, 2015, 33(29):3356-3364.
[6] Lee SY, Jeong EK, Ju MK, et al. Induction of metastasis, cancer stem cell phenotype, and oncogenic metabolism in cancer cells by ionizing radiation. Mol Cancer, 2017, 16(1):10.
[7] Hamidi H, Ivaska J. Every step of the way: integrins in cancer progression and metastasis. Nat Rev Cancer, 2018, 18(9):533-548.
[8] Kawauchi T. Cell adhesion and its endocytic regulation in cell migration during neural development and cancer metastasis. Int J Mol Sci, 2012, 13(4):4564-4590.
[9] Wells A, Chao YL, Grahovac J, et al. Epithelial and mesenchymal phenotypic switchings modulate cell motility in metastasis. Front Biosci (Landmark Ed), 2011, 16:815-837.
[10] Shnayder Y, Kuriakose MA, Yee H, et al. Adhesion molecules as prognostic factors in nasopharyngeal carcinoma. Laryngoscope, 2001, 111(10):1842-1846.
[11] Chua HH, Yeh TH, Wang YP, et al. Upregulation of discoidin domain receptor 2 in nasopharyngeal carcinoma. Head Neck, 2008, 30(4):427- 436.
[12] Yang PL, Wang JS, Cheng XM, et al. PPAR-γ ligand inhibits nasopharyngeal carcinoma cell proliferation and metastasis by regulating E2F2. PPAR Res, 2019, 2019:8679271.
[13] Wan XB, Long ZJ, Yan M, et al.Inhibition of Aurora-A suppresses epithelial-mesenchymal transition and invasion by downregulating MAPK in nasopharyngeal carcinoma cells. Carcinogenesis, 2008, 29(10):1930-1937.
[14] Tokunaga R, Zhang W, Naseem M, et al. CXCL9, CXCL10, CXCL11/
CXCR3 axis for immune activation - A target for novel cancer therapy. Cancer Treat Rev, 2018, 63:40-47.
[15] Tian P, Liu D, Sun L, et al.Cullin7 promotes epithelial‑mesenchymal transition of esophageal carcinoma via the ERK‑SNAI2 signaling pathway. Mol Med Rep, 2018, 17(4):5362-5367.
[16] Bertagnolo V, Benedusi M, Brugnoli F, et al. Phospholipase C-beta 2 promotes mitosis and migration of human breast cancer-derived cells. Carcinogenesis, 2007, 28(8):1638-1645.
Identification of signaling pathways and core genes in nasopharyngeal carcinoma metastases using bioinformatics analysis
GONG Wei, LI Tao
To provide a basis for the early diagnosis and targeted therapy potential markers of NPC metastasis, the core genes and related signal pathways related to NPC metastasis were screened out using gene chip technology and bioinformatics analysis method.
The expression profile of GSE103611 was downloaded from the Gene Expression Omnibus (GEO) database, which contained 48 samples including 24 NPC primary tumor samples and 24 NPC metastatic tumor samples. Microarray datasets were sequenced to obtain differentially expressed genes (DGEs). Common differentially expressed genes were obtained by comparison with the differentially expressed genes in contrast chips of the CNE-2SI cells constructed by our research group in the earlier stage, which were more capable of invasion and metastasis than CNE2. Gene ontology and DGEs enrichment of the Kyoto Encyclopedia Gene and Genome (KEGG) pathway were performed by DAVID online analysis. DGE’s protein-protein interaction (PPI) network was constructed from the STRING database. Hub gene analysis was made using the cytoHubba plugin in Cytoscape software. Survival analysis of key genes was obtain via Kaplan Meier-plotter database.
A total of 3301 differential genes were identified in the GSE103611, of which 506 genes were up-regulated and 2795 genes were down-regulated. There were 2691 differential expressed genes in our microarray, of which 1349 genes were up-regulated and 1342 genes were down-regulated. 47 up-regulated and 135 down-regulated common DEGs were identified, respectively. GO analysis indicated that the biological function of total 182 common DGEs were mainly concentrated in basic biological process and cell adhesion. The main cellular components include plasma membrane part and cytosol. Molecular functions included ATP binding. KEGG pathway analysis revealed that these DGEs were mainly involved in the glycerolipid metabolism signaling pathway, MAPK signaling pathway and cell adhesion molecules (CAMs) signaling pathway. Top 20 hub gene were screened out by cytoHubba plugin in Cytoscape software. Among the top 20 hub genes, 3 down-regulated hub genes (CXCL10, CUL7, PLCB2) had significant prognostic values in head and neck squamous cell carcinoma.
Bioinformatics analysis of DEGs and pathways related to nasopharyngeal carcinoma metastasis can help to understand the molecular mechanism of nasopharyngeal carcinoma metastasis, which has clinical significance for the early diagnosis of nasopharyngeal carcinoma metastasis.
Nasopharyngeal neoplasms; GEO data; Neoplasm metastasis
LI Tao, Email: 59889906@qq.com
10.3969/j.issn.1673-713X.2019.06.002
国家自然科学基金(31171351)
523808 东莞,广东医科大学广东省医学分子诊断重点实验室
李涛,Email:59889906@qq.com
Author Affiliation: Provincial Key Laboratory of Medical Molecular Diagnostics, Guangdong Medical University, Dongguan 523808, China
2019-07-26
