肝癌SMMC-7721细胞系HSF4调控的靶基因图谱分析
2018-08-02马汝海王天骄潘忠诚赵雨杰
马汝海,王天骄,潘忠诚,赵雨杰,何 群*
(中国医科大学a.公共基础学院化学教研室;b.基础医学院教育部细胞生物学重点实验室,中国辽宁沈阳 110122)
热休克转录因子(heat shock transcription factor,HSF)在热应激反应中与热休克基因相应的启动子结合,启动基因的转录过程,最终促进热休克蛋白(heat shock protein,HSP)的表达。目前发现HSF 家族有 5 个成员:HSF1、HSF2、HSF3、HSF4、HSF5;哺乳动物体内有3种热休克因子,即HSF1、HSF2 和 HSF4;家禽体内有 HSF3[1~3]。
作为热休克转录因子家族的成员,HSF4含有高度保守的DNA结合域[4],除调控HSP表达功能以外,也参与细胞的生长和分化过程。比如:HSF4表达与晶状体发育相关,可激活晶状体上皮细胞分化,HSF4缺失会影响晶状体上皮细胞热休克蛋白的表达[5,6];Hsf4突变与白内障发生相关[7,8];Hsf4基因缺失会抑制p53或Arf基因缺陷小鼠自发性肿瘤的发展,p53和Arf基因缺陷小鼠敲除Hsf4后其肿瘤图谱明显改变,淋巴瘤被显著抑制[9];Hsf4缺陷细胞中细胞衰老与细胞周期蛋白依赖性激酶抑制剂p21和p27蛋白的表达增加[10];HSF4通过调控RAD51参与DNA损伤修复[11];在Hsf4基因敲除小鼠中DNA损伤标志基因Trx1和Ddit3的表达异常[5,6];Miles等[12]报道在SUDHL-4细胞中,HSF4可与BCL6相互作用,BCL6有调节B淋巴细胞发育和生长的功能,最新研究表明BCL6在B细胞淋巴瘤和其他癌症的发病机制中起着重要作用[13,14]。这些研究结果提示HSF4参与了细胞生长发育等多种生理、病理过程,对HSF4可能调控的靶基因进行系统分析有助于了解HSF4潜在的生物学功能。因此,我们用染色质免疫共沉淀联合测序(chromatin immunoprecipitation sequencing,ChIP-Seq)方法对肝癌细胞系SMMC-7721中HSF4可能结合的靶序列以及可能调控的靶基因进行检测,对潜在的靶基因进行GO(gene ontology)分析和细胞传导通路分析,探讨HSF4可能参与的生物学过程,并用MEME SUITE软件对HSF4与DNA结合的序列模式进行了分析预测。
1 材料与方法
1.1 细胞培养
人肝癌SMMC-7721细胞购于中国科学院上海细胞库,RPMI 1640培养基(含10%胎牛血清)购于Gibco公司(USA),所有细胞置于37℃、5%CO2的培养箱中常规培养。
1.2 染色质免疫共沉淀
染色质免疫共沉淀由上海康成生物公司提供试剂盒并协助完成。细胞的交联和裂解:向细胞中加入终浓度为1%的甲醛交联细胞,37℃孵育10 min,加甘氨酸至终浓度为0.125 mol/L终止交联,吸尽培养基,用冰冷的PBS清洗细胞2次,收集细胞。加入裂解液和蛋白酶抑制剂复合物,超声破碎后将DNA剪切成300~400 bp的片段,分为两部分,分别加入HSF4(艾博抗上海贸易有限公司)和IgG(艾博抗上海贸易有限公司)于4℃过夜,采用HSF4特异性单克隆抗体(艾博抗上海贸易有限公司)收集HSF4与DNA结合的靶序列,磁珠分离,分别用低盐和高盐洗涤液、LiCl和TE洗涤液清洗沉淀复合物,加入洗脱液洗脱,每管中加入终浓度为0.2 mol/L的NaCl于65℃解交联过夜。DNA回收:解交联结束后,加入RNaseA和蛋白酶K处理,用QIAquick Gel Extraction Kit富集收集凝胶回收DNA片段,测定DNA浓度(Qubit®Fluorometer)。PCR分析验证:采用Actb作为阴性对照,Hsp70作为阳性对照,Actb-F:5′-CTAGGTCACCCACTAACGCC-3′;Actb-R:5′-GGGCACCTTTTACCCTGGAG-3′;Hsp70-F:5′-CCGCCACTCCCCCTTCCTC-3′;Hsp70-R:5′-CCGCCTTTTCCCTTCTGAGC-3′。加入T4 DNA聚合酶和Klenow polymerase对DNA进行钝化,在3′加入A末端。采用Illumina’s genomic adapters连接DNA片段,PCR扩增,凝胶电泳后用QIAquick Gel Extraction Kit富集收集DNA产物。使用Agilent 1000芯片试剂盒和Agilent 2100 Bioanalyzer验证DNA文库质量。最后加入0.1 mol/L NaOH产生单链DNA分子,按照TruSeq Rapid SBS Kit操作说明用Illumina HiSeq 2000进行测序。
1.3 ChIP-Seq数据分析
生成的序列图谱及图像分析结果提交到OLB V1.8软件读取测序结果,Solexa CHASTITY过滤后的数据使用BOWTIE software(V2.1.0)与人类基因组(UCSC hg19)进行比对,图谱峰值检测采用MACS V1.4.0。
1.4 富集分析
对启动子区有HSF4结合区域的基因进行GO分析(www.geneontology.org),P≤0.05。
1.5 细胞通路分析
根据KEGG(Kyoto Encyclopaedia of Genes and Genomes)最新数据,我们对启动子区有HSF4结合区域的基因进行细胞通路分析(P≤0.05)。
1.6 DNA基序分析
提取与HSF4结合的启动子区域的DNA序列,提交到MEME4.12.0(http://meme-suite.org/tools/meme)进行模式分析,探讨DNA序列与HSF4结合的序列模式。
2 结果
2.1 ChIP-Seq
将建立的DNA测序文库与hg19数据库进行对比,使用最新的UCSC RefSeq数据库进行基因注释。峰值区域按照最近的TSS被分成五类,即启动子区域:TSS上下游±2 000 bp;上游区域:从TSS上游2 000 bp起始最多达20 000 bp;内含子区域;外显子区域;不在上述4个区域的DNA集合区定义为基因间隔区。总共发现1 726个HSF4结合区域,约55.59%的HSF4结合区域位于基因间区,11.54%处于上游区域,25.57%位于内含子区,1.39%位于外显子区,5.91%位于启动子区(图1)。
2.2 GO分析结果
为了探讨HSF4在SMMC-7721细胞系中发挥的生物学功能,我们对在启动子区域有HSF4结合位点的102个基因序列进行聚类分析(GO分析,www.geneontology.org,P≤0.05),结果如表1和图2所示。Cellular component(CC)分析结果共有41个基因,主要位于细胞核,部分位于细胞器,少部分位于细胞膜;molecular function(MF)分析结果显示有18个基因,主要与核酸蛋白质结合以及蛋白质活性相关;biological process(BP)分析结果显示有31个基因,主要参与细胞增殖、系统发育、对化学药物以及外来刺激的反应和免疫应答过程,其中一些基因参与了多个生物学过程,隶属于不同亚项。表1列举了GO聚类分析中各项目的靶基因数目,很多基因参与一个聚类分析中两个或更多的亚项。
2.3 细胞传导通路分析
ChIP-Seq结果显示有102个基因在启动子区有HSF4结合区域,基于最新的KEGG数据库,我们对这些基因进行细胞通路富集分析,结果显示这些HSF4潜在的靶基因主要参与以下细胞通路:药物代谢、外源物代谢、化学致癌、视黄醇代谢、酪氨酸代谢、类固醇激素合成以及催乳激素的信号转导通路等(图3)。
2.4 DNA基序分析
目前尚没有看到与HSF4结合的DNA特定基序的有关报道,大多数转录因子(transcription factor,TF)的数据库有关HSF家族结合基序数据多依据HSF1的基序模式。我们将在启动子区域与HSF4结合的DNA序列提交到MEME4.12.0分析软件,以探讨在SMMC-7721细胞系中HSF4特定的DNA结合基序,结果如图4所示。我们将编码基因和非编码基因启动子序列分别进行分析,得出6个分析结果,编码基因和非编码基因各有3个基序模式,其中编码基因的启动子基序呈现AG或者CT的聚集簇。

表1 GO分析中参与各个功能的靶基因数量Table 1 The number of the target genes participating in GO categories
3 讨论
当细胞受到高温、毒物、自由基及感染等应激原的刺激时,会发生一种普遍的防御性适应反应——热休克反应(heat shock response,HSR)。当细胞受到应激性刺激时,细胞会通过HSR诱导热休克蛋白(heat shock protein,HSP)表达增强,引起基因表达谱的改变,减轻应激性刺激对细胞的不良影响。HSP作为一种分子伴侣,在调控细胞稳定性、提升细胞存活率方面发挥重要作用[15]。HSF4是HSF家族的重要成员,其发现较晚。已有研究显示,HSF4能启动HSP编码基因的转录过程,促进HSP的表达,发挥耐受不良刺激、保持细胞完整性等重要适应性保护作用[16]。近来相关研究发现,HSF4亦参与与应激性刺激无关的过程,例如:参与细胞周期、细胞分化,与HSF协同作用[17,18]。为了探讨HSF4在肝癌发生机制中所起的作用,我们采用ChIP-Seq方法对全基因组HSF4与DNA结合位点进行检测,寻找HSF4可能的靶基因,并对这些潜在的靶基因进行GO分析和细胞通路分析。分析结果显示:这些潜在的靶基因分别参与了药物代谢、外源性物质代谢、化学致癌、视黄醇代谢、酪氨酸代谢、类固醇激素的合成以及催乳激素的信号转导通路,参与了细胞发育、细胞增殖、药物刺激应答等多种生物学过程,其中多数基因具有与蛋白质或核酸结合的功能,与蛋白质的活性有关。在这些基因中,有部分基因与肿瘤的形成和细胞增殖关系密切,例如,Aldh3a1、Cyp2a6和Zbtb7b。文献报道,Aldh3a1在肝细胞癌中过表达,可激活Wnt/β信号传导通路[19];抑制Aldh3a1表达可抑制肿瘤细胞的增殖,在组织再生过程中ALDH3A1可刺激正常细胞增殖[20];此外,前列腺癌淋巴结转移患者中也发现了Aldh3a1表达上调[21]。CYP2A6是细胞色素P450酶家族的一个成员,P450是单加氧酶,参与药物代谢以及胆固醇、类固醇和其他脂类的合成反应。Cyp2a6主要在肝脏中表达,参与多种物质包括药物和有毒物质的代谢,如尼古丁代谢,并与吸烟引起的疾病密切相关[22]。Zbtb7b也称Thpok基因,编码产物为含有锌指结构的转录因子,该转录因子在未成熟的T细胞前体细胞系中起着调控作用[23],Thpok基因缺失会导致Cd8基因转录受到抑制[24],在早期结肠癌中Thpok起着免疫调节作用[25]。所有这些信息提示:HSF4可能通过调节这些潜在的靶基因,在肝癌的形成机制中发挥作用。
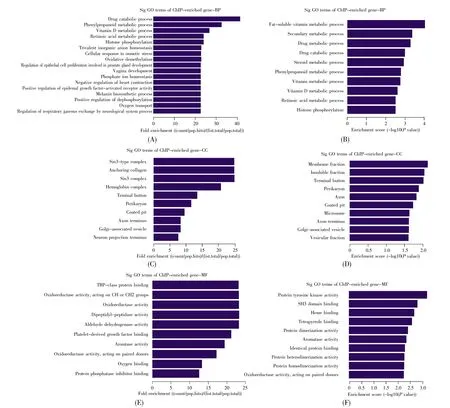
图2 GO分析结果Fig.2 GO analysis map

图3 启动子区域有HSF4结合位点的基因的细胞通路分析Fig.3 KEGG pathway analysis of genes with HSF4 binding sites in the promoter region
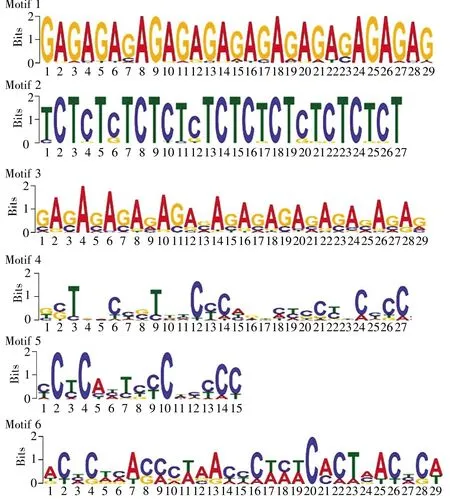
图4 SMMC-7721细胞ChIP-Seq实验中HSF4与DNA结合位点基序分析结果Fig.4 Motifs bound by HSF4 in SMMC-7721 cells in ChIP-Seq analysis
转录因子在基因转录起始点与特定的DNA序列相结合,这段DNA片段通常为6~18 bp,转录因子特异识别位点的确定一直具有挑战性。Hsf4的表达存在组织及细胞特异性,通常认为HSF4参与应激反应时,胞内或胞外的信号刺激活化HSF4,使HSF4结构改变,暴露DNA结合域,随后活化的HSF4识别并结合热休克基因上游的热休克元件,从而刺激HSP的表达。
目前,在所有热休克基因上游均发现“CTNGAANNTTCNAG”多个拷贝聚集区[26],一般认为热应答元件(heat response element,HSE)中至少有两个或以上的保守五核苷酸序列nGAAn反相交替排列,形成头-头(nGAAnnTTCn)和尾-尾(nTTC-nnGAAn)结构,为HSF的识别位点。但是目前有关人类HSF4与启动子区结合的DNA特征序列尚无报道,只有HSE启动子区的顺式作用元件(HSE:CGAATTCG)的报道[27,28]。Jaspar数据库中有关HSF结合motif只有HSF1的相关记录:ma0319.1(mouse)[29]和 ma04888886.1(yeast)[30]。ChIP-Seq 是确定转录因子与DNA结合区域的主要手段,但具体的motif还需要进一步分析。MEME是一种分析DNA motif的生物信息学软件[31],输入结合区域的DNA序列后,通过富集分析、定位分析和聚类分析可预测出潜在的motif。我们将ChIP-Seq结果中与HSF4结合的启动子区域的DNA序列提交给MEME4.12.0软件,最后得到与HSF4结合的motif,分析结果显示:潜在的靶基因DNA结合区域均存在agagag或tctctc聚集模式。
通过ChIP-Seq和生物信息学分析,我们对SMMC-7721细胞系中HSF4可能调控的靶基因以及这些靶基因在生物体中可能发挥的作用和可能参与的生物学过程进行了分析,并对HSF4与DNA结合的序列模式进行了预测,为进一步研究HSF4在肝癌形成机制中所发挥的作用提供了理论基础。
