2012—2016年中国南方地区帕利亚姆血清群病毒的分离与序列特征分析
2018-05-07李占鸿孟锦昕杨振兴吕敏娜林栩慧廖德芳牛保生李华春
杨 恒,肖 雷,李占鸿,孟锦昕,杨振兴,吕敏娜,林栩慧,廖德芳,牛保生,李华春*
(1. 云南省畜牧兽医科学院 热带亚热带动物病毒重点实验室,昆明650224;2. 广东省农业科学院动物卫生研究所,广州510640;3. 云南省师宗县动物疫病预防控制中心,师宗655799)
帕利亚姆血清群病毒(Palyam serogroup virus, PALV)为呼肠孤病毒科环状病毒属(Orbivirus)基因组分节段双链RNA(dsRNA)虫媒病毒,广泛分布于热带、亚热带与温带地区,可感染多种反刍动物,其中以牛最为易感。病毒主要通过吸血昆虫库蠓(Culicoides)对动物的吸血性叮咬传播[1-2]。PALV具有多种不同的血清型,由于历史原因,不同血清型的PALV往往以病毒首次分离地的地名进行命名,因此容易让人误认为它们是不同类型的病毒,但实际为同一种病毒的不同血清型。目前报道的PALV血清型有Chuzan virus(CHUV)、D’Aguilar virus(DAV)、Bunyip Creek virus(BCV)、CSIRO Village virus、Petevo virus、Gweru virus、Marondera virus、Marrakai virus、Abadina virus以及Vellore virus[3]。
CHUV于1985年首次从日本中山县库蠓C.oxystoma和牛血液中分离获得,因而命名为中山病毒[4]。CHUV为导致1985—1986年日本暴发的新生牛积水性无脑小脑发育不全综合征(hydranencephaly cerebellar hypoplasia syndrome, HCH)的病原[5],目前日本已经研发了针对CHUV的灭活疫苗,用于控制本病的流行[6-7]。2007年韩国的一项家畜虫媒病毒血清学调查研究显示,发生流产牛的CHUV抗体阳性率为22.8%,认为CHUV可能与牛的流产有一定的关系[8]。DAV与BCV最早分别于1970与1975年从澳大利亚的库蠓C.oxystoma与C.peregrinus中分离。2016年,日本从监控牛群与库蠓上分离出多株DAV与BCV,证实了两种病毒在日本的流行[9]。目前关于BCV与DAV致病性报道较少,推测两种病毒可能与发生在牛上的流产有一定关系,但仍需进一步通过动物试验证明[10]。
与环状病毒属的蓝舌病病毒(BTV)和鹿流行性出血热病毒(EHDV)类似[11],PALV的基因组大小约21 kb,由Seg-1(3.9 kb)至Seg-10(0.7 kb)等10个基因节段构成,可编码7种结构蛋白(VP1~VP7)与三种非结构蛋白(NS1~NS3)[12-14]。Seg-2编码的VP2蛋白决定着PALV的血清型,诱导产生病毒的血清型特异性中和抗体,其核酸与氨基酸序列在不同血清型毒株之间高度变异。Seg-7编码的VP7蛋白为PALV的群特异性抗原,可诱导产生病毒的群特异性抗体,其氨基酸序列在不同血清型之间高度保守,Seg-7核酸序列在不同地域分离的毒株之间存在着一定差异,体现出病毒的地域特征[15-16]。
2012年,我们从云南省师宗县的监控牛上首次分离出CHUV(SZ/187毒株),并完成其全基因组序列测定[17],随后王芳(F.Wang)等[18]在项目设立于广西壮族自治区马山市的监控牛中也分离出CHUV(GX871毒株)。通过对两株中国CHUV毒株基因组序列的比较显示,两个毒株Seg-1至Seg-10基因节段的核酸序列相似度均大于99%,表明在我国云南与广西流行的CHUV有着非常近的亲缘关系。
为掌握PALV在我国大陆地区的分布、血清型与病毒的遗传背景,2012—2016年我们在云南、广东与广西设立的监控动物牛上开展了PALV的分离工作,相继分离出19株PALV。本文对我国PALV的分离、鉴定、Seg-2与Seg-7的序列特征进行了报道,研究结果将有助于掌握我国PALV的分布、遗传背景以及与世界范围PALV毒株之间的关系,为PALV诊断试剂的开发、流行病学调查与致病性研究提供科学依据。
1 材料与方法
1.1 试剂
磁珠法病毒RNA抽提试剂盒MagMAX-96 Viral RNA Isolation Kit、MEM培养基、胎牛血清购自Thermo公司; RNA提取试剂RNAiso-Plus、病毒RNA/DNA抽提试剂盒、高分辨率琼脂糖、PrimeScritp逆转录酶、ExTaqDNA聚合酶、胶回收试剂盒、荧光定量qRT-PCR试剂盒,购自大连宝生物公司;氯化锂(LiCl)购自Sigma公司。
1.2 细胞株、毒株、抗体
BHK-21细胞,BTV-1型毒株(Y863)、EHDV-1型毒株(V128/2014)与CHUV毒株(SZ/187)由本实验室分离保存;兔抗CHUV多克隆抗体由本实验室制备保存;FITC标记羊抗兔IgG荧光二抗,购自武汉博士德公司。
1.3 监控点与监控动物的设立
通过对云南、广西、广东牛、羊虫媒病毒的血清学调查并结合当地的气候、地理环境的分析,作者在我国的云南省师宗县、广西壮族自治区马山市以及广东省汕头市郊区分别设定了三个牛、羊虫媒病毒监控点。在监控点设置BTV血清学阴性的牛和羊作为虫媒病毒监控动物,每年3至10月定期从监控动物上采血进行虫媒病毒的检测与分离工作。
1.4 血液样品的采集与病毒分离
从监控动物上采集的肝素钠抗凝血、EDTA抗凝血与常规血液三种血液样品于4 ℃保存,并送至实验室进行虫媒病毒的检测与病毒分离工作。取200 μL EDTA抗凝血3 000 r·min-1离心10 min收集红细胞(RBCs),以PBS洗涤1次,加入1 mL灭菌水作用10 min裂解RBCs。取50 μL RBCs 裂解液以MagMax Express核酸自动提取仪提取核酸,采用本实验室建立的牛羊虫媒病毒高通量Real Time qRT-PCR技术进行血液样本中BTV、EHDV、PALV、阿卡斑病毒与牛流行热病毒的核酸检测。对qRT-PCR检测为PALV阳性的血液样本(Ct值>38),取200 μL RBCs裂解液接种长为单层的BHK细胞并连续盲传5代,当细胞出现明显的细胞病变(CPE)时,收获细胞上清,冻存于-80 ℃备用。
1.5 引物设计与合成
依据我们前期获取的中国CHUV毒株(SZ/187与GX871)序列[17-18]以及GenBank公布的PALV毒株的序列,以PALV的群保守性Seg-7基因节段和血清型特异性Seg-2基因节段为靶序列,设计4对扩增引物分别用于PALVSeg-7以及BCV、CHUV和DAV三种血清型Seg-2的特异性RT-PCR扩增。引物由上海捷瑞公司合成,引物序列见表1。
表1PALV的Seg-7与BCV、CHUV和DAV的Seg-2RT-PCR扩增引物
Table1RT-PCRamplificationprimerstargetingSeg-7ofPALVandSeg-2ofBCV,CHUVandDAV

引物名Primersname引物序列(5'-3')Primerssequence位置Primerslocation产物大小/bpProductssizePALV-S7-FGCCAACTACACAACAAGAAAGSeg-7:171-191791PALV-S7-RCCGAATATAACAGTAAAGCCASeg-7:941-961BCV-S2-FGRGCTRTCATAGATTCGGAAGCSeg-2:987-10081251BCV-S2-RTACAAACYTTTACYAGAGCCATSeg-2:2215-2237CHUV-S2-FGGATTCACGCYATTACTCGGASeg-2:1175-11951004CHUV-S2-RATGGATTTCTTGTTGGGATTATSeg-2:2157-2178DAV-S2-FATTTTTCAACGAAGAGACGTGSeg-2:929-9491223DAV-S2-RATAGGAACACCTTTACCYAGGSeg-2:2131-2151
1.6 核酸提取、RT-PCR扩增与测序
采用病毒RNA/DNA抽提试剂盒提取病毒核酸, 94 ℃变性3 min后立即冰浴,进行病毒dsRNA的解链。以变性后的核酸为模板,使用Radoms 6 mers引物和PrimeScritp逆转录酶进行cDNA的合成。取2 μL合成的cDNA为模板,以ExTaqDNA聚合酶进行PALV的Seg-7与Seg-2的PCR扩增。PCR扩增程序:94 ℃预变性3 min,94 ℃变性30 s,55 ℃复性30 s,72 ℃延伸1 min 30 s,30个循环,72 ℃延伸10 min。取5 μL PCR扩增产物进行电泳,对扩增大小符合预期条带进行电泳胶回收与测序,使用Gontig Express软件对测序结果进行拼接。将获取的序列进行BLAST比对,对病毒的种属与血清型进行判定。
1.7 高分辨率琼脂糖凝胶电泳分析
将我国分离的BTV-1(Y863毒株),EHDV-1(V128/2014毒株)、CHUV(SZ/187毒株)、DAV(V106/YN/2014毒株)与BCV(V104/YN/2014毒株)分别接种单层BHK-21细胞,待细胞出现完全CPE后,离心收集细胞,使用RNAiso-Plus按说明书提取细胞总RNA。按文献[19]的方法进行基因组dsRNA的纯化。以高分辨率琼脂糖配制2%浓度的凝胶,取10 μL纯化后的病毒基因组dsRNA于4 ℃以90 V电压电泳4~5 h,比较分析BTV、EHDV、CHUV、DAV和BCV五种病毒基因组dsRNA的电泳带型。
1.8 系统发育树的构建
从GenBank中下载分离自中国、日本、印度、澳大利亚与津巴布韦PALV毒株的Seg-2与Seg-7序列。使用Mega 6.0软件[20]与本研究中获取的序列进行比对,将比对结果以软件中的邻近法(Neighbor-joining,NJ)进行系统发育树构建,选择模型参数为:P-distanc,自举检验(Bootstrap)取值1 000。
1.9 免疫荧光试验
将CHUV(SZ/187毒株)、DAV(V106/YN/2014毒株)、BCV(V104/YN/2014毒株)分别接种12孔板BHK-21细胞,并设定未接毒空白细胞作为阴性对照,培养72 h。待细胞开始出现CPE时,弃去培养液,以预冷的甲醇/丙酮混合液(1∶1)室温固定20 min,PBST洗涤细胞一次;以5%的脱脂奶于37 ℃封闭1 h;弃去封闭液,加入200 μL兔抗CHUV多克隆抗体(1∶200稀释)37 ℃孵育1 h,以PBST洗涤细胞三次;加入FITC标记羊抗兔IgG荧光二抗200 μL(1∶400稀释)37 ℃孵育1 h,以PBST洗涤细胞三次,于倒置荧光显微镜下观察细胞的荧光。
1.10 病毒中和试验
选择云南省分离的SZ/187毒株、V106/YN/2014毒株与V104/YN/2014毒株分别作为中国分离PALV的CHUV、DAV与BCV血清型参考毒株,进行病毒的噬斑纯化。将纯化后的三个参考毒分别进行10-1至10-7连续10倍稀释,每个稀释度按50 μL·孔-1的量,接种96孔板培养的BHK-21细胞,培养6 d观察CPE,采用Reed-Muench法计算病毒的TCID50。将自然感染SZ/187、V106/YN/2014与V104/YN/2014三个毒株动物的血清以56 ℃灭活30 min,并以1∶20~1∶640连续倍比稀释备用。取50 μL 100 TCID50的参考毒病毒液,采用“固病毒-稀释血清”的方法测定感染动物血清的中和抗体效价;采用交叉保护中和试验分析CHUV、DAV与BCV阳性血清之间是否存在交叉保护。
2 结 果
2.1 中国PALV毒株的分离
2012至2016年作者在我国云南省师宗县、广西壮族自治区马山市以及广东省汕头市送检的牛、羊血液样品中,通过PALV的特异性Real Time qRT-PCR共计检测出47份阳性血液样品。取PALV核酸阳性血液以BHK-21细胞进行病毒的连续盲传分离,在盲传的第3~5代,细胞出现明显的细胞病变(CPE),表现为大量的贴壁细胞变圆与脱落(图1)。本研究共计分离出19株PALV,病毒分离信息见表2。
2.2 中国分离PALV Seg-7与Seg-2的扩增与序列分析
对PALV的Seg7序列比对显示,中国分离的19株PALV的Seg-7核酸序列相似性在97.9%~100%,与中国台湾分离毒株相似性在98.1%~99.1%,与日本分离毒株相似度在96%~97.4%,与印度分离毒株的相似度在93.9%~94.9%,与澳大利亚和津巴布韦分离毒株的相似性最低,在83.8%~90.5%。
对PALV的Seg2序列比对结果显示,本研究分离的PALV分属CHUV、DAV与BCV三种血清型(表2)。云南与广西分离的13株CHUV的Seg-2核酸序列相似性在99%~100%,与日本分离CHUV毒株序列相似性在96.2%~98.3%。云南分离的3株DAV的Seg-2核酸序列相似性大于99.5%,与日本分离DAV毒株相似性在94.7%~98.4%,与澳大利亚和津巴布韦DAV毒株的相似性为91%。云南与广东分离的4株BCV的Seg-2与日本分离BCV毒株核酸序列相似性均大于99%,与澳大利亚分离BCV毒株的相似性在91%~92.2%。

A. 感染细胞;B. 对照细胞A. Infected cells; B.Control cells图1 PALV感染BHK-21细胞引起的CPEFig.1 CPE of BHK-21 cells caused by the infection of PALV
表22012至2016年我国分离PALV的分离信息
Table2IsolationinformationofChinesePALVform2012to2016

毒株号StrainNo.病毒/血清型Virus/serotype分离动物Isolatedanimal分离时间Isolatedtime分离地点IsolatedsiteSZ/187PALV/CHUV13号牛2012年8月云南省师宗县V144/YN/2012PALV/CHUV14号牛2012年9月云南省师宗县GX871PALV/CHUV水牛2013年8月广西壮族自治区马山市V104/YN/2014PALV/BCV10号牛2014年10月云南省师宗县V145/YN/2014BPALV/BCV/CHUV4号牛2014年9月云南省师宗县V103/YN/2014PALV/CHUV5号牛2014年8月云南省师宗县V101/YN/2014PALV/CHUV20号牛2014年8月云南省师宗县V100/YN/2014PALV/CHUV4号羊2014年8月云南省师宗县V099/YN/2014PALV/CHUV4号羊2014年8月云南省师宗县V078/YN/2014PALV/CHUV20号牛2014年8月云南省师宗县V098/YN/2014PALV/CHUV17号牛2014年9月云南省师宗县V142/YN/2014PALV/CHUV19号牛2014年9月云南省师宗县V106/YN/2014PALV/DAV4号牛2014年9月云南省师宗县V141/YN/2014PALV/CHUV4号牛2014年9月云南省师宗县V156/YN/2015APALV/CHUV水牛2015年8月云南省红河州V157/YN/2015APALV/DAV水牛2015年8月云南省红河州V256/GD/V2015PALV/BCV奶牛2015年11月广东省汕头市V203/YN/2016PALV/DAV牛2016年8月云南省师宗县V252/GD/V2016PALV/BCV奶牛2016年1月广东省汕头市
A. V156/YN/2015与V157/YN/2015毒株分离自红河州送检血液样品,而非监控动物;B. V145/YN/2014同时含有BCV与CHUV两种血清型病毒
A. The V156/YN/2015 and V157/YN/2015 strains were isolated from blood samples of Red River State, not monitoring animals;B. V145/YN/2014 contains two serotype viruses of BCV and CHUV
2.3 中国分离PALV的dsRNA电泳分析
基因组核酸电泳可用于初步判断基因组分节段dsRNA病毒的种类[20]。我们提取并纯化了BTV、EHDV、CHUV、BCV、DAV五种病毒的基因组dsRNA,以2%的高分辨率琼脂糖进行电泳。结果如图2显示,PALV的CHUV、DAV、BCV三种血清病毒dsRNA型电泳带型完全一致,呈现“3-3-4”的电泳带型特征,而BTV和EHDV呈现出“3-3-3”的电泳带型,与PALV基因组电泳带型有着明显的差异,这提示PALV的基因组节段大小与BTV和EHDV存在明显差异,可通过核酸电泳“带型”的差异对这三种病毒进行快速区分。

1.BTV; 2.EHDV; 3.BCV; 4.CHUV; 5.DAV图2 BTV、EHDV、BCV、CHUV、DAV基因组dsRNA高分辨率琼脂糖凝胶电泳结果Fig.2 The high-resolution agarose gel electrophoresis result of genomic dsRNA of BTV, EHDV, BCV, CHUV and DAV
2.4 中国分离PALV Seg-2与Seg-7的系统发生树分析
不同血清型PALV的Seg-7显示出明显的地域特征(图3),可分为四簇:来自中国、日本与印度的不同血清型PALV共同形成一个大簇,我们将其命名为“Asia Group”,来自澳大利亚的PALV可形成两个独立的簇,分别命名为“Australia Group 1”与“Australia Group 2”,而来自非洲津巴布韦的PALV形成另一独立的簇,命名为“Zimbabwe Group”。
不同血清型PLAV分离毒株Seg-2的分子系统发育分析显示(图4),中国CHUV、DAV、BCV三种血清毒株与日本、澳大利亚分离对应血清型毒株聚为一簇。值得注意的是,在同一血清型毒株中,Seg-2的序列也体现出了一定的地域性,日本与中国分离毒株聚为紧密的一簇,而来自澳大利亚的毒株形成一个独立于中国与日本毒株的分支。
2.5 中国分离BCV、CHUV与DAV毒株的免疫荧光分析
以实验室制备的兔抗CHUV阳性血清为一抗,对感染了CHUV、BCV与DAV的BHK-21细胞进行免疫荧光染色分析。结果如图5显示,制备的CHUV阳性血清可与病毒感染后细胞中产生的病毒蛋白发生特异性结合,使BHK细胞显现出特异性绿色荧光,而未感染病毒的对照细胞无荧光。
2.6 中国分离BCV、CHUV与DAV毒株的血清中和试验
自然感染SZ/187、V106/YN/2014与V104/YN/2014三个毒株的牛血清对100 个TCID50的CHUV、DAV与BCV病毒液的中和抗体效价均在1∶160以上,而1∶20稀释的对照阴性血清对病毒液未产生中和作用,表明动物发生了对应血清型病毒的感染。交叉中和试验结果显示,CHUV、DAV与BCV三个血清型的中和抗体之间无交叉中和保护作用。
3 讨 论
PALV广泛分布于热带与亚热带地区,非洲的几内亚、尼日尼亚、津巴布韦,大洋洲的澳大利亚,亚洲的中国台湾地区、印度、日本、韩国均有该病毒的分离报道[1, 3-4, 8-9 ]。长久以来,PALV在我国大陆地区的分布情况、与国外毒株之间的关系以及对动物的危害等问题一直不清楚。近10年来,由于全球气温升高等因素,新发和复发虫媒病毒病不断出现,对畜牧业生产和人类健康构成严重威胁[21]。为掌握我国牛、羊虫媒病毒的分布与流行特征,作者在我国的云南、广西、广东、湖北、江苏、山西、新疆、内蒙古等8个省建立了牛、羊虫媒病毒鉴定点并设定了监控动物。自2012年首次从云南省的监控牛上分离出CHUV以来[17],我们连续5年在云南、广西与广东省的监控动物上分离出PALV,表明该病毒很可能广泛流行于我国南方地区。
我们从监控动物与送检血液样品中检出47份PALV核酸阳性血液样品,然而将这些阳性血液样品在BHK-21细胞上连续盲传却只分离出19株PALV,仍有部分阳性血液未能分离出病毒。我们认为可能有两方面的原因:(1)PALV阳性血液在BHK细胞上盲传可能不是PALV最佳的分离方式。我们分离的第一株PALV(SZ/187),通过将血液样本在BHK-21细胞连续盲传获得成功分离,因此后继大部分PALV的分离工作采用该方式进行。王芳(F.Wang)等[18]采用C6/36盲传1代而后继续在BHK细胞连续盲传的方式也从牛血液样品中分离到了PALV毒株,提示PALV可以采用不同的方式进行病毒分离,至于哪种分离方式对病毒分离效率最高,我们可以参考BTV的分离方式[22]进行比较与摸索,从而提高PALV的分离效率。(2)当感染动物产生了感染病毒的中和抗体以后,血液中抗体与病毒的结合将导致病毒对接种细胞的感染能力大幅下降[22]。这可能是我们虽然检测到PALV核酸阳性,但病毒分离失败的一个原因。通过PALV的动物感染试验,分析在不同的时间点感染动物血液中病毒核酸的水平、病毒中和抗体水平以及病毒成功进行分离之间的关系,可帮助选择最佳的时间点对监控动物进行采血,有效开展PALV的分离工作。
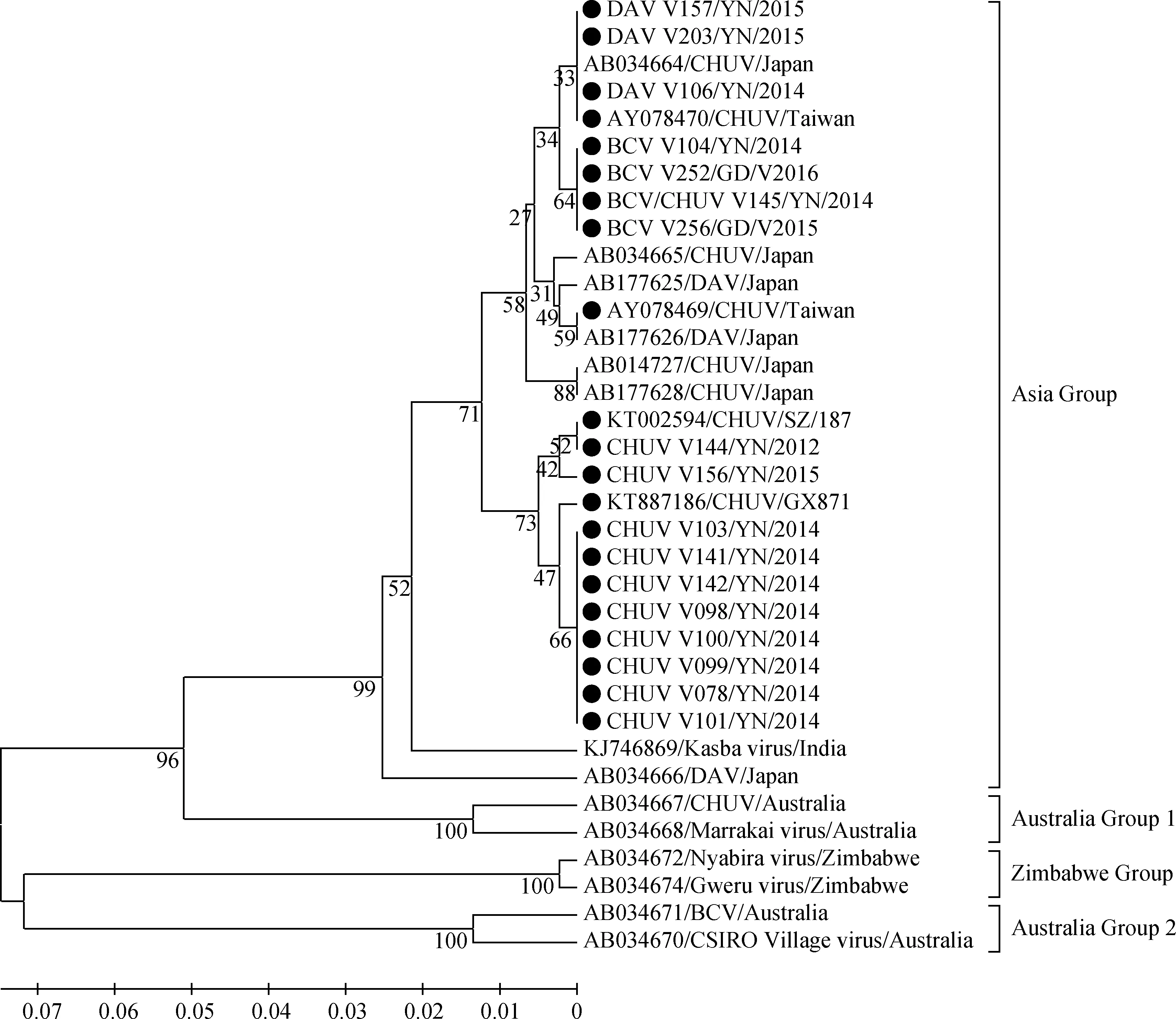
图中●表示本研究中分离的PALV毒株及其他中国分离株● represents the isolated PALV strains in this study and other Chinese isolates图3 邻近法构建的PALV Seg-7序列系统发育分析Fig 3 Phylogenetic analyses available Seg-7 of PALV with Neighbor-joining method

图中●表示本研究中分离的PALV毒株● represents the isolated PALV strains in this study图4 邻近法构建的PALV Seg-2系统发生树图Fig 4 Phylogenetic analyses available Seg-2 of PALV with Neighbor-joining method
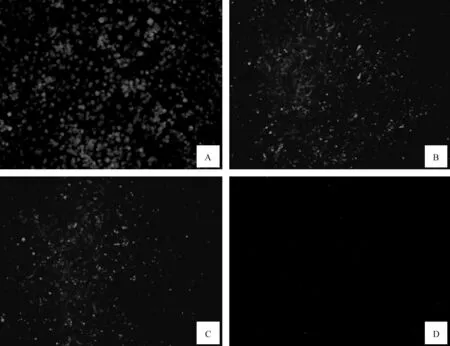
A. CHUV感染细胞;B.BCV感染细胞;C.DAV感染细胞;D.未感染BHK对照细胞A. CHUV infected cells; B.BCV infected cells; C.DAV infected cells; D. Uninfected BHK cells图5 BHK-21细胞感染BCV、CHUV和DAV 72 h后的免疫荧光观察结果Fig 5 Immunofluorescence result of BHK-21 cells after infected respectively with BCV, CHUV and DAV for 72 h
通过对PALV保守的Seg-7节段的测序与系统发育分析显示该基因节段体现出了明显的地域性。分离至中国、日本、印度三个国家的PALV聚为一个独立的“Asia Group”,而分离自澳大利亚与非洲的PALV则形成了各自单独的进化簇。这一方面表明PALVSeg-7节段的核酸序列可作为不同血清型PALV地域性(Topotype)判定的一个标准[15],另一方面也提示中国、日本、印度三个国家的PALV有着最近的共有祖先。
对BTVSeg-2的系统发育分析表明[23-25]:不同血清型的BTV毒株可聚在一起形成一个基因型,并据此将BTV 27个血清型分为A~L等十二个基因型;在同一个BTV血清型中,还可根据毒株分离地域的不同进一步分为“东方型”和“西方型”以及多个地域亚型。从本研究的结果来看,我们初步认为同一血清型PALV的Seg-2在一定程度上也体现了地域型的差异,但由于世界范围PLAV序列的缺乏,目前还难以确定PALV的Seg-2是否体现和BTV类似的进化特征。随着更多分离至亚洲、非洲与澳大利亚的不同血清型PALV毒株Seg-2的测序,将有助于我们从整体上进一步认知该病毒Seg-2与血清型之间的关系以及进化特征。
PALVSeg-2与Seg-6编码的VP2与VP5蛋白共同决定着病毒的血清型,激发感染动物产生血清型特异性抗体,Seg-7编码的VP7蛋白为PALV的群特异性抗原,诱导机体产生血清群特异性抗体[3,11-13]。我们以制备的CHUV多克隆抗体对感染CHUV、BCV与DAV的细胞进行免疫荧光染色,虽然感染细胞都可产生特异性荧光,但CHUV的荧光强度明显强于BCV与DAV。我们分析CHUV的阳性血清中既含有针对VP7的群特异性抗体,也含有针对CHUV VP2与VP5的特异性抗体,因此免疫荧光试验结果显示出CHUV感染细胞的免疫荧光信号强,而对BCV与DAV感染细胞的荧光信号相对较弱。血清中和试验显示CHUV、DAV与BCV阳性血清之间无交叉中和活性进一步证实了这一点。试验结果提示获取PALV VP7蛋白的单克隆抗体是研究PALV群特异性血清/抗原诊断检测方法的首选[26]。
本研究报道了2012—2016年中国PALV的分离与Seg-2、Seg-7序列特征,并首次报道了PALV的DAV与BCV两种血清型病毒在我国的分离。研究结果为我们进一步开展中国PALV的遗传学特征、感染与致病性、诊断方法的研究提供了材料。在下一步的研究中,我们将进行三方面的工作:(1)对中国分离的PALV代表毒株进行全基因组测序,获取中国分离CHUV、BCV与DAV代表毒株的遗传背景,分析病毒的变异特点以及毒株之间的基因重配[16]。(2)纯化出PALV病毒,建立PALV的血清学诊断方法,开展血清学调查。(3)开展不同血清型PALV毒株对牛羊等动物的感染特性与致病性分析,特别是研究动物发生多种血清型PALV感染或者与BTV混合感染时病毒的感染特性与致病性特点。
4 讨 论
2012—2016年在云南、广西、广东监控动物牛、羊血液中共计分离出19株 PALV,表明PALV流行于我国南方地区。对分离株的Seg-2序列分析与BHK-21细胞上的交叉中和试验结果显示,分离毒株分属Chuzan virus(CHUV)、D’Aguilar virus(DAV)与Bunyip Creek virus(BCV)三种血清型,分别分离到13、3和4株,PALV三种不同血清型的抗体之间无交叉保护作用。对Seg-7序列分析显示,该基因节段在不同血清型毒株间高度保守,基因序列的变异体出现明显的地域特征。首次报道了DAV与BCV两种血清型病毒在中国的分离。
参考文献(References):
[1] WHISTLER T, SWANEPOEL R, ERASMUS B J. Characterization of Palyam serogroup orbiviruses isolated in South Africa and serologic evidence for their widespread distribution in the country[J].EpidemiolInfect, 1989, 102(2): 317-324.
[2] 刘胜利, 黄冠胜, 赵增连, 等. 动物虫媒病与检验检疫技术[M]. 北京: 科学出版社, 2011.
LIU S L, HUANG G S, ZHAO Z L, et al. Animal vector-borne diseases and inspection & quarantine technology[M]. Beijing: Sciences Press, 2011. (in Chinese)
[3] WHISTLER T, SWANEPOEL R. Characterization of potentially foetotropic Palyam serogroup orbiviruses isolated in Zimbabwe[J].JGenVirol, 1988, 69(Pt 9): 2221-2227.
[4] MIURA Y, GOTO Y, KUBO M, et al. Isolation of Chuzan virus, a new member of the Palyam subgroup of the genusOrbivirus, from cattle andCulicoidesoxystomain Japan[J].AmJVetRes, 1988, 49(12): 2022-2025.
[5] TATEYAMA S, YAMAGUCHI R, UCHIDA K, et al. An outbreak of congenital hydranencephaly and cerebellar hypoplasia among calves in South Kyushu, Japan: a pathological study[J].ResVetSci, 1990, 49(2): 127-131.
[6] KIM Y H, KWEON C H, TARK D S, et al. Development of inactivated trivalent vaccine for the teratogenic Aino, Akabane and Chuzan viruses[J].Biologicals, 2011, 39(3): 152-157.
[7] HECHINGER S, WERNIKE K, BEER M. Evaluating the protective efficacy of a trivalent vaccine containing Akabane virus, Aino virus and Chuzan virus against Schmallenberg virus infection[J].VetRes, 2013, 44: 114.
[8] LIM S I, KWEON C H, TARK D S, et al. Sero-survey on Aino, Akabane, Chuzan, bovine ephemeral fever and Japanese encephalitis virus of cattle and swine in Korea[J].JVetSci, 2007, 8(1): 45-49.
[9] KATO T, SHIRAFUJI H, TANAKA S, et al. Bovine arboviruses inCulicoidesbiting midges and sentinel cattle in southern Japan from 2003 to 2013[J].TransboundEmergDis, 2016, 63(6): e160-e172.
[10] OHASHI S, YOSHIDA K, YANASE T, et al. Analysis of intratypic variation evident in an Ibaraki virus strain and its epizootic hemorrhagic disease virus serogroup[J].JClinMicrobiol, 2002, 40(10): 3684-3688.
[11] STUART D I, GRIMES J M. Structural studies on orbivirus proteins and particles[J].CurrTopMicrobiolImmunol, 2006, 309: 221-244.
[12] YAMAKAWA M, FURUUCHI S, MINOBE Y. Molecular characterization of double-stranded RNA segments encoding the major capsid proteins of a Palyam serogroup orbivirus that caused an epizootic of congenital abnormalities in cattle[J].JGenVirol, 1999, 80(Pt 1): 205-208.
[13] YAMAKAWA M, KUBO M, FURUUCHI S. Molecular analysis of the genome of Chuzan virus, a member of the Palyam serogroup viruses, and its phylogenetic relationships to other orbiviruses[J].JGenVirol, 1999, 80(Pt 4): 937-941.
[14] WHISTLER T, SWANEPOEL R. Proteins of Palyam serogroup viruses[J].JGenVirol, 1990, 71(Pt 6): 1333-1338.
[15] YAMAKAWA M, OHASHI S, KANNO T, et al. Genetic diversity of RNA segments 5, 7 and 9 of the Palyam serogroup orbiviruses from Japan, Australia and Zimbabwe[J].VirusRes, 2000, 68(2): 145-153.
[16] OHASHI S, MATSUMORI Y, YANASE T, et al. Evidence of an antigenic shift among Palyam serogroup orbiviruses[J].JClinMicrobiol, 2004, 42(10): 4610-4614.
[17] YANG H, XIAO L, MENG J X, et al. Complete genome sequence of a Chuzan virus strain isolated for the first time in mainland China[J].ArchVirol, 2016, 161(4): 1073-1077.
[18] WANG F, LIN J, CHANG J T, et al. Isolation, complete genome sequencing, and phylogenetic analysis of the first Chuzan virus in China[J].VirusGenes, 2016, 52(1): 138-141.
[19] MAAN S, RAO S J, MAAN N S, et al. Rapid cDNA synthesis and sequencing techniques for the genetic study of bluetongue and other dsRNA viruses[J].JVirolMethods, 2007, 143(2): 132-139.
[20] TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J].MolBiolEvol, 2013, 30(12): 2725-2729.
[21] SAEGERMAN C, BERKVENS D, MELLOR P S. Bluetongue epidemiology in the European Union[J].EmergInfectDis, 2008, 14(4): 539-544.
[22] CLAVIJO A, HECKERT R A, DULAC G C, et al. Isolation and identification of bluetongue virus[J].JVirolMethods, 2000, 87(1-2): 13-23.
[23] MAAN N S, MAAN S, BELAGANAHALLI M N, et al. Identification and differentiation of the twenty six bluetongue virus serotypes by RT-PCR amplification of the serotype-specific genome segment 2[J].PLoSOne, 2012, 7(2): e32601.
[24] MAAN S, MAAN N S, SAMUEL A R, et al. Analysis and phylogenetic comparisons of full-length VP2 genes of the 24 bluetongue virus serotypes[J].JGenVirol, 2007, 88: 621-630.
[25] MACLACHLAN N J. Bluetongue: history, global epidemiology, and pathogenesis[J]PrevVetMed, 2011, 102(2): 107-111.
[26] YAMAKAWA M, FURUUCHI S. Expression and antigenic characterization of the major core protein VP7 of Chuzan virus, a member of the Palyam serogroup orbiviruses[J].VetMicrobiol, 2001, 83(4): 333-341.
