ADIPOQ基因单体型与不同性别绵羊生长性状的关联分析
2018-05-07安清明周辉通李少斌罗玉柱JonHickford
安清明,周辉通,刘 秀,李少斌,罗玉柱*,Jon G.Hickford*
(1.铜仁学院乌江学院,铜仁 554300; 2.甘肃农业大学动物科学技术学院,甘肃草食动物生物技术重点实验室,兰州 730070; 3.林肯大学基因标记实验室,林肯 7646)
近年来,如何提高畜禽的生产性能并改善其产品质量已逐渐成为动物学家们的主要研究目标,而从人类营养学角度来分析,畜禽脂肪组成成分的研究已成一种有效的手段。其中,脂肪组织因能够分泌具有生物活性的脂肪因子而调节机体能量代谢,因此受到一定的关注,在脂肪因子调控过程中,包括了诸多相关基因的调控。脂联素(Adiponetin,ADIPOQ、Acrp30或APM1)是一种脂肪细胞因子家族的成员,主要由ADIPOQ基因编码,ADIPOQ基因最初于人类染色体上发现,定位于3q27,全长约17 kb,包含3个外显子和2个内含子[1-2],其CDS区共编码247个氨基酸。ADIPOQ蛋白分子量约为30 ku,主要包括4个结构域:信号肽、可变区、N-胶原蛋白三螺旋区域和C-球状区域。ADIPOQ主要分为两种不同的蛋白:低分子量(LMW:low-molecular-weight)与高分子量(HMW:hight-molecular-weight)蛋白,其不同的结构特性使得两种不同蛋白在机体内具有各自独特的生物学功能[3-4]。
ADIPOQ主要通过与ADIPOQ受体(ADIPOR-1和ADIPOR-2)结合协同调节其生理功能。研究表明,ADIPOQ通过与ADIPOR相结合,能够调节AMP激酶与PPAR配体活性,间接调节脂肪酸氧化和糖类摄取[5],也参与胰岛素的分泌调节[6]。同时,有研究表明,ADIPOQ可以通过调节PPAR-α刺激肌肉组织及肾中的脂肪酸氧化、减少甘油三酯的含量,从而改变生物体对胰岛素的敏感度[7]。在家畜中,ADIPOQ基因遗传变异对生产性状具有一定影响。研究发现,猪ADIPOQ基因定位于13号染色体,能够影响猪的背脂肪厚度、背最长肌脂肪酸链长度的相关QTL区域[8-9],同时发现,启动子区核苷酸c.-67G/A和c.-892C/T变异对猪胴体性状及肉质具有显著影响[10],内含子g.1735A/G与猪肩部脂肪量具有显著相关性[11];牛ADIPOQ启动子区核苷酸c.-176A/G与胴体性状有相关性,c.-199C/T,c.-34G/A与背膘厚、眼肌面积具有一定相关性[12-13]。
绵羊ADIPOQ基因多态性变异已有报道,但其相关变异与绵羊生产性状关联性和性别差异关联性的研究却少有报道。本研究以新西兰罗姆尼羊为研究对象,通过PCR-SSCP技术对罗姆尼羊群体进行遗传变异及相关性分析,探究ADIPOQ基因单体型对不同性别罗姆尼羊生长性状的影响。
1 材料与方法
1.1 试验样品及表型测定
试验于2014年6月—2015年8月在新西兰林肯大学基因标记实验室完成。1 185只新西兰罗姆尼羊来自于新西兰南岛同一牧场的17只优秀种公羊的子一代。羔羊出生12 h之内佩戴耳标,测定记录初生重、耳号、出生日期、性别和出生等级(单羔、双羔或三羔)。3周龄左右断尾并记录断尾重,同时用FTA卡收集血样。3月龄断奶,测定断奶重,并计算所有羔羊断奶前生长速度。由于在后期的相关性状测定及单体型分析中,部分羔羊缺乏相关表型数据,因此,参与结果分析的具体羔羊数量与总数量存在一定差异。
1.2 试验方法
1.2.1 基因组DNA提取 羔羊断尾时用FTA卡采集血液,父本公羊从颈静脉采血滴于FTA卡,自然晾干后置于阴凉处保存待用。基因组DNA采用H. Zhou等[14]所描述的两步法提取。
1.2.2 引物设计及PCR扩增 根据绵羊全基因组序列中ADIPOQ基因序列(GenBank 登录号:NC_019458.1),应用Primer 5.0自行设计2对不同区域引物(启动子区和部分外显子3区),用于基因多态性、单体型及相关性分析,引物由Integrated DNA Technologies(Coralville, IA, USA)合成。引物1:F:5′-TTCCTGCTTCTGATCTTGACC-3′;R:5′-CAGCCTAGAAATTGAATCAGTC-3′;引物2:F: 5′-GGTCTTCTTGTTCTCTAGGTC-3′;R:5′-TGGTCCACGTTCTGGTTCTG-3′。
PCR反应体系(20 μL):其中1.2 mm DNA disk模板1个,2 μL 10×buffer缓冲液,2 μL 5×Q溶液,1.2 μL 3 μmol·L-1MgCl2溶液,1.2 μL 150 μmol·L-1dNTPs,1 μL 0.25 μmol·L-1上下游混合引物,0.1 μL 0.5 UTaq聚合酶和12.5 μL ddH2O。
PCR扩增条件:94 ℃预变性2 min;94 ℃变性30 s,退火(引物1和引物2均为58 ℃)30 s,72 ℃延伸30 s,共37个循环;最后72 ℃延伸5 min,4 ℃保存。PCR产物用1%琼脂糖凝胶电泳检测。
1.2.3 PCR产物的SSCP检测 在20 μL PCR扩增产物中加入80 μL变性缓冲液(98%去离子甲酰胺、10 mmol·L-1EDTA、0.025%二甲苯氰、0.025%溴酚蓝),经105 ℃热变性5 min后立即置于冰水混合物中,然后将10 μL变性产物上样于不同浓度聚丙烯酰胺凝胶中,在相应引物所需条件下进行SSCP电泳(引物1:300 V电压17.0 ℃温控环境电泳19 h;引物2:200 V电压20.0 ℃温控环境电泳19 h)。结束后根据S. O. Byun等[15]描述具体步骤进行银染显色,判断相应基因型。
1.2.4 单体型判定 若所检测子代两个不同区域之间的基因型有任何一段区域的基因型为纯合型,可直接判定ADIPOQ基因两段区域之间的单体型。如:子代区域1的基因型为A1A1,而区域2的基因型为A2B2,那么子代的单体型可直接推断为A1-A2和A1-B2。若所检测的子代两个不同区域之间的基因型均为杂合型,那么可根据父本单体型及同一公羊生育子代单体型来判断该子代单体型。如父本的单体型为A1-B2/B1-C2,那么子代有50%的概率继承其中的一条单体型,同时根据同一父本产生后代来推断该子代单体型。
1.3 数据分析
本研究利用Mintab (Version 16,Minitab Inc.,Pennsylavania) 一般线性混合效应模型评估特定单体型的存在/缺失对生长性能的影响。对某一特定性状存在(1)、缺失(0)分析模型中,单体型、家系影响、出生等级/初生重为固定因素,遵从下列模型进行最小二乘方差分析:
Yiknm=μ+Gi+Mk+Fm+Xn+eiknm
其中,Yiknm为相应性状表型值,μ为群体均值,Gi为家系效应,Mk为出生等级效应/初生重(取决于谁对模型更具影响力),Fm为单体型或双体型,Xn为因素间互作效应,eiknm为随机误差。
在进行特定单体型存在/缺失分析时,对P<0.2的单体型要进行互作效应校正相关性数据。如进行ADIPOQ基因某一单体型对初生重的相关分析时,若单体型A1-A2、A1-B2和B1-C2的P值分别为0.003、0.014和0.450时,那么应在模型中同时校正分析单体型A1-A2和A1-B2对初生重的影响P值。在分析双体型对性状的影响时,只对频率大于10.00%的双体型进行分析。所有数据均采用“平均值±标准误”表示,P<0.05 为显著水平,P<0.2 为有影响趋势,P> 0.2 为无影响。
2 结 果
2.1 不同性别羔羊ADIPOQ基因单体型频率分析
在所检测的1 185只羔羊中,共检测到8种由区域1中的4个等位基因(A1、B1、C1和D1;已提交GenBank,序列号为:KP903754~KP903757)和区域2中的3个等位基因(A2、B2和C2;已提交GenBank,序列号为:KP903762~KP903764)构成的不同单体型,分别为A1-A2、A1-B2、A1-C2、B1-A2、B1-B2、B1-C2、D1-A2和D1-C2(其余推断也应存在的4种单体型在本试验没有检测到),单体型分型示意结果见图1,SSCP检测结果见图2。本试验所检测到的8种单体型中A1-A2、A1-C2、B1-A2和B1-C2最为普遍,其它4种单体型较为稀少(频率小于5.00%),且单体型D1-A2和D1-C2只在母羔中检测到,具体频率见表1。
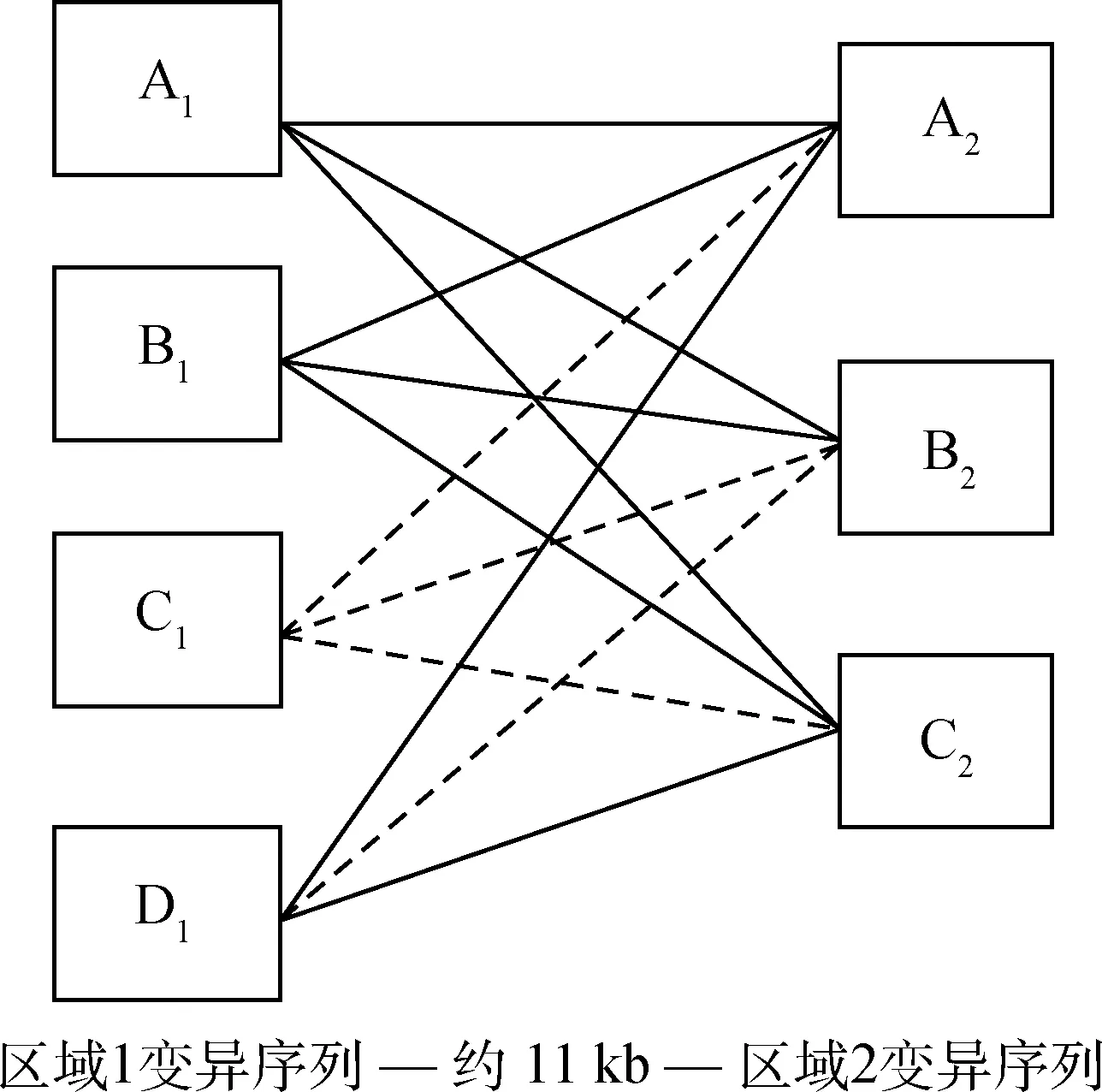

----代表本试验检测到的单体型;代表本试验没有检测到的单体型----Represents the haplotypes detected in this study; Represents the haplotypes not detected in this study图1 罗姆尼羊ADIPOQ基因单体型Fig.1 Haplotypes of ADIPOQ in Romney sheep
表1不同性别羔羊ADIPOQ基因横跨区域1与区域2的单体型频率
Table1HaplotypesfrequenciesofmaleandfemalelambsADIPOQspanningregion1toregion2
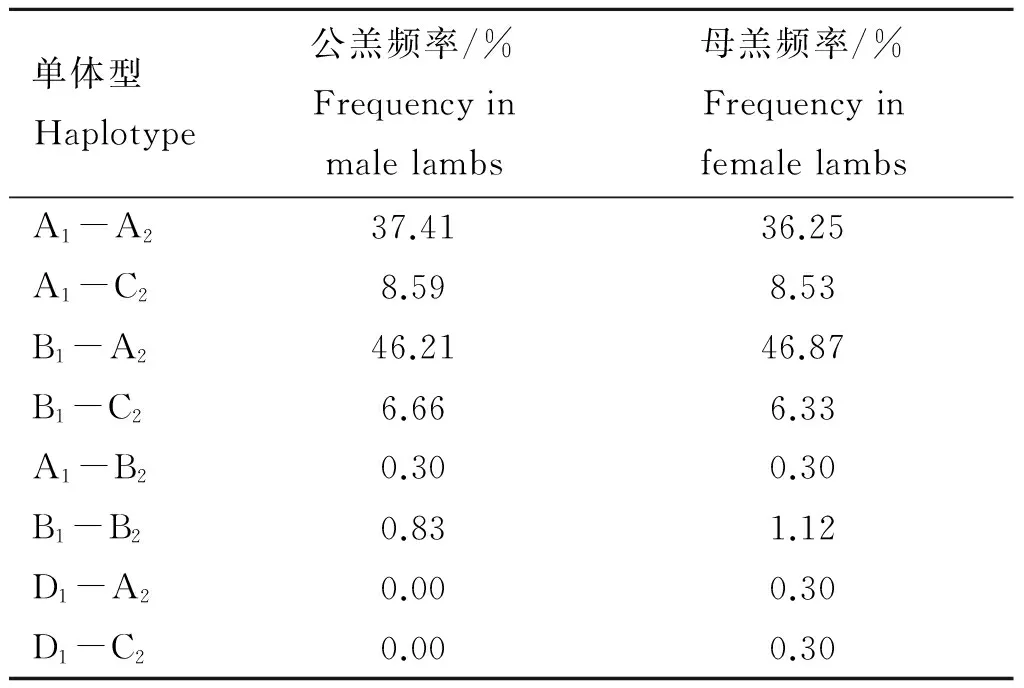
单体型Haplotype公羔频率/%Frequencyinmalelambs母羔频率/%FrequencyinfemalelambsA1-A237.4136.25A1-C28.598.53B1-A246.2146.87B1-C26.666.33A1-B20.300.30B1-B20.831.12D1-A20.000.30D1-C20.000.30
2.2 不同性别羔羊ADIPOQ基因单体型与生长性状关联性分析
本试验所检测到的8种单体型中,A1-B2、B1-B2、D1-A2和D1-C2在罗姆尼羊中的频率小于5.00%,样本较为稀少,不能用于相关性分析,因此,本试验只对单体型A1-A2、A1-C2、B1-A2和B1-C2进行性别差异的生长性状关联性分析,数据分析时将所有公羔和母羔置于同一群体模型中与本试验结果分析比较校正,将性别作为影响因素进行GLMs混合模型分析,结果表明,所有检测单体型均对羔羊生长性状无显著影响(结果未列出)。
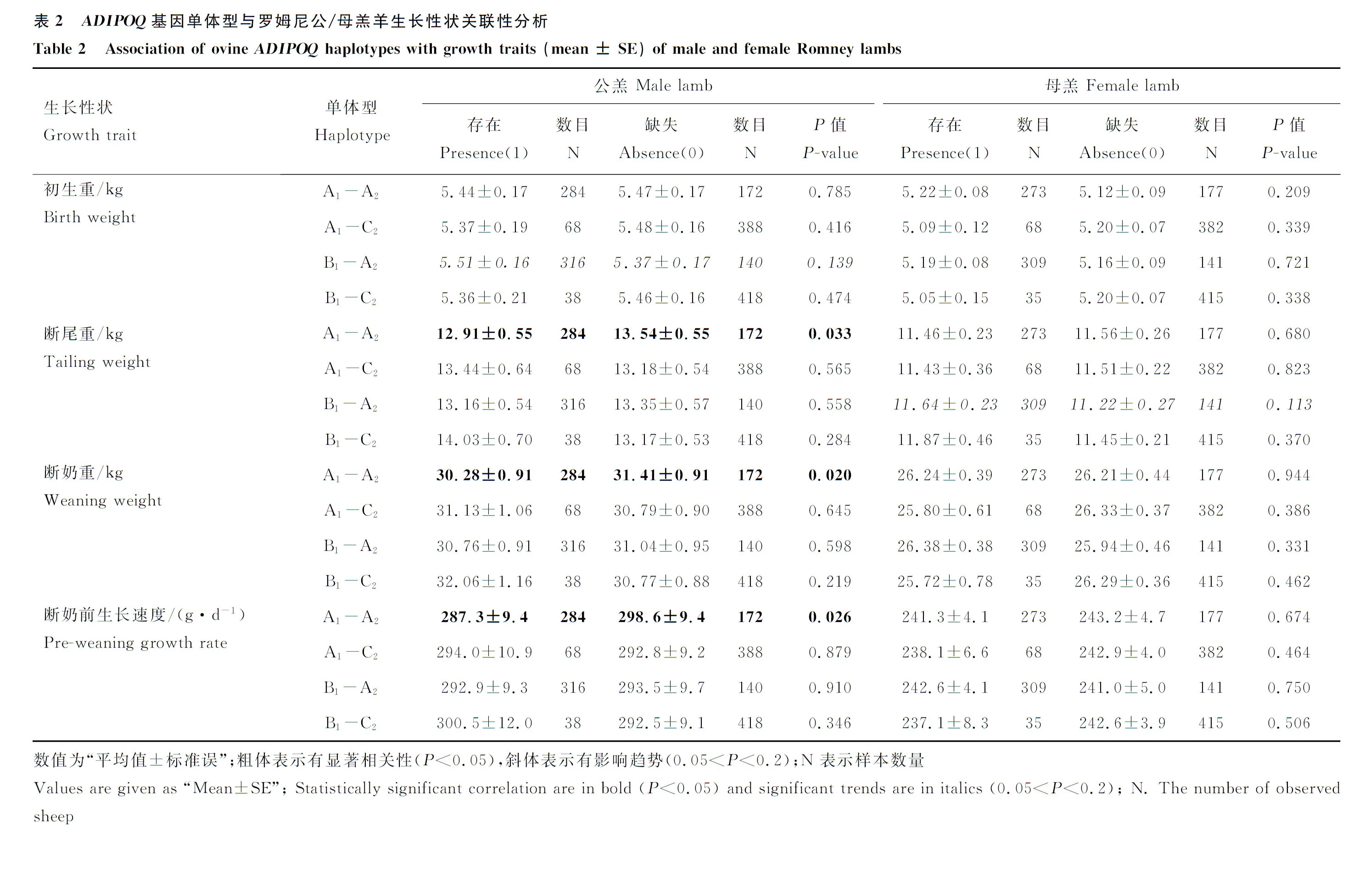
对具有完整数据及基因单体型特征的456只公羔和450只母羔进行单体型存在与缺失分析,结果表明,公羔中存在单体型A1-A2的群体具有较低的断尾重(存在:(12.91±0.55) kg,缺失:(13.54±0.55) kg;P=0.033),断奶重(存在:(30.28±0.91) kg,缺失:(31.41±0.91) kg;P=0.020)和断奶前生长速度(存在:(287.3±9.4) g·d-1,缺失:(298.6±9.4) g·d-1;P=0.026);未发现单体型A1-A2与羔羊初生重有显著相关性。其它3种单体型在公羔中均没有发现与罗姆尼羊生长性状具有显著相关性。在母羔中,4种单体型均没有发现与生长性状具有显著相关性(表2)。
双体型检测结果表明,公、母羔的生长性状与双体型之间均没有显著相关性(结果未列出)。
3 讨 论
单体型(Haplotype)是指在遗传学上同一染色体上多个基因座位或一个基因中多个位点共同连锁的变异,具有比单核苷酸变异更高的多态性信息含量,能够运用连锁的多个基因或位点进行协同基因标记,因此,单体型较单个SNP位点具有更高的遗传标记意义及可靠性[16-18]。
本研究检测了绵羊ADIPOQ基因不同区域单体型的变异情况,同时分析了不同性别羔羊ADIPOQ基因单体型差异对生长性状的影响。本试验选取间隔约11 kb的两段区域进行单体型检测,理论上来讲,两段区域可以共建12种不同单体型,但本试验中只检测到8种不同单体型,与Q.M.An等[19]已报道的单体型稍有不同,Q.M.An等检测到D1-C3的单体型在本研究中没有检测到与之相对应的单体型,且另外4种单体型本试验中亦没有检测到,这可能由于本研究只对单一绵羊品种(罗姆尼羊)进行单体型检测,也可能是由于该单体型在罗姆尼羊中存在的频率较低,本研究所选用的群体中没有包含,或者是该单体型对绵羊的经济性状有一定的影响,牧民在长期人工选择的过程中已将含有该单体型的羊只淘汰,因此,在更多绵羊群体或种群中进一步研究绵羊ADIPOQ基因单体型的进化史和变异是必要的,其或许能更进一步证实本研究的结果。
本试验检测得到的10个SNPs位点中7个位于启动子区、3个位于编码区,虽然目前很难具体确定启动子区域内核苷酸突变的具体生物学功能,但已有研究表明,在人类ADIPOQ基因启动子区域,存在许多具有一定生理学功能的位点,如SP1、SREBP、AP1及C/EBP等位点[20]。本研究在启动子区所检测到的突变位点与人类ADIPOQ基因检测到的突变位点(c.-11377C/G、c.-19166T/G、c.-11426A/G和c.-11391G/A)位置极其相近,其中核苷酸突变位点c.-11377C/G已被确认存在于SP1结合位点上,且其碱基G突变导致SP1结合位点的改变,能够降低ADIPOQ的生物活性[21];而其它3个 核苷酸突变位点(c.-19166T/G、c.-11426A/G和c.-11391G/A)已被检测与II型糖尿病及肥胖有一定的关联性[22]。3个位于编码区的核苷酸突变中2个核苷酸突变(c.225和c.387)为同义突变,另外1个(c.515G/A)为非同义突变,其突变导致了172位赖氨酸(Lys)/精氨酸(Arg)改变,且该氨基酸突变位于ADIPOQ蛋白的球状结构域中,其可能会改变ADIPOQ蛋白的空间结构,进而可能会影响到蛋白的生物学功能。如已有研究表明,人类ADIPOQ基因第2外显子rs2241766 G/T和第3外显子rs17366743 C/T的核苷酸突变与多个人群的ADIPOQ表达水平有一定关联性[23],因此,本研究中发现的3个编码区突变值得进一步确定其具体的生理功能。
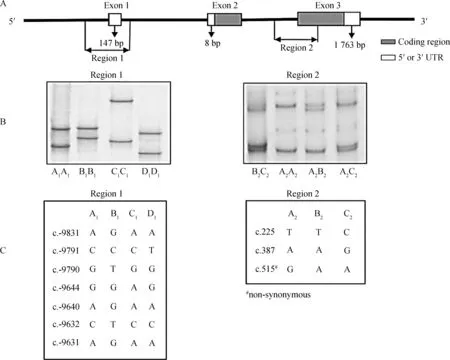
A. ADIPOQ基因扩增区域示意图;B. 2个不同区域的特异扩增ADIPOQ基因对应SSCP示意图;C. 所检测2个区域SNPs位点示意图A. Diagram showing the regions of ADIPOQ amplified; B. PCR-SSCP banding patterns for the two regions of ovine ADIPOQ amplified; C. The SNPs detected for ovine ADIPOQ in the two regions图2 ADIPOQ基因SSCP检测示意图Fig.2 Diagram of SSCP detection of ADIPOQ gene
本试验共检测到8种单体型,且其在公羔与母羔群体之间的分布存在差异。单体型D1-A2和D1-C2只在本试验母羔群体中检测到。这可能是本试验的公羔群体数量较少,没有检测到该单体型,或者是由于这两种单体型在绵羊群体中本身就存在性别差异。而单体型与生长性状关联性分析结果表明,单体型A1-A2与公羔群体的断尾重、断奶重及断奶前生长速度存在显著关联性,而对母羔群体的生长性状无显著影响,这进一步表明,ADIPOQ基因单体型在绵羊群体中存在性别差异。人类ADIPOQ
基因性别差异研究结果表明,在非洲裔美国人群体中,ADIPOQ基因变异对其在机体中表达水平与肥胖具有性别差异,其变异与非洲裔美国人群体的妇女肥胖有一定关联性[24]。同样,目前在一些对肌肉生长具有调控作用的基因中也发现其存在一定的性别差异性,如S. Reisz-Porszasz等[25]发现,MSTN基因只在雄性小鼠中发现,可能对其肌肉具有抑制作用,J. Han等[26]发现,MSTN基因变异在罗姆尼羊中存在性别比例差异。沈留红等[27]发现,脂联素(ADIPOQ基因)mRNA和蛋白表达水平对奶牛犊牛的初生重有较大影响,Y. S. Lee等[28]发现,WFIKKN2基因变异与不同时期小鼠的肌肉重存在性别关联性差异,J.Q.Wang等[29]发现,WFIKKN2基因变异与罗姆尼羊生长性状存在性别差异。因此,可以推断本研究分析所得ADIPOQ基因单体型对绵羊生长性状的影响具有性别差异有一定依据,有必要进一步加大群体数量研究其变异在在性别差异中的有效机制,从而验证本研究的结果。
单体型与生长性状关联性分析结果表明,单体型与公羔群体断尾重、断奶重和断奶前生长速度存在显著关联性,且存在单体型A1-A2的公羔群体较缺失群体有较低的断尾重、断奶重和断奶前生长速度。若羔羊在100日龄断奶,且忽略初生重之间的差异,那么存在单体型A1-A2的群体断奶重较缺失群体少1.13 kg,生长速度每天少增长11.3 g,即从出生到断奶每只绵羊平均少增长1.13 kg体重。而在实际育种生产过程中,一般在断奶期屠宰体重达标的公羔,降低群体饲养成本提高收益,而将母羔留作繁殖母体进行群体繁殖,从而提高实际收益及育种效率。因此,本研究结果在实际生产中,运用单体型A1-A2的存在与否,可在绵羊育种过程中筛选出断奶前生长速度较快的公羔群体,从而提高绵羊生产性能,增加经济收益。
4 结 论
本研究通过绵羊ADIPOQ基因单体型检测,共发现ADIPOQ基因的8种单体型构型,且其对绵羊生长性状的影响存在性别差异,结果表明,单体型只对绵羊公羔群体生长性能存在显著影响,该结果在生产育种中对提高公羔生长性能、增加屠宰产出具有理论指导意义。
参考文献(References):
[1] SCHERER P E, WILLIAMS S, FOGLIANO M, et al. A novel serum protein similar to C1q, produced exclusively in adipocytes[J].JBiolChem, 1995, 270(45): 26746-26749.
[2] HSUEH W C, ST. JEAN P L, MITCHELL B D, et al. Genome-wide and fine-mapping linkage studies of type 2 diabetes and glucose traits in the Old Order Amish: evidence for a new diabetes locus on chromosome 14q11 and confirmation of a locus on chromosome 1q21-q24[J].Diabetes, 2003, 52(2): 550-557.
[3] PAJVANI U B, DU X L, COMBS T P, et al. Structure-function studies of the adipocyte-secreted hormone Acrp30/adiponectin implications for metabolic regulation and bioactivity[J].JBiolChem, 2003, 278(11): 9073-9085.
[4] WANG Z V, SCHRAW T D, KIM J Y, et al. Secretion of the adipocyte-specific secretory protein adiponectin critically depends on thiol-mediated protein retention[J].MolCellBiol, 2007, 27(10): 3716-3731.
[5] YAMAUCHI T, KAMON J, ITO Y, et al. Cloning of adiponectin receptors that mediate antidiabetic metabolic effects[J].Nature, 2003, 423(6941): 762-769.
[6] KHARROUBI I, RASSCHAERT J, EIZIRIK D, et al. Expression of adiponectin receptors in pancreatic β cells[J].BiochemBiophysResCommun, 2003, 312(4): 1118-1122.
[7] YAMAUCHI T, HARA K, KUBOTA N, et al. Dual roles of adiponectin/Acrp30invivoas an anti-diabetic and anti-atherogenic adipokine[J].CurrDrugTargetsImmuneEndocrMetabolDisord, 2003, 3(4): 243-254.
[8] LIU G, JENNEN D G J, THOLEN E, et al. A genome scan reveals QTL for growth, fatness, leanness and meat quality in a Duroc-Pietrain resource population[J].AnimGenet, 2007, 38(3): 241-252.
[9] GUO T, REN J, YANG K, et al. Quantitative trait loci for fatty acid composition in longissimus dorsi and abdominal fat: results from a White Duroc × Erhualian intercross F2population[J].AnimGenet, 2009, 40(2): 185-191.
[10] CIESLAK J, FLISIKOWSKA T, SCHNIEKE A, et al. Polymorphisms in the promoter region of the adiponectin (ADIPOQ) gene are presumably associated with transcription level and carcass traits in pigs[J].AnimGenet, 2013, 44(3): 340-343.
[11] DAI L H, XIONG Y Z, DENG C Y, et al. Association of the A-G polymorphism in porcine adiponectin gene with fat deposition and carcass traits[J].Asian-AustralasJAnimSci, 2006, 19(6): 779-783.
[12] MORSCI N S, SCHNABEL R D, TAYLOR J F. Association analysis ofadiponectinandsomatostatinpolymorphisms on BTA1 with growth and carcass traits in Angus cattle[J].AnimGenet, 2006, 37(6): 554-562.
[13] SHIN S, CHUNG E. Novel SNPs in the bovineADIPOQandPPARGC1Agenes are associated with carcass traits in Hanwoo (Korean cattle)[J].MolBiolRep, 2013, 40(7): 4651-4660.
[14] ZHOU H, HICKFORD J G H, FANG Q. A two-step procedure for extracting genomic DNA from dried blood spots on filter paper for polymerase chain reaction amplification[J].AnalBiochem, 2006, 354(1): 159-161.
[15] BYUN S O, FANG Q, ZHOU H, et al. An effective method for silver-staining DNA in large numbers of polyacrylamide gels[J].AnalBiochem, 2009, 385(1): 174-175.
[16] MORRIS R W, KAPLAN N L. On the advantage of haplotype analysis in the presence of multiple disease susceptibility alleles[J].GenetEpidemiol, 2002, 23(3): 221-233.
[17] ZHOU H T, HICKFORD J G H, FANG Q, et al. Allelic variation of the ovine Toll-like receptor 4 gene[J].DevCompImmunol, 2007, 31(2): 105-108.
[18] GONG H, ZHOU H T, YU Z D, et al. Identification of the ovine keratin-associated protein KAP1-2 gene (KRTAP1-2)[J].ExpDermatol, 2011, 20(10): 815-819.
[19] AN Q M, ZHOU H T, HU J, et al. Haplotypes and sequence variation in the ovine adiponectin gene (ADIPOQ)[J].Genes, 2015, 6(4): 1230-1241.
[20] BARTH N, LANGMANN T, SCHÖLMERICH J, et al. Identification of regulatory elements in the human adipose most abundant gene transcript-1 (apM-1) promoter: role of SP1/SP3 and TNF-α as regulatory pathways[J].Diabetologia, 2002, 45(10): 1425-1433.
[21] ZHANG D Y, MA J, BRISMAR K, et al. A single nucleotide polymorphism alters the sequence of SP1 binding site in the adiponectin promoter region and is associated with diabetic nephropathy among type 1 diabetic patients in the Genetics of Kidneys in Diabetes Study[J].JDiabetesComplications, 2009, 23(4): 265-272.
[22] CHUNG H F, LONG K Z, HSU C C, et al. Adiponectin gene (ADIPOQ) polymorphisms correlate with the progression of nephropathy in Taiwanese male patients with type 2 diabetes[J].DiabetesResClinPract, 2014, 105(2): 261-270.
[23] GU H F. Biomarkers of adiponectin: plasma protein variation and genomic DNA polymorphisms[J].BiomarkInsights, 2009, 4: 123-133.
[24] RIESTRA P, GEBREAB S Y, XU R H, et al. Gender-specific associations betweenADIPOQgene polymorphisms and adiponectin levels and obesity in the Jackson Heart Study cohort[J].BMCMedGenet, 2015, 16: 65.
[25] REISZ-PORSZASZ S, BHASIN S, ARTAZA J N, et al. Lower skeletal muscle mass in male transgenic mice with muscle-specific overexpression of myostatin[J].AmJPhysiolEndocrinolMetab, 2003, 285(4): E876-E888.
[26] HAN J, ZHOU H, FORREST R H, et al. Effect of Myostatin (MSTN) g+6223G > A on production and carcass traits in New Zealand Romney Sheep[J].Asian-AustralasJAnimSci, 2010, 23(7): 863-866.
[27] 沈留红, 江 涛, 巫晓峰, 等. 奶牛胎盘脂联素、瘦素、内脂素与犊牛初生重相关性研究[J]. 畜牧兽医学报, 2017, 48(1): 185-192.
SHEN L H, JIANG T, WU X F, et al. The correlation between adiponectin, leptin, visfatin in placenta and calf birth weight[J].ActaVeterinariaetZootechnicaSinica, 2017, 48(1): 185-192. (in Chinese)
[28] LEE Y S, LEE S J. Regulation of GDF-11 and myostatin activity by GASP-1 and GASP-2[J].ProcNatlAcadSciUSA, 2013, 110(39): E3713-E3722.
[29] WANG J Q, ZHOU H T, FANG Q, et al. Effect of variation in ovineWFIKKN2 on growth traits appears to be gender-dependent[J].SciRep, 2015, 5: 12347.
