H1N1流感病毒HA蛋白抗原表位预测方法研究
2018-04-17郭春艳刘杨封青梁导艳孙晶莹李慧瑾赵向绒李研赵彭花胡军
郭春艳 刘杨 封青 梁导艳 孙晶莹 李慧瑾 赵向绒 李研 赵彭花 胡军
710068 西安,陕西省人民医院(郭春艳、刘杨、封青、梁导艳、孙晶莹、赵向绒、李研、赵彭花、胡军); 710068 西安交通大学医学院第三附属医院(郭春艳、刘杨、封青、梁导艳、孙晶莹、赵向绒、李研、赵彭花、胡军);710021 西安医学院(李慧瑾)
流感大流行对人类健康危害严重。流感病毒型别多,血凝素(Hemagglutinin, HA)抗原表位变异是造成流感反复流行的主要因素[1-4]。接种疫苗是预防流感的主要措施,但传统思路开发的疫苗往往滞后于疫情暴发[5]。研制能够诱导持久保护性免疫的通用流感疫苗对流感预防具有现实意义,而HA抗原表位预测工作是通用流感疫苗研究的重要基础之一。目前研究抗原表位的方法主要是计算机模拟技术和单克隆抗体(monoclonal antibody, mAb, 简称单抗)技术。Igaroshi等[6]采用计算机同源建模方法对2009年H1N1流感病毒HA抗原表位进行预测,并将HA抗原表位分为4个。应用生物信息学方法预测流感病毒HA的抗原表位成了主流,但是缺乏相应的实验室数据进行佐证[7]。
单抗能识别并结合单一的抗原表位,在生命科学研究领域中得到极为广泛的应用[8]。Rockman等[9]利用与H5N1各毒株具有广泛交叉反应性的单抗识别HA中和表位,详细说明了H5N1流感病毒抗原漂移的机制。本文以H1N1流感病毒裂解疫苗为免疫原,通过筛选制备3株识别HA蛋白的单抗,同时以单抗作为研究工具,从抗体相互阻断试验着手,并以计算机模拟方法对其验证,深入研究HA的抗原表位,其结果完善并补充了现有抗原表位预测方法,同时为阐明流感病毒免疫机制提供研究思路。
1 材料与方法
1.1主要试剂甲型H1N1流感病毒裂解疫苗(国药准字S20090015)购自华兰生物疫苗有限公司;RNA提取试剂盒(DP419)、cDNA第一链合成试剂盒(KR103)购自天根生化科技(北京)有限公司;DNA聚合酶、pMD19-T载体、DNA Marker为TaKaRa(日本)公司产品;SBA ClonotypingTMSystem/HRP单抗亚类鉴定试剂盒(5300-05)购自SouthernBiotech公司。
1.2单抗的制备和鉴定首次免疫:弗氏完全佐剂与流感裂解疫苗乳化后,进行皮下免疫,20~25μg/只;二次免疫:应用弗氏不完全佐剂,同首次免疫剂量和方法;加强免疫:融合前3 d,只用流感裂解抗原腹腔追加免疫。采用间接ELISA方法、有限稀释法进行3~4次亚克隆,筛选分泌抗体的阳性细胞株[10];BALB/c小鼠体内注入液体石蜡,7 d后腹腔注入筛选出的阳性杂交瘤细胞株制备腹水[11];然后采用辛酸-硫酸铵方法纯化腹水[12]。采用SBA ClonotypingTMSystem/HRP单抗亚类鉴定试剂盒测定,具体方法见说明书。
1.3单抗识别抗原位点分析用改良的过碘酸钠法将各株纯化的单抗标记HRP,间接ELISA方法测定酶标单抗的工作浓度(当待测单抗与抗原反应的OD450 nm值出现下降前的一个稀释度作为该抗体的工作浓度)。H1N1抗原包被96孔ELISA检测板,浓度为2 μg/ml;分别用过饱和的单抗与之反应进行封闭,150 μl/孔,37 ℃孵育1 h,常规洗涤;然后加入某一工作浓度的HRP标记单抗,100 μl/孔,以SP2/0作为阴性对照,37 ℃反应1 h后洗涤;TMB显色后2 mol/L浓硫酸终止反应,酶标仪检测OD450 nm值。根据(对照组-实验组)/对照组判定抑制率(IR)。其中IR小于或等于0.4为标记抗体和封闭抗体识别不相同的位点;IR大于或等于0.8为两株抗体识别的表位高度相关或一致;IR在0.4~0.8范围内代表识别的位点有相关性[13]。
1.4克隆3株单抗的轻链和重链可变区基因收集3株分泌抗H1N1流感病毒HA蛋白单抗的杂交瘤细胞株,提取RNA,逆转录合成cDNA,作为后续基因克隆的模板。对NCBI公布的小鼠单抗轻链和重链的可变区基因序列进行比对分析,共设计27条引物,见表1。用8条引物(7条上游引物+1条下游引物)PCR扩增单抗轻链可变区基因,用9条引物(5条上游引物+4条下游引物)PCR扩增单抗重链可变区基因。轻链基因约320 bp,重链基因约350 bp。将PCR扩增得到的产物再次进行PCR鉴定(轻链在扩增产物下游设计5条引物,重链在扩增产物上游设计5条引物)。轻链鉴定阳性目的条带约180 bp,重链鉴定为阳性的条带约150 bp。T载体与胶回收的阳性片段连接后转化DH5α大肠埃希菌,经PCR和酶切鉴定后测序,将测序正确的核苷酸序列翻译为氨基酸序列。
1.5计算机模拟分析HA蛋白与单抗结合的氨基酸位点操作方法如下:① 查找已经报告的抗体与流感病毒HA蛋白结合的晶体结构模板,并明确二者之间的结合表位;② 应用NCBI blast软件分别比对A1-12、H1-13、H1-15与晶体模板中抗体序列的同源性,以同源性最高的晶体模板中的抗体作为目标抗体;③ 找出该目标抗体与HA晶体结合的位点;④ 将本文所用的HA氨基酸序列与“③”中确定的HA晶体上的位点比对,以此分别确定三株单抗与HA蛋白的结合表位。
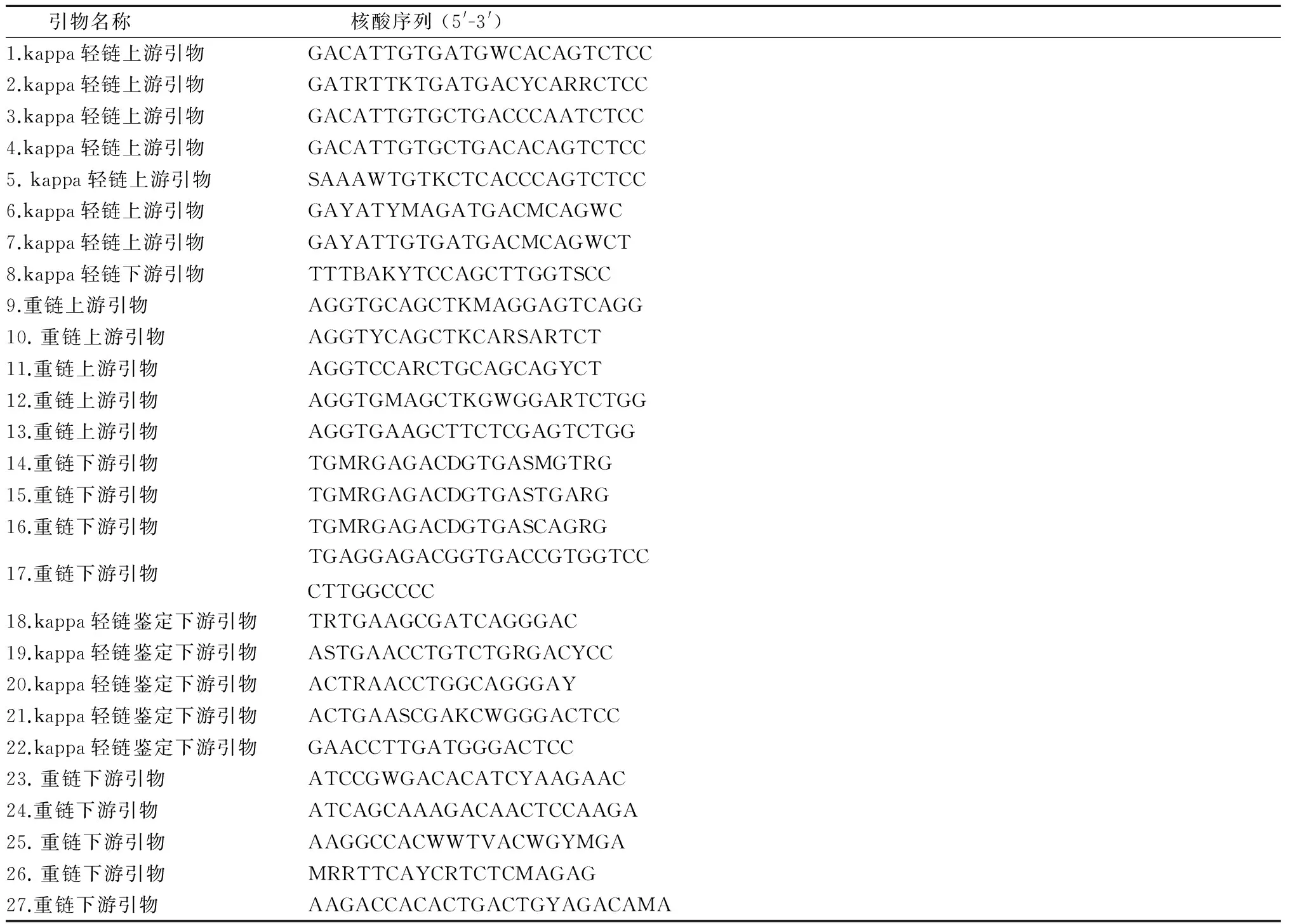
表1 小鼠单抗轻链和重链可变区引物
2 结果
2.1阳性单抗的建立及其特性鉴定应用现有的单抗融合技术,最终获得3株稳定分泌抗HA的单抗,分别命名为H1-13、H1-15、A1-12。应用单抗亚类鉴定试剂盒,测定3株单抗的型均为κ链,H1-15亚类为IgM,H1-13、A1-12亚类均为IgG1。
2.2单抗识别HA抗原表位分析将这3株单抗作为研究工具来研究抗原表位,具体结果见表2。H1-15与A1-12、H1-13间的反应抑制率均在40%以下,抑制不明显,抑制率越小,空间位置越远,表明其识别的位点可能不同,H1-15单独成为第一组。如先加H1-13,再加入HRP标记的A1-12,两者间的抑制率为27%,说明H1-13与A1-12识别两个不同的位点。如先加A1-12再加入HRP标记的H1-13,反应抑制率在93%,表明其识别的抗原表位具有相关性,推测其针对比较接近的2个不同的抗原表位,所以A1-12和H1-13成为第二组。
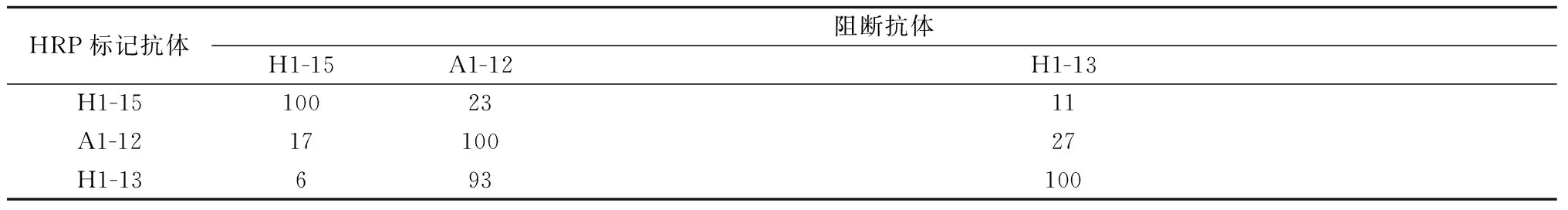
表2 3株单克隆抗体之间反应抑制率(%)
2.3抗体轻、重链可变区氨基酸序列分析分别克隆上述3株抗体细胞系的轻链和重链可变区基因序列VL和VH,见表3。
VH和VL配对时,各个高变区的环在分子表面形成抗原结合部位,由于抗体用这些高变环以表面互补的方式来结合抗原,一般又将这些高变环称为互补决定区(complementary-determining region, CDR),各个不同CDR的分别称作CDR1、CDR2和CDR3。在CDR之间的区域氨基酸序列的变化则较小,称为骨架区(framework region,FR),轻链和重链各有4个FR,即FR1,FR2,FR3,FR4。以H1-13为例进行说明,应用abYsis软件分析抗体的CDR区及FR区,具体见图1。
2.4分析单抗结合H1N1流感病毒HA上的氨基酸位点用计算机模拟方法分别分析3株单抗与2009年和1918年流感病毒结合的氨基酸位点,其中H1-13识别的位点与A1-12识别的位点非常相似,可将这两株抗体分为一组;H1-15识别的位点与另外两株截然不同,将其划为一组,具体预测结果见表4。
3 讨论
近几年,流感大流行严重威胁人们的生命安全,抗原表位预测是研究流感流行、致病及开发疫苗的重要工具,目前已经确定的HA表位非常有限。单克隆抗体技术在研究抗原表位方面应用日益广泛[14],计算机模拟技术研究抗原表位可以有效简化表位研究过程。因此,本研究以本实验室制备的3株抗H1N1流感病毒单抗为研究对象,联合应用ELISA阻断试验和计算机模拟技术,将这3株单抗识别的抗原表位区分为2类。两种方法预测的结果得到了相互印证,也进行了相互补充,间接验证了应用ELISA阻断试验预测抗体识别抗原表位方法的可行性[15]。

图1 H1-13抗体的轻链和重链可变区及骨架区基因序列Fig.1 Analysis of light and heavy chain variable region and skeleton region sequences of H1-13 antibody

单克隆抗体序列A1-12VHVQLVESGPGLVAPSQSLSITCTVSGFSLTDYGVNWVRQPPGKGLEWLGLIWGEGSTDYNSALKSRLSISKDNSKSQVFLKMNSLQTDDTARYYCARDKGINYWYFDVWGQGTTVTVSSVLDIVLTQSPLSLPVSLGDQASMSCRSSQSLVHSNGNTYLHWYLQKPGQSPKLLIHKVSNRFSGVPDRFSGSGSGTDFTLKISRVEAEDLGVYFCSQSTHVPPLTFGAGTKLEH1-13VHVQLEESGGGLVQPGGSLKLSCAASGFTFSSYTMSWVRQTPEKRLEWVAYISNGGGSTYYPDTVKGRFTISRDNAKNTLYLQMSSLKSEDTAMYYCARNYGYGFAYWGQGTTVTVSSVLDIVLTQSPATLSVTPGDSVSLSCRASQSISNNLHWYQQKSHESPRLLIKYASQSISGIPSRFSGSGSGTDFTLSINSVETEDFGMYFCQQSNSWPHTFGGGTKLELH1-15VHVQLQESGPELVKPGASVKISCKASGYSFTAYFMNWVMQSHGKSLEWIGRINPYNGDTFYNQKFKGKATLTVDKSSSTAHMELRSLASEDSAVYYCARHPFYAMDYWGQGTTVTVSSVLDVLMTQAPKFMSTSVGGRVSITCKASQDVNTAVAWYQQKPGQSPKLLIYSASYRYTGVPDRFTGSGSGTDFTFTISSVQAEDLAVYYCQQHYTTPYTFGGGTKLELK

表4 3株mAbs与H1N1流感病毒HA结合的氨基酸位点结果
阻断ELSIA试验中出现了叠加顺序不同,抑制率不同。从单抗与1918年流感病毒结合的位点可见,H1-13识别的位点位于A1-12识别的位点内;从与2009年流感病毒识别的位点可见,H1-13与A1-12识别的位点非常接近。原因可能是由于先加入H1-13作为封闭抗体后,导致了抗原表位结构的变构,Reynolds等[16]提出了变构数据库,利用这个数据库可以研究、分析变构蛋白的结构、功能。Alix等[17]利用PEOPLE程序来预测线性抗原表位,发现抗原与抗体发生反应的时候,抗原表位的构象发生了变化,因此抗原表位是有柔韧性的。此外,表面极性大、亲水性强的氨基酸残基通常也参与表位构成,所以须用其他实验方法来进一步验证。
1918年H1N1流感病毒与2009年H1N1流感病毒在HA抗原表位区域具有极高的相似度[18],对这3株单抗进行预测,共预测出二种结合模式,比对到1918、2009年流行的H1N1的血凝素HA蛋白上,分别预测出了结合位点。发现3株单抗结合HA上的氨基酸位点均分布在HA结构的头部,头部的抗原决定簇决定了病毒的抗原性,该结果为后期流感病毒HA表位疫苗的研究提供了实验数据。
利益冲突无
[1]Garten RJ, Davis CT, Russell CA, et al. Antigenic and genetic characteristics of swine-origin 2009 A(H1N1) influenza viruses circulating in humans[J]. Science, 2009, 325(5937):197-201. DOI:10.1126/science.1176225.
[2]Huang SS, Banner D, Fang Y, et al. Comparative analyses of pandemic H1N1 and seasonal H1N1, H3N2, and influenza B infections depict distinct clinical pictures in ferrets[J]. PLoS One, 2011, 6(11):1-14. DOI:10.1371/journal.pone.0027512.
[3]Maines TR, Jayaraman A, Belser JA, et al. Transmission and pathogenesis of swine-origin 2009 A(H1N1) influenza viruses in ferrets and mice[J]. Science, 2009, 325(5939):484-487. DOI:10.1126/science.1177238.
[4]Dreyfus C, Laursen NS, Kwaks T, et al. Highly conserved protective epitopes on influenza B viruses[J]. Science, 2012, 337 (6100):1343-1348. DOI:10.1126/science.1222908.
[5]Subbarao K, Murphy BR, Fauci AS. Development of effective vaccines against pandemic influenza[J]. Immunity, 2006, 24(1): 5-9. DOI:10.1016/j.immuni.2005.12.005.
[6]Igarashi M, Ito K, Yoshida R, et al. Predicting the antigenic structure of the pandemic (H1N1) 2009 influenza virus hemagglutinin[J]. PLoS One, 2010, 5(1):1-5. DOI:10.1371/journal.pone.0008553.
[7]周芳烨, 陈爱敏, 彭海燕, 等. 北半球季节性甲型流感疫苗株HA抗原表位的分析[J]. 现代预防医学, 2016, 43(1):167-170.
[8]Hajighasemi F, Gharagozlou S, Ghods R, et al. Private idiotypes located on light and heavy chains of human myeloma proteins characterized by monoclone antibodies[J]. Hyhbridoma(Larehmt), 2006, 25(6):329-335. DOI: 10.1089/hyb.2006.25.329.
[9]Rockman S, Camuglia S, Vandenberg K, et al. Reverse engineering the antigenic architecture of the haemagglutinin from influenza H5N1 clade1 and 2.2 viruses with fine epitope mapping using monoclonal antibodies[J]. Mol Immunol, 2013, 53(4):435-442. DOI:10.1016/j.molimm.2012.10.001.
[10]Naundorf S, Preithner S, Mayer P, et al. In vitro and in vivo activity of MT201, a fully human monoclonal antibody for pancarcinoma treatment[J]. Int J Cancer, 2002, 100(1):101-110. DOI: 10.1002/ijc.10443.
[11]Walls HH, Harmon MW, Slagle JJ, et al. Characterization and evaluation of monoclonal antibodies developed for typing influenza A and influenza B viruses[J]. J Clin Microbial, 1986, 23(2):240-245.
[12]Shibaguchi H, Kuroki M, Badran A, et al. Cloning and sequencing of variable region cDNA of a novel human monoclonal antibody to carcinoembryonic antigen, and generation of a single chain variable fragmented antibody[J]. Anticancer Res, 2004, 24 (5C):3355-3360.
[13]Jens Wrammert, Dimitrios Koutsonanos, Gui-Mei Li, et al. Broadly cross-reactive antibodies dominate the human B cell response against 2009 pandemic H1N1 influenza virus infection[J]. J Exp Med, 2010, 208(1): 181-193. DOI:10.1084/jem.20101352.
[14]Paul SS, Mok CK, Mak TM, et al. A cross-clade H5N1 influenza A virus neutralizing monoclonal antibody binds to a novel epitope within the vestigial esterase domain of hemagglutinin[J]. Antiviral Res, 2017, 144: 299-310. DOI:10.1016/j.antiviral.2017.06.012.
[15]李慧瑾, 郭春艳, 赵彭花, 等. 运用单克隆抗体分析流感病毒H1亚型HA蛋白的表位[J]. 西安交通大学学报(医学版), 2012, 33(3):291-295. DOI:10.3969/j.issn.1671-8259.2012.03.007.
[16]Reynolds KA, McLaughlin RN, Ranganathan R. Hot spots for allosteric regulation on protein surfaces[J]. Cell, 2011, 147(7): 1564-1575. DOI:10.1016/j.cell.2011.10.049.
[17]Alix AJ. Predictive estimation of protein linear epitopes by using the program PEOPLE[J]. Vaccine,1999, 18(3-4):311-314.
[18]Xu R, Ekiert DC, Krause JC, et al. Structural basis of preexisting immunity to the 2009 H1N1 pandemic influenza virus[J]. Science, 2010, 328(5976):357-360. DOI: 10.1126/science.1186430.
