甲型肝炎病毒疫苗株H2株的连续传代及其基因稳定性
2018-04-17王东宝曹晗郑勇史小军马忠飞段颖融邓燕孙明波
王东宝 曹晗 郑勇 史小军 马忠飞 段颖融 邓燕 孙明波
650118 昆明,北京协和医学院中国医学科学院医学生物学研究所 云南省重大传染病疫苗研发重点实验室
甲肝病毒(hepatitis A Virus)是一类能够引起人类肝功能异常的小RNA病毒,由它导致的传染病称为甲型肝炎,此病传播速度快,在所有肝炎中发病率最高,从90年代开始流行较为严重,导致数百万人发病,给患者和国家造成极大和经济负担,影响较为严重。[1]。
1992年,中国医学科学院医学生物学研究所(以下简称:生物所)与浙江省医学科学院、中国食品药品检定研究院三家单位共同合作,开发研制出甲型肝炎减毒活疫苗(H2株),该疫苗是我国首创,成功地控制了甲肝流行,取得了显著的社会效益及经济效益[2-3]。目前生物所生产的甲肝疫苗采用的是甲肝病毒H2株[4]进行生产,此毒株于1982年分离自我国杭州市郊农村一名12岁男性甲肝患者的粪便,经原代猴肾细胞传20代,人胚肺二倍体细胞KMB17传5代获得,经检定合格,用于疫苗生产。由于毒株是HAV野毒株经体外细胞传代适当减毒获得,根据疫苗生产的相关要求,用于生产活疫苗的毒种,必须保持减毒株的特性,其致病性的毒力和免疫原性应相对稳定,毒力不应当随着传代发生“返祖”或降低,尤其是重要毒力相关位点不能够发生重大突变。因此,在疫苗生产过程中,研究H2株毒种的遗传基因是否发生较大变异,具有重大的意义。
以往对HAV病毒基因的研究[5-8]都是采用一代测序原理 (即sanger 测序),进行部分片段或全基因组测序。这种直接测序方法具有高度的准确性和简单、快捷等特点,但由于此种测序方法有一定的具局限性,不能够全面反映出整个样品中HAV全部基因组突变概率和真实变化情况。而第二代高通量测序的基本原理是边合成边测序,它能够一次对几百万到几亿条DNA分子进行序列测定,具有高通量、超深度、高灵敏度并且速度快、准确率高、成本低和信息量丰富等优点,而且能够全面地反映基因组上变异图谱[9-10]。
我所学者己经采用第二代基因测序的方法,对脊髓灰质炎病毒进行了研究[11],受此启发,本试验对甲肝病毒H2M20K7主代毒种、工作种子、疫苗代次及传代后不同代次的病毒采用高通量测序(二代基因测序法)方法进行病毒的全基因序列测定,从分子水平对H2株不同代次病毒的基因稳定性和毒力基因位点变异率进行分析,以进一步考察H2株毒种在连续传代过程中的遗传稳定性和毒力特征, 也为疫苗株的选择、制备和稳定性考察提供参考和借鉴。
1 材料与方法
1.1病毒及细胞甲肝病毒原始种子H2M20K5(K5)由浙江省医学科学院提供,经生物所用KMB17细胞再传代2次,获得H2M20K7主代种子批(K7),批号为201201。K7再经KMB17细胞连续传代三次,建立了工作种子库H2M20K10(K10)。KMB17细胞种子来源于生物所二室,是生物所于1972建立的人二倍体细胞株[12],也是目前用于生产H2株减毒活疫苗的细胞,工作细胞代次为16代,批号为201501.
1.2主要试剂及仪器MEM培养基购自美国Gibico公司;新生牛血清购自兰州民海生物;细胞培养液、细胞维持液、病毒稀释液的其他成分均由本所提供;细胞培养容器为美国nunc细胞工厂;病毒RNA提取试剂盒(QIAamp Viral RNA Kit)为德国Qiagen公司提供;大容量高速离心机购自美国THERMO;超声破碎仪购自美国 Thermo;洗板机:美国BioTek ELX50;酶标仪:美国BioTek SynergyHT;HAV-Ag检测板:生物所生物制品九室提供。
1.3病毒连续传代及样品制备参照甲肝病毒生产工艺,取甲肝减毒活疫苗工作种子H2M20K10(K10),经冻融超声后,接种在培养成单层的KMB17细胞上(病毒接种MOI为0.1, 细胞代次为32代),进行培养,培养过程更换维持液,继续培养至病毒增值高峰期(22~26 d)收获病毒,得到疫苗代次病毒(K11)。 按上述方法,通过8个周期连续传代,得到K12~K18不同代次的病毒,冻存于-20 ℃待用。
1.4病毒感染性滴度检测取不同代次的病毒液(K7,K10,K11,K13,K15,K18),超声破碎后作10倍系列稀释,每个稀释度接种5个小方瓶,然后将小方瓶放入37 ℃恒温培养2 h取出,加入培养液,放入35 ℃恒温培养至24 d收获病毒,然后用ELISA法检测病毒感染性滴度(lgCCID50/ml)。
1.5病毒基因组RNA提取取不同代次的病毒经冻融、超声破碎后,8 000×g离心1 min,分别取上清150 μl,按QIAamp Viral RNA Kit说明书提取病毒RNA,洗脱得到病毒RNA。
1.6病毒基因组测序将提取得到的RNA,送上海源序生物科技有限公司进行全基因组测序。通过高通量测序技术(Illumina HiSeqTM2500)得到原始测序序列,对Raw Data进行统计分析,通过序列mapping统计碱基分布。
1.7基因序列对比分析将二代测序得到的数据,以主代毒种K7的全基因序列为基准,将不同代次的病毒(K10,K11,K13,K15,K18)的全基因组变化情况用PRISM软件进行比较分析, 研究HAV不同代次病毒在5′端非编码区、VP1-2 A、2B、2C区及重要神经毒力相关位点的碱基序列差异及变异情况。
2 结果
2.1感染性滴度检测H2株不同代次病毒的感染性滴度维持在7.76~8.50 lgCCID50/ml之间,P值为0.981差异无统计学意义,表明不同代次病毒在KMB17细胞中的感染性稳定,未发生较大变化。(见图1)

图1 H2株不同代次病毒的感染性滴度Fig.1 Infectious titers of different passages of H2 Virus
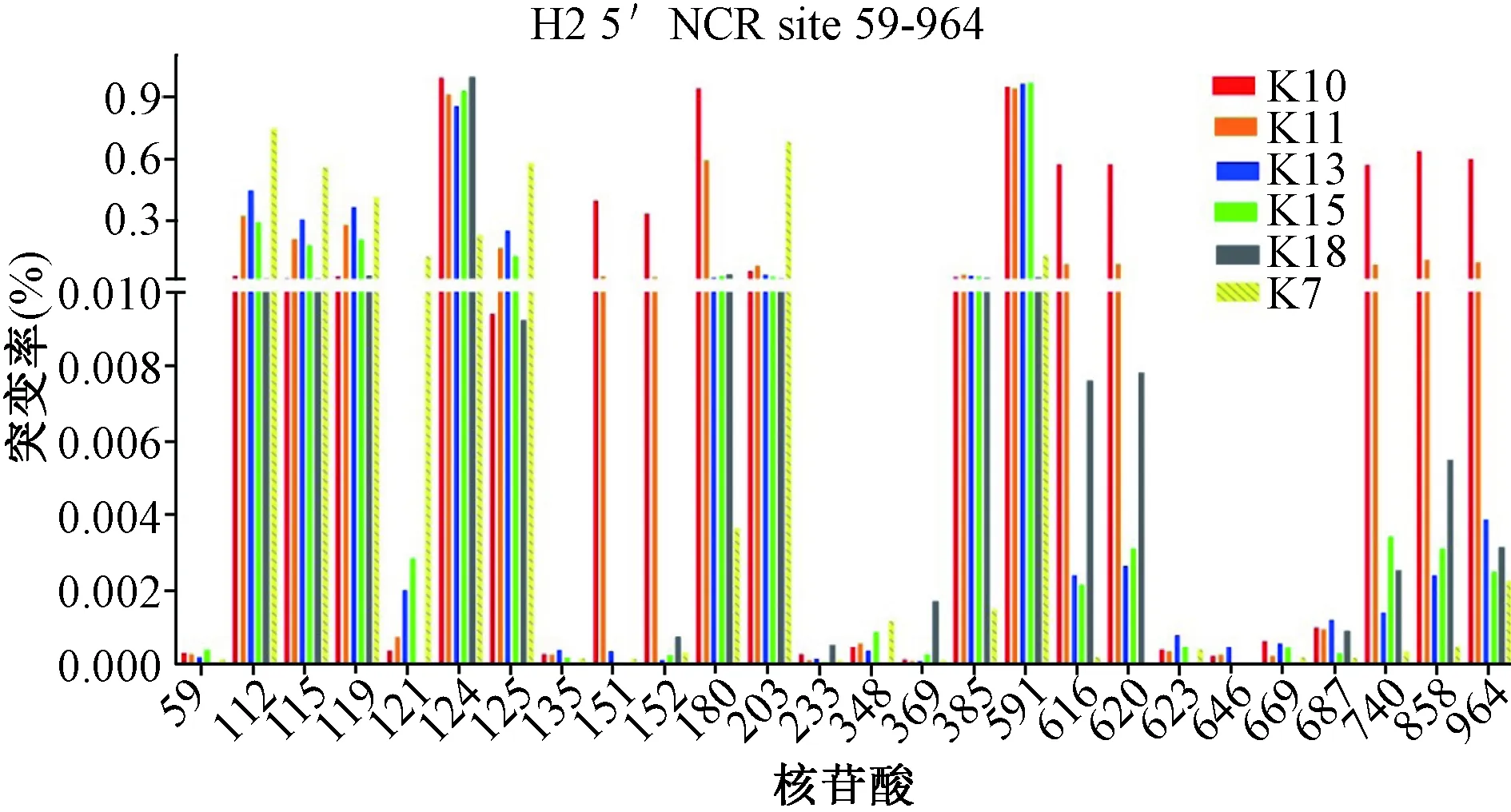
图2 H2株不同代次病毒在5′NCR重要位点的突变率Fig.2 Mutation rate of different passages virus of H2 strain at 5’non coding region
2.2碱基分布可信度分析分析碱基可信度,Q20数据可信度在92.94%~97.53%,Q30数据可信度在87.87%~94.18%,表明本试验数据可靠,可作进一步的分析。(见表1)
2.3基因突变率及位点统计通过smartools找到序列中每个碱基的覆盖度,计算每个位点的突变率,不同代次病毒在全基因段均有同义突变和非同义突变,但同义突变率均小于4.35%, 非同义突变变异均率小于0.58%(见表2)。

表1 Solexa(二代)测序结果统计碱基分布

表2 不同代次病毒与K7主代毒种相比较的突变率统计
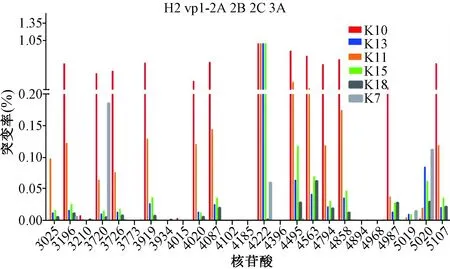
图3 H2株不同代次病毒在VP1/2 A,2B,2C,3 A区突变位点的突变率Fig.3 Mutation rate of different passages virus of H2 strain at VP1/2 A,2B,2C,3 A regions
2.4不同代次病毒在关键基因段及毒力位点的非同义突变情况H2株病毒不同代次的5′NCR非编码区基因序列有少量变异,整个病毒基因的5′ NCR区的序列相对同源性高、变异很小,最高突变率为:nt124位点0.999%(见图2)。病毒在连续传代后,在VP1/2 A,2B,2C和重要基因位点未发生明显突变,基因结构较为稳定,最高突变率为:nt4 222位点0.999%(见图3)。
3 讨论
3.1病毒感染性甲型肝炎减毒活疫苗H2株主代毒种K7在KMB17人胚肺二倍体细胞上经连续传代至K18,通过对不同代次病毒的感染性滴度进行检测和对比检测,病毒的感染性滴度维持在7.76~8.50 lgCCID50/ml之间,较为稳定,差异无统计学意义。结果表明,H2株主代毒种K7在经过KMB17细胞传代11次的过程中,不同代次的病毒感染性没有发生明显差异,感染性稳定。
3.25′NCR区基因基因突变率分析结合Cohen[13-15]、Funkhouser[16]和周德久[17]等进行的研究结果,我们将H2株不同代次病毒的5′ NCR基因序列区段(nt1~nt750)进行了比较分析,发现K7在传代过程中,不同代次病毒(K10,K11,K13,K15,K18)在整个5′NCR基因区段,与K7主代毒株同源性高、变异很小,除特定区段少数位点发生变化之外,大部份的基因未发生突变,总体突变率<0.1%。这可能是因为H2株在主代次毒种以前,经长期传代适应了KMB17细胞株后较为稳定,连续传代过程中变异率极低。
Schultz[18]的研究中,将HM175野毒株与HM175/P16细胞适应株进行对比,认为nt 203、204碱基缺失和nt 687位的碱基替换(U687G)是野毒株适应细胞株的特征,这几个变化能够增强核糖体进入位点的无帽翻译过程,增加病毒在细胞中的适应能力。我们将H2株不同代次病毒(K7,K10,K11,K13,K15,K18)的检测结果与之相对比,结果表明在nt 203未发生缺失,而在nt 204位碱基缺失情况与此前文献报导一致,各代次病毒在nt687位点100%为G,为适应细胞培养特征。
3.3VP1/2A、2B、2C毒力位特征位点基因突变率分析根据Emersor等[19-21]研究证明,HAV的毒力基因主要在VP1/2 A、2B、2C区, 毒力相关基因簇位与细胞适应相关的核酸位点主要分布在P2区。其中VP1/2 A连接区的(nt3 024~3 196)的168个核苷酸,是目前国际通过的HAV基因分型标准位点。我们将H2株各代次病毒(K10,K11,K13,K15,K18)测序结果与数据库中HM175的减毒株序列对比,在片段V P1/2 A 连接区nt3024 ~nt3191(168 bp,HM-175)与GenBank 库中全部HAV序列进行核苷酸/氨基酸同源性进行分析,结果证明K7主代毒种及经多次传后的各代次病毒的毒力特征,仍然与M-175/7MK-5 的同源性最高。按照目前国际通用的HAV基因分型标准,基因型属于IB亚型。与主代K7相比,不同代次病毒在全基因段均有同义突变和非同义突变,但同义突变率小于4.35%,非同义突变率小于0.58%,表明毒株经过连续传代后,基因变异情况非常小,保持稳定的减毒株型别特征。
在HAV病毒毒力位点中,椐Robertson[22]、Paulmann[23]等的文献报导其中比较关键的位点有:VP1/2 A区的nt3 025、3 196,2B区的nt3 889、3 919,2C区的nt 4 043、4 073、4 087、4 185、4 222、4 563、4 987。我们的研究结果显示上述位点突变率为0.0027%~0.675% 之间,变异率极低。本研究中H2株各代次病毒的2C 基因位点nt 4 185(2C/Lys64)与文献报道的其他疫苗株一致,并且非常稳定,此位点可能参与病毒的减毒[24-25]。检测结果与之相同,K18代病毒次在nt3 196、 4 043、4 073、4 222均发现较前代次基因发生变异相对较多,突变率在0.006%~0.99%之间,试验数据表明随着病毒传代次数的增加,病毒在多个基因位点突变率有所增加,但未超过1%。在K10,K11,K13,K15代病毒序列中, nt4 987位点的核苷酸发生A变为G(2C/A4987G),及2C区氨基酸由Glu变为Gly(2C/Gly331Glu)与刘建生等[26]的研究结果相同,推测与H2株细胞基质特异的适度减毒特征有关。
本试验用二代测序的方法对H2株各代次病毒进行全基因组测序,发现甲肝病毒H2株各代次病毒在KMB17细胞培养连续传代过程中,病毒的遗传稳定性良好,病毒感染性未发生明显改变。我们建立的方法和获得的数据,为今后进一步研究甲肝疫苗毒株及其他毒株的遗传稳定性打下了基础,也为研究疫苗的批间一致性提供了新的研究思路。
利益冲突:无
[1]赵铠,章以浩,李河民,等. 《医学生物制品学》[M]. 2007, ISNB 987-7-117-08474-1/R: 821-833.
[2]董德祥, 曹逸云,黄小琴,等. 甲型肝炎减毒活疫苗(H2株)的开发研究[J]. 中国疫苗和免疫, 2001, 7(3): 178-182.DOI:10.3969/j.issn.1006-916X.2001.03.031.
[3]董德祥, 曹逸云, 黄小琴,等. 甲型肝炎减毒活疫苗(H2株)的残余毒力及其返祖可能性的研究[J]. 医学研究杂志, 2003, 32(11): 19-20. DOI:10.3969/j.issn.1673-548X.2003.11.008.
[4]陈勇, 杨能宇, 洪艳, 等. 甲型肝炎病毒活疫苗H_2减毒株全基因文库的建立[J]. 中华医学杂志, 1996, (5): 342-344.
[5]井申荣. 甲型肝炎病毒的分子生物学研究进展[J]. 国际流行病学传染病学杂志, 2000, (5): 197-201.
[6]黄小琴, 杨净思, 周德久,等. H2株甲肝病毒经KMB17细胞培养的毒力及核苷酸序列[J]. 中国生物制品学杂志, 2000, 13(3): 133-135. DOI:10.3969/j.issn.1004-5503.2000.03.002.
[7]黄小琴, 周德久. 甲肝减毒活疫苗(H2株)接种后HAV的毒力和基因型[J]. 中国生物制品学杂志, 1998, (4): 200-203. DOI:10.13200/j.cjb.1998.04.200.huangxq.004.
[8]胡凝珠, 胡云章, 施海晶,等. 甲型肝炎减毒活疫苗(H2株)快速繁殖株不同代次全基因序列比较[J]. 中国生物化学与分子生物学报, 2001, 17(5): 541-546.DOI:10.3969/j.issn.1007-7626.2001.05.001.
[9]张欣欣, 王铭杰. 新一代测序技术在HBV变异研究中的应用[J]. 临床肝胆病杂志, 2015, (4): 514-519. DOI:10.3969/j.issn.1001-5256.2015.04.009.
[10]周帆, 林标扬,等. 第二代测序在检测人类基因突变中的应用[J]. 生命科学研究, 2012, 16(5): 451-456 DOI:10.3969/j.issn.1007-7847.2012.05.016.
[11]Neverov A, Purcell RH. Massively parallel sequencing for monitoring genetic consistency and quality control of live viral vaccines[J]. Proc Natl Acad Sci USA, 2010, 107(46): 20063-20068.DOI:10.1073/pnas.1012537107.
[12]郭仁, 曹逸云, 代振洲,等. 人二倍体细胞株KMB-17的特性[J]. 中国医学科学院学报, 1981, (4): 17-21.
[13]Cohen JI, Rosenblum B, Feinstone SM, et al. Attenuation and cell culture adaptation of hepatitis A virus (HAV): a genetic analysis with HAV cDNA[J]. J Virol, 1990, 63(12): 5364-5370.
[14]Cohen JI, Rosenblum B, Ticehurst JR, et al. Complete nucleotide sequence of an attenuated hepatitis A virus: comparison with wild-type virus[J]. Proc Natl Acad Sci U S A, 1987, 84(8): 2497-2501.
[15]Cohen JI, Ticehurst JR, Purcell RH, et al. Complete nucleotide sequence of wild-type hepatitis A virus: comparison with different strains of hepatitis A virus and other picornaviruses[J]. J Virol, 1987, 61(1): 50-59.
[16]Funkhouser AW, Purcell RH, D’Hondt E, et al. Attenuated hepatitis A virus: Genetic determinants of adaptation to growth in MRC-5 cells[J]. J Virol, 1994, 68(1): 148.
[17]周德久, 曹逸云, 黄小琴,等. H_2株甲肝减毒活疫苗K_7的核苷酸全序列分析[J]. 云南大学学报自然科学版, 1999, 21(3): 170-174.DOI:10.3321/j.issn:0258-7971.1999.03.002.
[18]Schultz DE, Honda M, Whetter LE, et al. Mutations within the 5’ nontranslated RNA of cell culture-adapted hepatitis A virus which enhance cap-independent translation in cultured African green monkey kidney cells[J]. J Virol, 1996, 70(2): 1041-1049.
[19]Emerson SU, Mcrill C, Rosenblum B, et al. Mutations responsible for adaptation of hepatitis A virus to efficient growth in cell culture[J]. J Virol, 1991, 65(9): 4882-4886.
[20]Emerson SU, Huang YK, Mcrill C, et al. Mutations in both the 2B and 2C genes of hepatitis A virus are involved in adaptation to growth in cell culture[J]. J Virol, 1992, 66(2): 650-654.
[21]Emerson SU, Huang YK, Nguyen H, et al. Identification of VP1/2A and 2C as Virulence Genes of Hepatitis A Virus and Demonstration of Genetic Instability of 2C[J].J Virol, 2002, 76(17): 8551-8559.DOI:10.1128/JVI.76.17.8551-8559.2002.
[22]Robertson BH, Jansen RW, Khanna B, et al. Genetic relatedness of hepatitis A virus strains recovered from different geographic regions[J]. J Gen Virol, 1992, 73(6): 1365-1377.DOI: 10.1099/0022-1317-73-6-1365.
[23]Paulmann D, Magulski T, Schwarz R, et al. Hepatitis A virus protein 2B suppresses beta interferon (IFN) gene transcription by interfering with IFN regulatory factor 3 activation[J]. J Gen Virol, 2008, 89(7): 1593-1604. DOI:10.1099/vir.0.83521-0.
[24]姜春来, 王鹏富, 刘景晔,等.甲肝减毒活疫苗L-A-1疫苗株的核酸序列测定与比较分析[J]. 中华实验和临床病毒学杂志, 2004, 18(4): 360-362.DOI:10.3760/cma.j.issn.1003-9279.2004.04.016.
[25]曹经瑗, 郭可謇, 詹美云,等. 中国甲型肝炎病毒龙甲株全基因序列的cDNA克隆及序列分析[J]. 中华实验和临床病毒学杂志, 2001, 15(4): 384-386 DOI:10.3760/cma.j.issn.1003-9279.2001.04.026.
[26]刘建生, 侯宗柳, 马绍辉,等. 甲型肝炎减毒活疫苗H2减毒株P2区基因序列测定与特征分析[J]. 中国疫苗和免疫, 2008, 14(1): 7-10.DOI:10.3969/j.issn.1006-916X.2008.01.002.
