碳青霉烯类耐药肺炎克雷伯菌多位点序列分型和不同ST分型感染患者的临床特点
2017-10-16田素飞褚云卓陈佰义刘丽文张智洁肖晓光
吴 娜, 田素飞, 褚云卓, 陈佰义, 刘丽文, 张智洁, 王 晶, 肖晓光, 路 娟
碳青霉烯类耐药肺炎克雷伯菌多位点序列分型和不同ST分型感染患者的临床特点
吴 娜1, 田素飞*, 褚云卓*, 陈佰义1, 刘丽文2, 张智洁3, 王 晶4, 肖晓光4, 路 娟5
目的对临床分离的碳青霉烯类耐药肺炎克雷伯菌(CR-KPN)进行多位点序列分型(MLST),同时研究不同ST分型感染患者的临床特点。方法收集2013年1月-2015年6月辽宁省为主5所医院临床分离的61株CR-KPN,采用微量肉汤稀释法测定受试菌株对14种抗菌药物的敏感性。应用PCR基因扩增技术及DNA测序方法对受试菌株进行碳青霉烯酶基因检测。通过对受试菌株进行MLST,探讨其克隆相关性和分子流行病学特征。同时分析不同ST分型下CR-KPN的耐药特点、耐药机制及菌株感染的临床特点。结果MLST分型显示61株CR-KPN共有18种ST分型,ST11为优势型别(53.3 %),研究同时发现这部分ST11型菌株更容易对碳青霉烯类抗生素耐药且MIC值较高,大部分产KPC-2型碳青霉烯酶。单因素分析提示ST11组患者在ICU接受治疗所占的比例及机械通气的使用率高于非ST11组,差异具有统计学意义。其他ST型中ST2033、ST2135、ST2193、ST2194、ST2195、ST2196为世界范围内首次注册,其中ST2193、ST2194、ST2195与ST11有密切的亲缘性。临床资料提示同一所医院同一时期以相同克隆流行为主。结论MLST显示CR-KPN共有18种ST型别,ST11为优势型别,结合临床资料发现同一所医院同一时期以相同克隆流行为主,提示CR-KPN有局部播散的风险。鉴于临床资料分析提示接受过ICU治疗和机械通气的患者容易发生CR-KPN菌株(特别是ST11型)感染的风险,此类患者应引起临床的广泛重视。
肺炎克雷伯菌; 多位点序列分型; 碳青霉烯酶
Abstract: ObjectiveTo investigate the homology of carbapenem-resistantKlebsiella pneumoniae(CR-KPN) isolates by multilocus sequence typing (MLST) method, and pro fi le CR-KPN infections in terms of MLST sequence types (STs).MethodsA total of 61 CR-KPN isolates were collected in fi ve hospitals from Liaoning Province from January 2013 to June 2015. The MICs of 14 antimicrobial agents against these isolates were determined by broth microdilution method. The genotypes of carbapenemases were analyzed by PCR and DNA sequencing techniques.The homology of CR-KPN isolates were analyzed by MLST method. The clinical data of patients with CR-KPN infection were reviewed to characterize CR-KPN infections.ResultsA total of 18 STs were identi fi ed among the 61 CR-KPN strains according to MLST data. More than 50 % of the isolates belonged to ST11 (53.3 %). ST11 strains showed higher resistance rate to carbapenems and higher prevalence of KPC-2 type carbapenemase. Univariate analysis indicated that more ST11-infected patients were treated in ICU and with mechanicalventilation than non-ST11 CR-KPN-infected patients (P<0.05). ST2033, ST2135, ST2193, ST2194, ST2195 and ST2196 were the STs fi rstly registered in the world. The eBURST analysis showed that ST2193, ST2194, ST2195 and ST11 were closely related.Clinical data indicated that the prevalent CR-KPN strains during the same period in the same hospital usually belonged to the same ST clone.ConclusionsMLST of CR-KPN showed 18 sequence types, of which ST11 was the predominant type. Clinical data indicated that the prevalent CR-KPN strains during the same period in the same hospital usually belonged to the same ST clone. This suggests the potential of local CR-KPN outbreak. The ICU patients and those receiving mechanical ventilation may be prone to CRKPN (especially ST11) infection. Such patients should be managed appropriately.
Key words:Klebsiella pneumoniae; multilocus sequence typing; carbapenemase
碳青霉烯类耐药肺炎克雷伯菌(carbapenemresistantKlebsiella pneumoniae,CR-KPN)不断增加的耐药趋势和播散形势引起广大临床工作者的关注,已成为全球性公共卫生问题。在对耐药机制研究的同时,研究人员采用多位点序列分型(multilocus sequence typing, MLST)方法对菌株的播散特征进行分子流行病学研究,结合菌株致病性或耐药性,追踪菌株间的遗传特性和宏观进化过程,为解决细菌耐药和播散提供指导。本研究将对本地区临床分离的碳青霉烯类耐药肺炎克雷伯菌进行MLST,掌握本地区的分子流行病学趋势。同时针对不同遗传来源的菌株进行药物敏感性、耐药机制及临床感染特点相结合的方式进行分析,探讨不同遗传来源细菌发生耐药的危险因素和感染的临床特点,指导本地区CR-KPN的感控防治和临床治疗。
1 材料与方法
1.1 材料
1.1.1 菌株来源 收集2013年1月-2015年6月5所医院的CR-KPN 61株,包括中国医科大学附属第一医院(30株)、中国医科大学附属盛京医院(4株)、辽宁省人民医院(9株)、大连医科大学附属第一医院(10株)、哈尔滨医科大学附属第一医院(8株),均为非重复菌株。质控菌株为大肠埃希菌ATCC25922,大肠埃希菌ATCC35218,铜绿假单胞菌ATCC27853。
1.1.2 仪器与试剂 PCR仪(美国Applied Biosystems公司,9700型),水平电泳仪(美国Bio-Rad公 司 );Mueller-Hinton琼 脂( 英国OXOID公司)、药敏试验所用抗菌药物购自U.S.Pharmacopeia、European Pharmacopoeia 和Dr.Ehrenstorefer GmbH,美罗培南纸片(10 μg,英国OXOID公司),乙二胺四乙酸(EDTA,pH=8,0.5 mol/L)、EB替代物为北京市普利莱基因技术
1.2 方法
1.2.1药敏试验 采用微量肉汤稀释法测定14种抗菌药物对受试菌株的最低抑菌浓度(MIC)。抗菌药物包括美罗培南、亚胺培南、厄他培南、头孢噻肟、头孢曲松、头孢他啶、头孢吡肟、哌拉西林-他唑巴坦、左氧氟沙星、环丙沙星、磷霉素、阿米卡星、多黏菌素、替加环素。
1.2.2PCR检测碳青霉烯类耐药基因
1.2.2.1DNA模版制备 从血琼脂培养皿挑取单个受试菌菌落加入5 mL的LB肉汤培养液中,37 ℃、100 r/ min恒温振荡培养18~20 h,搜集4 mL菌液。其余具体步骤参照DNA提取试剂盒说明书。
1.2.2.2PCR扩增目的基因反应体系及循环条件 PCR 扩增引物序列见表1。PCR 反应体系如下 :取基因组 DNA 为模板 5 μL(10 ng/μL),加入引物 F(20 μmol/L)0.5 μL,引物 R(20 μmol/L)0.5 μL,2×Taq PCR masterMix 12.5 μL, 无 菌纯水加至 25 μL,配制 25 μL 反应体系。PCR 反应条件:预变性94 ℃×5 min;循环94 ℃×30 s,61 ℃×30 s, 72 ℃×30 s,共 30个循环;最后延伸72 ℃×10 min。不同类型碳青霉烯酶基因的PCR反应条件有所不同,参考文献[1-6]。
1.2.2.3PCR产物纯化及测序 PCR产物交上海生工生物技术有限公司纯化及双向测序。测序得到的基因片段拼接后采用Bioedit软件进行序列比对和网上相似性检索分析(http://www.ncbi.nlm.nih.gov/blast/)。
1.2.3MLST PCR 引物参照MLST Pasteur专业网站设计的肺炎克雷伯菌7个管家基因引物(gapA、infB、rpoB、phoE、mdh、pgi、tonB),见有限公司产品,小量DNA抽提纯化试剂盒、DNA marker、2×Taq PCR MasterMix为北京天根生化科技有限公司产品,PCR引物由上海生工生物工程有限公司合成。表2。PCR总反应体系同前。反应条件:94 ℃预变性 2 min ;94 ℃变性 30 s,50 ℃复性 1 min,72 ℃延伸30 s,共35个循环;最后72 ℃再延伸7 min。PCR产物交上海生工生物技术有限公司纯化及双向测序,7个管家基因(gapA、infB、rpoB、phoE、mdh、pgi、tonB)的序列与 MLST数据库(http://www.mlst.net)中已知的肺炎克雷伯菌的等位基因序列相比,获得相应的序列号。ST序列数据间的亲缘关系使用eBURST V3软件(http://eburst.mlst.net)分析。
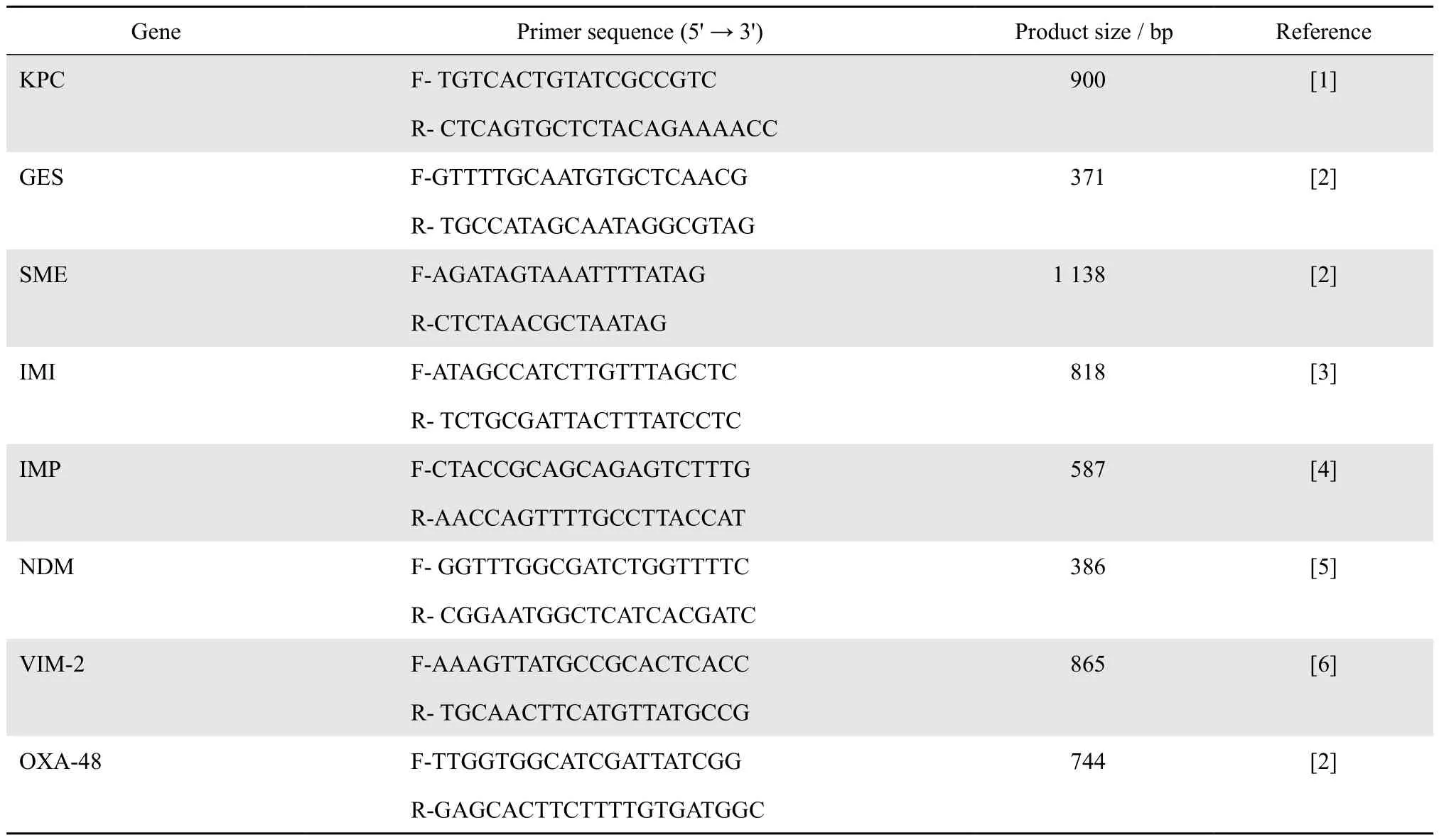
表1 碳青霉烯酶基因引物名称、序列、长度及参考文献Table 1 PCR primers for amplifying carbapenemase genes

表2 肺炎克雷伯菌多位点序列分型7个管家基因所用引物Table 2 PCR primers for amplifying the 7 housekeeping genes inK. pneumoniaeisolates
1.2.4临床资料的收集 查阅本次收集的61份感染CR-KPN菌株患者的临床资料,对每例病史进行详细的信息记录和收集。
1.2.5统 计 分 析 利 用Microsoft Excel 2003 、SPSS16.0软件统计分析。比较ST11型和其他ST分型各自的临床特点,进行单因素分析,P<0.05为差异有统计学意义。
2 结果
2.1 菌株对14种抗菌药物的敏感性分析
61株CR-KPN对头孢菌素及β内酰胺-酶抑制剂复合制剂(哌拉西林-他唑巴坦)的耐药率均>80 %;对碳青霉烯类抗生素厄他培南耐药率为100 %,对美罗培南和亚胺培南的耐药率分别为78.7 %和70.5 %,有29株对两者同时耐药;耐药率最低的是多黏菌素和替加环素,分别为14.8 %和19.7 %。见表3。
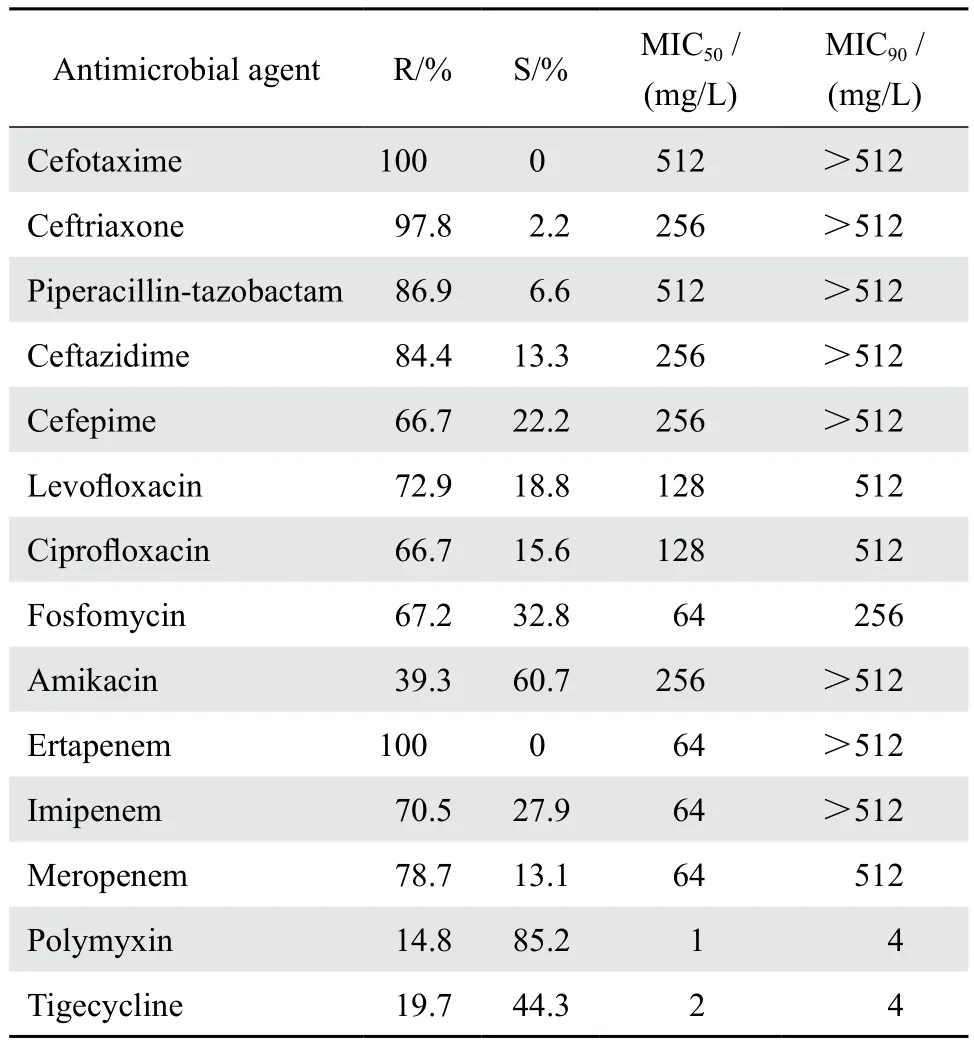
表3 菌株对14种抗菌药物的敏感性分析Table 3 Susceptibility ofK. pneumoniaeisolates to antimicrobial agents
2.2 碳青霉烯酶耐药基因检测结果
经PCR检测和DNA测序,61株临床分离菌中,共检出51株携带碳青霉烯酶基因。其中,产KPC型碳青霉烯酶有40株,均为KPC-2型。产金属酶包括IMP型酶基因9株,均为IMP-4型酶基因;产NDM型酶基因4株,均为NDM-6型。有3株菌株同时产KPC-2及IMP-4型酶基因。所有菌株中GES、SME、IMI、VIM-2、OXA-48碳青霉烯酶基因检测均为阴性。
2.3 肺炎克雷伯菌的MLST
61株CR-KPN进行MLST,对其内部7个管家基因片段:rpoB、gapA、mdh、pgi、phoE、infB和tonB进行PCR扩增和DNA测序,与Pasteur网站内的标准序列进行比对,结果见表4。除去3株测序失败,根据MLST技术检测,58株CRKPN共18种ST序列型,其中8株细菌无法与数据库中已知的ST序列比对成功,已向http://www.pasteur.fr/recherche/genopole/PF8/mlst/index.html网站提交该序列,获得新的序列型别,分别为 ST2033、ST2135、ST2193(3株 )、ST2194、ST2195、ST2196。
ST序列数据间的亲缘关系使用eBURST V3软件(http://eburst.mlst.net)分析,如果在7个管家基因中仅相差l~3个差异基因型,则认为这2种ST型有亲缘关系,在eBURST图上会呈现各个点之间的连线(图1)。ST1711、ST2193、ST2194、ST2195起源于ST11,并且亲缘性很近。结合菌株ST序列型的时间和空间分布,同一时间段同一所医院以相同克隆流行为主(表4)。结合药敏试验结果,美罗培南对ST11型菌株的MIC50、MIC90均高于非ST11型。亚胺培南的药敏结果显示ST11型的MIC50、MIC90亦高于非ST11型。结合碳青霉烯酶的检测,大部分ST11型菌株携带KPC-2型碳青霉烯酶(29/32,90.6 %),4株ST1型检测出NDM-6碳青霉烯酶,3株ST17携带IMP-4型碳青霉烯酶。
2.4 临床资料统计分析结果
61株CR-KPN菌株感染的患者中,剔除5份患者的病史资料信息不全和同一患者多次分离的菌株(其中包括5 株菌株均分离自同1例患者、4株菌株均分离自同1例患者、3 株菌株均分离自同1例患者、2株菌株均分离自同1例患者4例),余43份病史资料保存完整。
CR-KPN菌株感染的患者男28例,女15例,平均年龄64岁。临床分离的菌株标本类型以呼吸道分泌物和尿液为主,分别占58.1 %(25 / 43)和14.0 %(6 / 43),分离自血液、腹水、引流液、脑脊液、分泌物、灌洗液、胸水的标本分别占9.3 %(4 / 43)、4.7 %(2 / 43)、4.7 % (2 / 43)、2.3 % (1 / 43)、2.3 %(1 / 43)、2.3 % (1 / 43)、2.3 % (1 / 43)。送检的临床科室主要集中在重症医学科(23 例,53.5 %)和神经外科(6例,14.0 %)。65.1 %患者在住院期间更换过床位。超过60 %患者于ICU接受过治疗,治疗过程中患者接受过的有创性操作包括:静脉穿刺(72.1 %)、导尿管留置(72.1 %)、引流管留置(48.8 %)和气管切开(46.5 %)。菌株分离前使用最多的抗菌药物为第三代头孢菌素及酶抑制剂复合制剂(88.4 %)和碳青霉烯类抗菌药物(60.5 %)。67.4 %患者同时还携带其他类型的耐药菌株,其中以鲍曼不动杆菌和铜绿假单胞菌最常见。
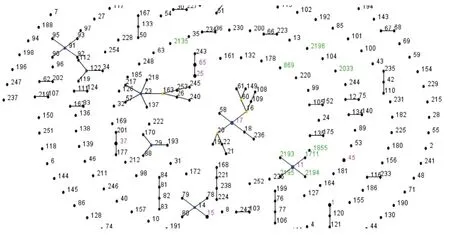
图1 利用eBURST软件分析18种ST型相互关系Figure 1 The eBURST diagram showing the relationship between the 18 sequence types ofK. pneumoniaeisolates
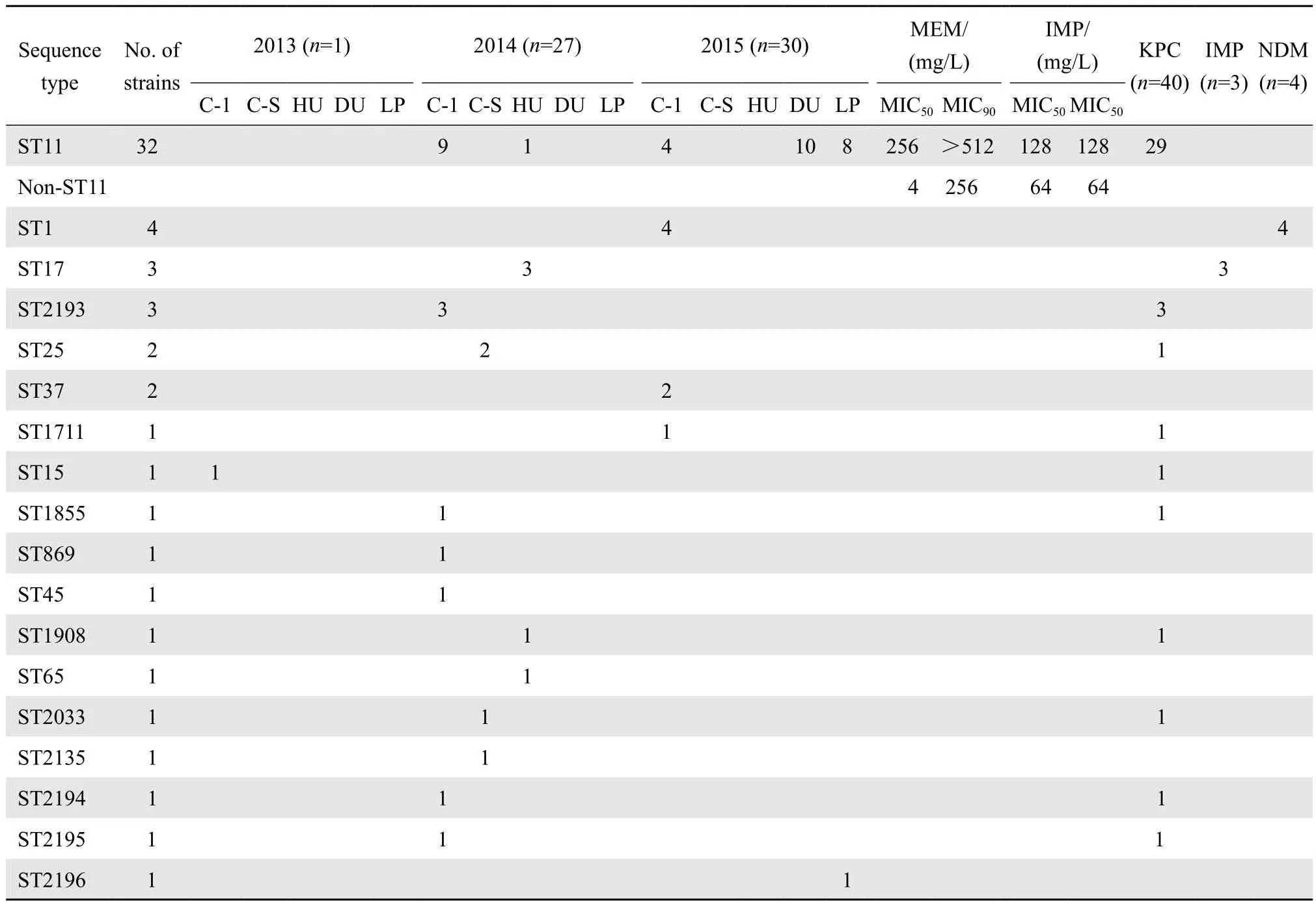
表4 58株肺炎克雷伯菌的MLST分型Table 4 Multilocus sequence typing of 58Klebsiella pneumoniaeisolates
2.5 单因素分析
剔除3例ST测序失败的临床资料,将收集到的40份临床资料分为ST11组(n=23)和非ST11组(包括ST1,ST37等)(n=17),分别进行统计,两组患者在年龄、性别、基础疾病方面差异无统计学意义。ST11组患者在ICU接受治疗所占的比例及机械通气的使用率高于非ST11组,差异均有统计学意义(P值分别为0.015、0.042)。两组患者大部分在治疗过程中接受过包括静脉穿刺、导尿管留置、引流管留置、气管切开等有创性操作,在菌株分离前应用过头孢菌素和碳青霉烯类抗生素,然而两组间这些临床特点差异均无统计学意义。两组的预后方面,ST11组的病死率高于非ST11组(43.4 %,17.6 %),但差异无统计学意义。见表5。
3 讨论
目前世界范围内肺炎克雷伯菌的ST分型已经超过2 000种,美国、挪威、瑞典等欧美国家CRKPN的ST型主要为ST258[7],国内Qi等[8]2012年的研究发现我国CR-KPN MLST主要的流行序列型为ST11, 而研究分析发现ST11和ST258仅有1个管家基因(tonB)的差别,具有较近的亲缘关系,属于同一个克隆复合体CC258。与国内的研究相一致,本实验菌株仍以ST11(32 / 61, 53.3 %)为主,结合前期的药敏试验结果ST11型菌株对美罗培南、亚胺培南的MIC50、MIC90均高于非ST11型,由此推断ST11型对碳青霉烯类抗生素的耐药率偏高,需要进一步大样本试验证实。碳青霉烯酶的检测提示ST11型中以KPC-2酶为主,说明STl1型肺炎克雷伯菌可能非常容易获得编码KPC-2基因的质粒,并且大规模流行,应引起重视。
研究中还发现了6种ST序列型世界范围内尚未有报道,已将数据提交注册,分别给予命名ST2033、ST2135、ST2193、ST2194、ST2195、ST2196。 同 时eBURST分 析 发 现ST2193、ST2194、ST2195与ST11紧密相连,提示有很密切的亲缘性。新型ST的发现提示CR-KPN存在克隆性和多样性,应随时进行密切关注和监测。
结合本研究的临床资料发现在同一时期同一所医院以某一ST序列型为主,如2014年中国医科大学附属第一医院分离的菌株以ST11(9 / 17)为主,中国医科大学附属盛京医院分离的菌株以ST25(2 / 4)为主,哈尔滨医科大学附属第一医院以ST17(3 / 6)为主。同时发现在某些区域集中时间段内以相同克隆流行为主,如2014年中国医科大学附属第一医院分离的17株肺炎克雷伯菌(ST分 型 分 别 为 ST11、ST2193、ST2194、ST2195、ST1855、ST869、ST45), 其 中 在 2014年 3月至6月ICU集中分离了14株菌株,ST序列分别为 ST11、ST2193、ST2194和 ST2195, 通 过 等位基因的比较发现,ST2193、ST2194、ST2195与ST11仅有1个等位基因存在差异, eBURST分析提示具有很近的亲缘性,属于相同克隆,提示ST11型该克隆有在该区域流行的趋势,应引起临床的高度重视,及早进行该克隆的监测和预防,避免广泛传播。
研究同时回顾性收集了 CR-KPN 菌株感染患者的临床资料,发现CR-KPN感染的患者年龄均偏大(平均年龄65岁),高龄患者通常基础疾病较多、免疫力低下、住院时间长,容易成为肠杆菌科细菌感染侵袭的主要对象。研究同时发现超过60 %患者接受过ICU的治疗,由于入住ICU患者多存在多脏器受累,呼吸机辅助通气、侵袭性操作的应用很频繁,治疗上经常给予多种抗菌药物、糖皮质激素及静脉营养液,这些都有可能作为危险因素促成耐药菌的出现。相关研究也已经表明长时间留住ICU是感染CR-KPN的独立危险因素[9-11]。
研究还发现CR-KPN菌株感染的患者常携带以鲍曼不动杆菌、铜绿假单胞菌、白念珠菌为代表的多种病原体。一方面,临床上针对这部分病原体通常给予多种广谱抗生素的治疗,而抗生素的广泛使用已被公认是导致耐药菌感染的危险因素之一[9,12-13],本次调查中,菌株分离前使用最多的抗菌药物为第三代头孢菌素及酶抑制剂复合制剂(88.4 %)和碳青霉烯类抗菌药物(60.5 %),使得肠杆菌科细菌更易于定植或感染,细菌耐药性不断增强。另一方面,一些研究还发现耐药基因可通过质粒在不同的细菌种属间进行转移播散[2],导致耐药菌属扩大,给临床治疗造成极大的困难。
结合ST分型,分别研究ST11组和非ST11组的临床资料发现,ST11组患者在ICU接受治疗所占的比例及机械通气的使用率高于非ST11组,差异均有统计学意义(P值分别为0.015、0.042)。由此可以推断入住ICU、接受机械通气是CR-KPN(特别是ST11型菌株)感染的危险因素,结合前期的药敏试验结果ST11型菌株更容易对碳青霉烯类抗生素耐药且MIC值较高,给治疗带来极大的挑战。
[1] WOODFORD N, TIERNO PM JR, YOUNG K JR, et al.Outbreak ofKlebsiella pneumoniaeproducing a new carbapenem-hydrolyzing class A β-lactamase, KPC-3, in a New York Medical Center[J]. Antimicrob Agents Chemother,2004,48(12):4793-4799.
[2] QUEENAN AM,BUSH K. Carbapenemases:the versatile beta-lactamases[J]. Clin Microbiol Rev, 2007,20(3):440-458.
[3] AUBRON C, POIREL L, ASH RJ,et al. CarbapenemaseproducingEnterobacteriaceae, U. S. rivers[J]. Emerg Infect Dis,2005,11(2):260-264.
[4] SENDA K,ARAKAWA Y,ICHIYAMA S,et al. PCR detection of metallo- lactamase gene(bla-IMP) in gramnegative rods resistant to broad-spectrum β-lactmas[J]. J Clin Microbiol,1996,34(12): 2909-2913.
[5] POIREL L, REVATHI G, BERNABEU S, et al. Detection of NDM-1-producingKlebsiella pneumoniaein Kenya[J].Antimicrob Agents Chemother,2011,55(2): 934-936.
[6] YAN JJ,HSUEH PR,KO WC,et al. Metallo-beta-lactamases in clinicalPseudomonasisolates in Taiwan and identi fication of VIM-3, a novel variant of the VIM-2 enzyme[J]. Antimicrob Agents Chemother,2001,45(8):2224-2248.
[7] ADLER A,HUSSEIN O,BEN-DAVID D,et al. Persistence ofKlebsiella pneumoniaeST258 as the predo minant clone of carbapenemase-producingEnterobacteriaceaein post-acute-care hospitals in Israel, 2008-13[J]. J Antimicrob Chemother,2014,70(1):89-92.
[8] QI Y,WEI Z,JI S,et al. ST11, the do minant clone of KPC-producingKlebsiella pneumoniaein China[J]. J Antimicrob Chemother,2011,66(2):307-312.
[9] GASINK LB,EDELSTEIN PH,SYNNESTVEDT E,et al. Risk factors and clinical impact ofKlebsiella pneumoniaecarbapenemase-producingK. pneumoniae[J]. Infect Control Hosp Epidemiol,2009, 30(12):1180-1185.
[10] ORSI GB,GARCÍA-FERNÁNDEZ A,GIORDANO A,et al. Risk factors and clinical signi fi cance of ertapenem-resistantKlebsiella pneumoniaein hospitalised patients[J]. J Hosp Infect,2011,78(1):54-58.
[11] LING ML,TEE YM, TAN SG,et al. Risk factors for acquisition of carbapenem resistantEnterobacteriaceaein an acute tertiary care hospital in Singapore[J]. Antimicrob Resist Infec Control,2015, 4(1):26.
[12] MARCHAIM D, CHOPRA T, BHARGAVA A,et al.Recent exposure to antimicrobials and carbapenem-resistantEnterobacteriaceae: the role of antimicrobial stewardship[J].Infect Control Hosp Epidemiol,2012,33(8): 817-830.
[13] SCHECHNER V,KOTLOVSKY T,TARABEIA J,et al. Predictors of rectal carriage of carbapenem-resistantEnterobacteriaceae(CRE) among patients with known CRE carriage at their next hospital encounter[J]. Infect Control Hosp Epidemiol,2011,32(5):497-503.
Multilocus sequence typing of carbapenem-resistant Klebsiella pneumoniae isolates and its utility in pro filing clinical infections
WU Na, TIAN Sufei, CHU Yunzhuo, CHEN Baiyi, LIU Liwen, ZHANG Zhijie, WANG Jing, XIAO Xiaoguang,LU Juan. (Department of Infectious Diseases, the First Affiliated Hospital of China Medical University,Shenyang 110001, China)
R378.996
A
1009-7708 ( 2017 ) 05-0509-07
10.16718/j.1009-7708.2017.05.005
2016-11-30
2017-01-17
1. 中国医科大学附属第一医院感染科,沈阳 110001;*检验科;
2. 辽宁省人民医院检验科;
3. 中国医科大学附属盛京医院;
4. 大连医科大学附属第一医院检验科;
5. 哈尔滨医科大学附属第一医院检验科。
吴娜(1983—),女,博士,讲师,主要从事感染性疾病研究和诊治。
陈佰义,E-mail:Chenbaiyi63@163.com。
