生物素标记的甲基化间区位点扩增技术分辨不同组织DNA甲基化水平方法初探
2017-02-16王小怡王博王雪伟高哲郭大玮
王小怡,王博,王雪伟,高哲,郭大玮
生物素标记的甲基化间区位点扩增技术分辨不同组织DNA甲基化水平方法初探
王小怡,王博,王雪伟,高哲,郭大玮
DNA 甲基化是重要的表观遗传修饰之一。在哺乳动物细胞中,DNA 甲基化参与基因表达和染色质结构的调控[1],与多种癌症的发生发展相关[2-3]。DNA 甲基化在胚胎发育早期、个体生长发育及多种疾病的发生发展过程中起着重要的作用,是当前表观遗传研究领域的热点之一[4-5]。分析基因组中甲基化水平能够了解不同组织之间、正常与疾病状态以及不同年龄引起的甲基化水平的差异。Frigola 等[6]通过扩增甲基化间区位点(amplification of intermethylated sites,AMIS)分析了结肠癌组织与癌旁组织基因组中 CCCGGG位点的甲基化水平。该法通过 [α-33P]dATP 进行放射自显影,对机体有很大的损害。有文献报道可以用生物素标记的引物进行 PCR 扩增[7],因此本研究尝试用生物素标记引物进行 PCR 扩增代替放射性核素法分析基因组 DNA 甲基化水平(图 1)。
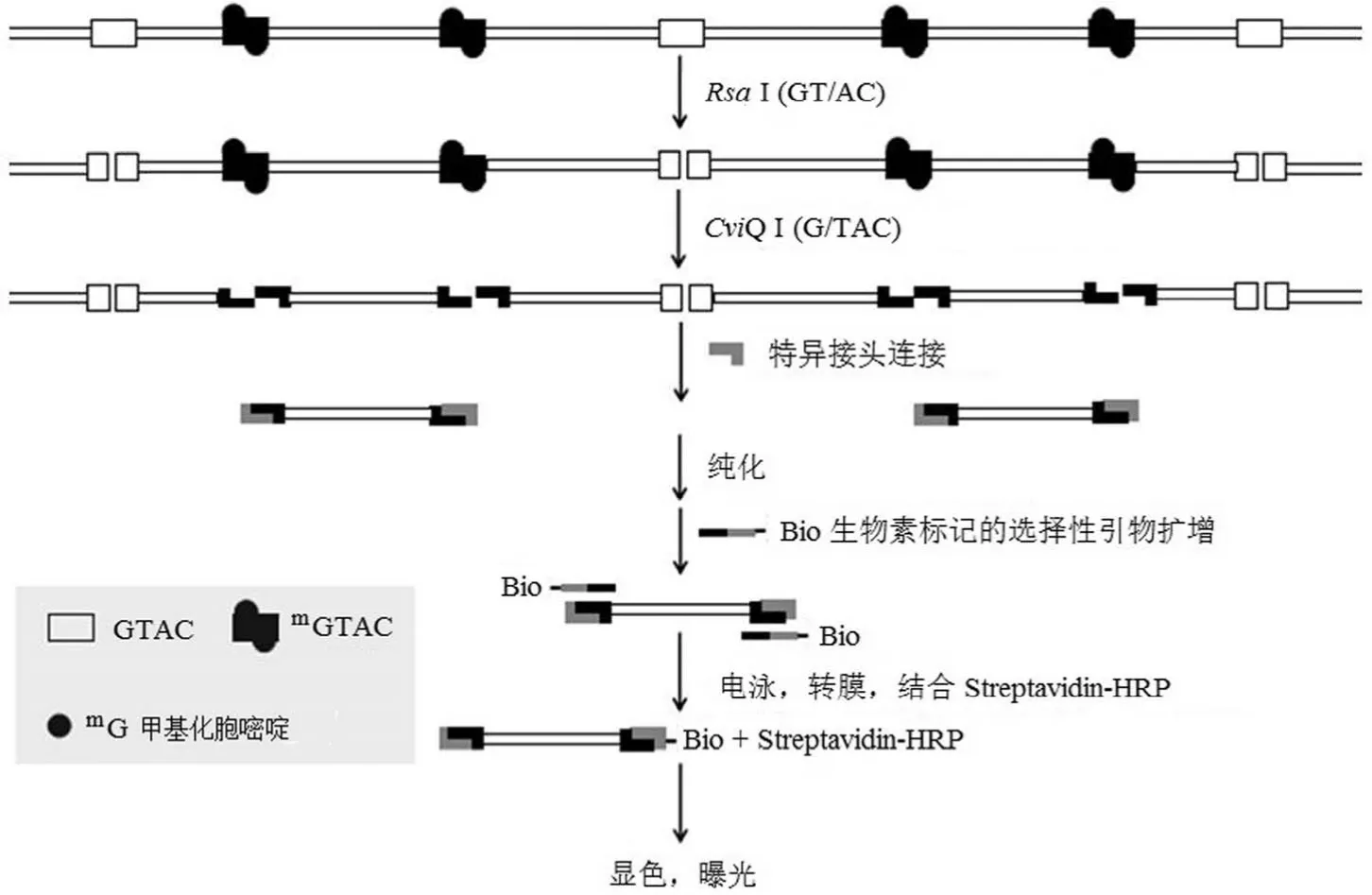
图 1 生物素标记引物进行 PCR 扩增分析基因组 DNA 甲基化水平原理示意图(双线代表基因组 DNA)
1 材料与方法
1.1 材料
1.1.1 实验动物 SPF 级 SD 大鼠 9 只,标记为 1 ~9 号,雄性,体重 180 ~ 200 g,购于中国人民解放军军事医学科学院实验动物中心(许可证编号:SCXK-(军)2012-0004),饲养于山西医科大学实验动物中心。
1.1.2 主要试剂和仪器 限制性内切酶 Rsa I 和 CviQ I、T4 连接酶均购自美国 NEB 公司;PCR 产物纯化试剂盒购自生工生物工程(上海)股份有限公司;rTaq 聚合酶购自日本 Takara 公司;化学发光试剂盒购自上海碧云天生物技术有限公司;TC-96/G/H(b)B 型基因梯度扩增仪购自杭州博日科技有限公司。
1.2 方法
1.2.1 9 只大鼠随机分为两组,1 ~ 4 号正常饲养;5 ~ 9 号脑部局部双侧给予 2 nmol/L 的 δ-阿片受体激动剂 1 μl。一周后处死大鼠,取心、脑、肝、肺、肾、脾、胰组织,置于–80 ℃ 备用。
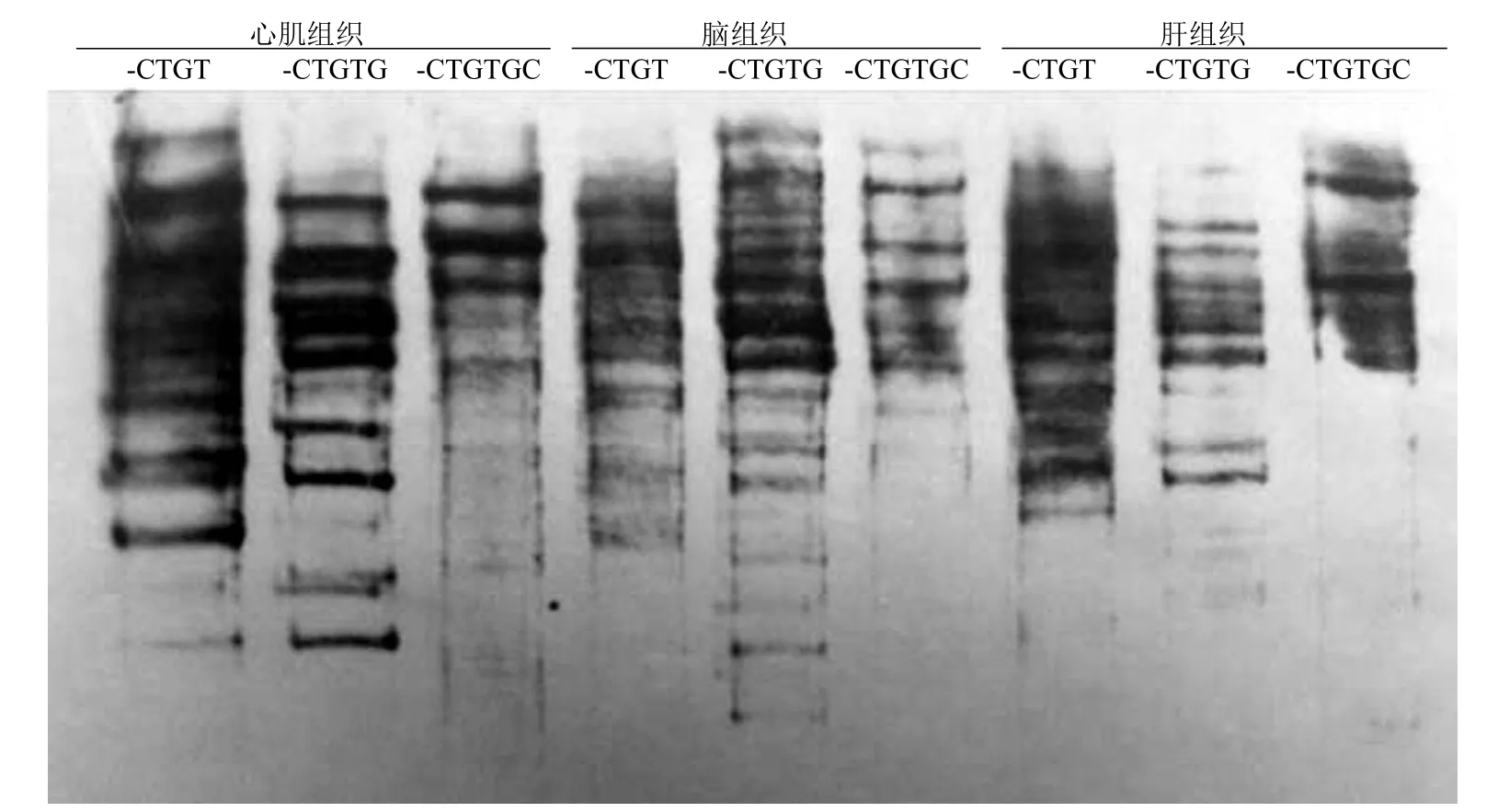
图 2 5 号大鼠心、脑、肝组织以不同引物扩增时甲基化图谱
1.2.2 大鼠组织基因组 DNA 提取 酚氯仿法提取大鼠组织的基因组 DNA(100 ng/μl),A260/280在 1.8 ~ 1.9。
1.2.3 甲基化间区位点扩增 10 U 的甲基化敏感性内切酶 Rsa I 消化 1 μg 基因组 DNA,37 ℃ 过夜酶切,留下钝性末端(GT/AC);酶切产物用 10 U 甲基化非敏感性内切酶 CviQ I 消化,25 ℃ 过夜酶切,留下粘性末端(G/TAC)。特异接头准备:100 μmol/L MCA-V(5' TATCAG AGCTTTGCGAAT 3')和 100 μmol/L Blue(5' ATTCGCAAA GCTCTGA 3')等体积混合置于 95 ℃ 10 min,室温约 2 h退火;1 nmol的接头与 1 μg 双酶切的产物在 T4 连接酶的作用下连接。PCR 产物纯化试剂盒纯化连接产物。
1.2.4 生物素标记的引物扩增两端连接特异接头的 DNA序列 用 5'-Bio-Blue-TAC 末端加 4 ~ 6 个随机核苷酸的引物进行 PCR 扩增,引物序列为 5'-Bio-Blue-TAC-CTGT-3,5'-Bio-Blue-TAC-CTGTG-3',5'-Bio-Blue-TAC-CTGTGC-3'。扩增体系:30 ng 连接产物,4 μmol/L 生物素标记的引物,1 U rTaq,2 μl dNTP,2.5 μl 10 × PCR buffer,灭菌水补足25 μl。扩增条件:95 ℃ 10 min;94 ℃ 15 s,68 ℃ 45 s,72 ℃ 1 min,35 个循环;72 ℃ 10 min。
1.2.5 聚丙烯酰胺凝胶电泳 PCR 产物稀释 5 倍,3 μl稀释后产物与 12 μl 的变性上样缓冲液 DLB(95% 甲酰胺、20 mol/L EDTA、0.09% 溴酚蓝、0.09% 二甲苯青)混匀后 95 ℃ 变性 10 min,置于 –20 ℃ 备用。变性后样品上样 5 μl,8 mol/L 尿素、6% 的聚丙烯酰胺凝胶 200 V 恒压电泳 2 h。使用半干转印仪,以 0.5 × TBE 为转膜液,将凝胶上的 DNA 转移到尼龙膜上,电压保持在 6 ~ 11 V 的条件下恒流转膜 45 min。按化学发光 EMSA 试剂盒使用说明书进行显色。
2 结果
2.1 引物的选择以及同一只大鼠不同组织之间甲基化水平的比较
3' 末端随机核苷酸数目的增加能够减少扩增产物的条带数目,使甲基化图谱条带清晰且容易分析。条带灰度值越高说明甲基化水平越高。5 号大鼠的心、脑、肝组织分别以3' 末端增加 4、5、6 个核苷酸为引物扩增得到的甲基化图谱,随着末端核苷酸数的增加,条带数目逐渐减少(图 2)。选择 3' 末端增加 4 个碱基(即 Bio-Blue-TAC-CTGT)为引物来分析大鼠组织的甲基图谱,表明同一个引物同一只大鼠的不同组织的甲基化图谱有明显差异。
2.2 不同大鼠脑组织甲基化水平图谱基本一致
1 ~ 4 号大鼠的脑组织甲基化图谱基本一致。5 号大鼠经过局部脑阿片受体激动剂处理,其脑组织甲基化水平图谱与 1 ~ 4 号大鼠脑组织有较大差异(图 3)。
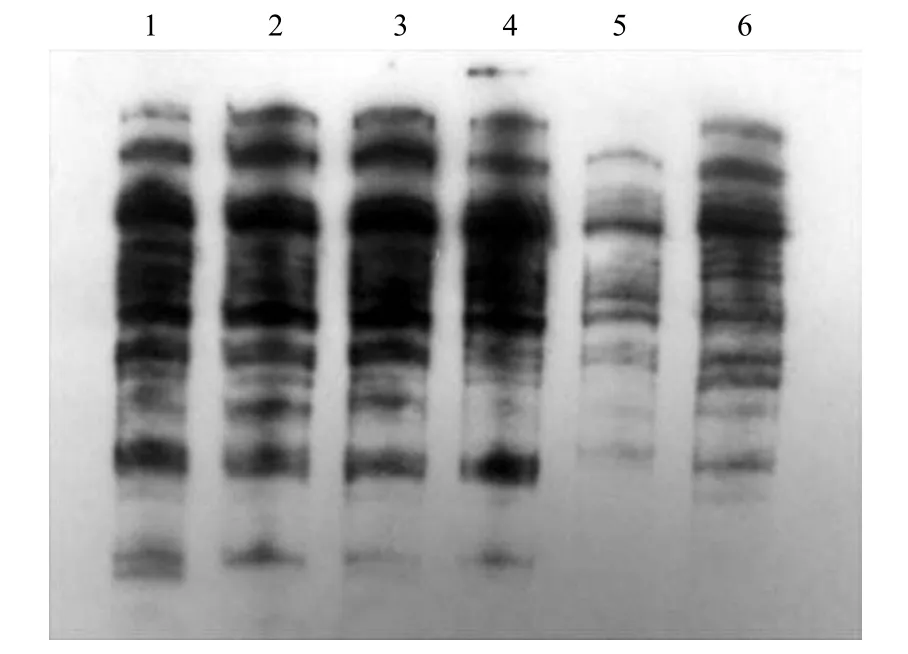
图 3 不同大鼠组织甲基化图谱
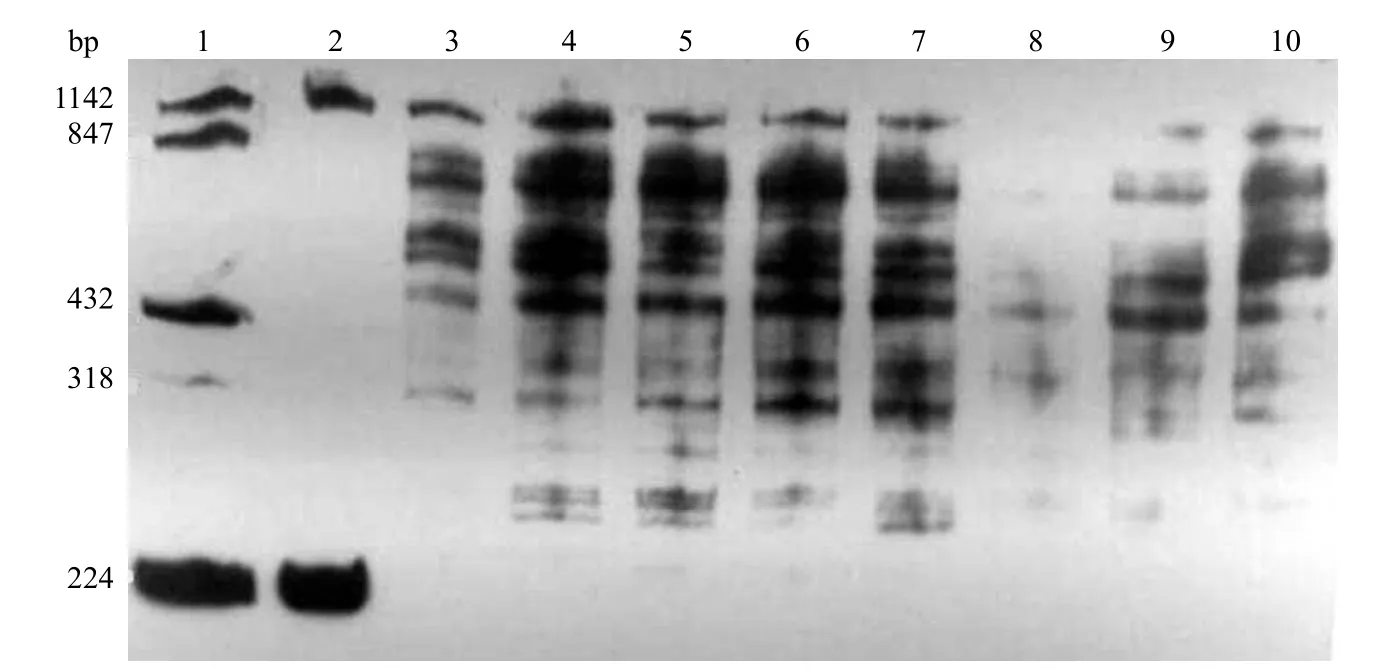
图 4 DNA 片段长度范围分析
2.3 甲基化位点之间 DNA 片段长度范围分析
采用本实验室现有的 H19 基因的引物,共用的上游引物 5' 端进行生物素标记,选择 5 个不同的下游引物扩增后产物等比例混合制成标准品,电泳时和样品一起上样。由图 4 可知电泳条带在 200 ~ 1200 bp。
3 讨论
用 AMIS 方法对基因组范围内的甲基化水平进行检测首先由 Frigola 等于 2002 年提出。实验中作者采用SmaI/PspA1(CCCGGG)这对内切酶及接头 DNA 巧妙地分离了甲基化与非甲基化状态的胞嘧啶。由于在全基因组范围内进行检测,因此,与预设甲基化位置的基因芯片检测方法相比,检测范围更加宽泛,例如:Illumina 推出的 Infinium Methylation EPIC BeadChip 一次可以检测超过 850 000 个甲基化位点[8],但这些甲基化位点倾向于与已注释的基因表达调控相关,不免有检测局限性;根据 Fazzari 和 Greally[9]对人类基因组计划的资料分析表明,甲基化敏感酶 Hpa II的识别位点分布于人类已注释的 CpG 岛区域,约占总切点的 12%,其余大部分切点分布于其他未知的区域;Xu 等[10]在人类长片段重复序列检出了可能作为分辨同卵双生子DNA 甲基化位点,这些说明立足于整个基因组范围内检测DNA 甲基化的 AMIS 方法有其独特的优点。
另一方面,由于 AMIS 检测所涉及的片段较多,用普通显色方法灵敏度不够,因此,Frigola 等[6]采用了放射性检测方法 。本文用生物素标记的引物检测出了不同脏器的不同 DNA 甲基化水平。据 Frigola 的报道,同位素方法的检测灵敏度为 1/16,我们在实验过程中将样品稀释到 1/10仍能检测到甲基化条带。
与放射性同位素标记相比,生物素标记的引物 AMIS操作安全、简单,适用于多个样本,并能达到检测出不同脏器的不同 DNA 甲基化水平的目的。该方法的灵敏度较高,少量的产物即可得到清晰的基因组 DNA 甲基化水平图谱。因此,我们认为虽然非放射性 AMIS 的检测灵敏度低于放射性标记,但 1 μg 的 DNA 模板用生物素标记的AMIS 法已然能够检出足够条带,从而满足多数实验的要求。如此可以避免放射性核素的使用。
本文通过非放射性 AMIS 替代放射性方法,得到了较为清晰的酶切位点基因组 DNA 甲基化水平图谱。同一只大鼠的不同组织甲基化水平有明显差异,而不同大鼠同一个组织的甲基化水平基本一致。唐韶青等[11]用 MSAP 技术对猪、牛、羊、小鼠、鸡和鸭的基因组 DNA 甲基化水平检测,发现同一动物的不同组织基因组 DNA 甲基化水平有差异。另有文献报道,人的不同组织之间甲基化水平也是有差异的[12-13],这与本研究结果一致。
本文中采用 Rsa I/CviQ I(GTAC)内切酶对进行检测,观察了基因组中非 CpG 二核苷酸处的甲基化修饰情况。与Frigola 等所用的 6 核苷酸内切酶相比,我们采用的识别4 核苷酸的内切酶理论上可能检出更多的甲基化位置,采用3 端增加 4 ~ 6 个碱基的引物扩增,仍然能够得到较多的条带,更适用于两个样本间甲基化差异较少的检材。此外,4 核苷酸内切酶分析甲基化水平得到的甲基化间区的 DNA 范围 200 ~ 1200 bp,较 Frigola 等所用的 6 核苷酸内切酶得到的范围窄,这是因为 4 核苷酸比 6 核苷酸出现的频率高。
该方法的使用也存在一些局限性。图 3 中 1 ~ 4 脑组织是鼠龄、体重情况一致的大鼠,其甲基化水平基本一致。若在脑局部给阿片受体激动剂(5 号大鼠),则其脑组织甲基化图谱与 1 ~ 4 鼠脑组织有明显不同,慢性使用阿片导致脑组织的 DNA 甲基化水平发生改变已有报道[14],本实验也观察到经上述处理的鼠甲基化水平图谱异于未处理鼠。可见药物等会影响个体基因组甲基化格局的改变。此外,基因组水平 DNA 甲基化与年龄、疾病等亦有着密切的关系[15-17]。上述问题在使用该方法鉴别不同组织时应加以考虑。此外,由于该方法基于内切酶酶切,因此对 DNA 质量要求较高,对于 DNA 降解的样本则无法进行检测。脾脏 DNA 提取后琼脂糖凝胶电泳发现 DNA 降解严重,这可能是导致图 4 四个脾组织之间的甲基化格局不同的原因。因而,使用该方法应注意 DNA 提取质量,避免 DNA降解;同时,比较样品间的 DNA 甲基化差异应保持实验条件的齐同。
本次研究初步探索了用生物素标记的 AMIS 法替代放射性 AMIS 法进行大鼠脑组织甲基化水平检测的可能性。后续我们期待用该方法探索其他组织的甲基化图谱,找出各种组织之间基因组 DNA 的甲基化差异,以期用甲基化水平来区分不同组织。
[1] Jones PA, Takai D. The role of DNA methylation in mammalian epigenetics. Science, 2001, 293(5532):1068-1070.
[2] Plass C, Smiraglia DJ. Genome-wide analysis of DNA methylation changes in human malignancies. Curr Top Microbiol Immunol, 2006, 310:179-198.
[3] Verma M. The role of epigenomics in the study of cancer biomarkers and in the development of diagnostic tools. Adv Exp Med Biol, 2015, 867:59-80.
[4] Reik W, Dean W, Walter J. Epigenetic reprogramming in mammalian development. Science, 2001, 293(5532):1089-1093.
[5] Robertson KD. DNA methylation and human disease. Nat Rev Genet, 2005, 6(8):597-610.
[6] Frigola J, Ribas M, Risques RA, et al. Methylome profiling of cancer cells by amplification of inter-methylated sites (AMIS). Nucleic Acids Res, 2002, 30(7):e28.
[7] Niemeyer CM, Adler M, Pignataro B, et al. Self-assembly of DNA-streptavidin nanostructures and their use as reagents in immuno-PCR. Nucleic Acids Res, 1999, 27(23):4553-4561.
[8] Illumina, Inc. Comprehensive coverage for epigenome-wide association studies -- Quantitatively interrogate methylation sites across the genome at single-nucleotide resolution. [2016-09-10] http:// www.illumina.com/techniques/microarrays/methylation-arrays.html.
[9] Fazzari MJ, Greally JM. Epigenomics: beyond CpG islands. Nat Rev Genet, 2004, 5(6):446-455.
[10] Xu J, Fu G, Yan L, et al. LINE-1 DNA methylation: A potential forensic marker for discriminating monozygotic twins. Forensic Sci Int genet, 2015, 19:136-145.
[11] Tang SQ, Zhang Y, Xu Q, et al. Analysis of methylation level of genome in various tissues of different animal species. J Agric Biotechnol, 2006, 14(4):507-510. (in Chinese)唐韶青, 张沅, 徐青, 等. 不同动物部分组织基因组甲基化程度的差异分析. 农业生物技术学报, 2006, 14(4):507-510.
[12] Li W, Liu M. Distribution of 5-hydroxymethylcytosine in different human tissues. J Nucleic Acids, 2011, 2011:870726.
[13] Khare T, Pai S, Koncevicius K, et al. 5-hmC in the brain is abundant in synaptic genes and shows differences at the exon-intron boundary. Nat Struct Mol Biol, 2012, 19(10):1037-1043.
[14] Doehring A, Oertel BG, Sittl R, et al. Chronic opioid use is associated with increased DNA methylation correlation with increased clinical pain. Pain, 2013, 154(1):15-23.
[15] Hernandez DG, Nalls MA, Gibbs JR, et al. Distinct DNA methylation changes highly correlated with chronologicalage in the human brain. Hum Mol Genet, 2011, 20(6):1164-1172.
[16] Steegenga WT, Boekschoten MV, Lute C, et al. Genome-wide age-related changes in DNA methylation and gene expression in human PBMCs. Age (Dordr), 2014, 36(3):9648.
[17] Kang JT, Liang QJ, Liang C, et al. Overview on epigenetics and its progress. Sci Technol Rev, 2013, 31(19):66-74. (in Chinese)康静婷, 梁前进, 梁辰, 等. 表观遗传学研究进展. 科技导报, 2013, 31(19):66-74.
国家发改委、科技部印发高级别生物安全实验室体系建设规划(2016 – 2025年)
按照国务院《病原微生物实验室生物安全管理条例》等有关要求,国家发展和改革委员会会同有关部委联合编制了《高级别生物安全实验室体系建设规划(2016 – 2025 年)》。
详情请登录国家发展和改革委员会网站 http://www.ndrc.gov.cn/zcfb/zcfbtz/201612/t20161220_830455.html 查阅。
10.3969/cmba.j.issn.1673-713X.2017.01.016
030001 太原,山西医科大学法医学院
郭大玮,Email:guo8dawei@aliyun.com
2016-09-20
