蓝点马鲛鱼内参基因的克隆及表达稳定性评价
2016-06-29张家玲薛良义史钧信
张家玲,薛良义,史钧信
(宁波大学 海洋学院, 宁波 315211)
蓝点马鲛鱼内参基因的克隆及表达稳定性评价
张家玲,薛良义,史钧信
(宁波大学 海洋学院, 宁波 315211)
摘 要合适内参基因的选择是实时荧光定量PCR技术准确测定目的基因表达量的前提。首先克隆了蓝点马鲛鱼3个内参基因β-actin、GAPDH和EF1-α的部分序列,并通过实时荧光定量PCR分析β-actin、GAPDH、EF1-α和18S rRNA 4个内参基因在蓝点马鲛鱼各组织中的Ct值,应用3种内参基因筛选软件(geNorm,NormFinder和Bestkeeper)综合分析这4个基因的表达稳定性。结果表明最稳定的内参基因是EF1-α,其次是18S rRNA。研究结果为今后蓝点马鲛鱼合适内参基因的选择提供了依据。
关键词蓝点马鲛鱼;实时荧光定量PCR;内参基因;geNorm软件;表达稳定性
实时荧光定量PCR(Quantitative Real-time PCR,qPCR)技术比传统的半定量及Northern杂交等方法具有灵敏度高、特异性强、快速准确等优势,可以更准确分析基因表达差异,成为基因表达水平测定的最常用的方法[1, 2]。但是,这种方法的准确性受到多种因素的影响,包括RNA的质量、cDNA反转录酶的活性、PCR扩增效率及合适的内参基因等[3, 4],其中内参基因的选择是一个至关重要的环节。内参基因在生物体不同组织、不同发育时期或不同实验条件下具有相对稳定的表达特性[5-7]。但是,近年的研究表明,在不同实验体系中,内参基因的表达稳定性是不一样的。Olsvik等对大西洋鲑鱼8种组织中6个内参基因的表达稳定性进行分析,结果表明:在肌肉、肝脏、腮、头肾、脾脏、胸腺中EF1AB是最稳定的;在脑和肠中最稳定的分别是EF1AA和β-actin。GAPDH在肝脏、头肾、脾脏、脑和胸腺中稳定性最差。建议EF1AA和EF1AB可作为大西洋鲑鱼基因表达定量研究的内参基因[8]。Ma等在许氏平鲉qPCR内参基因筛选中,发现8种内参基因在心、肝脏、肠、腮、肌肉、脑、肾脏、脾脏、卵巢和睾丸等组织中表现出不同的稳定性,RPL17和EF1A是最稳定的,GAPDH是最不稳定的[9]。所以对于不同的实验体系,必须进行最适合内参基因的筛选,以减少实验的误差。蓝点马鲛鱼(Scomberomorusniphoius)隶属于鲈形目、鲅科、马鲛属,是一种重要的经济鱼类。近几年人工养殖马鲛鱼已经初步成功,但是蓝点马鲛鱼基因功能方面的研究非常少,NCBI数据库只报道了18SrRNA序列,Zhang等在运用分子生物学方法识别中国南海鱼类时,蓝点马鲛鱼18SrRNA序列被用于DNA条形码技术中的一种工具[10]。
本实验选取qPCR最常使用的β-actin,GAPDH,EF1-α和18SrRNA这4 个内参基因作为筛选蓝点马鲛鱼qPCR最佳内参基因的候选基因。利用geNorm软件[11]、NormFinder软件[12]和BestKeeper软件[13]分析这4个候选内参基因在各组织中表达的稳定性,筛选出最合适的内参基因。
1 材料与方法
1.1 实验材料
蓝点马鲛鱼全部捕自浙江象山港,平均体重(0.6±0.15)kg。
1.2 方法
1.2.1总RNA提取及cDNA第一链合成
蓝点马鲛鱼活体解剖后取样(每组样品分别取自3条鱼),取脑、眼、腮、肾、脾、肝脏、肠、性腺、胃、心和肌肉等11个组织,每个组织约100 mg于1.5 mL RNasefree管中,加入1 mL Trizol(Invitrogen,USA)试剂,参照Trizol说明书抽提RNA。1%琼脂糖凝胶电泳检测 RNA的完整性,超微量紫外分光光度计测定RNA浓度及纯度。每份组织取2 μg RNA,按照Prime Script RT reagent Kit with gDNA Eraser(Takara,大连宝生物)说明书反转录合成cDNA第一链。得到的cDNA 样本-20 ℃保存。
1.2.2基因组DNA的提取
采用SQ Tissue DNA Kit(Omega,USA)从肌肉组织提取蓝点马鲛鱼基因组DNA。具体步骤参照说明书。
1.2.3蓝点马鲛鱼内参基因的克隆
采用同源克隆的方法克隆β-actin、GAPDH和EF1-α这3个内参基因。在NCBI数据库中,搜索已登录的鲈形目的大黄鱼(Larimichthyscrocea,GenBank number:GU584189.1);重牙鲷(Diplodussargus,GenBank:JN210581.1);点带石斑鱼(Epinepheluscoioiaes,GenBank: AY510710.2)等的β-actin基因序列,利用Primer5.0和DNAstar软件,设计扩增保守区的兼并引物,命名为β-actin-f和β-actin-r。
EF1-α和GAPDH的克隆步骤参照上述β-actin。引物EF-f/EF-r克隆EF1-α;GAPDH-f/GAPDH-r克隆GAPDH。分别以蓝点马鲛鱼肌肉组织DNA和cDNA为模版进行PCR扩增,程序如下:94 ℃预变性4 min;94 ℃变性45 s,58 ℃退火45 s,72 ℃延伸90 s,共32个循环;72 ℃再延伸10 min。PCR产物经1%的琼脂糖凝胶电泳分离,切胶回收,连接到pMD19-T载体上,转化大肠杆菌DH5α感受态细胞,挑取阳性克隆送上海华大生物技术有限公司测序。测序结果用DNAstar和blast等软件进行分析比较。
1.3 定量引物的设计
在β-actin、GAPDH和EF1-α内部设计1对跨内含子的特异性引物作为定量引物,分别命名:β-ref-f/β-ref-r;GAPDH-ref-f/GAPDH-ref-r和EF-ref-f /EF-ref-r。根据GenBank上已报道的蓝点马鲛鱼18SrRNA(Scomberomorusniphonius,GenBank number:JN211933.1)序列设计特异性的定量引物:18S-ref-f和18S-ref -r。引物相关信息见表1。

表1 实验引物
1.4 qPCR分析
采用Fast Start Universal SYBR Green Master (Rox)(罗氏,上海)和Eppendorf Mastercycler ep Realplex2PCR仪(Eppendorf,德国)进行qPCR实验。具体步骤参照说明书。在延伸阶段检测荧光强度,收集信号。之后进行55℃~95℃熔解曲线分析。每个样品设3 个重复。
2 结果
2.1 蓝点马鲛鱼β-actin、GAPDH和EF1-α的克隆与鉴定
引物β-actin-f/β-actin-r分别以蓝点马鲛鱼肌肉cDNA和DNA为模板,克隆得到蓝点马鲛鱼β-actin的cDNA序列1 128 bp,DNA序列2 114 bp(图1)。cDNA和DNA的序列比对显示蓝点马鲛鱼β-actin含有5个外显子和4个内含子,外显子和内含子的边界符合GT-AG规律。其中第1个内含子长度363 bp,第2个内含子长度415 bp,第3个内含子长度112 bp,第4个内含子长度96 bp。序列处理在线工具包(SMS)分析发现其cDNA编码375个氨基酸,预测其蛋白质分子质量为41.733 ku,理论等电点(pI)为5.28。序列已提交GeneBank(GeneBank number:KT009015)。
引物GAPDH-f/GAPDH-r和EF-f/EF-r以蓝点马鲛鱼肌肉DNA为模板,克隆得到蓝点马鲛鱼GAPDH和EF1-α部分DNA序列,片段大小分别为:272 bp和383 bp(图1),GAPDH和EF1-α序列已提交GeneBank(GeneBank number:KT009013;KT009014)。
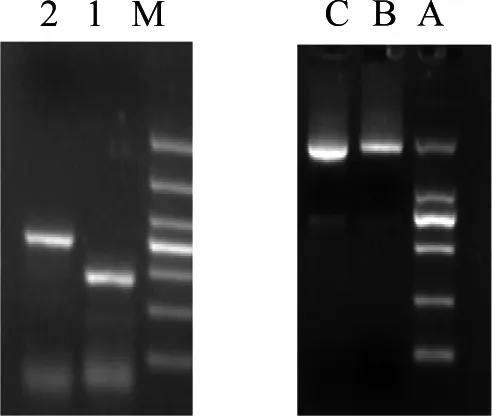
图1 β-actin、GAPDH和EF1-α PCR产物
M:2000 bp Marker;1、2:GAPDH和EF1-α的DNA片段;A:2000 bp Marker;B、C:β-actin的DNA片段
2.2 内参基因定量引物的检测
qPCR预试验检测引物特异性(图2)。从熔解曲线中可以看出,引物扩增片段的特异性很好,并且无引物二聚体。
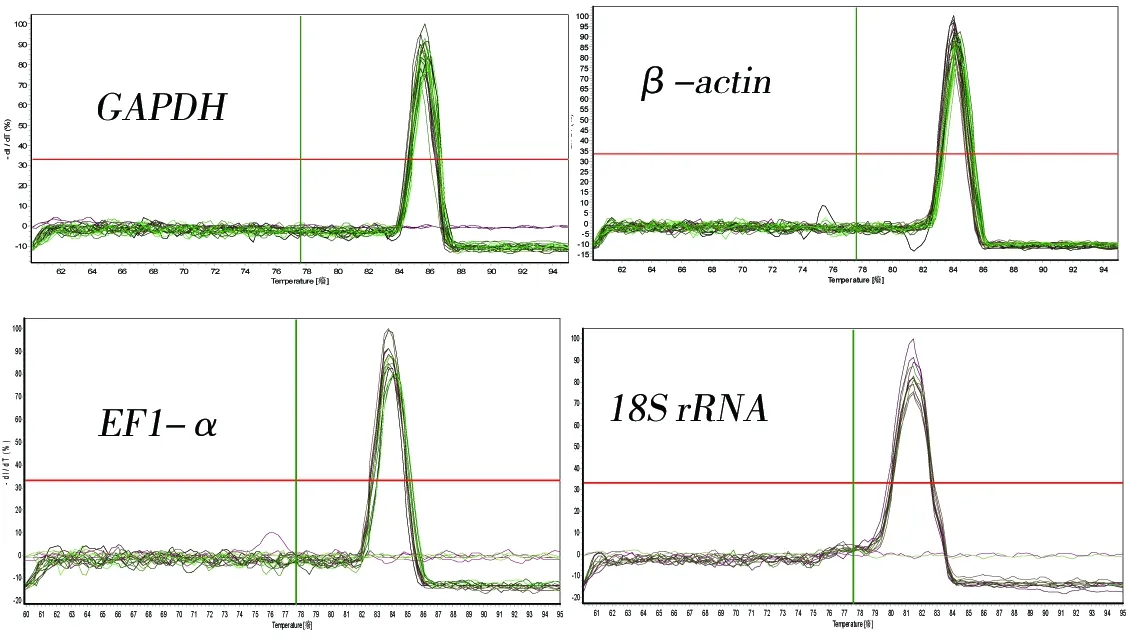
图2 4对引物的溶解曲线
2.3 不同组织中各内参基因的Ct值
4个内参基因在各组织中的Ct值对比显示如图3。总体来说,18SrRNA在11种组织中的表达量最高(CtMean=13.04±0.105);脑、眼、腮、肾、性腺、心和胃组织中EF1-α次之(CtMean=20.52±0.09),脾和肝中β-actin次之,肌肉和肠中GAPDH次之;GAPDH在脑、眼、脾、肝、性腺、腮和肾脏中的表达量最低(CtMean=25.27±0.15),但是在心、肌肉、胃和肠中β-actin的表达量是最低的。
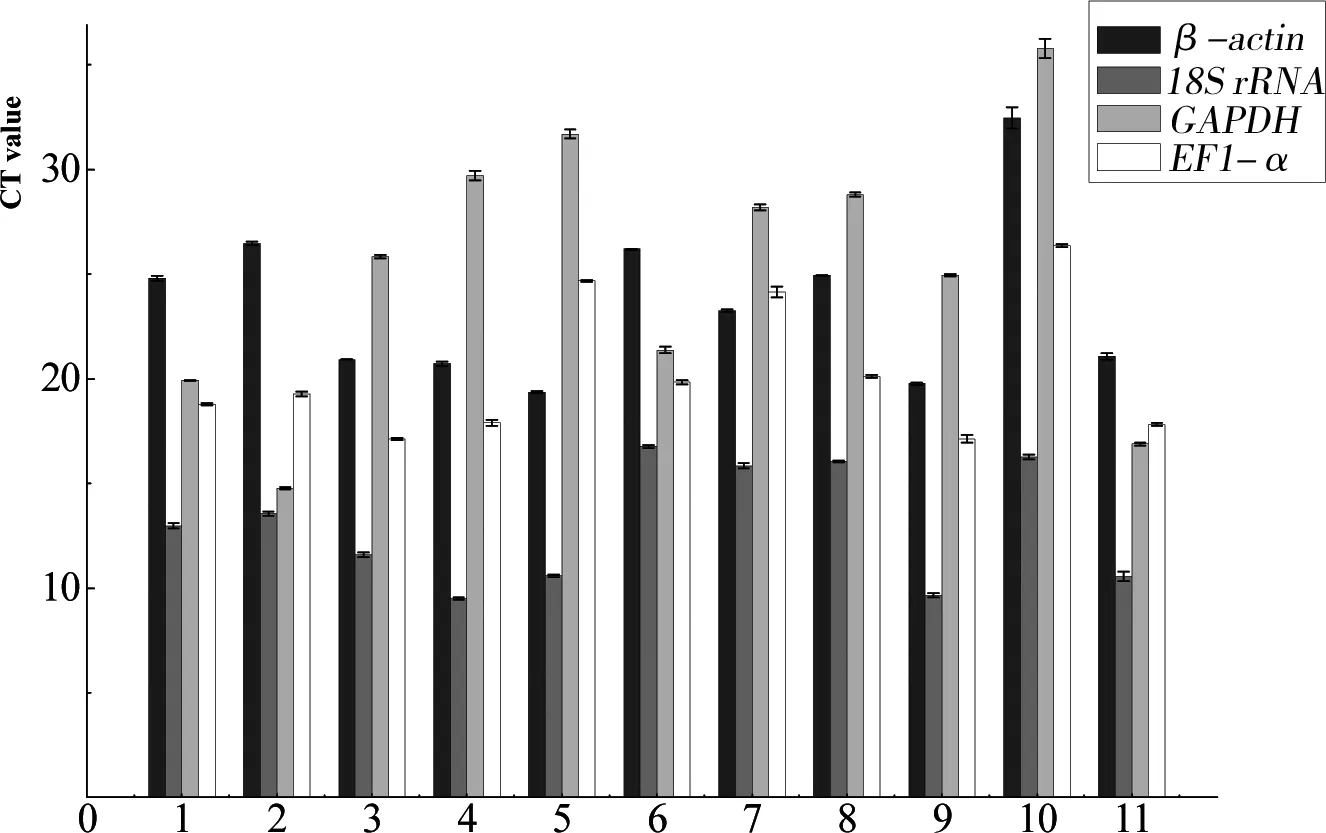
图3 4个内参基因在蓝点马鲛鱼11种不同组织中
1:心;2:肌肉;3:脑;4:眼;5:脾脏;6:胃;7:肝脏;8:性腺;9:腮;10:肾脏;11:肠
2.4 内参基因的稳定性
运用geNorm软件对4个内参基因稳定值进行计算,内参基因β-actin、18SrRNA、GAPDH和EF1-α的稳定值M依次为:0.440、0.406、0.630及0.393,基因稳定性排序为EF1-α>18SrRNA>β-actin>GAPDH。
经过NormFinder程序计算,本试验中蓝点马鲛鱼候选内参基因稳定性排序:EF1-α>β-actin>18SrRNA>GAPDH(表2)。

表 2 NormFinder 软件计算出的内参基因稳定值
BestKeeper程序根据所有被研究的基因的Ct值计算基因表达变化量。从表3中可以看出,EF1-α基因有最高的相关系数(r=0.84),它自身的变异系数(CV)最低;标准偏差(SD)较低。所以BestKeeper分析结果与GeNorm软件基本一致,基因稳定性排序为:EF1-α>18SrRNA>β-actin>GAPDH。所以,综合这3种软件分析得出:最适内参基因为EF1-α,其次为18SrRNA。
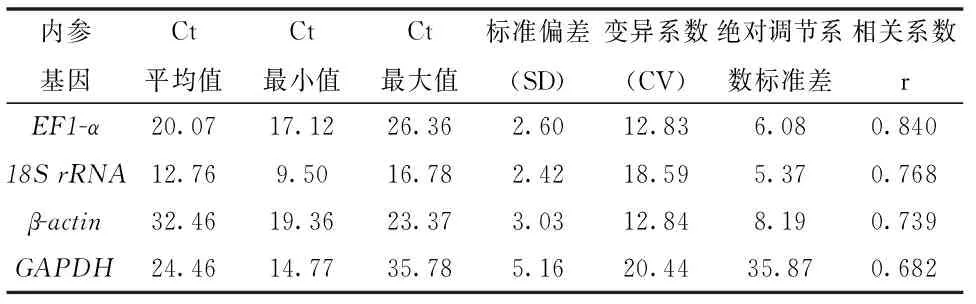
表3 BestKeeper分析4个内参基因在蓝点马鲛鱼不同组织中的表达稳定性
3 讨论
本试验首先从蓝点马鲛鱼中克隆了β-actin、GAPDH和EF1-α这3个常用内参基因,其核苷酸序列分别为2 114 bp、272 bp和383 bp。blast比对发现与鲈形目的条石鲷、尖吻鲈和南方黑鲔的β-Actin、GAPDH和EF1-α核苷酸序列的相似性分别为98%、99%和96%,物种间高度保守。这些基因片段补充了NCBI上蓝点马鲛鱼基因数据,为今后研究蓝点马鲛鱼基因表达奠定了基础。
荧光定量PCR技术中,Ct值是指每个反应管内的荧光信号到达设定的域值时所经历的循环数。研究表明,起始拷贝数越多,Ct值越小。利用已知起始拷贝数的标准品可作出标准曲线,只要获得未知样品的Ct值,即可从标准曲线上计算出该样品的起始拷贝数。比较蓝点马鲛鱼各组织中4个内参基因的Ct值发现,18SrRNA的Ct值最低,表明蓝点马鲛鱼中18SrRNA表达量最高,其次是EF1-α。
在qPCR分析过程中,评价作为校准和标准化的内参基因有多种分析方法。目前,已开发的用于内参基因筛选的软件包括:GeNorm软件、NormFinder软件和BestKeeper软件。分别使用这三个软件对数据进行分析,选出稳定度最高的内参基因,最后综合这三个软件得到最稳定的内参基因[14]。geNorm软件将某一看家基因与其他看家基因表达水平的两两比值经对数变换后,计算其平均标准差作为基因表达稳定度的平均值M,M值越小,说明候选内参基因的表达稳定性越好,反之稳定性越差[15]。geNorm用于qPCR内参基因稳定性以及最适数目的判定,已经在动物、微生物和植物等物种的基因表达研究中得到广泛应用[16]。Eileen等通过geNorm软件分析蝾螈各组织中11 个内参基因的稳定性,发现ACTB是最稳定的内参基因[17]。BestKeeper数据的初步分析是根据原始Ct值计算所有样本中的所有内参基因的标准偏差SD(±Ct)和变异系数CV (%CT),SD值和CV值越小,稳定性越好。NormFinder 程序则是通过对荧光定量数据分析得出基因的表达稳定值,根据稳定值的大小排序,具有最小稳定值的基因是最为稳定的基因。Deloffre综合GeNorm、NormFinder和BestKeeper这3 种软件分析硬骨鱼类卵子发生过程中定量内参基因表达稳定性时,发现尽管这3种软件对6个内参基因(β-actin、CTSD、CTSZ、EF1A、TBP和TUBA1A)的稳定性排序不同,但是都选择相同最佳内参基因CTSD和ACTB[18]。因此比较不同算法,对内参基因选择可以提供更精确地评估。
本实验通过这3种软件分析候选内参基因在蓝点马鲛鱼不同组织中的表达稳定性,发现EF-1α在各组织中表达最稳定,18SrRNA次之。GAPDH是最不稳定的,证实了这个基因作为内参基因的普遍怀疑论[19]。EF-1α和18SrRNA都可以作为蓝点马鲛鱼qPCR的内参基因。但是,一般我们不选择18SrRNA,主要是因为rRNA占总RNA量的80%左右,呈高丰度表达,可能会比目的基因的表达丰度高很多,容易造成试验过程中和数据分析时易放大误差。且有研究表明18SrRNA的转录易受到各种生物因素和药物的影响,Spanakis等发现28SrRNA、18SrRNA在有丝分裂期间表达量会发生变化[20]。因此建议在蓝点马鲛鱼qPCR实验时选取EF1-α基因作为内参基因,而不是18SrRNA。本研究为今后蓝点马鲛鱼基因定量表达研究中合适内参基因的选择提供了依据。
参考文献:
[1]VALASEK M A, REPA J J. The power of real-time PCR[J]. Adv Physiol Educ, 2005, 29(3): 151-159.
[2]PARK Y, KIM J, CHOI J R, et al. Evaluation of multiplex PCR assay using dual priming oligonucleotide system for detection mutation in the Duchenne muscular dystrophy gene[J]. Korean J Lab Med, 2008, 28(5): 386-391.
[3]MAHONEY D J, CAREY K, FU M H, et al. Real-time RT-PCR analysis of housekeeping genes in human skeletal muscle following acute exercise[J]. Physiol Genomics, 2004, 18(2): 226-231.
[4]BUSTIN S A, BENES V, GARSON J A, et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments[J]. Clin Chem, 2009, 55(4): 611-622.
[5]LØVDAL T, LILLO C. Reference gene selection for quantitative real-time PCR normalization in tomato subjected to nitrogen, cold and light stress[J]. Anal Biochem, 2009, 387(2): 238-242.
[6]GUÉNIN S, MAURIAT M, PELLOUS J, et al. Normalization of qRT-PCR data: The necessity of adopting a systematic, experimental conditions specific, validation of references[J]. J Exp Bot, 2009, 60(2): 487-493.
[7]张 岗, 赵明明, 张大为, 等. 铁皮石斛实时定量PCR内参基因的筛选[J]. 中国药学杂志, 2013, 48(19): 1664-1668.
[8]OLSVIK P A, KAI K L, JORDAL A E O, et al. Evaluation of potential reference genes in real-time RT-PCR studies of Atlantic salmon[J]. BMC Mol Biol, 2005, 6(22): 1-9.
[9]LIMAN M, WENJI W, CONGHUI L, et al. Selection of reference genes for reverse transcription quantitative real-time PCR normalization in black rockfish (Sebastesschlegeli)[J]. Mar Genom, 2013, 11: 67-73.
[10]ZHANG J, HANNER R. Molecular approach to the identification of fish in the south China sea[J]. PLoS ONE, 2012, 7(2): e30621.
[11]VANDESOMPELE J, DE PRETER K, PATTYN F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biol, 2002, 3(7): 1-34.
[12]ANDERSEN C L, JENSEN J L, RNTOFT T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Res, 2004, 64(15): 5245-5250.
[13]PFAFFL M W, TICHOPAD A, PRGOMET C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: Bestkeeper-excel-based tool using pair-wise correlations[J]. Biotechnol Lett, 2004, 26(6): 509-515.
[14]ZHANG Y, ZHANG X D, LIU X, et al. Reference gene screening for analyzing gene expression across Goat tissue[J]. Asian Austral J Anim Sci, 2013, 26(12): 1665-1671.
[15]张艳君, 朱志峰, 陆融, 等. 基因表达转录分析中内参基因的选择[J]. 生物化学与生物物理进展, 2007, 34(5): 546-550.
[16]黄雪玲, 冯 浩, 康振生. 小麦条锈菌实时荧光定量 PCR 分析中内参基因的选择[J]. 农业生物技术学报, 2012, 20(2): 181-187.
[17]GUELKE E, BUCAN V, LIEBSCH C. Identification of reference genes and validation for gene expression studies in diverse axolotl (Ambystomamexicanum) tissues[J]. Gene, 2015, 560(1): 114 123.
[18]DELOFFRE L A, ANDRADE A, FILIPE A I. Reference genes to quantify gene expression during oogenesis in a teleost fish[J]. Gene, 2012, 506(1): 69 75.
[19]HUGGETT J, DHEDA K, BUSTIN S, et al. Real-time RT-PCR normalisation; strategies and considerations[J]. Genes Immun, 2005, 6(4): 279 284.
[20]SPANAKIS E. Problems related to the interpretation of autoradiographic data on gene expression using common constitutive transcripts as controls[J]. Nucleic Acids Res, 1993, 21(16): 3808-3819.
Cloning and expression stability evaluation of reference genes in Spanish Mackerel (Scomberomorus niphonius)
ZHANG Jia-ling, XUE Liang-yi, SHI Jun-xing
(School of Marine Science, Ningbo University, Ningbo 315211, China)
AbstractSelection of a suitable reference gene is an important prerequisite for precise analysis of target gene expression by Real-time quantitative PCR(qPCR). In this paper, the partial sequence of three reference genes, β-actin and GAPDH, EF1-α, from spanish mackerel Scomberomorus niphonius were firstly cloned, and then the Ct values of the four reference genes in various tissues were measured by qPCR. Three different statistical algorithms including geNorm, NormFinder and BestKeeper were used to analyze the expression stability of the four reference genes in different tissues. The result showed that the most stable reference gene was EF1-a, followed by 18S ribosomal RNA. It provides a useful basis for selecting of the appropriate reference gene in Spanish Mackerel.
Key wordsScomberomorus niphonius; qPCR; reference gene; geNorm; expression stability
收稿日期:2015-10-08;修回日期:2015-10-27
基金项目:宁波市科技局项目(编号:2012C10035)
作者简介:张家玲,硕士研究生,主要从事海洋生物基因资源,E-mail:xin.jialing@163.com; 通信作者:薛良义,教授,博士生导师,主要从事鱼类分子生物学研究,E-mail:xueliangyi@nbu.edu.cn。
中图分类号Q344+.13
文献标识码A
文章编号2095-1736(2016)03-0020-04
doi∶10.3969/j.issn.2095-1736.2016.03.020
