流感病毒M1蛋白与M2蛋白相互作用以及其在病毒出芽中的作用
2016-03-03侯天龙包旦奇李泽君刘芹防刘焕奇
侯天龙,包旦奇,李泽君,刘芹防,刘焕奇
(1.青岛农业大学动物科技学院,青岛266109;2.中国农业科学院上海兽医研究所,上海200241;3.内蒙古农业大学,呼和浩特 010018)
·研究论文·
流感病毒M1蛋白与M2蛋白相互作用以及其在病毒出芽中的作用
侯天龙1,2,包旦奇3,李泽君2,刘芹防2,刘焕奇1
(1.青岛农业大学动物科技学院,青岛266109;2.中国农业科学院上海兽医研究所,上海200241;3.内蒙古农业大学,呼和浩特 010018)
流感病毒为囊膜病毒,以出芽的方式释放病毒。M1蛋白是流感病毒的基质蛋白,在维持病毒颗粒形态与病毒的致病性中发挥重要作用。M2蛋白是离子通道蛋白,在病毒出芽过程中发挥重要作用。研究显示M1蛋白与M2蛋白存在相互作用,但是这种互作在病毒出芽中的功能尚不清楚。根据M1蛋白的结构,M1蛋白分成N端、C端和中间区域3个区域,与M2蛋白相互作用的区域也不清楚。本文利用双分子荧光互补实验与病毒出芽实验方法研究了M1蛋白与M2蛋白互作的区域及这种互作在病毒出芽中的作用。研究结果显示,M2蛋白通过与M1蛋白的N端1~160个氨基酸相互作用,从而帮助M1蛋白出芽;M1蛋白的1~20 氨基酸与140~160氨基酸在决定M1蛋白的稳定性与功能发挥中具有重要作用,缺失这2个氨基酸序列可导致M1蛋白N端1~160个氨基酸蛋白不稳定,并不能与M2蛋白互作。本研究揭示了3H1N1流感病毒M1蛋白与M2蛋白互作功能区域、以及他们之间相互作用在流感病毒出芽中发挥的重要作用。
流感病毒;M1蛋白;M2蛋白;出芽
流感病毒为正黏病毒科、流感病毒属的有囊膜病毒,基因组分为分节段的、单股负链的RNA(SsRNA)[1]。流感病毒中存在核蛋白(NP)和基质蛋白(M),根据其抗原性的不同,可将流感病毒划分为A、B、C型。通过对流感病毒表面的血凝素(HA)和神经氨酸酶(NA)进行分类,发现其拥有多个亚型。目前已经鉴定的HA亚型有18种,NA亚型11种,常见亚型有H1N1、H5N1、H7N9、H9N2等。H17N10与H18N11病毒是从蝙蝠中新发现的A型流感病毒[2]。
流感病毒基因组包含8个分节段的负链RNA,至少编码12~13种蛋白,包括HA、NA、NP、MP、PB2、PB1、PA、NP、NS等蛋白。流感病毒是具有囊膜包被的RNA病毒,形态多为球形结构,大小在80~120 nm,也存在数千纳米的丝状。病毒囊膜出自于宿主细胞脂膜,包括糖蛋白、类脂和内膜蛋白共3层。糖蛋白形成HA分子,其形态为棒状三聚体构成;NA分子由四聚体构成,其形态为蘑菇状[3]。M1蛋白是流感病毒颗粒中含量最多的蛋白之一,由252个氨基酸组成,包含3个区域:N端、C端与中间区域[4]。M1蛋白在病毒囊膜下形成一个病毒基质层,在维持病毒颗粒的形态与结构方面起着重要的作用[5]。M1蛋白也具有多重功能,与病毒基因组(RNPs)相互作用从而影响其出核过程,在病毒的复制过程中起到十分重要的作用[6],但是M1蛋白在流感病毒出芽过程中的具体作用尚存在争议[7]。
研究表明M1蛋白必须与M2蛋白相互作用才能完成M1蛋白的出芽[8],但是M1蛋白与M2蛋白相互作用的功能区域尚不清楚[9]。M2蛋白是流感病毒颗粒中的一个离子通道蛋白[10],在病毒感染早期,M2蛋白通过调节病毒颗粒中的pH值,从而使病毒释放其基因组RNPs到感染的细胞中。在病毒感染后期,M2蛋白在流感病毒出芽后期,剪切细胞膜进一步释放病毒中也起到重要作用[11]。M2蛋白由97个氨基酸组成,在膜上形成同源四聚体[12]。最近研究证明M2蛋白的胞质尾区,特别是分子螺旋46~62位残基,可能在病毒的装配和出芽中扮演着重要的作用[8]。目前,M1蛋白与M2蛋白相互作用的功能区域以及这种互作在病毒出芽中的作用尚不清楚,本研究利用双分子荧光互补实验(bimolecular fluorescence complementation,BIFC)与病毒出芽实验(viruslike particle,VLP)对此问题进行探索研究。
1 材料方法
1.1 细胞与毒株 293T细胞、A/California/04/2009(H1N1)由上海兽医研究所动物流感病原生态学创新团队保存。
1.2 载体与主要试剂 VN、VC、3Flag与2HA真核表达载体为本实验室保存;PCR试剂购自大连宝生物有限公司(TaKaRa);限制性内切酶Xho I与Not I,T4 DNA连接酶购自NEB公司;大肠杆菌DH5α感受态细胞购自北京全式金生物技术有限公司;胎牛血清、Opti-MEM购自Gibco公司;Phanta® Super-Fidelity DNA Polymerase购自诺唯赞公司;QIA quick Gel Extraction Kit凝胶回收试剂盒购自北京华绿源科技有限公司;大剂量质粒抽提试剂盒购自QIAGEN公司;转染试剂Mirus购自Bio公司。
1.3 双分子荧光互补实验
1.3.1 质粒构建 提取H1N1流感病毒基因组RNA,提取的RNA反转录后获得cDNA待下一步研究使用。利用特异性引物扩增M1、M1-1-160、M1-1-140、M1-20-160、M1-60-252、M2基因片段,然后将这些片段克隆进入BIFC系统中工作VC质粒的Xho I与Not I酶切位点之间,命名为VC-M1、VC-M1-1-160、VC-M1-1-140、VC-M1-20-160、VC-M1-60-252;扩增的M2片段需要单独克隆于BIFC系统中工作VN质粒的Xho I与Not I酶切位点之间,将其命名为VN-M2。
1.3.2 细胞培养与质粒转染 用腔室载玻片培养293T细胞,培养时间为12~20 h。将用于BIFC系统中构建成功的质粒VC-M1、VC-M1-1-160、VC-M1-1-140、VC-M1-20-160、VC-M1-60-252分别(100 ng/质粒)与VN-M2质粒(100 ng)利用转染试剂Mirus共转染293T细胞,转染后,细胞继续培养20 h。
1.3.3 细胞固定、染色与观察 转染后20 h,弃掉上清,PBS清洗细胞。弃掉PBS洗液,再加入4%多聚甲醛固定为10 min。固定结束后弃掉固定液,用DAPI染料对细胞进行核染色5 min。弃掉染液用PBS清洗细胞后在载玻片上滴加甘油,盖玻片固定封片。固定好的封片用倒置荧光显微镜进行观察。
1.4 出芽实验(virus-like particle,VLP)
1.4.1 质粒构建 在VLP出芽实验中,利用特异性引物进行扩增M1、M1-1-160、M1-1-140、M1-20-160、M1-60-252、M2基因片段,将扩增完成的这些片段克隆进入3Flag载体的Xho I与Not I酶切位点之间,命名为3Flag -M1、3Flag -M1-1-160、3Flag -M1-1-140、3Flag -M1-20-160、3Flag -M1-60-252。扩增的M2片段克隆于2HA真核表达质粒的Xho I与Not I酶切位点之间,将其命名为2 HA -M 2。1.4.2 细胞培养与质粒转染 用100 mm细胞培养皿培养293T细胞20 h。转染质粒3Flag-M1、3Flag-M1-1-160、3Flag-M1-60-252(10 μg/质粒)分别与2HA-M2质粒(10 μg)共转染293T细胞。
1.4.3 VLP纯化 转染后48 h分别收集细胞上清液与细胞,细胞上清液4℃、500×g离心30 min。离心后吸取上清移至新的离心管,4℃、超速离心5 h。弃掉上清,仅留底部200 μL,将沉淀溶解后冻存于-80℃冰箱备用。293T细胞用800 μL RIPA的裂解液进行裂解,裂解条件为4℃、10 min。裂解完成后收集于1.5 mL离心管中,冻存于-80℃冰箱中。
1.4.4 Western blot 取VLP纯化物与细胞裂解液各20 μL,分别加入2×SDS上样缓冲液20 μL,100℃煮沸10 min,使蛋白样品充分变性,进行12%SDSPAGE凝胶电泳。电泳结束后,取出蛋白凝胶,将蛋白转移至硝酸纤维素膜(恒流250 mA、2 h)。
1.4.5 封闭、抗体孵育及显色 蛋白转移至硝酸纤维素膜后,用PBST溶液配制5%脱脂奶,摇床室温封闭2 h。孵育结束弃掉脱脂奶,PBST清洗后,用抗Flag或HA标签蛋白的鼠源抗体(1∶1000稀释)室温孵育2 h,再用PBST漂洗膜3次(10 min/次),洗去未结合抗体。加入HRP标记兔抗鼠IgG的二抗(1∶2000稀释)室温孵育2 h,PBST漂洗膜3次(10 min/次),洗去未结合的二抗。漂洗结束进行显色。
2 结果
2.1 M1蛋白通过N端1~160氨基酸与M2蛋白相互作用通过双分子荧光互补实验(BIFC)研究M1蛋白和M2蛋白之间的相互作用,我们将VC-M1和VN-M2两种质粒共同转染293T细胞。在转染20 h后观察293T细胞,发现只有胞浆和细胞膜上有绿色荧光,这说明了在M1蛋白和M2蛋白之间存在较强的相互作用(图1)。
为了进一步研究M1蛋白与M2蛋白相互作用的区域,我们将M1蛋白分为2个区域,即1~160氨基酸的结构功能区和60~252氨基酸的C端区域,并将 2个区域克隆到BIFC系统中,将VC-M1-1-160、VCM1-60-252分别与VN-M2质粒共转染293T细胞。转染后20 h,倒置荧光显微镜下观察结果。我们发现VC-M1-1-160组在观察中可清晰见到绿色荧光(图2),但在VC-M1-60-252组中几乎无荧光表达(图2)。结果说明,M1蛋白N端的1~160的氨基酸残基能与M2蛋白相互作用,而C端的60~252氨基酸残基不与M2蛋白作用。

图1 利用BIFC检测M1蛋白与M2蛋白相互作用Fig.1 Interaction between M1 and M2 protein in 293T cell by using BIFC system
为了验证是否存在更小的作用区域,即对M1蛋白与M2蛋白相互作用区进行精细定位,将M1蛋白的1~160区域N端与C端各截短20个氨基酸,分别构建质粒VC-M1-1-140和VC-M1-20-160。将VCM1-1-140、VC-M1-20-160和VN-M2分别共转染293T细胞后分析BIFC结果,结果显示这两组中都没有产生绿色荧光,说明M1蛋白N端的1~160氨基酸残基是与M2蛋白相互作用的最小功能区域(图2)。
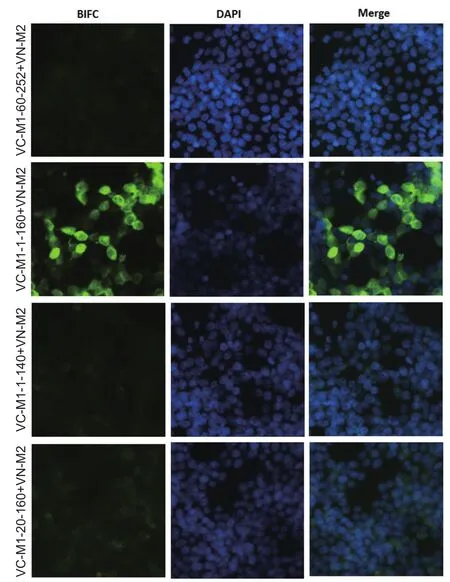
图2 利用BIFC检测M1蛋白不同区域与M2蛋白相互作用Fig.2 Interaction between truncated M1 and M2 protein in 293T cell by using BIFC system
2.2 M1蛋白与M2蛋白相互作用在出芽中的作用 前期结果已证明M1蛋白通过N端的160个氨基酸残基与M2蛋白相互作用,为了研究这个相互作用在蛋白出芽中的功能,用3Flag-M1质粒单独转染293T细胞,或者3Flag-M1和2HA-M2质粒共转染到293T细胞中,48 h后收集细胞上清进行VLP纯化。Western blot检测结果显示,单独转染3Flag-M1的细胞上清的VLP中检测不到M1蛋白,说明M1蛋白显然不具备单独出芽能力(图3)。而对3Flag-M1和2HA-M2质粒共转染的细胞上清进行检测后发现两种蛋白都可以在上清中被检测到,说明M2蛋白可以协助M1蛋白完成出芽,释放到细胞上清中。

图3 M1蛋白与M2蛋白共转染293T细胞蛋白出芽情况Fig.3 Budding of M1 proteins with M2 protein in 293T cells
2.3 M1的N端1~160氨基酸与M2相互作用在出芽中的作用 BIFC实验已经验证M1-1-160的区域是与M2蛋白相互作用的,那么在病毒出芽过程中,M2蛋白是否通过与M1蛋白的1~160区域互作而帮助其出芽的尚不清楚。将3Flag-M1-1-140、3Flag-M1-20-160、3Flag-M1-1-160、3Flag-M1-60-252质粒分别与2HA-M2质粒共转染293T细胞,48 h后检测其细胞上清VLP和细胞裂解液中蛋白水平。结果表明M2蛋白无论在上清还是裂解液中表达水平都很高,但是截断M1蛋白中只有M1-1-160在细胞上清中被检测到。结果证明,M1蛋白N端1~160氨基酸是与M2蛋白相互作用的功能区域,并且在病毒出芽过程中,M2蛋白通过与M1蛋白N端1~160氨基酸区域互作从而帮助M1蛋白完成出芽。
3 讨论
流感病毒作为一种人畜共患病病原,在国内外都受到很高的重视,但是目前在H1N1流感病毒出芽机制方面的研究不多。M1蛋白作为流感病毒颗粒中含量最多的蛋白之一,它能够与病毒基因组相互作用从而影响其出核过程,在病毒复制中扮演十分重要的角色,但是M1蛋白在H1N1流感病毒出芽过程中是否也具有重要作用尚不清楚。研究人员对M1蛋白的出芽能力存在争议,有研究表明单独M1蛋白可以独立完成出芽,另外一些研究表明单独的M1蛋白不具备出芽能力[7,9]。
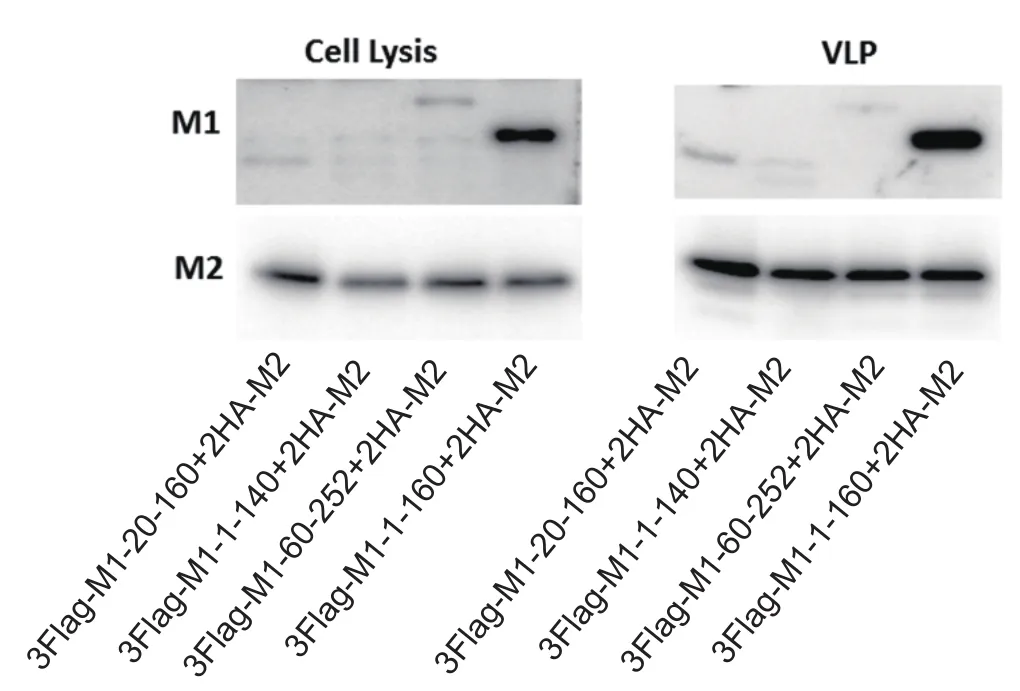
图4 截断M1蛋白与M2蛋白共转染293T细胞蛋白出芽情况Fig.4 Budding of truncated M1 proteins with M2 protein in 293T cells
本研究利用体外出芽实验与BIFC方法验证了单独的M1蛋白在293T细胞中不能完成出芽,它可以通过与M2蛋白的互作,在M2蛋白的协助下完成出芽。本研究首次确定了M1蛋白的N端160个氨基酸是与M2发生相互作用的功能区域,M2蛋白通过与此功能区域互作从而协助M1蛋白出芽,而且M1蛋白的N端160氨基酸残基是互作的最小功能区域,缺失N或者C端的20个氨基酸都会导致M1蛋白的N端结构不稳定,Western blot检测不到蛋白。M1蛋白的N端1~160氨基酸残基蛋白的晶体结构图也显示1~20与140~160两端氨基酸残基正好构成两个B螺旋,在维持蛋白结构上发挥重要作用[4]。另外,M1蛋白的1~20氨基酸与140~160氨基酸这两个区域也有可能是与M2蛋白直接互作的功能区域,此推测尚需要进一步研究证实。本研究首次揭示了M1蛋白与M2蛋白相互作用的功能区域,以及其在病毒出芽中的作用,为流感的相关研究与预防提供理论基础,也为新型抗流感病毒药物设计提供新的靶点与思路。
[1] Perdue M L, GarcıˊA M, Senne D, et al.Virulence associated sequence duplication at the hemagglutinin cleavage site of avian influenza viruses[J]. Virus Res, 1997, 49(2)∶ 173-186.
[2] Sux T, Xue Y Z, Yan L, et al. New world bats harbor diverse influenza A viruses[J]. PLos Pathogens, 2013, 9(10)∶ 1078-1084.
[3] Lamb R A. Genes and proteins of the influenza viruses[M]//King R M. The Influenza Viruses, Springer US, 1989∶ 1-87.
[4] Harris A F, Forouhar S, Qiu B S, et al. The crystal structure of the influenza matrix protein M1 at neutral pH∶M1-M1 protein interfaces can rotate in the oligomeric structures of M1[J]. Virology, 2001, 289∶ 34-44.
[5] Burleigh L M, Calder L J, Skehel J J, et al. Influenza a viruses with mutations in the m1 helix six domain display a wide variety of morphological phenotypes[J] J Virol, 2005,79∶1262-1270.
[6] Bui M, Wills E G,Helenius A, et al. Role of the influenza virus M1 protein in nuclear export of viral ribonucleoproteins[J]. J Virol, 2000,74(4)∶ 1781-1786.
[7] Wang D A, Harmon J, Jin D H, et al. The lack of an inherent membrane targeting signal is responsible for the failure of the matrix (M1) protein of influenza A virus to bud into virus-like particles[J]. J Virol, 2010, 84∶ 4673-4681.
[8] Chen B J, Leser G P, Jackson D, et al. The influenza virus M2 protein cytoplasmic tail interacts with the M1 protein and influences virus assembly at the site of virus budding[J]. J Virol, 2008, 82∶ 10059-10070.
[9] Rossman J S, Lamb R A. Influenza virus assembly and budding[J]. Virology, 2011, 411∶ 229-236.
[10] Pinto L H, Holsinger L J, Lamb R A. Influenza virus M2 protein has ion channel activity[J]. Cell, 1992, 69∶ 517-528.
[11] Schmidt N W, Abhijit M, Jun W, et al. Influenza virus A M2 protein generates negative Gaussian membrane curvature necessary for budding and scission[J]. J Ame Chem Soc, 2013, 135 (37)∶ 13710-13719.
[12] Sakaguchi T, Tu Q, Pinto L H, et al. The active oligomeric state of the minimalistic influenza virus M2 ion channel is a tetramer [J]. Proc Nat Acade Sci USA, 1997, 94∶ 5000-5005.
INTERACTION BETWEEN M1 AND M2 PROTEINS OF INFLUENZA VIRUS AND ITS ROLE IN VIRUS BUDDING
HOU Tian-long1,2, BAO Dan-qi3, LI Ze-jun2, LIU Qin-fang2, LIU Huan-qi1
(1. College of Animal Science and Technology, Qingdao Agricultural University, Qingdao 266109, China; 2. Shanghai Veterinary Research Institute, CAAS, Shanghai 200241,China; 3. Inner Mongolia Agricultural University, Hohhot 010018, China )
Inf uenza virus is an enveloped virus and released from infected cells by budding. M1 protein, a matrix protein of inf uenza virus, plays an important role in the pathogenicity of the virus and maintenance of the viral morphology. M2 protein, an ion channel protein, plays an important role in virus budding process. Previous studies have shown that M1 protein interacts with M2 protein but the function of this interaction in virus budding remains unclear and the interacting regions of M1 and M2 is unknown. M1 is divided into three regions, N terminal, C end and middle region according to its structure. In the present study, BiFC( biomolecular f uorescence complementation) and VLP(virus-like particle) experiments were conducted to investigate the interaction regions of M1 and M2 and functions of such interactions in virus budding. The results showed that M2 protein interacted with M1 protein through N terminal 1-160amino acids of M1 protein and M2 protein facilitated budding of M1 protein by interacting with this region of M1 protein. The regions consisting of 1-20 and 140-160 amino acids of M1 protein determined the stability and function of this protein. This study revealed the interacting regions between M1 protein and M2 protein and the function of this interaction in virus budding of inf uenza virus.
Inf uenza virus; M1 protein; M2 protein; budding
S852.659.5
A
1674-6422(2016)06-0001-05
2016-04-06
国家自然科学基金面上项目(31572543);中央级公益性科研院所基本科研业务费专项资金项目(2016JB13);中国农业科学院青年英才项目
侯天龙,男,硕士研究生,预防兽医学专业;包旦奇,男,硕士研究生,预防兽医学专业
刘焕奇,E-mail:Huanqiliu@126.com;刘芹防,E-mail:liuqinfnag@shvri.ac.cn
