人分泌型磷脂酶A2-IIA的功能性动力学特征研究*
2022-07-25巩卫康李春华
张 珊 巩卫康 张 娜 李春华
(北京工业大学环境与生命学部,北京 100124)
人 分 泌 型 磷 脂 酶 A2-ⅠⅠA (secretory phospholipase A2group ⅠⅠA,sPLA2-ⅠⅠA)是一种脂解酶,在细胞脂代谢和信号传导中起重要作用[1]。sPLA2-ⅠⅠA 通过催化甘油磷脂sn-2 位酯键水解,产生众多信号介质前体,参与许多炎症性疾病的发生,如哮喘、关节炎和癌症等[2]。研究sPLA2-ⅠⅠA的结构动力学和功能之间的关系可更好地理解其相关疾病的分子机制。
到目前为止,有16 种亚型的sPLA2从蛇、人、猪以及植物等生物中纯化出来。它们的结构具有6对以上二硫键和保守的催化二联体His/Asp 及钙结合 环(Xxx-Cys-Gly-Xxx-Gly-Gly)。 大 部 分 的sPLA2具有相似的折叠结构:3个α螺旋(α-helix)、2 个β 折叠(β-sheet)和1 个保守的Ca2+结合环(loop2)(图1)。在哺乳动物中研究最多的sPLA2是人ⅠⅠA型的sPLA2(sPLA2-ⅠⅠA),该型酶含有7对二硫键,结构稳定,还具有防御细菌感染和杀菌的能力[3]。sPLA2-ⅠⅠA 催化循环包括4 个步骤:a. 酶通过底物结合区与膜结合;b.从膜上提取单个磷脂分子到酶的结合口袋;c.水解磷脂的sn-2 位酯键;d. 释放水解产物[4]。酶与底物结合是催化循环中重要的环节,其中膜作为变构剂,它的结合使酶从关闭构象转变为打开构象,促进酶结合磷脂分子而发生催化反应[4]。因此,sPLA2-ⅠⅠA 的结构动力学以及别构过程一直是人们研究的热点。
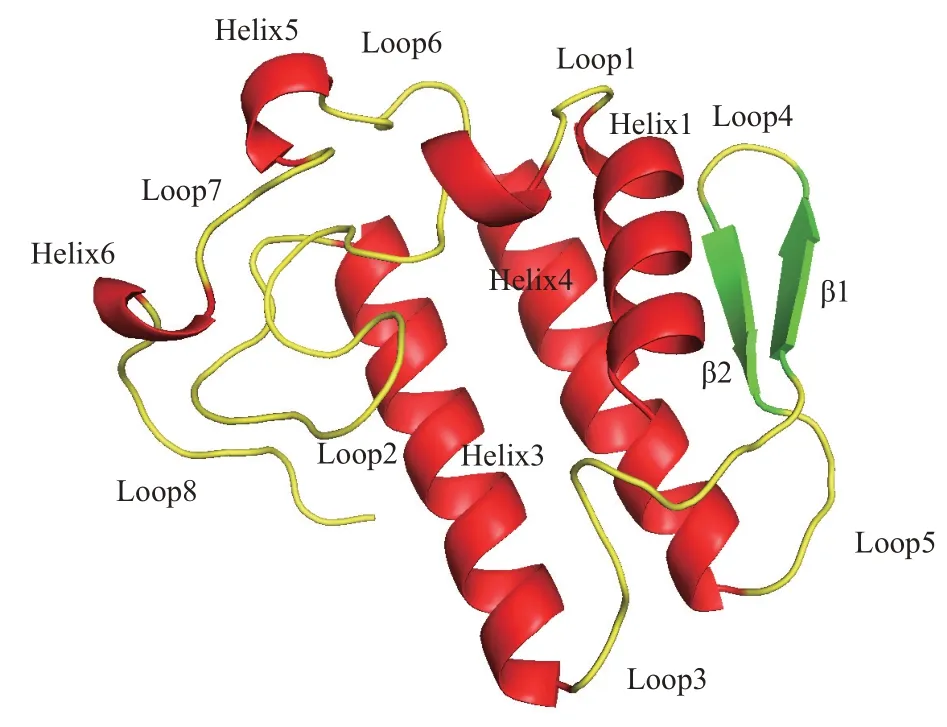
Fig.1 Structure of sPLA2(PDB ID:3u8b)
2015 年Mouchlis 等[4]应 用 分 子 动 力 学(molecular dynamics,MD)模拟方法研究了不同类型sPLA2酶结合口袋的功能差异,并对其催化循环提出了新的见解。2020年Manukyan[5]用MD模拟方法研究了人类sPLA2在有无膜环境下的构象变化。众所周知,MD模拟可以在原子水平探测生物分子的动力学性质,但是它非常耗时,很难对蛋白质大尺度的功能性运动进行采样分析。针对该问题,研究人员提出了一些粗粒化的模型,其中弹性网络模型(elastic network model,ENM)是研究蛋白质功能性运动最为有效的方法之一。两种常用的ENM是高斯网络模型(Gaussian network model,GNM)[6]和 各 向 异 性 网 络 模 型(anisotropic network model,ANM)[7]。结 合ENM,Atilgan等[8]提出了微扰响应扫描(perturbation-response scanning,PRS)方法来评价蛋白质残基对外界微扰的响应程度,该方法已被广泛应用于识别蛋白质变构调控中潜在的别构位点[9]。另外,蛋白质结构网络(protein structure network,PSN)方法也被广泛应用于蛋白质折叠和关键位点预测的研究。
本研究使用基于结构的模型和方法,包括ENM、PRS和PSN分析了人sPLA2-ⅠⅠA型结构的共享和特异性和动力学与其功能的关系,并识别了关键残基,加深了对酶催化机制的理解。
1 研究方法
1.1 数据集的建立
从Protein Data Bank(PDB)数据库中下载来自人分泌型磷脂酶(sPLA2)ⅠⅠA 型的31 个X 射线(X-ray)衍射晶体结构作为研究对象,其PDB ⅠD分别为:1ayp-A,1ayp-B,1ayp-C,1ayp-D, 1ayp-E,1ayp-F,1db4-A,1db5-A,1dcy-A,1j1a-A,1j1a-B,1kqu-A,1kvo-A,1kvo-B,1kvo-C,1kvo-D,1kvo-E,1kvo-F,1n28-A,1n28-B,1n29-A,1pod-A,1poe-A,1poe-B,3u8b-A,3u8d-A,3u8d-B,3u8h-A,3u8h-B,5g3n-A,5g3n-B。
1.2 弹性网络模型
弹性网络模型(ENM)将蛋白质的三维结构简化为粗粒化的弹性网络,每个残基简化为网络中的一个节点,用Cα原子表示;如果两个Cα原子间的距离小于某一截断半径,则认为它们之间存在相互作用,用倔强系数相同的弹簧相连。本工作选取7.0 Å为截断半径。这样,该弹性网络的总势能为:

其中,γ是弹簧的弹性系数,ΔR(3N维的列向量)代表N个Cα原子偏离平衡位置的位移,E是单位矩阵,⊗为矩阵的直积,Γ为N×N阶的Kirchhoff 矩阵,其元素为:
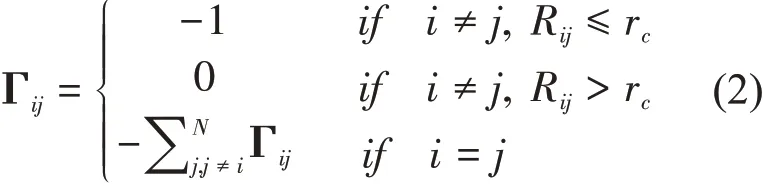
其中,Rij是第i个和第j个Cα原子之间的距离,rc为截断半径。每个残基的均方涨落以及不同残基间涨落的交叉相关性分别表示为:
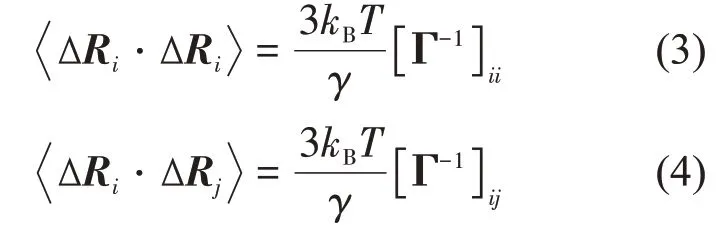
其中kB为Boltzmann常数,T为绝对温度。
GNM 模型可以提供残基运动的幅度,但不包括方向信息,而ANM 模型可以提供该信息。在ANM 中,选取14.0 Å 为截断半径,网络的总势能为:

其中,Rij和Rij0分别为节点i和j之间的瞬时距离以及处于平衡位置时的距离。蛋白质的运动模式由一个Hessian 矩阵H决定,它的元素是大小为3×3 的子矩阵。子矩阵的元素hij为体系势能函数(5)对位置的二阶偏导数,当i≠j时,hij为:
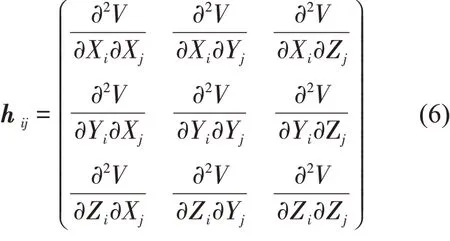
当i=j时,hij为:

在ANM中,残基的均方涨落以及残基间涨落的交叉相关性为:

归一化的交叉相关性为:

交叉相关性的取值范围为-1到1,正值表示残基的运动方向相同,负值表示它们的运动方向相反。该值的绝对值越大表示两个残基间的运动相关性越强。
1.3 微扰响应扫描模型
微扰响应扫描(PRS)[8]是基于线性响应理论(linear response theory,LRT)[10]发 展 的 模 型。PRS可以计算扰动某个残基时所有残基的响应,从而来推断蛋白质的变构特性。由胡克定律F=HΔR,可以计算在受到外力扰动时所有残基位置 的 改 变 量ΔR,其 中H是ANM 中3N×3N的Hessian 矩阵(截断半径仍为14.0 Å)。在PRS 中,每次对网络中的一个节点施加外力,观测其他所有残基的响应。以对残基i施加外力为例:
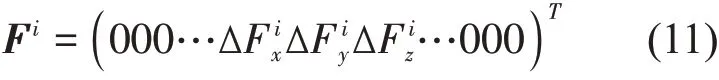
得到的响应为:

其中,ΔRi是在外力对残基i扰动后所有残基位置的改变量。对于力的方向,有研究表明外力的方向对于结构的扰动不是非常重要[8],因此在本文中,施加在i残基3个方向上的力为(1,1,1)。
依次扰动所有残基可以得到一个N×N阶的响应矩阵P,其第j列代表扰动残基j其他残基的响应。将P矩阵进行归一化后,每一列的平均值代表了该列对应残基的敏感性,如第j列的平均值即为残基j的敏感性,所有残基的敏感性就组成了残基敏感性特征曲线。该曲线峰值对应的残基被认为是敏感残基,在蛋白质变构中起关键作用[11]。
1.4 蛋白质结构网络方法
蛋白质结构的形成和功能的发挥在一定程度上依赖于残基间复杂的相互作用网络。在传统的蛋白质结构网络(PSN)方法中,蛋白质被简化成一个无权的图,图中的节点是残基,边是残基间的相互作用。这里,当残基间的距离小于截断半径8.0 Å时,认为它们有相互作用。这样,蛋白质的PSN可以用邻接矩阵来描述,其元素为:
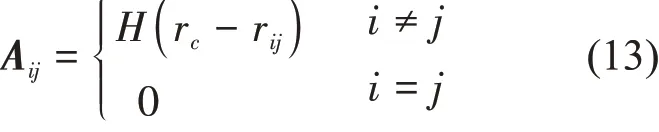
其 中rij表 示 节 点i和j之 间 的 距 离。H(x) 是Heaviside 函数,当x>0 时,H(x)=1,当x≤0 时,H(x)=0。节点i的度(degree)是与该节点连接的其他节点数量。
2 结果与讨论
2.1 高斯网络模型的慢运动模式分析
在ENM 中,蛋白质的低频运动模式提供了分子大幅度的集合运动信息,与功能运动有关。为了解sPLA2的功能性动力学特征,本文对人sPLA2-ⅠⅠA型31 个结构分别构建了GNM 模型,计算了前12个最慢运动模式(对涨落的贡献大于50%)的均方涨落,以及其平均值和标准差(图2a)。
图2a 中的残基涨落与前人MD 模拟[5]得到的涨落曲线相似,说明GNM是获得蛋白质柔性的有效方法。从图2a 发现处于涨落极小值的残基,包括Cys26、Cys28、Cys43、Cys44、His47、Asp48、Cys49、 Tyr51、 Tyr66、 Cys77、 Cys83、 Cys90、Asp91、Cys97 和Cys117,也具有较小的标准差,说明它们在不同的结构中都具有低运动性,是这些结构的动力学共同特征。其中,His47、Asp48、Tyr51和Asp91构成了催化网络(图2b),其通过与水分子发生相互作用稳定了催化反应所必需的氧离子中间体[12]。保守的催化二联体His47-Asp91占据涨落的极小值,这符合催化残基精确定位的要求[13]。另外,上述10个高度保守的半胱氨酸也处于极小值处,参与了二硫键的形成,起到了维持结构稳定的作用。残基Tyr66 也高保守,有低运动性,参与了与68 位Tyr/Phe/Trp 的芳香环堆叠相互作用,起到了稳定酶结构的作用[14]。
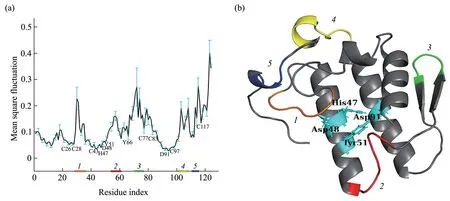
Fig.2 Residue fluctuations obtained by GNM for sPLA2 family member structures
sPLA2的C 端是loop 区,这可能是其具有较大涨落的原因。目前尚未发现它有重要的生物学功能,因此在分析涨落峰值的区域时没有考虑C 端。将涨落中峰值的区域标记为1~5(图2a),并将其展示在结构(PDB ⅠD:3u8b)中(图2b)。区域1(Gly29~Ser35)属于Ca2+结合环,主要负责Ca2+的结合。钙离子通过与区域1中残基的氧原子、催化残基Asp48以及水分子的相互作用,得以稳定在催化位点附近,为实现催化功能提供条件[5]。区域2(Gly58~Phe63)、区域4(Lys102~Tyr109)和区域5(Ser113~Lys115)主要参与了酶与膜的相互作 用, 特 别 是 残 基Thr61、 Phe63、 Lys102、Lys107、Asn114 和Lys115 通过疏水或静电相互作用与膜结合,其突变会影响酶的活性[15]。区域3(Ser71~Arg74)属于连接两个β 折叠的loop4,其功能还不是很明确。以前的MD模拟工作也显示此区域的柔性较大,并且在有无膜的环境下,其残基涨落的差异较为明显[5],因此推测该区域可能参与了膜的识别。另外,这些区域的涨落标准差较大,这是由于不同的结构结合的配体有所不同导致的,体现了结构对配体识别的特异性,这也说明了这些区域对不同配体结合的调控。
总之,从GNM 对慢运动模式分析的结果可发现,处于涨落极小值处的残基是对结构稳定或催化功能至关重要的残基,这些残基的低涨落是该酶成员共享的动力学特征,而位于涨落峰值处的残基可能与底物的结合有关,不同的结构间存在显著差异。
2.2 运动相关性分析
为了理解蛋白质结构中残基运动的关联性,通过式(10)用ANM 模型计算了所有残基涨落间的交叉相关系数的平均(图3)。由于慢运动模式对应着功能性运动,所以本文选取前21 个慢运动模式进行计算(对涨落的贡献大于50%)。
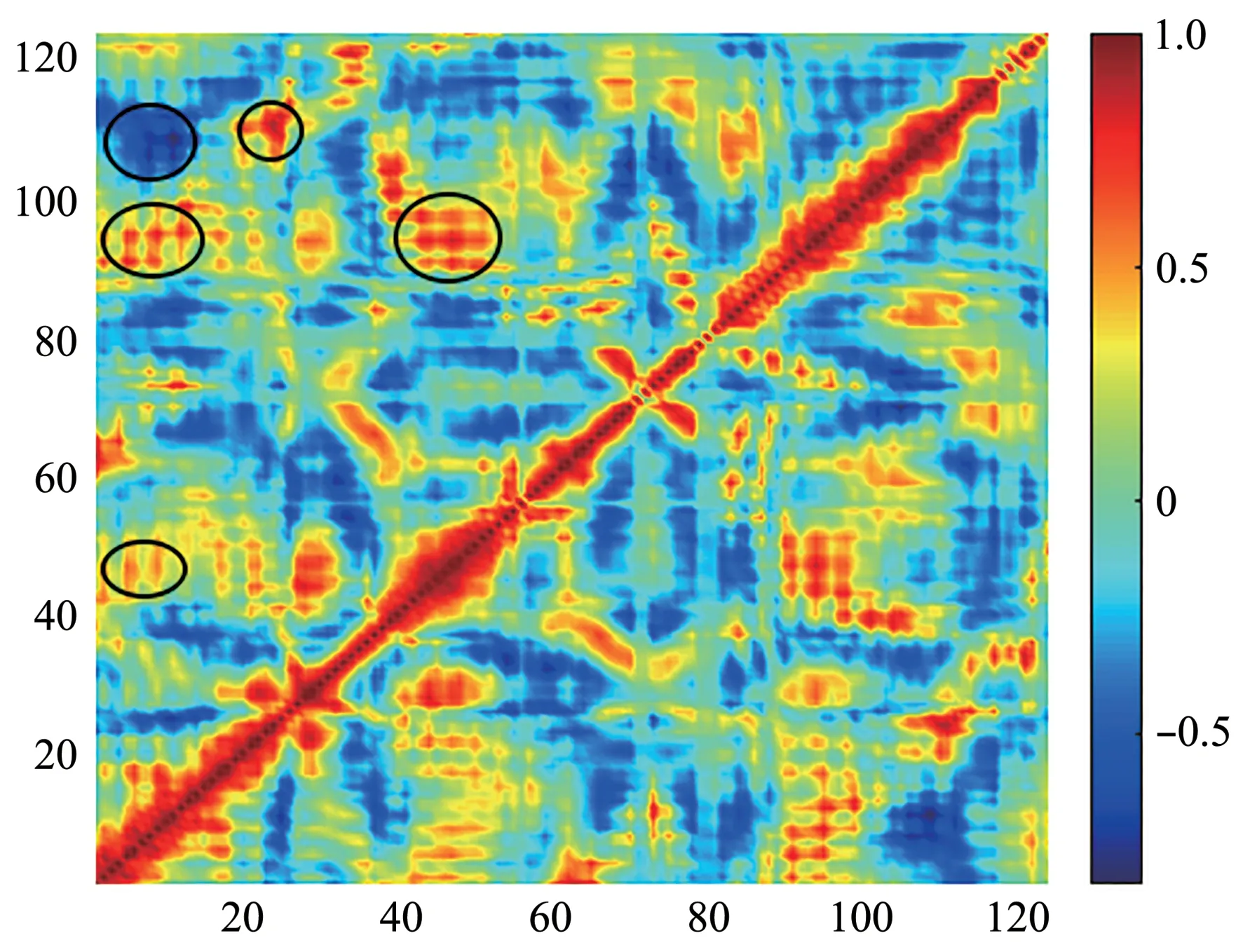
Fig.3 Average residue cross-correlations for human 31 sPLA2-IIA members
从 图3 可 见,残 基Thr104~Asn114 (位 于helix5和loop6上)与残基Asn1~Thr13(位于helix1上)呈较强负相关,而与残基Tyr21~His27(位于loop2 上)呈正相关。这些残基在催化位点周围,这种强相关运动有助于酶催化残基的暴露及催化反应的发生。此外,残基Ala39~Arg53 和残基Cys88~Asn100 之间存在较强的同向运动,它们分别位于承担催化作用的helix3 和helix4 上,其中His47、Asp48、Tyr51 和Asp91 构成了催化网络。这些残基的同向运动保证了催化功能的稳定发挥。另外还发现,残基Asn1~Thr13 (helix1)、残基Ala39~Arg53 (helix3) 和 残 基 Cys88~Asn100(helix4)呈现同向运动。helix1 和4 的同向运动部分是由于形成了疏水三联体(Phe5-Ala94-Phe98)而形成的。该三联体在空间上彼此接近,参与了高度保守的底物结合疏水通道的形成,促进了磷脂与催化位点的结合[16]。总之,酶在催化位点周围的强相关运动有助于酶与膜和磷脂的结合,促进催化反应的发生。
2.3 微扰响应扫描(PRS)分析
PRS 方法将ENM 与LRT 相结合,用来评估在蛋白质上施加微扰对其构象的影响。计算了31 个结构的平均PRS热图(图4a),及其各列元素平均后得到的残基敏感性曲线(图4c)。发现敏感性较高的残基恰好对应于从GNM慢运动模式分析识别到的5个区域。区域1负责Ca2+结合,敏感性较高,这是酶实现催化功能的要求。区域2、4和5均参与了酶与膜的相互作用,膜作为变构剂调控酶的构象。也就是说,这些区域在酶的变构中起到了重要作用。总之,PRS是识别蛋白质变构过程中起关键作用残基的有效方法。
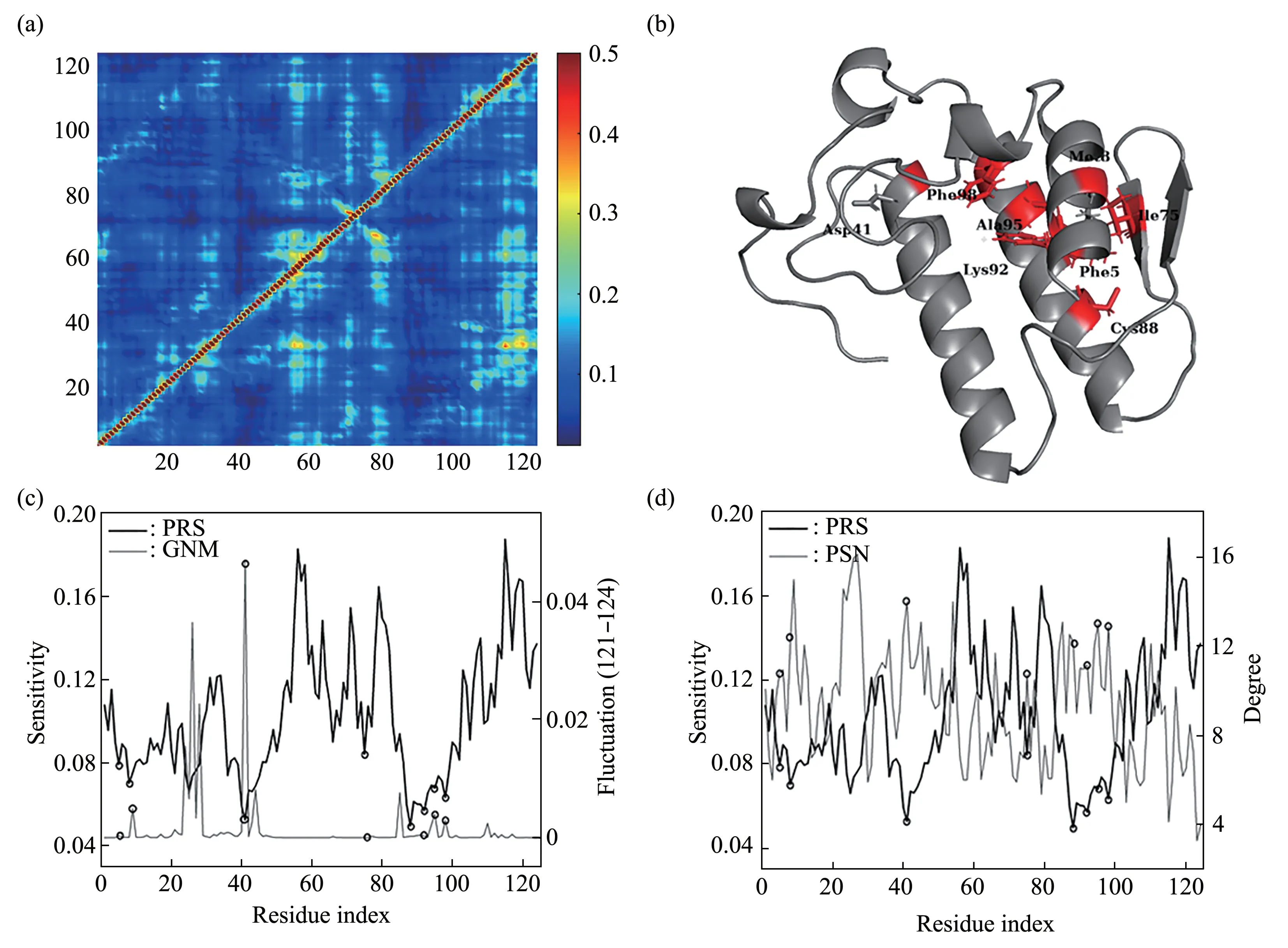
Fig.4 Evaluation of the role of sPLA2 residues sensitivity and variation among family members
除此之外,关注到一些具有较低敏感性的残基Phe5、Met8(位于helix1 上)、Asp41(位于helix3上)、Ⅰle75 (位于β 折叠上)、Cys88、Lys92、Ala95、Phe98(位于helix4 上),它们包埋在结构中(图4b)。有意思的是,这些低敏感残基几乎占据了快运动模式下(最快的3 个121~124)涨落极大值的位置(图4c)。GNM 的快运动模式反映了蛋白质局部结构的几何不规则性,在蛋白质折叠中,相较于慢运动模式下涨落,快运动模式下的涨落伴随着更大的熵减[17]。同以往的研究结果一致,本研究发现快运动模式下活跃的残基往往处于蛋白质折叠核区域,并且这些残基对结构稳定起重要作用[17-18]。同时也发现它们在蛋白质结构网络中具有高的平均度值(图4d)。残基度值(degree)反映了残基在局部网络中的连通性,也反映了残基对结构稳定的作用[19]。因此,这些残基在结构上的紧密堆积(高的连通性)以及在快运动模式下的高活动性表明它们在稳定结构方面具有重要作用。
3 结 论
本工作探索了人sPLA2中ⅠⅠA 型结构动力学和变构特性,探索了其动力学共性和特异性与功能的关系。根据GNM分析,发现结构中动力学高度受限的残基,即催化残基和半胱氨酸残基,与酶的共有功能有关,它们分别对酶的催化和结构稳定至关重要,在不同成员之间共享。另外,识别到柔性较大的5个区域,它们参与了保证酶催化活性的钙离子的结合,以及与别构剂膜的结合。这些区域的涨落在不同成员中差异较为显著,体现出了成员的特异性。ANM 分析结果暗示了酶催化位点周围的强相关运动,这有利于配体的结合及酶催化功能的实现。最后PRS 方法对酶变构特性的分析表明,对扰动高度敏感的残基对应上述柔性较大的5 个区域,说明这些残基作为传感器在变构通信中发挥重要作用,而低敏感性残基在快运动模式下较为活跃,对酶的结构稳定有大的贡献。本工作有助于深入理解sPLA2酶共有和特异性功能背后动力学的机制,对同系物设计和药物设计有一定的指导意义。
