COMP特异性单抗15A11的表位特征研究①
2016-01-08王瑞玲韩魏巍邓灵福袁永泽刘德立
王瑞玲 韩 冬 韩魏巍 邓灵福 袁永泽 熊 丽 刘德立 耿 辉
(华中师范大学生命科学学院,湖北省遗传调控与整合生物学重点实验室,武汉430079)
[摘 要] 目的:鉴定RA相关自身抗原COMP的一株特异性单克隆抗体15A11的表位特征。方法:选用随机十二肽噬菌体库对mAb 15A11进行三轮筛选,随机挑取40个单噬菌斑,提取DNA,测序;ELISA检测每个噬菌体克隆与mAb 15A11结合的特异性;通过ClustalW2对特异性结合的噬菌体展示的十二肽和COMP进行氨基酸序列比对,PyMOL分析一致氨基酸所在肽链的二级结构及表位氨基酸之间的距离,初步确定mAb 15A11的表位;变性和非变性Western blot、EDTA螯合Ca2+后ELISA分析以及合成多肽的ELISA实验进一步确定表位序列。结果:得到的40个测序噬菌体中,共有5个噬菌体克隆,ELISA确定克隆1和克隆2与mAb 15A11特异性结合,其他克隆均为非特异性结合的噬菌体;氨基酸序列比对,在COMP上未发现与克隆1和克隆2噬菌体相同的连续氨基酸序列,提示mAb 15A11的抗原表位可能为非线性表位;PyMOL分析表位氨基酸在COMP上的定位及距离,显示构象表位的合理性;变性Western blot分析为阴性,而非变性Western blot条件下为阳性;EDTA螯合Ca2+破坏COMP的构象后不能与mAb 15A11结合,而未经处理的与mAb 15A11结合,均说明mAb 15A11表位是构象表位;合成多肽与mAb 15A11的ELISA结果进一步确定了mAb 15A11的构象表位序列。结论:鉴定了mAb 15A11的表位是构象表位序列,且确定了该构象表位的氨基酸组成,为研究COMP抗体与抗原反应机制具有重要理论价值,并对类风湿性关节炎的检测有重要应用意义。
[关键词] COMP;单克隆抗体;类风湿性关节炎;噬菌体展示技术;表位
doi:10.3969/j.issn.1000-484X.2015.11.006
COMP特异性单抗15A11的表位特征研究①
王瑞玲韩冬韩魏巍邓灵福袁永泽熊丽刘德立耿辉
(华中师范大学生命科学学院,湖北省遗传调控与整合生物学重点实验室,武汉430079)
[摘要]目的:鉴定RA相关自身抗原COMP的一株特异性单克隆抗体15A11的表位特征。方法:选用随机十二肽噬菌体库对mAb 15A11进行三轮筛选,随机挑取40个单噬菌斑,提取DNA,测序;ELISA检测每个噬菌体克隆与mAb 15A11结合的特异性;通过ClustalW2对特异性结合的噬菌体展示的十二肽和COMP进行氨基酸序列比对,PyMOL分析一致氨基酸所在肽链的二级结构及表位氨基酸之间的距离,初步确定mAb 15A11的表位;变性和非变性Western blot、EDTA螯合Ca2+后ELISA分析以及合成多肽的ELISA实验进一步确定表位序列。结果:得到的40个测序噬菌体中,共有5个噬菌体克隆,ELISA确定克隆1和克隆2与mAb 15A11特异性结合,其他克隆均为非特异性结合的噬菌体;氨基酸序列比对,在COMP上未发现与克隆1和克隆2噬菌体相同的连续氨基酸序列,提示mAb 15A11的抗原表位可能为非线性表位;PyMOL分析表位氨基酸在COMP上的定位及距离,显示构象表位的合理性;变性Western blot分析为阴性,而非变性Western blot条件下为阳性;EDTA螯合Ca2+破坏COMP的构象后不能与mAb 15A11结合,而未经处理的与mAb 15A11结合,均说明mAb 15A11表位是构象表位;合成多肽与mAb 15A11的ELISA结果进一步确定了mAb 15A11的构象表位序列。结论:鉴定了mAb 15A11的表位是构象表位序列,且确定了该构象表位的氨基酸组成,为研究COMP抗体与抗原反应机制具有重要理论价值,并对类风湿性关节炎的检测有重要应用意义。
[关键词]COMP;单克隆抗体;类风湿性关节炎;噬菌体展示技术;表位
doi:10.3969/j.issn.1000-484X.2015.11.006
中图分类号①本文受湖北省自然科学基金重点项目(2014CFA079 ) 和国家自然科学基金(No.30972693,21473065)资助。
作者简介:王瑞玲(1990年-),女,主要从事免疫耐受与耐受失调研究,E-mail:ruilingWangss@163.com。
通讯作者及指导教师:耿辉(1971年-),女,博士后,副教授,硕士生导师,主要从事免疫耐受与耐受失调研究,E-mail:genghui@mail.ccnu.edu.cn。
[Abstract]Objective:To identify the epitope of mAb15A11 which is specific against RA associated autoantigen cartilage oligomeric matrix protein(COMP).Methods: A filamentous phage library displaying random linear dodecapeptides was used to mapping the epitope of mAb15A11.After three rounds of screenings,40 phage clones were selected at random and sequenced.The specificity of phages was confirmed by enzyme immunoassays.Homology search by ClustalW2 and structure analysis by PyMol to identified the epitope amino acid sequence.Western blot analysis of COMP and ELISA analysis of COMP-derived peptides were used to confirm epitope′s characterization.Results: After repeated screenings using bio-panning method,2 clones were identified,which interacted specifically with mAb 15A11.Homology search did not find succession consensus sequence within COMP molecular,which indicated that the epitope was not linear.PyMol Structure analysis identified the rationality of conformational epitope.Western blot analysis and ELISA of EDTA-treated COMP further prove an conformational structure of the epitope recognized by mAb 15A11.ELISA analysis of COMP-derived peptides demonstrated both disulfide bonds between229C-243C and237C-253C and every epitope amino acid of232G,238H,240H,241A,244V,247R and251R were essential to the binding of mAb 15A11 with COMP.Conclusion: In this study,the potential B cell antigentic epitopes of mAb 15A11 was identified by phage display library.The epitope amino acids sequence and characterization were also recognized.It may have important theoretical value for the study of reaction mechanism of COMP antibody and antigen and may also show application significance in the detection of rheumatoid arthritis.
Identification of epitope recognized by mAb 15A11 sepecific against cartilage oligomeric matrix protein
WANGRui-Ling,HANDong,HANWei-Wei,DENGLing-Fu,YANGYong-Ze,XIONGLi,LIUDe-Li,GENGHui.CollegeofLifeScience,HuazhongNormalUniversity,HubeiKeyLaboratoryofGeneticRegulationandIntegrativeBiology,Wuhan430079,China
[Key words]Cartilage oligomeric matrix protein;Monoclonal antibody;Rheumatoid arthritis;Phage display;Antigenic epitope
类风湿关节炎(Rheumatoid arthritis,RA)是一种自身免疫性疾病,具有慢性、多发性、进行性、侵袭性特点,主要临床表现为:关节滑膜炎和关节外病变。它由滑膜巨噬细胞、T细胞、B细胞以及成纤维细胞的复合作用导致关节侵蚀性炎症的发生[1]。与RA相关的蛋白质包括软骨寡聚基质蛋白(Cartilage oligomeric matric protein,COMP)、蛋白聚糖、Ⅱ/Ⅸ/Ⅺ型胶原蛋白、纤维蛋白原、α-烯醇化酶、葡萄糖-6-磷酸异构酶及波形蛋白,而且这些RA相关自身抗原与HLA-DR4的结合有关[2,3]。
COMP是一种钙结合糖蛋白,为主要在软骨中表达的组织特异性非胶原蛋白,有研究表明它也在肌腱、半月板以及滑膜中表达[4]。它是细胞外基质的主要组成成分,在细胞的行为和软骨形成中起到重要作用[5,6]。目前研究发现,COMP是RA相关的自身抗原[1,7]。COMP免疫引起的关节炎症反应和人类类风湿性关节炎相似。抗COMP血清转移及COMP特异性单抗过继性输注能够导致受体鼠发生急性关节炎症。我们最近制备并鉴定了一株导致类风湿性关节炎的单克隆抗体15A11,初步鉴定它识别COMP的EGF4-Like结构域。
蛋白质表位是蛋白质抗原性的基础,深入研究抗原表位对于设计多肽和新型疫苗以及开发诊断试剂具有重要意义。B细胞表位研究方法包括理论预测和实验鉴定。最经典的表位预测参数为Hopp等[8]学者在20世纪80年代提出的亲水性参数及二级结构预测方案等[9]。它们单独应用时准确率低,有一定的局限性。现在广泛使用的很多预测B细胞表位的网站或者软件都是基于综合多种参数或方案的预测模式,如ABCpred、Bcepred、DiscoTope、EPSVR、Ellipro、BepiPred、CEP等[10-16]。虽然可以通过表位预测使表位研究过程有效的简化,但还需要实验验证其真实性。研究B细胞表位的实验方法有肽库途径、核磁共振光谱(NMR)、表面等离子共振(SPR)、化学裂解法或生物酶解法、杂交肽裂解法、肽序列扫描法、肽芯片高通量筛选法[17-23]。肽库途径包括噬菌体随机肽库展示技术和有机合成肽库法,与生物肽库相比,有机合成肽库法,包括氨基酸序列分析在内,其成本很高而且不能扩增以及重复使用。因此,噬菌体随机肽库展示技术最广泛应用于肽库途径[24]。噬菌体展示技术被证明是确定酶、蛋白质甚至非蛋白质的配体,抗体表位的有力工具[25,26]。本文首先通过噬菌体展示技术确定了mAb 15A11的抗原表位以及在COMP中的构象状态,用PyMol分析一致氨基酸的二级结构及表位氨基酸之间的距离,再经变性、非变性Western blot以及合成多肽的ELISA实验法进一步确定表位序列。本文为研究COMP抗体与抗原反应机制具有重要理论价值,并对类风湿性关节炎的检测有重要应用意义。
1材料与方法
1.1材料随机十二肽噬菌体库购自New England Biolabs。四环素购自大连宝生物,Eag I-HF、Kpn I-HF购自NEB(北京)、抗-M13抗体购自GE Healthcare。羊抗鼠 IgG-HRP 酶标二抗购自 Sigma。预染蛋白Marker购自Fermentas。PVDF膜购自Amersham。Taq-mix、DNA Marker及蛋白标准Marker购自东盛生物。回收试剂盒购自博大泰克。其他试剂均为国产或进口分析纯试剂。COMP蛋白的晶体结构来自于PDB 数据库,登录号:3FBY。
1.2方法
1.2.1mAb 15A11的纯化及重组COMP蛋白的制备实验室早期制备的抗-COMP mAb 15A11的杂交瘤细胞并得以鉴定[27],复苏冰冻细胞,并在加入10%低-IgG胎牛血清的DMEM培养基中培养(37℃,5% CO2)。上清用蛋白G柱层析分离并纯化mAb 15A11。ELISA测定其效价,并用Nano drop 测定抗体的浓度。按照先前的方法制备重组COMP蛋白[27,28]。
1.2.2噬菌体滴度测定在微量离心管中加入对数期宿主菌ER2738 200 μl,分别加入10 μl不同稀释度的噬菌体,快速震荡混匀,室温下温育1~5 min;将感染细胞迅速加入预温的顶层琼脂培养管中,快速摇匀,立即注倒于37 ℃预温的LB/IPTG/Xgal平板上。倒置于37 ℃培养过夜;对平板上生长出的噬菌斑计数,乘以稀释因子,得到每10 μl噬菌体的空斑形成单位(pfu)滴度,计算得出噬菌体的滴度。
1.2.3随机十二肽噬菌体库的筛选用10 μg/ml mAb 15A11包被96孔板,4℃孵育过夜,挑取ER 2738单克隆于10 ml LB+Tet 液体培养基中,37 ℃恒温摇床中过夜培养;倒掉包被液,用力拍甩除去残余的溶液;加封阻缓冲液[(0.1 mol/L NaHCO3,5 g/L 牛血清白蛋白(BSA),0.2 g/L NaN3,pH8.6],4℃封闭2 h;除去封阻液,用0.1 % TBST[TBS+0.1 %(v/v)Tween-20]缓冲液快速洗板6次;加入100倍稀释的原始噬菌体文库(1.5 ×1013pfu/ml)100 μl,温和摇动60 min;吸去未结合噬菌体,TBST缓冲液洗板10次;加入100 μl非特异性缓冲液[0.2 mol/L Glycine-HCl(pH2.2),1 mg/ml BSA]96孔板,温和摇动10~20 min,收集洗脱液加入15 μl 1 mol/L HCl(pH9.1)中和。取2 μl洗脱液用于滴度测定,其余噬菌体加入处于对数生长期的宿主菌ER2738中,37℃剧烈摇动(250 r/min)培养4.5~5 h进行扩增;扩增噬菌体转入灭菌大离心管中,于冷冻离心机4℃ 12 000 r/min离心10 min,上清液转入另一灭菌的离心管,再次离心以除去残留的沉淀,80%上清液转入新鲜无菌的大离心管,加入体积为上清液1/6体积的20% PEG/2.5 mol/L NaCl,4 ℃沉淀过夜;4℃12 000 r/min离心15 min,弃上清,得到噬菌体沉淀。TBS溶液重悬噬菌体沉淀,转入微量离心管4℃,14 000 r/min离心5 min,除去残留细胞沉淀;上清液转入新微量离心管,加入1/6体积的20%PEG/2.5 mol/L NaCl,冰上孵育60 min;4℃,14 000 r/min离心10 min,弃上清,200 μl TBS重悬噬菌体,离心除去所有残余的不溶物沉淀,上清即为扩增的洗脱物。10 μg/ml mAb 15A11重新包被96孔板,进行噬菌体的第二轮和第三轮筛选。第二轮洗脱液为TBST+0.3% Tween,第三轮洗脱液为TBST+0.5% Tween,其余方法步骤均与第一轮筛选相同。第三轮噬菌体洗脱物的滴度测定时得到的LB/IPTG/Xgal平板的噬菌斑,4℃贮存,用于测序。
1.2.4噬菌斑测序模板的快速纯化收集1 000 μl含噬菌体的上清液,加入400 μl 20% PEG/2.5 mol/L NaCl,颠倒混匀,室温静置10~20 min;14 000 r/min 4 ℃离心10 min,弃上清;噬菌体沉淀用100 μl碘化物缓冲液彻底重悬,加入250 μl乙醇,颠倒混匀,室温温育10 min,14 000 r/min 4 ℃离心10 min,弃上清,沉淀中含有单链噬菌体DNA。用0.5 ml冰育的70% 乙醇洗沉淀,重新离心,除上清,室温干燥。沉淀的DNA用30 μl灭菌ddH2O重悬,取1 μl DNA进行琼脂糖凝胶电泳,检测噬菌体DNA是否提取成功,片段大小及纯度,以便测序。
1.2.5噬菌斑DNA序列测定选用New England Biolabs试剂盒中提供的-96 gIII测序引物:5′-HOCCC TCA TAG TTA GCG TAA CG-3′,送至金斯瑞生物公司进行自动测序。
1.2.6噬菌体ELISA扩增噬菌体克隆,对每个克隆进行滴度测定。100 μg/ml 的mAb 15A11包被96孔板,4℃过夜。除去多余靶分子,加封阻液,置于室温1~2 h;甩出封阻液,TBS/Tween洗板6次,Tween浓度与淘选清洗步骤中所用浓度相同;分别将稀释好的100 μl含有1011、109、107个病毒粒子的TBS 以及空M13噬菌体分别加入包被孔,及未包被有靶分子的孔中,室温震荡作用1-2 hrs。TBST洗板6次;以1∶5 000的比例用封阻液稀释HRP标记的抗-M13抗体,每孔加入200 μl稀释抗体,4 ℃孵育1 h;1×TBS/Tween洗板6次;每孔加入200 μl碱性磷酸酶和四甲基联苯胺为底物,室温避光孵育15 min,每孔加入25 μl 2 mol/L H2SO4,用酶标仪读取450 nm处的吸光值。
1.2.7噬菌体插入十二肽序列在COMP上的定位及结构分析筛选的噬菌体克隆通过测序找出一致序列,并将一致序列通过噬菌体ELISA确定其结合的特异性,对于特异性结合的噬菌体,用在线生物软件ClustalW2对筛选噬菌体的插入十二肽与COMP进行氨基酸序列比对,PyMol对可能的表位氨基酸进行COMP定位,并分析氨基酸所在肽链的二级结构及氨基酸之间的距离。
1.2.8Western blot分析(1)变性Western blot:分别制备12%的分离胶和5%的浓缩胶;取重组COMP蛋白5 μl,加入蛋白质变性缓冲液混合,沸水浴10 min,再冰浴2 min;取10 μl样品及5 μl预染蛋白Marker 分别点样,用80 V电压电泳30 min,至指示带到达分离胶,调节电压至120 V,继续电泳90 min,待指示带到达分离胶底部而且预染蛋白Marker中的条带分离后停止电泳;转膜;封闭1 h,用TBST洗3次,每次5 min,与1∶200比例稀释的COMP的单克隆抗体 mAb 15A11孵育1 h,TBST洗3次,每次5 min,与1∶200比例稀释的羊抗鼠酶标二抗孵育1 h,TBST洗3次,加入底物DAB孵育15 min指示条带的位置。
(2)非变性Western blot制备8 %的分离胶和5 %的浓缩胶;取COMP蛋白5 μl(在检测Ca2+对COMP构象的影响时,COMP一组经EDTA处理,一组未经处理),加入蛋白质非变性缓冲液(4×),电泳除了加入的电泳缓冲液为未加SDS的非变形电泳缓冲液,转膜时用未加SDS的转膜缓冲液转膜2.5 h,其余条件和方法与变性Western blot相同。
1.2.9合成多肽氨基酸序列比对初步确定的一致序列,在强耀生物公司通过SYMPHONY 12-通道肽合成仪(Protein Technologies Inc,Tucson,USA)用基于fluorenylmethoxycarbonyl的化学方法来合成,末端氨基酸通过Gly-Gly-Gly与生物素连接。粗产品在C18柱子(30*250 mm;Venusi MRC-ODS C18)上用反向高压液相色谱纯化,用含有0.1%三氟乙酸的乙腈-水洗脱,最终成分经过质谱分析和氨基酸分析。
1.2.10多肽ELISA 1.5 μg/ml的生物素化的合成多肽加入5 μg/ml链霉亲和素包被的96孔板,4 ℃包被过夜,封阻缓冲液封闭2 h;TBST洗板6次;加入1∶200的比例稀释mAb 15A11,结合的mAb 15A11通过羊抗鼠HRP-酶连IgG多克隆抗体检测,方法与噬菌体ELISA相同。
2结果
2.1mAb 15A11的合成及纯化复苏抗-COMP mAb 15A11的杂交瘤细胞,置于10%低-IgG胎牛血清的DMEM培养基中培养,收集上清液经蛋白G柱层析分离并纯化得到mAb 15A11,免疫印迹检测其与COMP结合的特异性(结果未显示)。并测定抗体的浓度约为1 mg/ml。
2.2随机十二肽噬菌体库的筛选结果用mAb 15A11对随机十二肽噬菌体库进行连续三轮筛选,每一轮筛选后均测定洗脱噬菌体的滴度,并测定第一轮和第二轮洗脱噬菌体扩增后的滴度,即为第二轮和第三轮噬菌体筛选时加入噬菌体的滴度,表1显示随着噬菌体筛选轮数的增加,富集度逐渐增加,说明特异性结合的噬菌体得到很好的富集。
2.3mAb 15A11筛选噬菌体展示的十二肽先找出测序噬菌体DNA的KpnⅠ以及EagⅠ的酶切位点,再从互补链的5′-3′方向,找到GGCCGA核苷酸序列,其随后的3′方向的序列即为包括间隙中三个Gly以及插入十二肽在内的十五肽序列,用EditSeq将序列反转,并翻译成氨基酸序列,通过三轮噬菌体的亲和筛选,得到五个克隆,获得十二肽的氨基酸序列如表2所示。结果显示,测序的40个噬菌体克隆中,GLHTSATNLYLH出现了28次,HLFNHNKNLP KR出现了8次,其他噬菌体克隆的氨基酸序列仅出现1~2次。
2.4筛选噬菌体与mAb 15A11结合特异性通过ELISA鉴定筛选得到的5个噬菌体克隆与mAb15A11的结合特异性,用不包被任何抗体的孔作为对照组。图1显示,展示GLHTSATNLYLH氨基酸序列的G1噬菌体(图1A)、展示HLFNHNKNLPKR氨基酸序列的G2噬菌体(图1B)与mAb 15A11相对结合能力明显高于没有插入任何肽的空M13噬菌体,以及未包被任何抗体的阴性对照组,且此两个噬菌体克隆均为浓度依赖型,随着噬菌体浓度的增加,与mAb 15A11结合力增强,说明G1和G2与mAb 15A11是特异性结合的,并且虽然G2克隆较少,但是其与 mAb 15A11的结合能力更强。而G3、G4、G5噬菌体克隆的结合能力和空M13噬菌体以及阴性对照均较弱,说明G3、G4、G5均为非特异性结合噬菌体。结果初步表明,G1和G2展示的十二肽是mAb 15A11结合的表位序列。
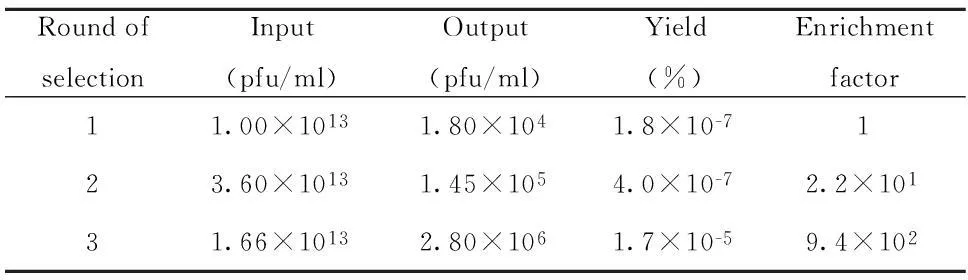
表1 mAb 15A11筛选噬菌体的投入/产出率Tab.1 Ratio of input/output of biopanning with mAb 15A11
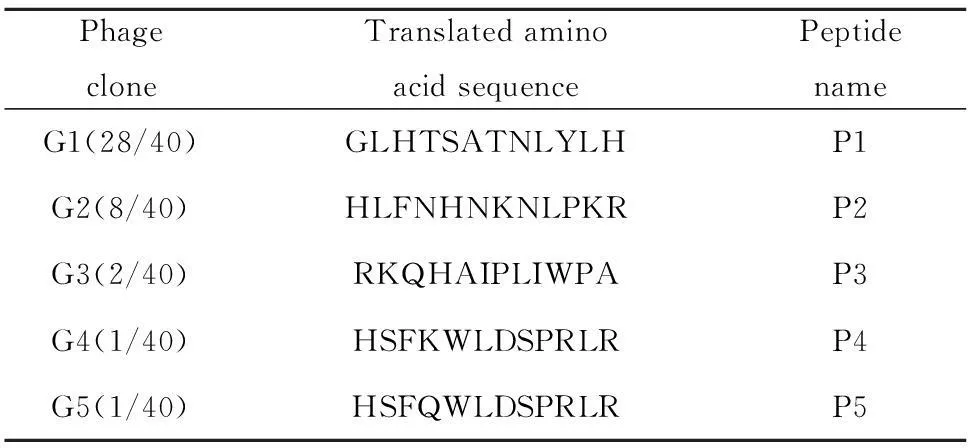
表2 mAb 15A11筛选噬菌体展示的十二肽Tab.2 mAb 15A11-binding peptides selected from random phage-displayed library

图1 筛选噬菌体与mAb 15A11结合的特异性Fig.1 Binding specificity of selected phages with mAb 15A11 in ELISA
2.5mAb 15A11结合表位在COMP上的定位我们早期的研究表明,mAb 15A11的结合表位在COMP的EGF-Like4结构域[28],从UniProt数据库获得该结构域的氨基酸序列,并与P1和P2进行氨基酸序列比对,图2A表明,P1和P2与COMP的EGF-Like4结构域虽有相同氨基酸(红色表示)以及性质相同的氨基酸(绿色表示),但未发现连续的一致氨基酸序列,提示mAb 15A11的表位是非线性表位。从PDB 数据库下载得到COMP蛋白的晶体结构(登录号:3FBY),用PyMol将相同或性质相似的氨基酸定位到COMP分子上,图2B和2C显示该七个氨基酸从不同角度观察的结果,绿色表示232G、蓝绿色表示238H、蓝色表示240H、红色表示241A、淡蓝色表示244V、洋红色表示247R、橘红色表示251R。可以看出,这七个氨基酸均处于蛋白质COMP的表面,可以构成构象表位。
2.6表位结构分析mAb 15A11结合表位相应肽链在COMP分子EGF-Like4结构域的结构如图3A表示,229C-243C和237C-253C 均有一个二硫键(黄色表示),232G、238H、240H均处于无规卷曲;241A处于β转角;244V、247R和251R处于β折叠(颜色表示与表位在COMP上的定位一致),与先前报道的表位特征吻合:β转角为凸出结构,多出现在蛋白质抗原表面,有利于与抗体结合,较可能成为抗原表位。进一步分析发现,这七个氨基酸均不处于α螺旋,且六个氨基酸与中间251R的距离均小于15 Å(如图3B),可以形成构象表位。这与α螺旋则不易变形、较难结合抗体、一般不作为抗原表位的研究报道结果一致。
2.7表位性质鉴定为了进一步鉴定表位的性质,用变性Western blot和非变性Western blot方法检测COMP结构改变对于mAb 15A11与COMP结合的影响。在变性SDS-PAGE胶试验中,由于SDS、加热等条件下,使得COMP的结构完全破坏而呈线性状态。而在非变性PAGE胶中COMP保持构象结构。图4B显示,在非变性PAGE胶实验中,免疫反应为阳性(图4A),而在变性SDS-PAGE实验中,免疫反应为阴性,即COMP构象破坏后与mAb 15A11的结合能力丧失。这些结果进一步表明,mAb 15A11的表位为构象表位。
研究表明,COMP构象的维持必须有Ca2+参与[4,30],COMP经EDTA处理螯合Ca2+后,Native PAGE结果表明,和未经EDTA螯合Ca2+的对照组相比,COMP电泳速度慢(结果未显示)。说明,EDTA螯合Ca2+后导致COMP的构象破坏。图4C说明,结构破坏的COMP和mAb 15A11的结合能力明显降低,与多克隆抗体作为一抗的对照组结果一致。
2.8合成多肽及ELISA为了进一步验证构象表位的正确性,采用化学方法合成225A-255C的一系列多肽(表3),末端氨基酸通过Gly-Gly-Gly与生物素连接。
合成多肽与mAb 15A11的结合能力通过ELISA进行鉴定。图5表明,合成三十肽226Q-C255的结合能力最强,说明表位位于该段多肽内;缺少229C和235的序列C232G-R251结合能力明显降低,表明229C-243C和237C-253C 之间的二硫键对于构象维持起关键作用;缺少232G的237C-R251和235S-V244结合能力更低,表明232G对于mAb 15A11和COMP的结合起重要作用,240H-R251、241A-R251、241A-S250、242D-R251、241A-S250和238H-R247没有结合能力,说明241A、244V、247R和251R对于结合也很重要。235SECHEHADCV244进一步说明238H、240H、241A和244V对于mAb 15A11和COMP结合的作用。以上结果均表明,232G、238H、240H、241A、244V、247R和251R是抗原表位,229C-243C和237C-253C 之间的二硫键对于构象维持以及mAb 15A11与COMP的结合具有关键作用。

图2 mAb 15A11结合表位在COMP上的定位Fig.2 Localization of mAb 15A11 binding epitope on COMP

图3 表位结构分析Fig.3 Structure analysis of epitope
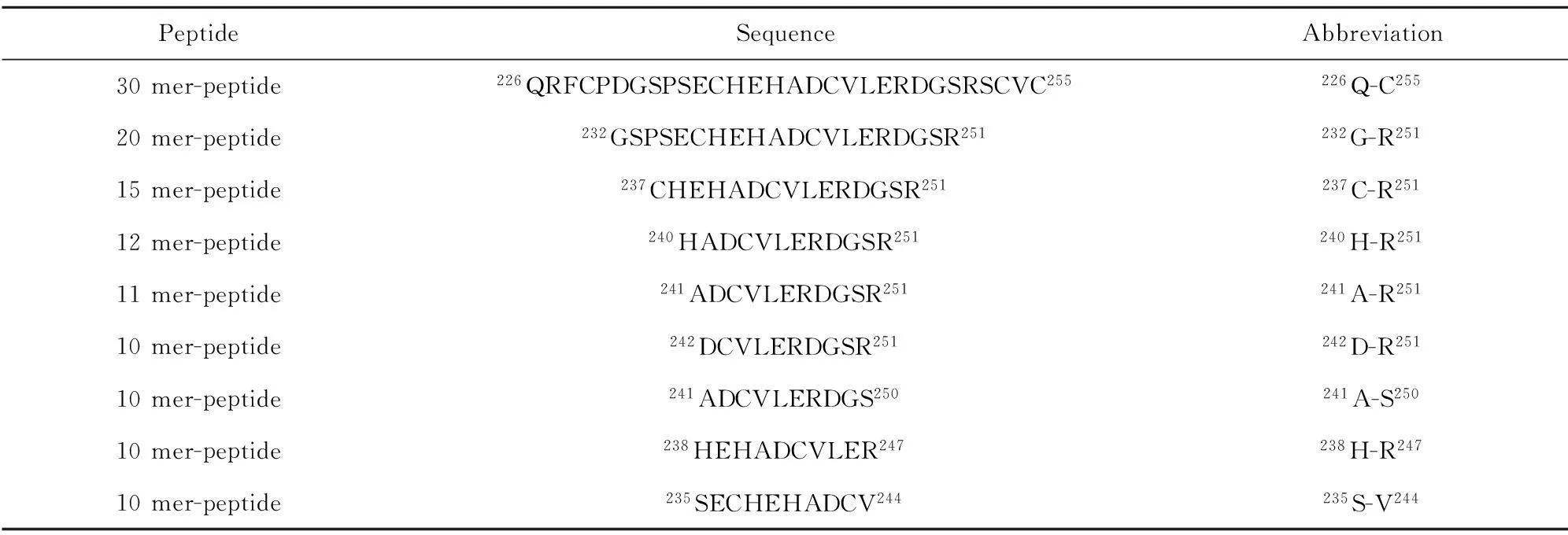
表3 合成多肽的COMP氨基酸序列Tab.3 Amino acid sequences of synthetic peptides deprived from COMP molecule
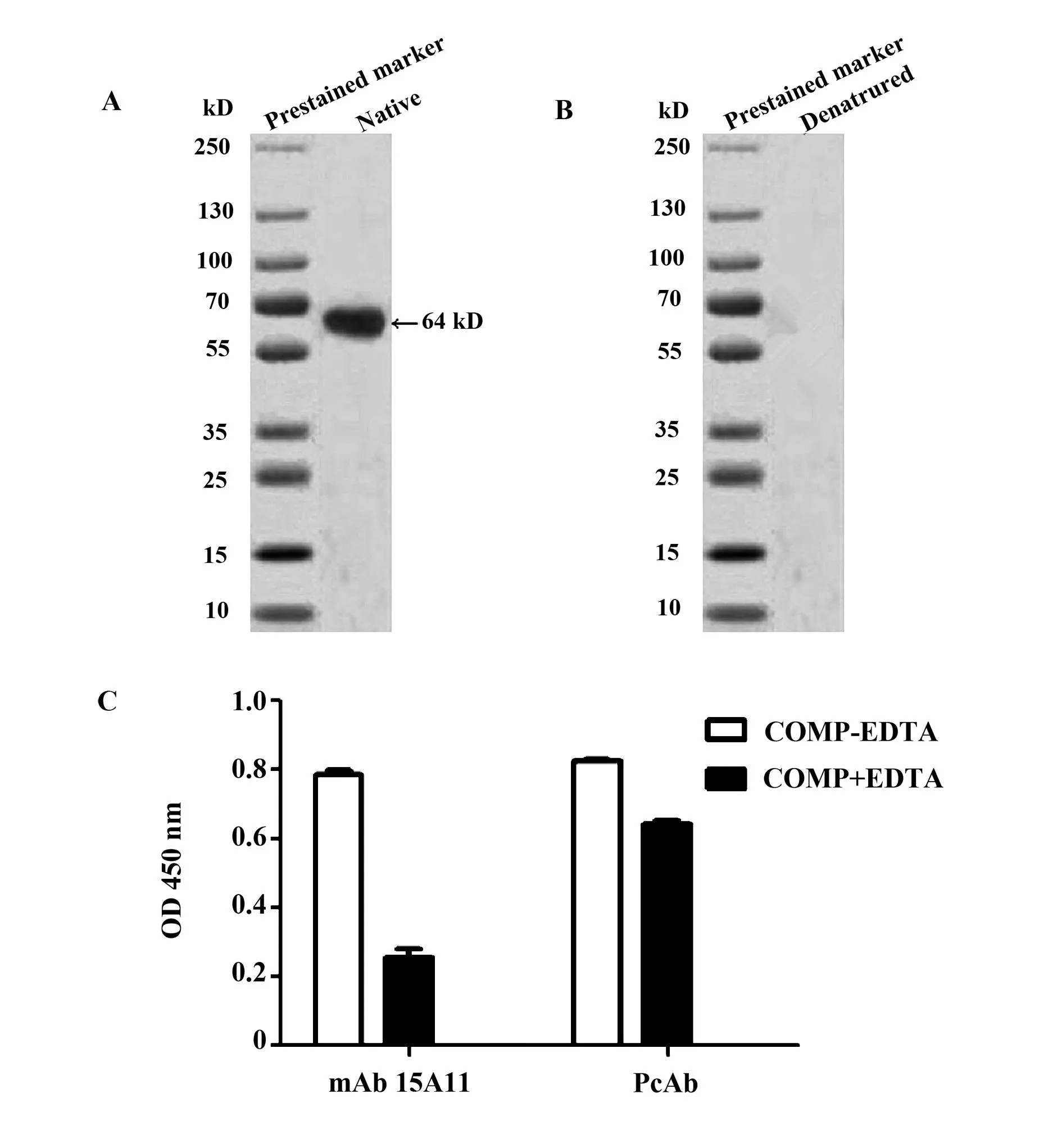
图4 表位性质鉴定Fig.4 Characterization identification of epitope

图5 合成多肽ELISAFig.5 ELISA analysis of COMP-derived peptides
3讨论
软骨寡聚基质蛋白(COMP)是一种类风湿性关节炎相关的自身抗原,mAb 15A11特异性识别COMP的一种单克隆抗体。为了鉴定mAb 15A11的COMP抗原表位,本研究首先采用随机十二肽噬菌体库进行三轮筛选,随着筛选次数的增加,噬菌体的投入产出率逐渐增加,说明特异性结合的噬菌体得到很好的富集。对第三轮筛选的噬菌体进行滴度测定,并随机挑取40个噬菌斑克隆,对其DNA进行测序,共得到五个噬菌体克隆,其中克隆1和克隆2为mAb 15A11特异性的。我们之前已经证实mAb 15A11结合COMP的EGF4-like结构域[27],翻译该两个克隆的插入十二肽氨基酸序列并与EGF4-like结构域氨基酸序列比对未发现连续一致氨基酸序列,表明该表位为构象表位。比对结果有七个不连续的相同或性质相同的氨基酸:232G、238H、240H、241A、244V、247R和251R。PyMol分析,该七个氨基酸均位于蛋白质的表面,且距离中间的251R均小于15Å,证明该七个氨基酸形成构象表位的可能性。Western blot以及EDTA处理COMP后的ELISA试验进一步证明该表位是构象表位。合成多肽与mAb 15A11的ELISA结果确定了mAb 15A11的构象表位序列。 本研究利用随机十二肽噬菌体展示技术筛初步确定表位序列及表位性质,再结合生物信息学进行分析,最后又回归试验,不仅进一步确定mAb 15A11的表位是构象表位,而且确定了表位序列的正确性。在炎症反应中,胞外基质发生改变,很多软骨成分作为酶裂解片段释放。其中,COMP更加容易裂解成50~70 kD的片段。除此之外,COMP片段还在软骨发生改变时释放到滑膜液和血清中[29-31]。在软骨中,基质金属蛋白酶(MMP)-1,-3,-9,-13和蛋白聚糖酶(ADAMTS)家族的成员如ADAMTS-4,-7 和 -12 与COMP的降解有关[32,33]。因此,这个抗体适合诊断类风湿性关节炎病人滑膜液和血清中的COMP片段,从而可以监视关节的损坏以及治疗的效果。不仅对研究COMP抗体与抗原反应机制具有重要理论价值,并对类风湿性关节炎的检测有重要应用意义。
参考文献:
[1]Souto-Carneiro MM,Burkhardt H,Müller EC,etal.Human monoclonal rheumatoid synovial B lymphocyte hybridoma with a new disease-related specificity for cartilage oligomeric matrix protein [J].J Immunol,2001,166(6):4202-4208.
[2]Trentham DE,Townes AS,Kang AH.Autoimmunity to type II collagen an experimental model of arthritis [J].J Exp Med,1977,146(3):857-868.
[3]Carlsen S,Lu S,Holmdahl R.Arthritis induced with minor cartilage proteins [J].Methods Mol Med,2007,136(2):225-242.
[4]Hedbom E,Antonsson P,Hjerpe A,etal.Cartilage matrix proteins.An acidic oligomeric protein(COMP)detected only in cartilage [J].J Biol Chem,1992,267(9):6132-6136.
[5]Chen FH,Thomas AO,Hecht JT,etal.Cartilage oligomeric matrix protein/thrombospondin 5 supports chondrocyte attachment through interaction with integrins [J].J Biol Chem,2005,280(38):32655-32661.
[6]Kipnes J,Carlberg AL,Loredo GA,etal.Effect of cartilage oligomeric matrix protein on mesenchymal chondrogenesis in vitro [J].Osteoarthritis Cartilage,2003,11(6):442-454.
[7]Carlsen S,Nandakumar KS,Backlund J,etal.Cartilage oligomeric matrix protein induction of chronic arthritis in mice [J].Arthritis Rheum,2008,58(7):2000-2011.
[8]Hopp TP,Woods KR.Prediction of protein antigenic determinants from amino acid sequences [J].Proc Natl Acad Sci USA,1981,78(6):3824-3828.
[9]来鲁花.蛋白质的结构预测和分子设计[M].北京大学出版社,1993:49.
[10]Saha S,Raghava G.Prediction of continuous B-cell epitope in an antigen using recurrent neural network [J].Protein,2006,65(1):40-48.
[11]Saha S,Raghava GPS.BcePred:Prediction of continuous B-cell epitopes in antigenic sequences using physico-chemical properties.In:Artificial immune systems [J].Springer Berlin Heidelberg,2004,3239(16):197-204.
[12]Jens Vindahl Kringelum1,Claus Lundegaard1,Ole Lund,etal.Reliable B cell epitope predictions:impacts of method development and improved benchmarking [J].PLoS Comput Biol,2012,8(12):e1002829.
[13]Liang S,Zheng D,Standley DM,etal.EPSVR and EPMeta:prediction of antigenic epitopes using support vector regression and multiple server results [J].BMC Bioinformatics,2010,11:381.
[14]Ponomarenko J,Bui H-H,Li W,etal.ElliPro:a new structure-based tool for the prediction of antibody epitopes[J].BMC Bioinformatics,2008,9:514.
[15]Larsen J,Lund O,Nielsen M.Improved method for predicting linear B-cell epitopes [J].Immunome Research,2006,2:2.
[16]Kulkarni-Kale U,Bhosle S,Kolaskar AS.CEP:a conforma-tional epitope prediction server [J].Nucleic Acids Res,2005,33(Web Server issue):W168-W171.
[17]Rosen O,Anglister J.Epitope mapping of antibody-antigen complexes by nuclear magnetic resonance spectroscopy [J].Methods Mol Biol,2009,524:37-57.
[18]Pär Säfsten.Epitope mapping by surface plasmon resonance [J].Methods Mol Biol,2009,524:67-76.
[19]Morris GE.Epitope mapping by chemical fragment [J].Methods Mol Biol,1996,66:121-127.
[20]Mazzoni MR,Porchia F,Hamm HE.Proteolytic fragmentation for epitope mapping [J].Methods Mol Biol,2009,524:77-86.
[21]Dhungana S,Williams JG,Fessler MB,etal.Epitope mapping by proteolysis of antigen-antibody complexes [J].Methods Mol Biol,1996,66:97-108.
[23]Maier RH,Maier CJ,Önder K,etal.Epitope mapping of antibodies using a cell array-based polypeptide library [J].J Biomol Screen,2010,15(4):418-426.
[24]Su qp,Huai yy,Wang yc,etal.The use of hybrid phage displaying antigen epitope and recombinant protein in the diagnosis of systemic Candida albicans infection in rabbits and cancer patients [J].Diagn Microbiol Infect Dis,2010,68(4):382-389.
[25]Smith GP,Petrenko VA.Phage display [J].Chem Rev,1997,97(2):391- 410.
[26]杨乔欣,马文煜,余颖.单纯疱疹病毒糖蛋白模拟表位的研究[J].中国免疫学杂志,2002,18(2):146-148.
[27]Geng H,Nandakumar KS,Pramhed A,etal.Cartilage oligomeric matrix protein specific antibodies are pathogenic [J].Arthritis Res Ther,2012,14(4):1478-6362.
[28]董佳慧,揭瑞,余金辉,等.抗人软骨寡聚基质蛋白单克隆抗体的研制及表位鉴定[J].中国免疫学杂志,2013,29(4):370-376.
[29] Brun J.Proteasome inhibition as a novel therapy in treating rheu-matoid arthritis [J].Med Hypotheses,2008,71(1):65-72.
[30] Mansson B,Carey D,Alini M,etal.Cartilage and bone metaboli-sm in rheumatoid arthritis.Differences between rapid and slow progression of disease identified by serum markers of cartilage metabolism [J].J Clin Invest,1995,95(3):1071-1077.
[31] Dickinson SC,Vankemmelbeke MN,Buttle DJ,etal.Cleavage of cartilage oligomeric matrix protein(thrombospondin-5)by matrix metalloproteinases and a disintegrin and metalloproteinase with thrombospondin motifs [J].Matrix Biol,2003,22(3):267-278.
[32] Luan Y,Kong L,Howell DR,etal.Inhibition of ADAMTS-7 and ADAMTS-12 degradation of cartilage oligomeric matrix protein by alpha-2-macroglobulin [J].Osteoarthritis Cartilage,2008,16(11):1413-1420.
[33] Kashiwagi M,Enghild JJ,Gendron C,etal.Altered proteolytic activities of ADAMTS-4 expressed by C-terminal processing [J].J Biol Chem,2004,279(11):10109-10119.
[收稿2015-02-11修回2015-04-17]
(编辑张晓舟)
