棕色棉DFR基因的克隆与生物信息学分析
2014-07-12肖向文朱奇朗刘海峰王俊铎罗城曾闻梁亚军龚兆龙李晓波
肖向文 朱奇朗 刘海峰 王俊铎 罗城 曾闻梁亚军 龚兆龙 李晓波
(1. 中国科学院新疆理化技术研究所 干旱区植物资源化学重点实验室,乌鲁木齐 830011;2. 中国彩棉(集团)股份有限公司,乌鲁木齐830016;3. 新疆农业科学院经济作物研究所,乌鲁木齐 830091)
天然彩色棉作为环境友好型的纯天然绿色产品,其纤维具有自然色彩,可省略化学染色,既节省生产成本,降低环境污染,又可避免纺织品中的化学染料对人体健康可能存在的某些不良影响,是真正意义上的“绿色、生态、环保”天然纤维产品。彩棉纺织品被誉为“人类第二健康肌肤”、“天然的护肤品”,符合人们对衣着需求的发展趋势和回归自然的潮流,迎合了市场的需求,是21世纪国际绿色纺织品市场上最具发展潜力的产品之一,开发前景十分广阔[1-3]。近几年经过科研工作者的努力,选育出了一批产量、色泽、品质等综合性状较优的彩色棉新品种,在产量、抗性水平以及纤维品质等方面取得了一定进展,但彩色棉花一直存在着颜色单一、色谱狭窄、着色不均匀及色泽不稳定等问题,此外彩棉与白色棉相比,纤维品质仍然有一定差距,这些因素都制约了彩棉的进一步发展[4,5]。因此,提高彩棉的纤维品质及获得稳定优良的色泽特性,对于彩棉产业的可持续发展具有重要意义。
彩棉色素物质是彩棉纤维中独特的次生代谢产物,此前的相关研究表明,彩棉色素物质的合成同植物类黄酮合成途径存在着密切关系[6],但具体机制尚不清楚。因此,开展彩棉类黄酮合成途径中关键基因的克隆、表达特性研究及转基因功能分析,对于揭示彩棉色素合成及调控的分子机制以及利用基因工程技术进行彩棉纤维色彩及品质的分子改良有着重要的意义。
类黄酮(flavonoids)是植物次生代谢产物,类黄酮化合物中的花青素等物质是许多植物花、果实和种子色素的主要成分。迄今为止,在一些高等植物中,如拟南芥、玉米、矮牵牛等,有关类黄酮物质合成途径的主要步骤已基本探明[7],类黄酮途径已成为研究植物次生代谢基因表达及调控的模式途径,也是植物次生代谢基因工程、代谢工程的主要目标。Xiao等[8]利用分子生物学方法,从棕色棉中分别克隆了类黄酮途径中的5个关键酶基因查尔酮异构酶(CHI),黄烷酮羟基化酶(F3H),二氢黄酮-4-还原酶(DFR),花青素合成酶(ANS),花青素还原酶(ANR);通过RT-PCR 的方法检测在棉纤维不同发育时期这5个基因的表达情况,结果表明这几个基因在棕色棉花中有较高的表达,而在白色和绿色棉花中表达量较低。此研究表明类黄酮途径参与了彩色棉纤维中色素的合成。
为了研究DFR基因及类黄酮途径在棕色棉纤维色素形成中的作用,本研究根据GenBank上登录的棕色棉品种T586的DFR基因序列[5]设计引物,以新彩棉6号(XC-6)纤维的RNA以及DNA为模板克隆得到GhDFR基因的CDS全长编码序列及带有内含子的基因组序列。通过生物信息学的方法对GhDFR的氨基酸序列组成成分、理化性质、疏水性、二级结构、信号肽、亚细胞定位、结构域等方面进行较为全面的分析。此项研究为探究DFR基因在棕色棉色素形成过程中的可能的功能及作用机制奠定前期理论基础,同时也为后期利用基因工程的技术对彩色棉的色彩进行遗传改良提供依据。
1 材料与方法
1.1 材料
1.1.1 植物材料 选择棕色棉品种新彩棉6号(XC-6)作为试验材料,2012年4月种植于新疆天然彩色棉花研究所实验基地,取开花后(DPA)16 d的纤维。所取新鲜材料迅速放入液氮中,-80℃保存备用。
1.1.2 菌种、质粒与试剂 大肠杆菌DH5α为本实验室保存;克隆载体pGEM-T Easy购自Promega;琼脂糖凝胶 DNA回收试剂盒购自天根公司;反转录试剂盒、DNA分子量Marker、ExTaqDNA聚合酶等购自TaKaRa。引物合成及测序由上海生工生物工程公司完成。其他试剂均为分析纯。
1.2 方法
1.2.1 基因组DNA、总RNA提取及cDNA第一链合成 基因组DNA的提取采用CTAB法[9],总RNA提取方法参照热硼酸/蛋白酶K法[10-12],总RNA经琼脂糖凝胶电泳检测,质量完好的RNA置-80℃保存备用。DNaseⅠ消化残留的DNA后,参照TaKaRa公司提供的反转录试剂盒说明书以Oligo(dT)18为引物,使用M-MLV、37℃ 温浴2 h,进行RT-PCR扩增,获得cDNA第一条链。
1.2.2GhDFR基因的克隆与测序 根据GenBank登录的序列EF187441,利用Primer 5.0进行引物设计,FP(Forward Primer):5'-GGTCTTTCTTTATGCCAAC-TG-3',RP(Reverse Primer):5'-GACATGGGTAGGCACTCAATT-3'。PCR的扩增体系为20 μL,包括10×PCR Buffer 2 μL,0.2 mmol/L dNTP,1.5 mmol/L MgCl2,模板cDNA(或基因组DNA)约50 ng,上下游引物各0.2 μmol/L,1 U Ex Taq DNA聚合酶(宝生物)。扩增程序:95℃ 预变性3 min;94℃ 45 s,58℃ 45 s,72℃ 1.5 min,35 个循环;72℃延伸10 min。PCR 产物经1%琼脂糖凝胶电泳后,用天根公司的胶回收试剂盒回收DNA,连入Promega 公司pGEM-T Easy载体,转化DH5α感受态细胞,将鉴定正确的阳性克隆质粒送上海生工公司测序。
1.2.3 引物的设计与生物信息学软件 应用ExPASy工具包(http://expasy.org/tools/)中的ProtParam(http://web.expasy.org/protparam/)在线分析GhDFR氨基酸序列的组成和理化性质。利用SignalP 3.0 Server(http://www.cbs.dtu.dk/services/SignalP/)在线分析蛋白质的信号肽。ProtScale(http://web.expasy.org/protscale/)在线预测DFR蛋白的亲水性/ 疏水性。应用PSORTII Prediction(http://psort.hgc.jp/form2.html)进行亚细胞定位。SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page =npsa_sopma.html)预测二级结构元件。序列相似性搜索用NCBI数据库中的Blast完成(http://blast.ncbi.nlm.nih.gov/)。DFR核苷酸和氨基酸序列的同源性多重比对用DNAMAN完成。Pfam 27.0(http://pfam.sanger.ac.uk/)和NCBI的CDD(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb. cgi)在线预测DFR蛋白的功能结构域。利用Clustal X软件(http://bips.u-strasbg.fr/fr/Documentation/ClustalX/)对基因组序列进行相似性比较,MEGA软件(http://www.megasoftware.net/)进行聚类分析。
2 结果
2.1 GhDFR基因的克隆与测序
核苷酸序列的测定结果如图1,图2,所克隆获得的GhDFR基因组序列为1 620 bp,CDS序列的长度为1 068 bp,编码355个氨基酸。序列结果已经提交GenBank,登录号为:KF749429。根据核苷酸序列测定结果,利用生物信息学与比较基因组学的方法,推测出该基因基因组序列有5个内含子和6个外显子,5个内含子以GT开始并以AG结尾的序列,而且3'端富含嘧啶核苷酸,基本符合内含子的类型。新彩棉6号中克隆的GhDFR基因CDS编码区与克隆自彩棉品种T586的GhDFR基因EF187441的CDS编码序列完全相同,与GenBank中已经登录的FJ713480序列相比,其编码区在5'端多出15个氨基酸编码序列。内含子可能是提高基因表达的一种原因,通过分析基因内部内含子的组成,对理解基因的表达调控机制具有非常重要的意义。内含子还可以作为来研究物种进化的分子标记。
2.2 GhDFR编码产物与其他植物GhDFR同源性比较
利用DNAMAN软件将得到的GhDFR基因所编码的氨基酸序列与GenBank上公布的其他物种DFR的氨基酸序列完全比对,结果如图3,发现该基因与圆叶葡萄VrDFR、毛果杨PtrDFR、葡萄VvDFR、天竺葵PzDFR、槿麻TcDFR、芍药PlDFR、甜樱桃PaDFR的氨基酸同源性较高,分别为80.8%、80.2%、79.6%、80.6%、79.0%、79.3%、76.9%,与拟南芥DFR同源性为72.7%。在GhDFR 蛋白质序列N端存在21个氨基酸残基的 NADP(H)结合位点“VTGGSGFIGSWLIKLLLERGY”,该序列在不同植物中具有一定的保守性,另外还存在一个由26个氨基酸构成的底物特异性结合保守区域“TIDVAEQQKPCYDETCWSDLEFIQAK”,决定其底物的特异性[13,14]。NCBI的BLAST比对结果表明,DFR 蛋白属于NADB-Rossmann超基因家族(NADBRossmann super-family)。
2.3 GhDFR氨基酸序列的理化性质
ProtParam分析得出:GhDFR蛋白的理论分子量为39.65 kD,等电点(pI)为5.67,其中Lys最多,有32个,占9.0%。其次为Leu,有29个,占8.2%。而Tyr较少只有7个,占2.0%。最少的是Trp仅有5个,占1.4%。其中带负电荷的氨基酸有48个,带正电荷的氨基酸有40个。不稳定指数为35.88,是一个相对稳定的蛋白。脂肪族氨基酸指数较高达到了85.15,这是因为GhDFR基因的蛋白含有较多的Leu。总的平均亲水值Grand average of hydropathicity(GRAVY)为-0.168,是亲水性蛋白。

图1 GhDFR基因的全长序列及推断的氨基酸序列
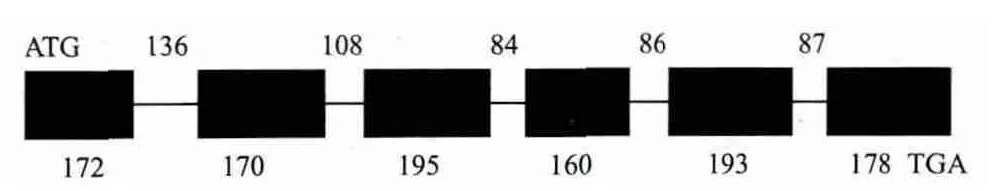
图2 GhDFR内含子及外显子分析
2.4 GhDFR的亲水性/疏水性、亚细胞定位
蛋白质亲水性/ 疏水性的预测是蛋白质二级结构预测以及功能域划分的重要的过程。ProtScale的分析结果(图4)表明GhDFR蛋白的亲水性/ 疏水性的最大值为208位的脯氨酸2.378,最小值为265位的丙氨酸-2.478,亲水/ 疏水趋势图形也基本一致,结果如图4所示。总体而言,与前面所有氨基酸序列理化性质预测的结果一致,GhDFR蛋白属于亲水性蛋白。

图3 GhDFR与其他物种DFR基因编码的氨基酸序列的多重比对
SignalP的分析结果(图5)表明GhDFR蛋白不具有信号肽,说明GhDFR蛋白不是一个分泌蛋白。
PSORT Ⅱ Prediction 对GhDFR蛋白亚细胞定位,结果表明,GhDFR定位于细胞质的可能性最高,为43.5%;其次为线粒体的26.1%,细胞核8.7%,内质网8.7%,最低为分泌囊泡、高尔基体和过氧化物酶体,可能性均为4.3%。
2.5 GhDFR蛋白的二级结构和功能结构域的预测
SOPMA对GhDFR进行二级结构的预测结果显示(图6),共有α-螺旋(Alpha helix)133处,占二级结构的37.46 %,延伸链(Extended strand)50处,占总二级结构的14.08%,β-转角(Beta turn)26处,占二级结构的7.32%,无规卷曲(Random coil)146处,占二级结构的41.13%。该蛋白中α-螺旋和无规则卷曲为该蛋白二级结构中的主要结构元件,分散于整个蛋白质中。
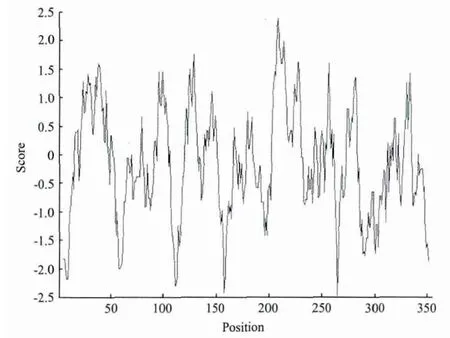
图4 GhDFR蛋白的亲水性/疏水性分析
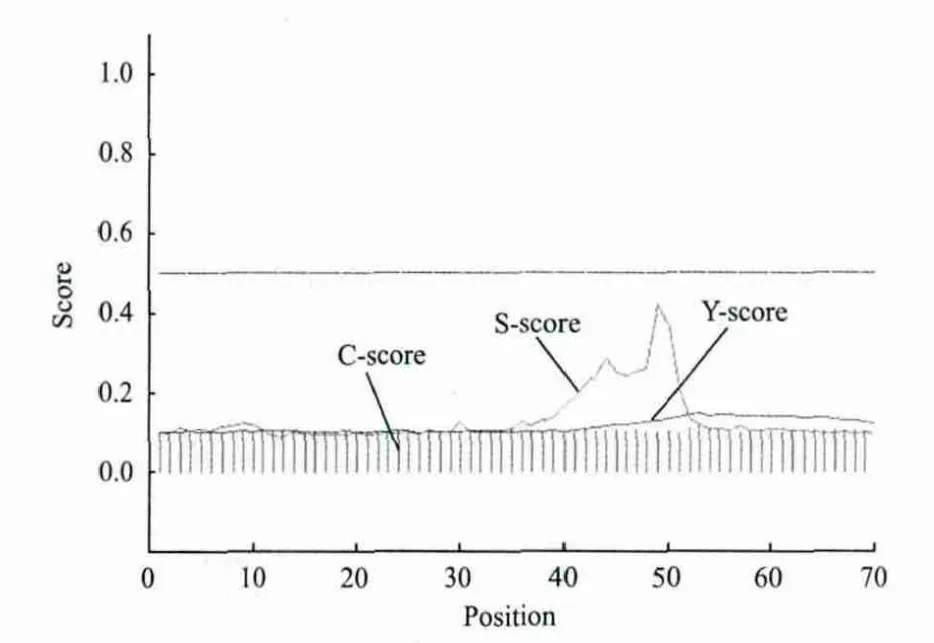
图5 GhDFR蛋白的信号肽预测分析
蛋白质结构域是蛋白质执行功能的结构基础。Pfam27.0预测GhDFR蛋白的结构域的结果(图7)表明,GhDFR蛋白只有一个明显的结构域,即NAD dependent epimerase/dehydratase family,与NCBI的CDD在线预测结果一样,都证实了GhDFR 属于NADB-Rossmann superfamily。
2.6 GhDFR蛋白系统进化分析
应用Clustalx1.83软件将编码的氨基酸序列及从GenBank获取的其他植物的DFR基因推导的氨基酸序列进行比对分析,然后利用 MEGA5.2软件按邻接法构建系统进化树,结果如图8所示,途中所示数字为相对遗传距离。GhDFR与天竺葵PzDFR蛋白以及芍药PlDFR的亲缘关系最近,可以聚为一类,与拟南芥AtDFR亲缘关系较远。
3 讨论
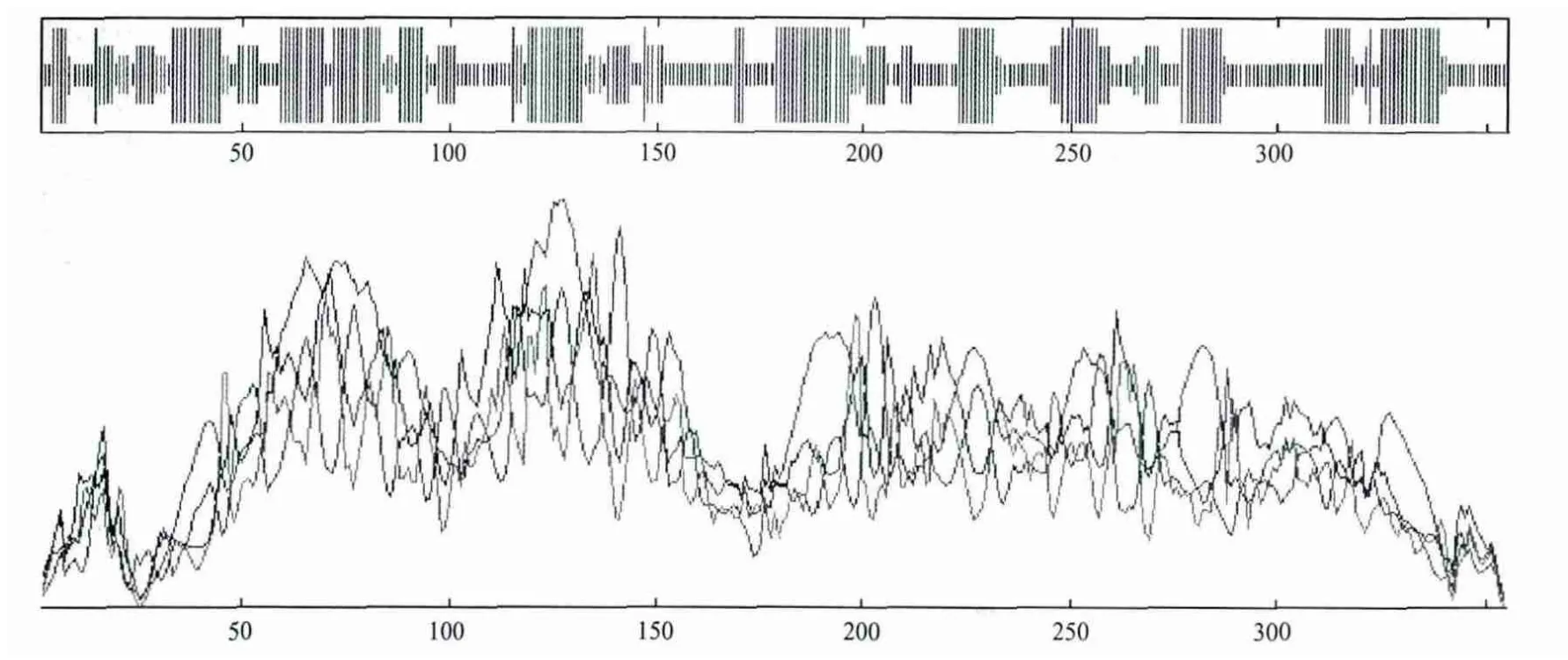
图6 GhDFR蛋白的二级结构预测图

图7 GhDFR 蛋白的结构域分析

图8 GhDFR与其他高等植物 DFR蛋白的进化树分析
花色苷合成的类黄酮途径是研究的最为清楚的植物次生代谢途径之一[7],类黄酮类化合物是植物花青素的主要成分,是花卉、果实、种皮重要的成色物质。DFR即二氢黄酮醇4-还原酶(dihydroflavonol 4-reductase)属于细胞色素P450家族,是类黄酮/花青素生物合成途径的一个关键酶,在类黄酮途径中,DFR催化二氢黄酮醇,如二氢堪非醇(dihydrokaempferol,DHK)、二氢槲皮素(dihydroquercetin,DHQ)和二氢杨梅素(dihydromyricetin,DHM),在C4位发生立体特异的还原反应,分别生成无色天竺葵素、无色矢车菊素和无色飞燕草素(翠雀素),这些产物都是合成有色的花色苷类物质的前体物质[7,13]。从O’Reilly第一次克隆得到DFR基因开始,利用同源克隆的方法分别在矮牵牛(Petunia hybrida)、拟南芥(Arabidopsis thaliana)、金鱼草(Antirrhinum majus)、番茄(Lycopersicon esculentum)、康乃馨(Dianthus caryophyllus)、葡萄(Vitis vinifera)、草莓(Fragaria ananassa)、玉米(Zea mays)等物种中都已经克隆出来,其分子特征和调控机制已在很多植物中得到深入研究,研究发现来自不同物种的DFR有非常高的同源性,但是不同植物的DFR结合底物的特异性有所不同。在矮牵牛中,DFR优先转化DHM生成无色翠雀素,其次是DHQ,不能转化DHK,因此矮牵牛中缺乏天竺葵色素,将玉米的DFR基因A1转化矮牵牛,A1能够催化DHK生成无色天竺葵素,进而在花青素合成酶(ANS)的作用下生成桔红色的天竺葵素,矮牵牛花色由淡红色变为桔红色[14]。关于底物选择特异性的分子机理,Beld等[15]根据对矮牵牛DFR的研究提出了一个由26个氨基酸组成的底物特异性结合区域,该区域的氨基酸序列决定DFR对底物的特异性结合。随后,Johnson等[16]利用DFR单氨基酸突变体证明改变底物特异性结合区域的某些氨基酸能够改变矮牵牛DFR的底物特异性,而有些位置的氨基酸具有高度保守性,并确定了与底物特异性直接相关的几个氨基酸残基,如133位、134位、142位和145位,其中第134位氨基酸直接决定底物特异性,矮牵牛DFR第134位是Asp,其最佳底物为DHM,对DHQ的还原效率很低,不能以DHK为底物生成天竺葵素,其他134位是Asn的植物都能以DHK为底物。将矮牵牛134位的Asp替换为Leu,则DFR从只以DHQ和DHM为底物变为优先转化DHK,而不能有效转化DHM。根据DFR蛋白该相应位置氨基酸残基不同,发现植物DFR分为几种类型,即Asn型、Asp型及非Asn/Asp型,在植物中Asn型DFR分布广泛,单子叶植物都是Asn型DFR,而Asp型DFR只分布在矮牵牛等部分双子叶植物中。此外,只有少数植物含有非Asn/Asp型。根据蛋白比对结果,本研究克隆得到的棉花GhDFR属于第二种类型(即Asp类型),推测其不能以DHK为底物生成无色天竺葵素,但目前还没有关于棉花中花青素组成成分的报道。
内含子是在转录后的加工中,从最初的转录产物除去的内部的核苷酸序列。内含子可能含有“旧码”,就是在进化过程中丧失功能的基因部分。正因为内含子对翻译产物的结构无意义,它比外显子累积有更多的突变。但越来越多的试验结果表明,内含子在基因的表达与调控中起着非常大的作用。拟南芥、矮牵牛和金鱼草等双子叶植物的DFR基因都由6个外显子和5个内含子组成,而且它们的内含子位置都大致相同[17]。本研究从棉花中得到的GhDFR基因的DNA序列也是由6个外显子和5个内含子组成,说明该基因在进化上具有一定的保守性,这类基因可能具有较为重要的功能。
目前关于DFR在类黄酮/花青素合成途径中的作用及相关转录调控方面已有较多研究,影响DFR基因表达的因素可分为两类:内部因素(各种转录因子的调控,如MYB、bHLH和WD40蛋白等)和外界因素(如光照、温度等环境诱导因素),但目前对彩色棉类黄酮/花青素相关方面的研究还很少。本研究从彩棉DFR基因入手,利用生物信息学手段对棉花GhDFR基因的相关蛋白性质、结构及系统进化关系等进行了初步研究,为将来研究彩色棉类黄酮/花青素生物合成以及利用基因工程技术进行彩棉种质资源的创新奠定了重要基础。
4 结论
从棕色棉中克隆到GhDFR基因的全长编码序列及内含子序列,该基因含有6个外显子,5个内含子,其编码的氨基酸序列包含具有高度保守性的NADP(H)的结合位点以及底物特异性结合位点,系统进化分析结果表明其与天竺葵、芍药的DFR亲缘关系较近。
[1] 邓福军, 陈谦, 汤振江, 张哲锋. 彩色棉的科研现状与发展前景(上)[J] . 世界农业, 2005(1):48-50.
[2] 邓福军, 陈谦, 汤振江, 张哲锋. 彩色棉的科研现状与发展前景(下)[J] . 世界农业, 2005(2):42-44.
[3] 杨伯祥, 周宜军, 王冶斌. 彩色棉主要经济性状研究[J] . 中国棉花, 1999, 26(1):9-10.
[4] 杜雄明, 石玉真. 天然彩色棉纤维特性及开发利用[J] . 针织工业, 2002(1):29-33.
[5] 孙东磊, 孙君灵, 杜雄明, 马峙英. 彩色棉种质资源农艺性状和纤维品质鉴定与分析[J] . 植物遗传资源学报, 2008, 9(4):469-474.
[6] Hua SJ, Wang XD, Zhao XQ, et al. Dynamics of carbohydrate and pigment content during fiber development in brown-colored cotton[J] . Cotton Science, 2008, 20(3):239-241.
[7] Nishihara M, Nakatsuka T. Genetic engineering of flavonoid pigments to modify flower color in floricultural plants[J] . Biotechnology Letters, 2011, 33:433-441.
[8] Xiao YH, Zhang ZS, Yin MH, et al. Cotton flavonoid structural genes related to the pigmentation in brown fibers[J] . Biochemical and Biophysical Research Communications, 2007, 358(1):73-78.
[9] Paterson AH, Brubaker CL, Wendel JF. A rapid method for extraction of cotton(Gossypiumspp.)genomic DNA suitable for RFLP or PCR analysis[J] . Plant Molecular Biology Reporter,1993, 11:122-127.
[10] Wan CY, Wilkin TA. A modified hot borate method significantly enhances the yield of high-quality RNA from cotton(Gossypium hirsutumL.)[J] . Analytical Biochemistry, 1994, 223:7-12.
[11] 梁明炜, 刘海峰, 陆雪莹, 等. 棕色棉类黄酮 3'-羟化酶基因(F3'H)的克隆及色素合成途径中相关基因表达特性研究[J] .农业生物技术学报, 2011, 19(5):808-814.
[12] 宋洋, 吴巧雯, 郭三堆. 制备棉花幼蕾高质量总RNA的方法比较[J] . 棉花学报, 2008, 20(3):231-234.
[13] Holton TA, Cornish EC. Genetics and biochemistry of anthocyanin biosynthesis[J] . The Plant Cell, 1995, 7:1071-1083.
[14] Meyer P, Heidmann I, Forkmann G, Saedler H. A new petunia flower colour generated by transformation of a mutant with a maize gene[J] . Nature, 1987, 330(6149):677-678.
[15] Beld M, Martin C, Huits H, et al. Flavonoid synthesis inPetunia hybrida:partial characterization of dihydroflavonol-4-reductase genes[J] . Plant Molecular Biology, 1989, 13:491-502.
[16] Johnson ET, Ryu S, Yi HK, et al. Alteration of a single amino acid changes the substrate specificity of dihydroflavonol 4-reductase[J] .Plant Journal, 2001, 25:325-333.
[17] Inagaki Y, Johzuka-Hisatomi Y, Mori T, et al. Genomic organization of the genes encoding dihydroflavonol 4-reductase for flower pigmentation in the Japanese and common morning glories[J] .Gene, 1999, 226:181-188.
