Cell-SELEX技术研究进展
2014-07-12赵仲麟李燕袁超
赵仲麟 李燕 袁超
(1.河南农业大学理学院,郑州 450002;2.郑州轻工业学院食品与生物工程学院,郑州 450002)
适配子是从组合核酸库中筛选到的、与靶分子具有高度亲和性的分子[1]。与抗体类似,适配子对目标分子具有高度亲和性和特异性。然而适配子还有很多额外的优点,如可以抵抗还原条件和热变性等。典型的适配子一般少于40个核苷酸,同时还要易于化学合成。另外,适配子可以在一些生理条件下,可通过反义寡核苷酸杂交使其失活。因此,可以用于设计解毒剂。适配子在分子生物学和医学已经被广泛应用[2]。适配子通过SELEX技术,即指数富集配体系统进化技术,经反复的放大筛选得到的[3,4]。这个过程中,单链核酸库与靶分子孵育,单链核酸包括DNA、RNA和修饰的核酸,库由30-100个核苷酸的随机序列组成,约含1014-1015变异体。与目标有结合活性的变异体将被回收,然后通过RT-PCR进行文库富集扩增,然后利用富集的PCR产物,通过模板链除去或体外转录重建单链库。该过程将重复几轮至20轮不等,最终找到目标适配子。
SELEX程序最早于1990年建立[3,4],适配子可与多种目标作用,包括可与大分子蛋白作用的小分子化合物。SELEX过程中使用高纯度的目标分子,一种理论研究认为复合目标也可以用于寻找特异的适配子[5]。这种观点已经通过实验得到了确认[6,7]。如果与负选择相结合进行识别,那些可与非目标结合的变异体可被除去,得到目标特异的适配子(图1)。如果SELEX过程使用活细胞,则可对活细胞的变种进行检验,称为Cell-SELEX 技术。本文对Cell-SELEX 技术的发展进行综述。
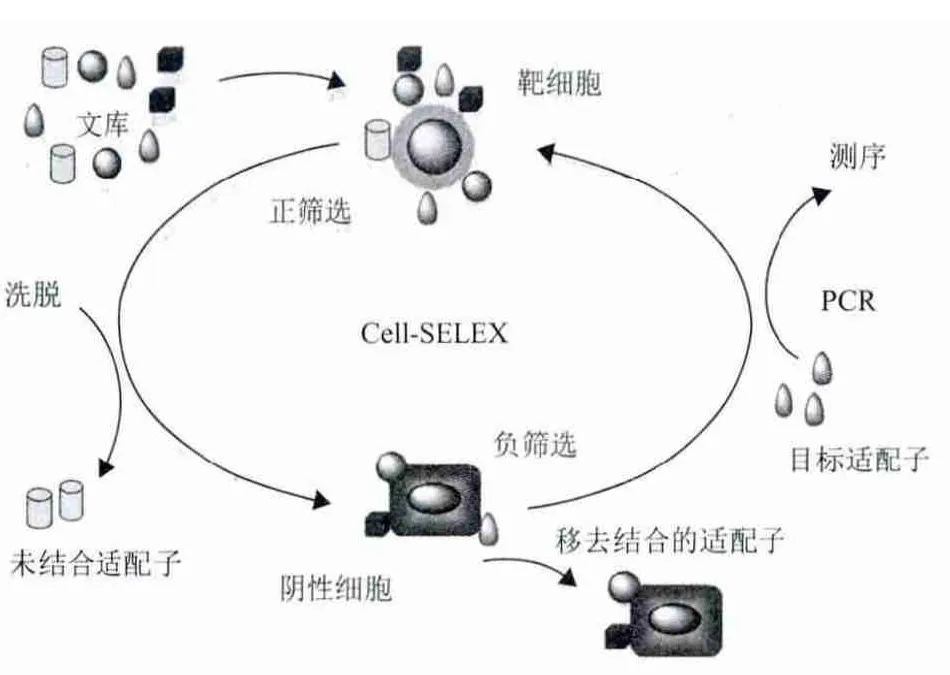
图1 Cell-SELEX
1 Cell-SELEX用于活病原微生物适配子筛选
适配子可以用于开发诊断试剂和药物,而且活的病原微生物变种也可以作为Cell-SELEX技术的操作对象[7]。最早Cell-SELEX技术的应用例子是使用活病原微生物,非洲布氏锥虫Trypanosoma brucei。T.brucei是单细胞原生动物寄生虫,有两个寄主,昆虫和哺乳动物寄主。寄生虫可以引起非洲昏睡病,一种人类慢性病和牲畜那加那病。该寄生虫的一个特点是其表面具有被称为可变表面糖蛋白(VSG)构成的保护壳,可逃脱宿主的免疫系统。Homann等[8]利用SELEX技术,将血液中活的布氏锥虫作为目标,筛选到了几个与寄生虫具有高度亲和力的RNA适配子。同时他们还发现得到的适配子能结合到VSG变种上。他们利用氟代嘧啶修饰的RNA库,通过SELEX技术与VSG变异体VSG117作用,然后再与活布氏锥虫稳定表达的VSG变异体作用。经过若干轮SELEX后,纯化得到一个不同的变异体VSG221,最后再与寄生虫作用。筛选得到的适配子显示出了与变异体高亲和力。
Ulrich等[9]使用SELEX策略,以另一种活的寄生虫热带南美洲锥虫Trypanosoma cruzi为目标筛选适配子。T. cruzi可引起chagas巨食管症,是一种人类的致命疾病。现已知寄生虫表面与宿主细胞基质分子进行相互作用,这恰巧与寄生虫侵染相关。研究者想找到能与基质分子竞争,并可与寄生虫结合的适配子。他们将T. cruzi与RNA库孵育,然后通过添加过量的基质分子,如纤维连接蛋白、粘连蛋白、硫酸乙酰肝素、血管内皮抑制因子等,来特异的洗脱目的适配子。接下来他们使用短膜虫期的寄生虫,这个阶段属于非传染阶段,利用负筛选除去不需要的适配子。最终成功的得到了能与目标寄生虫作用的适配子,最后通过体外侵染试验证实这些适配子具有抑制活性。
2 癌细胞特异性适配子筛选
由于适配子可以识别多种特定的哺乳动物细胞,因此,可以将其作为诊断和基础研究的分子探针。癌细胞是最广泛的检测目标,目前已发现多种适配子可以与癌细胞进行相互作用[10,11]。通过荧光标记,适配子可以作为癌症检测探针。Shangguan等[12]报道了一系列DNA适配子,可作为分子探针用于癌症研究。他们使用人急性淋巴细胞白血病T淋巴细胞(CCRF-CEM)和人源Burkitt淋巴瘤B细胞系分别进行正负筛选。使用几种癌细胞和经蛋白酶处理的CCRF-CEM细胞进行结合分析试验,结果显示每种适配子只识别一种特定的细胞表面分子。目前,已通过Cell-SELEX技术筛选到多种用于检测癌细胞的适配子,其中包括淋巴细胞白血症、粒细胞性白血病、肝癌、小细胞肺癌和非小细胞肺癌。这些适配子对靶细胞都具有较高的特异性[10],这些结果均表明在不知道任何细胞表面蛋白种类和数量的情况下,也可通过该技术筛选适配子。
Cell-SELEX技术也用于体细胞和胚胎干细胞研究中[13]。Iwagawa等[14]利用RNA库,以胚胎干细胞为目标进行筛选,得到的适配子能与胚胎干细胞高效结合。此外,鼠胚胎干细胞的分化过程也可以通过荧光标记的适配子进行监控。干细胞研究对于再生医学和发育生物学非常重要,因此开发新的、可用于干细胞研究的分子探针是及其必要的。
此外,Cell-SELEX技术也可以筛选一些能与病毒感染的细胞进行特异结合的DNA适配子。病毒感染后,宿主细胞表面被病毒蛋白所修饰,这些变化正好可作为病毒特异性目标,通过设计特异性分子探针来识别被病毒感染的细胞。Tang等[15]通过Cell-SELEX方法,以牛痘病毒感染的腺癌上皮细胞A549为目标,结合负筛选过程,成功得到特异性DNA适配子。分离得到的适配子可结合到几种被病毒感染的细胞系,证实适配子能识别宿主细胞表面的病毒蛋白。
3 生物标记物筛选
Cell-SELEX可以用于靶细胞新生物标记物的识别。Blank等[16]报道利用Cell-SELEX技术进行大鼠YPEN-1内皮细胞和小鼠N9噬神经细胞的正负筛选。许多筛选到的适配子能结合到胶质母细胞瘤的病理性微管上,其中一个适配子只在实体瘤生长区域显示出与脉管非常强的结合力,然后他们将该适配子的靶蛋白通过质量指纹图谱和多肽测序进行分析,最终发现该蛋白是内皮分化的调控蛋白,在肿瘤的微血管中过表达。Berezovski等[17]发展出多种系统程序用于目标细胞的新生物标记物的识别,他们将该程序命名为 AptaBiD,包括3步:首先,通过Cell-SELEX技术构建适配子库;接下来利用适配子库的结合活性,亲和分离候选的生物标记物。最后,通过肽质量指纹图谱鉴定分离到的候选蛋白。Shoji等[18]构建了DNA适配子库,利用Cell-SELEX技术作用于未成熟和成熟的树突细胞,使用生物素标记的DNA库,从目标细胞裂解液中分离候选的生物标记物,通过LC-MS分析经胰蛋白酶处理的候选蛋白,最终分别从未成熟的细胞中得到6个,从成熟的细胞中得到3个生物标记物,其中6个是以前未知的生物标记物。
4 基于SELEX技术的细胞摄取
Cell-SELEX 筛选得到的适配子不仅可以结合靶细胞,还可以进行胞内运输。例如从活的T. brucei细胞筛选的一个适配子能够通过胞吞作用迅速内化,通过小泡输送转运到溶酶体[7,10]。适配子这种内化特性可以使其进行胞内的药物传递[19],利用适配子介导的短RNA干扰已被试验证实[20,21]。为产生能介导向目标特异细胞进行胞内药物运输的适配子,Wu等[22]开发出一套独特的SELEX策略,正是基于胞内运输。他们利用一个随机库与靶细胞孵育,通过严苛条件(0.2 mol/L glycine-HCl,pH4.0)洗脱,而后将这些洗脱的变异体合并,通过加入蛋白酶和清洗剂细胞裂解,进行选择性回收。应用这种方法得到的DNA基元可以高效的运输到人慢性淋巴细胞白血病B细胞[23]。相同的策略也被用于生产tRNA衍生物,可被分离的线粒体吸收[24]。这些RNA基元可以被构建成与线粒体疾病治疗相关的新载体。
对于用于胞内药物投递的适配子,不仅要细胞摄取效率高,而且对于其吸收、分布、代谢和排泄等药代动力学参数有一定的限制。Mi等[25]发展出基于SELEX的细胞摄取,用于活肿瘤动物模型的体内筛选。他们将随机的RNA库静脉注射进肝癌移植小鼠中,循环20 min后,收集肝癌细胞后提取肿瘤细胞中的变异体,然后进行扩增,进而进行下一轮的注射。发现一个变异体对于肿瘤细胞的粗提物显示出增强的亲和力,而对正常的结肠细胞无亲和性。然后他们将荧光标记的适配子进行尾部静脉注射发现,适配子只定植于肝癌细胞,从而证实了该程序的可行性。最终的肽质量指纹图谱鉴定分析结果显示,得到的适配子能够识别RNA解旋酶p68。
5 给药系统开发
细胞结合适配子可以用于研发目标细胞特异性分子探针和进行药物投递系统的开发。Douglas等[26]使用DNA适配子进行药物给药研究。他们将药物分子,主要是可与细胞表面结合的抗体,包裹在一个三维DNA纸盒(DNA origami box)中,适配子与其反义的DNA寡聚物杂交使盒子关闭。当细胞上的目标分子与适配子结合后,适配子构象改变使杂交解链,从而打开盒子,暴露出药物。只有当细胞展示适配子的靶分子时,药物才会暴露出来,这是一种高度特异性给药系统。通过Cell-SELEX 技术得到的适配子在该系统中作为开关而存在。
另外,与细胞结合适配子也可用于开发细胞操作系统。例如,适配子固定的树脂可被用于特定目标细胞的回收[27,28]。利用适配子的优点是它们可以在温和条件下,如反义杂交和二价阳离子的螯合作用下进行可逆失活。因此,可以在温和条件下使用树脂反复分离结合的细胞。同时,细胞特异性适配子还能够用于特定细胞的粘附[29,30]。Schroeder等[31]开发出一个活细胞芯片,由肽配体和DNA芯片组成。靶细胞结合的肽配体与DNA寡聚物连接,通过杂交排列在DNA芯片上,当细胞加入芯片上后,靶细胞可通过与排列的肽配体亲和附着在特定位置上。Gartner和Bertozzi 将多种类型的细胞进行空间排布,通过DNA杂交固定到细胞表面,这种细胞操作系统可用于细胞通讯研究[32]。
6 展望
适配子凭借其独特的性质已应用于生物学和医学研究中,利用Cell-SELEX技术,筛选得到的可与细胞结合的适配子应用程序为人们发展新的医药和生物技术打开了一扇新的大门,其作用是传统的生物亲和物抗体所不能实现的。Cell-SELEX技术提供一种高效的生产大量适配子的方法,这些适配子可作为探针特异性锚定各种癌细胞。适配子已用于癌细胞生物学研究的多个领域中,未来的研究也会演化更多的研究方向,如位点特异性化学修饰可使适配子与金纳米颗粒或量子点交联,使用比色或荧光的方法检测癌细胞。由于具有较高的灵敏度,因此可以用于癌症的早期诊断。同时,通过适配子对癌细胞的高度特异性,可以用于药物靶向试剂的研发,从而得到低毒、高效的治疗性药物。另外,使用Cell-SELEX筛选的适配子可以钓取目的蛋白,用于新癌症生物学标记物的发现。同时,也可作为调控子抑制或激活某些信号途径从而进行癌症发育研究。这种基于适配子的化学生物学方法也可以用于其他疾病的研究。不同的适配子可以特异的与各种不同类型的疾病细胞结合,未来的个性化用药将成为现实。
[1] Klussmann S. The Aptamer handbook:functional oligonucleotides and their applications[M] . Weinheim:Wiley-VCH, 2006.
[2] Stoltenburg R, Reinemann C, Strehlitz B. SELEX-a(r)evolutionary method to generate high-affinity nucleic acid ligands[J] . Biomol Eng, 2007, 24:381-403.
[3] Ellington A, Szostak JW.In vitroselection of RNA molecules that bind specific ligands[J] . Nature, 1990, 346:818-822.
[4] Tuerk C, Gold L. Systematic evolution of ligands by exponential enrichment:RNA ligands to bacteriophage T4 DNA polymerase[J] .Science, 1990, 249:505-510.
[5] Vant-Hull B, Payano-Baez A, Davis RH, et al. The mathematics of SELEX against complex targets[J] . J Mol Biol, 1998, 278:579-597.
[6] Morris KN, Jensen KB, Julin CM, et al. High affinity ligands fromin vitroselection:complex targets[J] . Proc Natl Acad Sci USA,1998, 95:2902-2907.
[7] Homann M, Goringer HU. Combinatorial selection of high affinity RNA ligands to live African trypanosomes[J] . Nucleic Acids Res,1999, 27:2006-2014.
[8] Lorger M, Engstler M, Homann M, et al. Targeting the variable surface of African trypanosomes with variant surface glycoproteinspecific, serum-stable RNA aptamers[J] . Eukaryot Cell, 2003, 2:84-94.
[9] Ulrich H, Magdesian MH, Alves MJ, et al.In vitroselection of RNA aptamers that bind to cell adhesion receptors ofTrypanosoma cruziand inhibit cell invasion[J] . J Biol Chem, 2002, 277:20756-20762.
[10] Zhang Y, Chen Y, Han D, et al. Aptamers selected 1 by cell-SELEX for application in cancer studies[J] . Bioanalysis, 2010, 2:907-918.
[11] Phillips JA, Lopez-Colon D, Zhu Z, et al. Applications of aptamers in cancer cell biology[J] . Anal Chim Acta, 2008, 621:101-108.
[12] Shangguan D, Li Y, Tang Z, et al. Aptamers evolved fromlive cells as effective molecular probes for cancer study[J] . Proc Natl Acad Sci USA, 2006, 103:11838-11843.
[13] Guo KT, Schafer R, Paul A, et al. Aptamer-based strategies for stem cell research[J] . Mini Rev Med Chem, 2007, 7:701-705.
[14] Iwagawa T, Ohuchi SP, Watanabe S, et al. Selection of RNA aptamers against mouse embryonic stem cells[J] . Biochimie,2012, 94:250-257.
[15] Tang Z, Parekh P, Turner P, et al. Generating aptamers for recognition of virus-infected cells[J] . Clin Chem, 2009, 55:813-822.
[16] Blank M, Weinschenk T, Priemer M, et al. Systematic evolution of a DNA aptamer binding to rat brain tumor microvessels. Selective targeting of endothelial regulatory protein pigpen[J] . J Biol Chem, 2001, 276:16464-16468.
[17] Berezovski MV, Lechmann M, Musheev MU, et al. Aptamerfacilitated biomarker discovery(AptaBiD)[J] . J Am Chem Soc,2008, 130:9137-9143.
[18] Shoji O. Cell-SELEX technology[J] . Bio Research Open Access,2012, 1(6):265-272.
[19] Farokhzad OC, Jon S, Khademhosseini A, et al. Nanoparticleaptamer bioconjugates:a new approach for targeting prostate cancer cells[J] . Cancer Res, 2004, 64:7668-7672.
[20] Chu TC, Twu KY, Ellington AD, et al. Aptamer mediated siRNA delivery[J] . Nucleic Acids Res, 2006, 34:e73.
[21] McNamara JO, Andrechek ER, Wang Y, et al. Cell type-specific delivery of siRNAs with aptamer-siRNA chimeras[J] . Nat Biotechnol, 2006, 24:1005-1015.
[22] Wu CC, Castro JE, Motta M, et al. Selection of oligonucleotide aptamers with enhanced uptake and activation of human leukemia B cells[J] . Hum Gene Ther, 2003, 14:849-860.
[23] Mende M, Hopert A, Wunsche W, et al. A hexanucleotide selected for increased cellular uptake in cis contains a highly active CpG-motif in human B cells and primary peripheral blood mononuclear cells[J] . Immunology, 2007, 120:261-272.
[24] Kolesnikova O, Kazakova H, Comte C, et al. Selection of RNA aptamers imported into yeast and human mitochondria[J] . RNA,2010, 16:926-941.
[25] Mi J, Liu Y, Rabbani ZN, et al.In vivoselection of tumor-targeting RNA motifs[J] . Nat Chem Biol, 2010, 6:22-24.
[26] Douglas SM, Bachelet I, Church GM. A logic-gated nanorobot for targeted transport of molecular payloads[J] . Science, 2012,335:831-834.
[27] Smith JE, Medley CD, Tang Z, et al. Aptamer-conjugated nanoparticles for the collection and detection of multiple cancer cells[J] . Anal Chem, 2007, 79:3075-3082.
[28] Schafer R, Wiskirchen J, Guo K, et al. Aptamer-based isolation and subsequent imaging of mesenchymal stem cells in ischemic myocard by magnetic resonance imaging[J] . Rofo, 2007, 179:1009-1015.
[29] Guo K, Wendel HP, Scheideler L, et al. Aptamer-based capture molecules as a novel coating strategy to promote cell adhesion[J] .J Cell Mol Med, 2005, 9:731-736.
[30] Guo KT, Schafer R, Paul A, et al. A new technique for the isolation and surface immobilization of mesenchymal stem cells from whole bone marrow using high-specific DNA-aptamers[J] . Stem Cells,2006, 24:2220-2231.
[31] Schroeder H, Ellinger B, Becker CF, et al. Generation of live-cell microarrays by means of DNA-directed immobilization of specific cell surface ligands[J] . Angew Chem Int Ed Engl, 2007, 46:4180-4183.
[32] Gartner ZJ, Bertozzi CR. Programmed assembly of 3-dimensional microtissues with defined cellular connectivity[J] . Proc Natl Acad Sci USA, 2009, 106:4606-4610.
