一株宽宿主谱裂解性大肠杆菌噬菌体的进化分析
2014-04-15包红朵张莉莉
周 艳,包红朵,张 辉,张莉莉,王 冉
(江苏省农业科学院食品质量安全与检测研究所 江苏省畜禽产品安全性研究重点实验室 江苏省食品质量安全重点实验室 省部共建国家重点实验室培育基地,南京 21004)
一株宽宿主谱裂解性大肠杆菌噬菌体的进化分析
周 艳,包红朵,张 辉,张莉莉,王 冉
(江苏省农业科学院食品质量安全与检测研究所 江苏省畜禽产品安全性研究重点实验室 江苏省食品质量安全重点实验室 省部共建国家重点实验室培育基地,南京 21004)
裂解性噬菌体能够特异性裂解细菌,本研究分析了一株从自然界中分离的宽宿主谱裂解性大肠杆菌噬菌体的种属和进化关系。前期用Adams双层平板琼脂法从猪场污水中分离纯化出一株裂解性噬菌体vB_EcoM_JS09,裂解谱分析能够裂解禽致病性大肠杆菌和产肠毒素大肠杆菌。扩增分析gene18、gene 23序列,并根据gp18和gp23氨基酸序列构建系统进化树,分析JS09的遗传进化关系。结果表明噬菌体vB_EcoM_JS09基因组为dsDNA,属于有尾噬菌体目、肌尾噬菌体科、T4-like噬菌体、T-evens亚群。其gp18氨基酸序列与大肠杆菌噬菌体RB69同源性最高,为100%;gp23氨基酸序列与大肠杆菌T4噬菌体同源性最高为96%。该结果为禽致病性大肠杆菌和产肠毒素大肠杆菌的防控提供了生物学基础。
禽致病性大肠杆菌;产肠毒素大肠杆菌;宽宿主谱裂解性大肠杆菌噬菌体;T4-like噬菌体;进化分析
噬菌体(bacteriophage)广泛存在于自然界,是数量和种类最多的生物体[1-5]。噬菌体能够特异性的裂解并杀死细菌,分离并研发其作为新型抗菌生物制剂已成为各国研究的热点之一[4-8]。研究较多较详细,并成为模式噬菌体的是T偶数噬菌体,如T2、T4和T6噬菌体[3,9,10]。近年来人们逐渐分离并研究了很多T4-like噬菌体。T4-like噬菌体代表为T4噬菌体,基因组为dsDNA,属于有尾噬菌体目、肌尾噬菌体科。基于基因序列比对,可把T4-like分为四个亚群:T-evens亚群、pseudo T-even亚群、schizo T-even亚群和exo T-even亚群[3]。T-even亚群中的噬菌体分离自肠道菌,其DNA序列与T4噬菌体相似性较高。然而有研究发现T4-like噬菌体的种类较多,超出了这几个亚群分类[11]。T4-like噬菌体具有一个较长多面体头部,长且可收缩的尾部,形态以及基因组大小接近于T4噬菌体[3,9]。尽管T4-like噬菌体之间有差异,它们在遗传进化上具有亲缘关系,如衣壳蛋白、尾蛋白和DNA复制酶基因中仍保留了的保守同源区域,其中gene 18和gene 23与T4噬菌体存在同源性较高序列[3]。因此,在本研究中将这两个基因为基准进行序列比对和噬菌体的进化分析。
禽致病性大肠杆菌(Avain pathogenicE.coli,APEC)是一类致禽类急性的、全身性疾病的肠道外的病原性大肠杆菌,禽类感染后主要表现为气囊炎、肺炎、心包炎、肝周炎、腹膜炎、输卵管炎等,或表现为急性败血症、肿头综合症或雏鸡脐炎,给世界范围内的畜禽养殖业带来了巨大经济损失,禽致病性大肠杆菌也是重要的人畜共患病原菌[12]。产肠毒素大肠杆菌(EnterotoxigenicE.coli,ETEC)是一类能够导致人和幼畜如初生仔猪等腹泻最常见的病原性大肠杆菌[13]。宽宿主谱大肠杆菌裂解性噬菌体vB_EcoM_JS09(简称JS09),用Adams双层平板琼脂法[14]分离自江苏省射阳市某猪场污水中,经鉴定能够裂解禽致病性大肠杆菌和产肠毒素大肠杆菌。对其gp18和gp23氨基酸序列进行遗传进化分析,鉴定JS09为一株T4-like噬菌体。本研究对这一株新分离的宽宿主谱大肠杆菌噬菌体的种属和进化关系进行了分析,为禽致病性大肠杆菌和产肠毒素大肠杆菌的防控提供了生物学基础。
1 材料和方法
1.1 菌种和噬菌体样品来源禽致病性大肠杆菌菌株和产肠毒素大肠杆菌均分离自病鸡肝脏;宽宿主谱裂解性大肠杆菌噬菌体JS09分离自猪场污水(GenBank登录号:KF582788),均由本实验室保存。
1.2 引物和主要试剂DNaseI、RNaseA和MungBeanNuclease购自Sigma生物工程技术服务有限公司;蛋白酶K购自Amresco公司;引物由上海生工公司合成;EcoRI、HindIII、DNAMarker、LATaq酶和PCR所用试剂均购自TaKaRa公司;T4连接酶购自上海生工生物工程技术服务有限公司;胶回收试剂盒和质粒提取试剂盒购自广州东盛生物科技有限公司;酵母提取物、胰蛋白胨、NaCl、PEG8000、琼脂糖、氨苄青霉素等化学试剂为进口和国产分析纯。
1.3 噬菌体的纯化和基因组的提取扩增培养JS09噬菌体至104pfu/mL,APEC菌株浓度至109cfu/mL。按照1:10比例接种噬菌体和菌株,混合至锥形瓶,室温静置吸附15min,加入40mLLB液体培养基,37℃过夜振荡培养。将含有噬菌体JS09的裂解培养物冷却至室温,PEG8000沉淀法纯化培养物中的噬菌体颗粒,保存于SM缓冲液(NaCl5.8g、MgSO4·7H2O2g、pH7.5、Tris·Cl1mol/L、2%gelatin5mL,ddH2O补足至1L)中[15-17]。苯酚/氯仿/异丙醇法提取SM中噬菌体的基因组,单酶切鉴定噬菌体基因组,10μL酶切体系为EcoRI/HindIII1μL、10×bufferH1μL、基因组模板(10倍稀释)5μL,补足ddH2O至10μL,涡旋混匀后37℃酶切3h。用DNaseI、RNaseA和MungBeanNuclease酶切处理鉴定基因组类型[15-17]。
1.4 噬菌体gene18与gene23的扩增参照文献[4]引物,分别扩增噬菌体gene18与gene23序列(表1)。以噬菌体JS09基因组DNA为模板。PCR反应体系:10×PCRbuffer5μL,dNTPmix(2.5mM)4μL,MgCl25μL,上下游引物各1μL,基因组模板(10倍稀释)2μL,补足灭菌ddH2O50μL。PCR程序:94℃预热4min;94℃变性30s,60℃退火30s,72℃延伸1min,30个循环;72℃延伸10min。用胶回收试剂盒纯化回收目的片段,送交上海生工生物工程技术服务有限公司测序。

表1 gene 18和gene 23扩增所用引物Table 1 Primers used in PCR of gene 18 and gene 23
1.5 噬菌体遗传进化分析在NCBI分别获取11株噬菌体RB32、RB69、T4、RB49、RB42,RB16、RB43、KVP40、Aeh1、nt-1、vB_EcoM_CBA120的gp18氨基酸序列,用DNAStar将测得的JS09的gene18核苷酸序列转换为氨基酸序列。将这11株序列与测得的JS09的gp18氨基酸序列用DNAStar中的MegAlign软件进行gp18氨基酸序列比对,TreeView1.6 .6软件构建噬菌体系统进化树。
2 结果与讨论
2.1 噬菌体JS09的分离和其基因组的特点EcoR I和Hind III分别单酶切纯化的噬菌体JS09基因组,1%琼脂糖凝胶电泳表明提取出了JS09基因组(图1)。经DNase I、RNase A和Mung Bean Nuclease酶切处理后,电泳结束表明基因组为双链DNA。除大肠杆菌T4噬菌体之外,近年来已发现了很多T4-like噬菌体。据报道在电镜观察下,已分离到了5500株噬菌体,但仅获得了555株噬菌体的基因组全序列,其中肠杆菌噬菌体仅约为70株[1]。随着从自然界分离的噬菌体种类的增多,对噬菌体的形态、生物学特性和基因的了解已不再局限于几种模式噬菌体[18]。抗生素的滥用产生了许多危害,抗药性细菌给人类健康和畜禽养殖业带来了危害以及巨大的经济损失,分离鉴定能够裂解病原菌的噬菌体成为首要解决的问题。因此分离新的噬菌体,建立不同噬菌体之间的进化关系,有助于鉴定一株新分离的噬菌体,也为进一步研究噬菌体的生物多样性和进化变异提供了途径[18-20]。
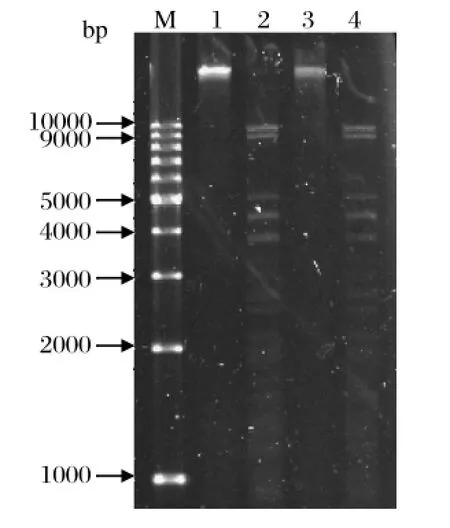
图1 JS09基因组单酶切鉴定Fig.1 Identif cation of JS09 genome digested by resitriction endonucleases enzyme
2.2 gene 18与gene 23的扩增和序列比对结果以纯化的噬菌体JS09基因组为模板,两对引物分别扩增出目的片段gene 18和gene 23,与预期片段大小一致,分别约为1330 bp和850 bp(图2)。在本研究中,基于gene 18和gene 23保守序列扩增,将所测的序列结果,通过NCBI Blast搜索同源性较高的序列,共有9株噬菌体的gene 18核苷酸序列与JS09同源性较高,为76%~98%,其中与大肠杆菌噬菌体HX01核苷酸同源性最高,为98%。将获得的部分gene 18核苷酸序列转换为333个氨基酸序列后,与大肠杆菌噬菌体RB69同源性最高,为100%;而S09 gene 23核苷酸序列与大肠杆菌噬菌体HX01核苷酸同源性最高,为99%,将获得的部分gene 23核苷酸序列转换为264个氨基酸序列后,与大肠杆菌噬菌体T4同源性最高,为96%。参考GenBank上已公布的噬菌体基因组序列和文献中的噬菌体形态,噬菌体JS09的gene 18和gene 23核苷酸序列与大肠杆菌噬菌体HX01同源性最高,HX01属于有尾噬菌体目、肌尾噬菌体科、T4-like噬菌体。HX01的宿主菌也为禽致病性大肠杆菌,该噬菌体分离自鸭场[21],而本研究中JS09分离自猪场污水。根据已报道的文献,噬菌体RB69、T4与vB_EcoM_JS09宿主谱不同,RB69的宿主为E.coliB/5[10],T4的宿主为E.coliK-12株系[23]。然而尚未有报道表明RB69和T4噬菌体能够裂解禽致病性大肠杆菌和产肠毒素大肠杆菌。
在随后的全基因组测序研究中,JS09的gp18蛋白即gp18 tail sheath protein 含有660个氨基酸,其氨基酸序列与大肠杆菌RB69同源性为100%,属于蛋白家族18 superfamily。JS09的gp23蛋白即major capsid protein含有155个氨基酸,其氨基酸序列与大肠杆菌RB69同源性为100%,属于蛋白家族gp23。在T4-like噬菌体中,18 superfamily 和gp23均为保守的蛋白结构域,属于T4-like噬菌体。这表明JS09的gene 18和gene 23在遗传进化上很保守。而在分类上噬菌体HX01、T4噬菌体和噬菌体RB69都属于有尾噬菌体目,肌尾噬菌体科、T4-like噬菌体、T-evens亚群,基因组均为双链DNA。
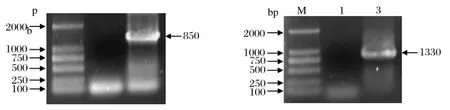
图2 gene 18和gene 23的PCR扩增结果Fig. 2 PCR amplif cation results of gene 18 and gene 23
2.3 JS09遗传进化分析基于gp18氨基酸序列构建系统进化树(图3),JS09与T4噬菌体、RB69关系较近;而基于gp23氨基酸序列构建的系统进化树与之基于gp18构建的(图省略)。本研究中新分离的JS09属于有尾噬菌体目、肌尾噬菌体科、T4-like噬菌体、T-even亚群。T-even亚群与T4噬菌体在基因组序列上具有较高相似性,而pseudo T-even亚群、schizo T-even亚群和exo T-even亚群与T4噬菌体在序列相似性上较低。与T4噬菌体比对,T-even亚群、pseudo T-even亚群、schizo T-even亚群和exo T-even亚群gp23氨基酸序列相似性分别为90%、60%、50%和30%。研究表明RB69与T4噬菌体的基因组结构图谱大部分相对应,且大多数基因接近[11]。因此对于RB69以及T4噬菌体的研究有助于改造或克隆JS09相关基因,从而将JS09应用于致病性大肠杆菌病的防治上。
结合我们对JS09的电镜形态观察,以及对其高通量测序获得的全基因组序列(GenBank登录号:KF582788)比对分析表明,基于gene 18和gene 23序列对JS09的分类是可靠的。虽然在自然界的进化变异中,噬菌体形态、宿主特异性和基因组也在发生演变[22]。但在这些T4-like噬菌体中,仍保留着高度保守的基因序列。因此在噬菌体电镜形态观察的基础上,基于保守的gene 18和gene 23序列,可以初步鉴定一株T4-like噬菌体。在第九次国际病毒分类委员会上,将RB69归为未界定的T4-like噬菌体,但在国外报道的文献中将RB69作为T4-like噬菌体来研究,因此JS09至少是与噬菌体T4相关的噬菌体。
目前已知的大部分T4-like噬菌体分离自肠道菌,这些噬菌体主要属于T-evens亚群。在T4-like噬菌体中,T4噬菌体作为模式噬菌体,研究最为详细。在自然界分离的一些噬菌体,由于在形态和基因组序列上与T4噬菌体相似,故称为T4-like噬菌体[9]。目前仅得到了约40株T4-like噬菌体的基因组序列[9]。研究认为,T4-like噬菌体来源于T4噬菌体。而本研究中,JS09株形态和基因组大小类似于T4噬菌体,因此参照已有的模式噬菌体,有助于研究JS09噬菌体与宿主之间的相互作用、抗性机制以及进化关系等。
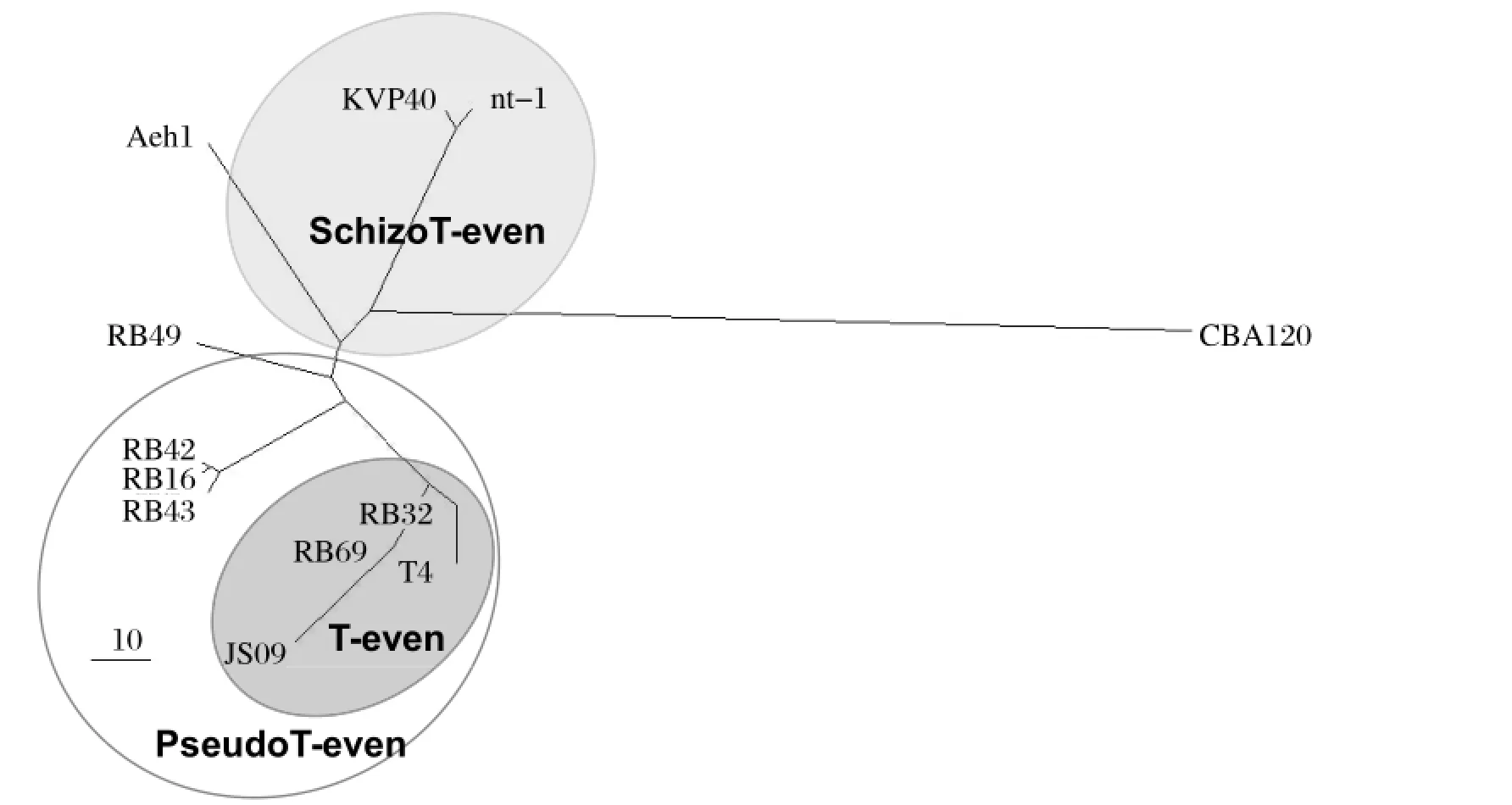
图3 基于gp18氨基酸序列构建的系统进化树Fig. 3 Phylogenetic tree constructed by the TreeView1.6.6 program using the amino sequence of gp18 from 12 T4-like phages
[1] Li S, Liu L, Zhu J, et a1. Characterization and genome sequencing of a novel coliphage isolated from engineered Escherichia coli[J]. Intervirology, 2010, 53(4): 211-220.
[2] Liao W C, Ng W V, Lin I H, et a1. T4-like genome organization of the Escherichia coli O157: H7 lytic phage AR1[J]. J Virol, 2011, 85(13): 6567-6578.
[3] Tétart F, Desplats C, Kutateladze M, et a1. Phylogeny of the major head and tail genes of the wide-ranging T4-type bacteriophages[J]. J Bacteriol, 2001, 183(1): 358-366.
[4] Nho S W, Ha M A, Kim K S, et a1. Complete genome sequence of the bacteriophages ECBP1 and ECBP2 isolated from Two different Escherichia coli strains[J]. J Virol, 2012, 86(22): 12439-12440.
[5] 姜焕焕, 王盛, 李存, 等. 一株新分离的肠杆菌噬菌体的快速遗传鉴定及其宿主识别基因的快速克隆[J]. 生物工程学, 2011, 27(6): 884-890.
[6] Collins J J, Lu T K T. Engineered bacteriophages as adjuvants for antimicrobial agents and compositions and methods of use thereof: WIPO Patent Application PCT/ US2009/030755[P]. 2009-1-12.
[7] Fenton M, Keary R, McAuliffe O, et a1. Bacteriophage-Derived Peptidase Eliminates and Prevents Staphylococcal Biofilms[J]. Int J Microbiol, 2013: 625341.
[8] Lu T K, Collins J J. Engineered bacteriophage targeting gene networks as adjuvants for antibiotic therapy[J]. Proc Natl Acad Sci USA, 2009, 106(12): 4629-4634.
[9] Petrov V M, Ratnayaka S, Nolan J M, et a1. Genomes of the T4-related bacteriophages as windows on microbialgenome evolution[J]. Virol J, 2010, 7: 292-292.
[10] Driukas A, Nivinskas R. Substitution in vivo of the holin inhibitor RI of bacteriophage T4 by the homologous protein of T-even phage RB69 [J]. Biologija, 2009, 55(1): 14-17.
[11] Kutter E, Sulakvelidze. A. Bacteriophages: biology and applications[M]. Boca Raton, FL, USA: CRC Press, 2004.
[12] 马兴树, 范翠蝶, 夏玉龙. 禽致病性大肠杆菌研究进展.中国畜牧兽医[J]. 2013, 40(2): 169-174.
[13] 陆承平, 兽医微生物学第[M]. 5版. 中国农业出版社, 2013.
[14] Adams M H. Bacteriophages[M]. New York: Interscience Publishers, 1959.
[15] 王冉, 韩晗, 张辉, 等. 大肠杆菌 K88 噬菌体的分离鉴定及其生物学特性[J]. 华北农学报, 2012, 27(4): 163-167.
[16] Sambrook J, Lasel D W. 分子克隆实验指南[M]. 黄培堂,等译. 3 版. 北京: 科学出版社, 2002: 147-243.
[17] Jamalludeen N, Johnson R P, Friendship R, et a1. Isolation and characterization of nine bacteriophages that lyse O149 enterotoxigenie Escherichia coli [J]. Vet Microbiol, 2007, 124(1-2): 47-57.
[18] Kulikov E, Kropinski A M, Golomidova A, et a1. Isolation and characterization of a novel indigenous intestinal N4-related coliphage vB_EcoP_G7C [J]. Virology, 2012, 426(2): 93-99.
[19] Lévesque C, Duplessis M, Labonté J, et a1. Genomic organization and molecular analysis of virulent bacteriophage 2972 infecting an exopolysaccharideproducing Streptococcus thermophilus strain [J]. Appl Environ Microbiol, 2005, 71(7): 4057-4068.
[20] Chibani-Chennoufi S, Bruttin A, Dillmann M L, et a1. Phage-host interaction: an ecological perspective [J]. J Bacteriol, 2004, 186(12): 3677-3686.
[21] Tang F, Li Y, Zhang W, et a1. Complete Genome Sequence of T4-Like Escherichia coli Bacteriophage HX01 [J]. J Virol, 2012, 86(24): 13871-13871.
[22] Samson J E, Magadán A H, Sabri M, et a1. Revenge of the phages: defeating bacterial defences [J]. Nature Reviews Microbiol, 2013, 11(10): 675-687.
[23] Petrov V M, Ratnayaka S, Nolan J M, et a1. Genomes of the T4-related bacteriophages as windows on microbial genome evolution [J]. Virol J, 2010, 7: 292.
PHYLOGENETIC ANALYSIS OF A BROAD-HOST-RANGE LYTIC COLIPHAGE
ZHOU Yan, BAO Hong-duo, ZHANG Hui, ZHANG Li-li, WANG Ran
(Key Lab of Animal-derived Food Safety of Jiangsu Province, Key Lab of Food Quality and Safety of Jiangsu Province-State Key Laboratory Breeding Base, Institute of Food Safety and Quality, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China)
Lytic bacteriophage (phage) can kill bacteria. This study analyzed the classification and phylogeny of a broad-host-range lytic coliphage isolated from the nature. In a previous study, a strain of phage vB_EcoM_JS09 was isolated by using Adams doublelayer agar plate method and purif ed from the sewage of a swine farm. The genome was extracted through phenol chloroform isopropanol method, then treated with DNase I, RNase A and Mung Bean Nuclease. The conserved tail sheath gene 18 and major capsid protein gene 23 of T4-like phages were amplif ed using the reported primers and JS09 as a template and their deduced amino acid sequences were used for phylogenetic tree analysis. Results showed that lytic coliphage JS09 had the genome of double stranded DNA, belonging to the Caudovirales, Myoviridae, T4-like phage, T-even subgroups. The homology of amino acid sequence of gp18 was 100% with E. coli phage RB69 while that of gp23 shared 96% identity to E. coli T4 phage. The results provide a good foundation for the biology of Avian pathogenicEscherichia coliand enterotoxigenicEscherichia coliprevertion Cuel control.
Avain pathogenicE.coli;enterotoxigenicE.coli;broad-host range lytic coliphage; T4-like phage; phylogenetic analysis
S852.612
B
1674-6422(2014)06-0071-06
2014-11-06
国家自然科学基金(31402234);江苏省自然科学基金(BK2012788);江苏省农业科技自主创新基金(CX(13)3086)
周艳,女,助理研究员,主要从事噬菌体防控致病菌研究
王冉,E-mail:wangran2001@126.com
