一株Nsp2蛋白部分氨基酸自然缺失的猪繁殖与呼吸综合征病毒的分离和鉴定
2014-04-13洪绍锋韦苗苗覃陆英覃艳然黄伟坚韦祖樟
洪绍锋,韦 莹,韦苗苗,覃陆英,覃艳然,陈 樱,黄伟坚,韦祖樟
(广西大学动物科学技术学院,南宁 530005)
一株Nsp2蛋白部分氨基酸自然缺失的猪繁殖与呼吸综合征病毒的分离和鉴定
洪绍锋,韦 莹,韦苗苗,覃陆英,覃艳然,陈 樱,黄伟坚,韦祖樟
(广西大学动物科学技术学院,南宁 530005)
将RT-PCR检测为猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus, PRRSV)阳性的肺组织碾磨、过滤后接种MARC-145细胞,在细胞上连续传代,上清传至第二代后能产生细胞病变。通过RT-PCR方法可以检测到释放在细胞上清中PRRSV RNA。间接免疫荧光试验也能检测细胞内PRRSV N蛋白的表达,表明该PRRSV毒株成功地在MARC-145细胞中分离,命名为PRRSV NN1396株。对NN1396 分离株GP5基因进行克隆测序分析,结果表明该毒株与NCBI登录的PRRSV GP5核苷酸同源性为62.9%~98.2%,与PRRSV原型毒株VR-2332的同源性为87.9%,与高致病性毒株PRRSV JXA1同源性为98.0%,通过GP5的遗传进化分析发现所分离的PRRSV为北美型PRRSV,且与高致病性PRRSV分布在同一个小分支上;对部分nsp2基因的测序、比对结果表明,该部分基因与NCBI登录的参考序列VR-2332氨基酸同源性为73.0%,与JXA1的同源性为94.8%。NN1396分离株Nsp2蛋白含有高致病性PRRSV特异的1+29个氨基酸的缺失,此外,在缺失29个氨基酸区域的上游,含有连续19个氨基酸缺失。该PRRSV毒株的分离和Nsp2蛋白部分序列的缺失鉴定,为下一步研究Nsp2部分氨基酸缺失对病毒生物学特性影响奠定基础。
高致病性猪繁殖与呼吸综合征病毒;分离鉴定;GP5;Nsp2部分氨基酸自然缺失
猪繁殖与呼吸综合征(porcine reproductive and respiratory syndrome,PRRS)是猪繁殖与呼吸综合征病毒(Porcine reproductive and respiratory syndrome virus, PRRSV)引起的一种危害严重的接触性传染病,俗称“蓝耳病”。该病于1987年在北美首次爆发,主要临床表现为仔猪呼吸困难,体重下降,生长缓慢及母猪繁殖障碍[1]。根据遗传特性的差异, 可将PRRSV 划分为两个基因型(Genotype),即欧洲(1)型(代表株Lelystad virus,LV)和美洲(2)型(代表株VR2332)。前者主要流行于欧洲地区,而后者主要流行于美洲和亚太地区。两者之间基因组差异约40%,同型病毒间的亚型基因组差异也可高达20%[2,3]。PRRSV是一种有囊膜不分节段的单股正链RNA病毒,全长约15 kb,具有5′端帽状结构和3′poly(A)尾。病毒粒子呈球型,直径为55~60 nm。蛋白编码区包含至少8个部分首尾重叠的开放阅读框(open reading frame,ORF)[4],其中ORF1a和ORF1b编码病毒的复制酶蛋白,这些蛋白在病毒基因组的复制和亚基因组转录以及拮抗干扰素中起到重要作用[5]。ORF2a、ORF2b、ORF3和ORF4 分别编码GP2a、GP2b、GP3、GP4等次要结构蛋白。ORF5、ORF6 和ORF7分别编码病毒主要囊膜蛋白GP5、膜基质蛋白M和核衣壳蛋白N。
Nsp2为PRRSV编码的最大的病毒蛋白,在各基因型中和同一基因型之间变异很大。北美洲型的HB-2株、BJ-4株、VR-2332 株和CH-1a株,其Nsp2的氨基酸序列同源性为78.3%~88.9%[6]。特别的是,在Nsp2的高变区,常有氨基酸的缺失或插入,其病毒生物学意义还有待进一步研究。本研究利用MARC-145细胞对从广西省南宁市某猪场采集的RT-PCR检测PRRSV为阳性的组织器官(肺)进行病毒分离,分离到1株病毒,并命名为NN1396株。利用RT-PCR方法扩增NN1396 株Nsp2、ORF5基因并测序分析,并发现该毒株Nsp2中有49个氨基酸的缺失。
1 材料与方法
1.1 病料、细胞、菌株病料为采自广西省南宁市某猪场发病死亡猪的组织(肺);MARC-l45细胞、大肠杆菌DH5α由本实验室保存。
1.2 主要试剂新生胎牛血清、MEM、胰蛋白酶购自GIBCO公司;TaqMix、dNTPMixture、RnaseInhibitor、M-MLV等均购自宝生物(大连)有限公司。
1.3 PCR引物本研究参考已发表的HUN株和中国标准株CH-la株的核昔酸序列,用生物软件Oligo6.0分别在部分nsp2、ORF5、ORF7基因前后的保守区设计了3对引物:nsp2-F/nsp2-R、ORF5-F/ORF5-R和N1/N2(见表1)。

表1 本研究设计的PRRSV 扩增引物Table 1 Primers for PRRSV in this study
1.4 毒株分离及鉴定方法
1.4.1 PRRSV检测 将肺组织剪碎研磨,600×g 离心8 min ,取上清液抽提病毒的总RNA,以病毒RNA作为模板进行逆转录,合成与RNA模板互补的cDNA链。cDNA在Taq Mix酶的作用下,以N1/N2(表1)为引物进行PCR反应,同时设阴、阳性对照。PCR扩增条件:95℃预变性3 min;95℃变性1 min,59℃退火1 min,72℃延伸1 min,共32个循环;72℃再延伸10 min。用1.2%琼脂糖凝胶电泳检测PCR产物。
1.4.2 毒株分离 组织研磨成浆液,100×g 离心5 min感染细胞。MARC-145细胞培养于六孔板中,待长到约90%,弃去培养基,用病毒液(组织液)在37℃、5% CO2温箱孵育1 h,再加2%维持培养基,感染细胞放在37℃、5% CO2温箱中培养4~7 d,观察细胞病变(cytopathic effect,CPE)。当CPE达80%时,收获上清,-70℃ 冻存。上清(记作F1)接种正常细胞,同上连续传代,待其收毒时间稳定,保存备用。
1.4.3 毒株鉴定
1.4.3.1 RT-PCR 收集的病毒上清液抽提病毒的总RNA,方法同1.4.1。
1.4.3.2 间接免疫荧光 将病毒上清感染单层 MARC-145细胞,感染后48 h弃去培养基。用冰甲醇固定10 min,1%BSA封闭30 min,用 PRRSV N 蛋白的特异性单抗室温孵育2 h ,再加入Alexa Fluor 568标记羊抗鼠的二抗室温孵育1 h,PBS洗3遍后,在荧光显微镜下观察结果。
1.5 ORF5基因、部分nsp2基因的扩增将已鉴定为阳性的PRRSVNN1396株F2代细胞毒提取病毒总RNA。取16μLRNA,加入5×buffer(Mg2+)5μL、dNTPMixture(2.5mmol/L)2μL、RNaseinhibitor(40U/μL)0.5μL、反转录引物1μL、AMV酶(5U/μL)0.5μL,混匀,42℃1h,-20℃保存备用。
1.5.1 ORF5基因扩增 cDNA在Taq Mix酶的作用下,以ORF5-F/ORF5-R(表1)为引物进行PCR反应。PCR扩增条件:95℃预变性3 min;95℃变性1 min,59℃退火1 min,72℃延伸1 min,共32个循环;72℃再延伸10 min。用1.2%琼脂糖凝胶电泳检测PCR产物。
1.5.2 部分nsp2基因扩增 cDNA在Taq Mix酶的作用下,以nsp2-F/nsp2-R(表1)为引物进行PCR反应,PCR扩增条件:95℃预变性3 min;95℃变性l min,60℃退火l min,72℃延伸l min,共32个循环;72℃再延伸10 min。用1.2%琼脂糖凝胶电泳检测PCR 产物。
1.5.3 ORF5部分nsp2基因序列分析 检测为阳性的目的片段进行目的基因回收,克隆,测序。测序所获得的基因序列应用生物软件Lasergene、MEGA4.1分析。
2 结果
2.1 PRRSV的检测和病毒分离将阳性病料碾磨,抽提总RNA,通过特异的诊断引物(表1)进行检测,发现该肺脏组织样品检测结果为PRRSV阳性,目的片段在443 bp左右,与预期相符(图1)。将PCR产物直接进行测序分析,通过核苷酸比对发现该序列属于我们预期设计扩增PRRSV片段(数据未显示),表明所检测的组织中含有PRRSV。
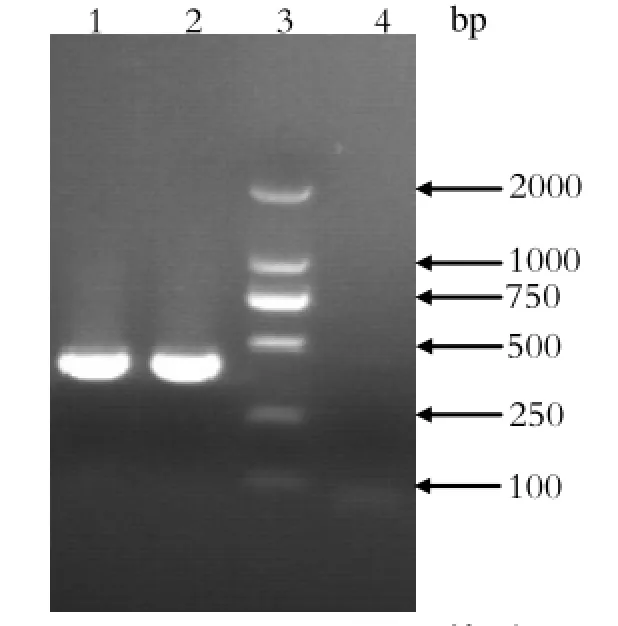
图1 PRRSV PCR检测Fig.1 Detection of PRRSV by PCR
为了从组织中分离到PRRSV,将碾磨的病料过滤,接种到MARC-145细胞上,4 d后取细胞上清200 μL接种到新鲜的MARC-145细胞上,如此进行连续传代。结果发现接种病料上清的 MARC-145 细胞单层从第 2 代(F2)开始出现CPE,3代后病变趋于稳定,表现为接种 MARC-145 细胞 48 h 后即可产生明显 CPE,细胞折光性增强,聚集、皱缩,72 h后细胞呈灶状脱落,形成空斑。4~5代细胞与第3代病变相似,细胞在接毒 48 h 后开始出现CPE(图2)。

图2 MARC-145细胞感染PRRSV分离株后出现的细胞病变Fig.2 Cytopathic effect of MARC-145 infected with PRRSV isolate
2.2 分离病毒的鉴定为鉴定导致MARC-145产生CPE是否由PRRSV感染引起,我们对细胞上清中病毒核酸进行检测,取感染细胞的上清进行RNA抽提,通过PRRSV诊断引物(表1)进行检测,也能扩增出预期大小的PCR片段,测序结果也表明扩增的片段为PRRSV的基因组序列(结果未显示)。为确定PRRSV是否在细胞中复制,我们用间接免疫荧光(indirect immunofluorescence,IFA)对细胞中表达的病毒蛋白进行鉴定。对传代的MARC-145细胞进行固定,用PRRSV特异的N蛋白单克隆进行检测。结果显示,PRRSV分离株感染的 MARC-145 细胞中有特异荧光产生(图3A),而未感染的空白对照细胞中没有特异红色荧光出现(图3B),由此,签定所分离到的病毒为PRRSV。
2.3 GP5基因的克隆和遗传进化分析为对病毒进行遗传进化分析,设计的ORF5基因引物(引物序列见表1)。对PRRSV NN1396分离株进行RT-PCR扩增,扩出与预期一致的片段,如图4所示。对GP5基因进行克隆测序,结果表明其与NCBI上登录的参考序列相应GP5的同源性为62.9%~98.2%,与PRRSV 原型株VR2332的同源性为87.9%,与高致病性PRRSV JXA1株同源性为98.0%。遗传进化表明所分离的PRRSV为北美型PRRSV,如图6,其与高致病性蓝耳病分布在同一个小分支上。
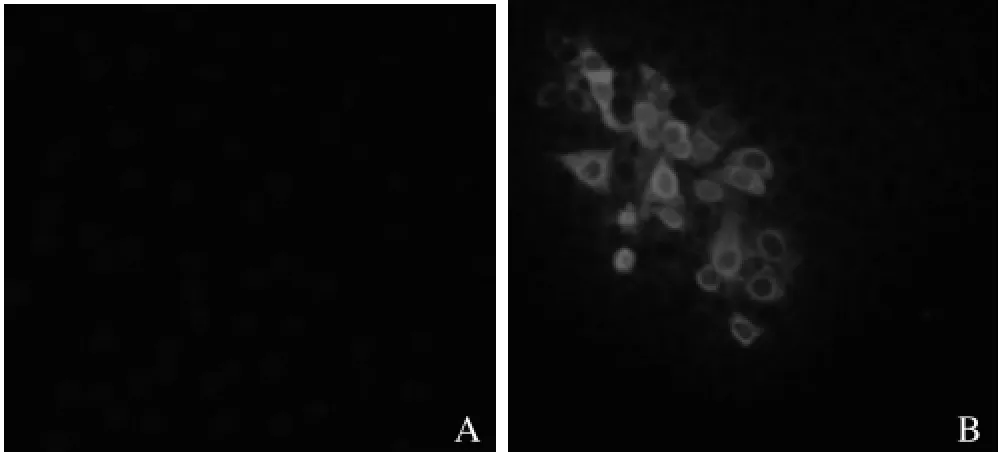
图3 间接免疫荧光检测PRRSV分离株Fig.3 Detection of PRRSV isolate by indirectimmun of uorescence(IFA)
2.4 Nsp2高变区部分氨基酸缺失的鉴定Nsp2蛋白编码区是PRRSV变异最大的区域,为研究nsp2的遗传变异规律,我们对nsp2高变区进行测序分析。通过特异的引物(引物序列见表1),进行PCR扩增,如图5所示,所获得的目的基因长度为739 bp,比预期片段少了57个核苷酸,截去引物后,获得的核苷酸序列为690 bp,核苷酸与NCBI登录的参考序列对应部分核苷酸序列同源性为81.3%~96.5%;推导编码230个氨基酸,推导编码的氨基酸与NCBI登录的参考序列对应部分氨基酸同源性为72.6%~94.8%,与VR2332氨基酸同源性为73.0%,与JXA1的同源性为94.8%;NN1396分离株缺失49个氨基酸,1+29个氨基酸缺失的位置与JXA1株的一致,此外,NN1396分离株在30个氨基酸缺失位点之间,还缺失19个氨基酸,NN1396分离株氨基酸缺失位置如图7。
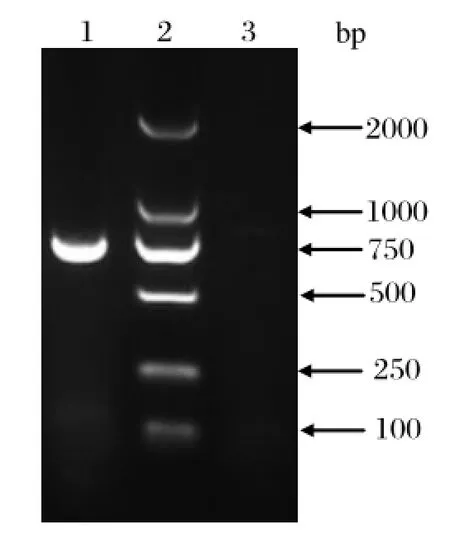
图5 PRRSV分离株的部分nsp2基因扩增Fig.5 Amplif ed part of nsp2 of PRRSV isolate by PCR
3 讨论
Nsp2是PRRSV编码的最大的蛋白,它至少含有4个结构域:N端的CP/OUT结构域、中间的高变区、跨膜区和C端的富含半胱氨酸的区域[7]。Nsp2的半胱氨酸蛋白酶(cysteine proteinse,CP2)具有顺式(cis-)或者反式(trans-)的切割活性,介导Nsp2/Nsp3切割。研究表明,Nsp2含有多个B细胞表位,这些表位能够激发机体在病毒感染早期产生很高的抗体[8]。2006年我国爆发“猪高热综合征”,研究表明高致病性PRRSV是导致该“猪高热综合征”的主要病原之一。高致病性PRRSV一个遗传特征就是在Nsp2高变区含有1+29个氨基酸的缺失[9],2007年,这一高致病性PRRSV开始广泛流行。然而也有研究表明,30个氨基酸的缺失与病毒毒力强弱没有关系[10]。本研究分离的NN1396毒株,其Nsp2蛋白高变区除了存在1+29个氨基酸缺失外,还在29个氨基酸缺失的上游缺失19个氨基酸。几乎所有强毒力毒株如MN184、08RB1、HP-PRRSV等都在Nsp2部位存在缺失,NN1396毒株Nsp2中19个氨基酸的缺失对病毒生物学特性的是否有影响,还需要进一步研究。
总之,本研究中,我们成功分离到一株PRRSV NM1396株,并鉴定其Nsp2蛋白存在49个氨基酸的缺失,为进一步研究Nsp2氨基酸缺失对病毒生物学特性的影响奠定了基础。

图6 NN1396株GP5核苷酸序列与参考株相应序列遗传进化树Fig.6 GP5 of NN1396 genetic evolutionary tree
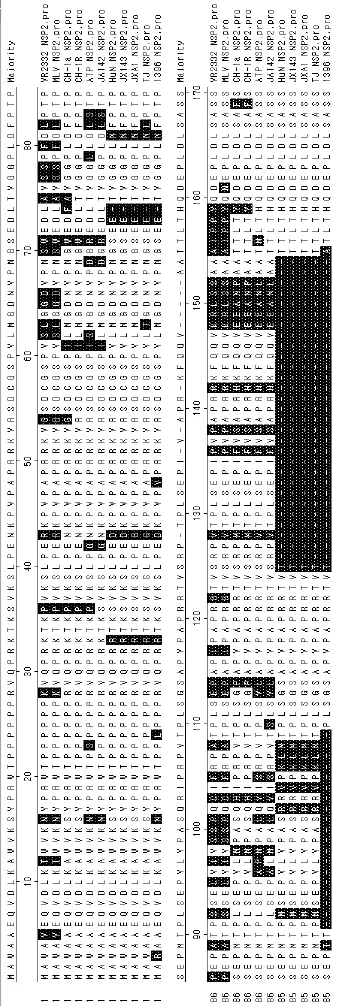
图7 NN1396 株部分Nsp2 蛋白序列与参考序列对比结果Fig.7 The deduced amino acid sequence comparison of the nsp2 gene between the NN1396 and the reference sequence
参考文献
[1] Rossow K D. Porcine reproductive and respiratory syndrome[J]. Vet Pathol, 1998, 35(1): 1-20.
[2] Wu W H, Fang Y, Rachel F,et al.A 10 kDa structural protein of porcine reproductive and respiratory syndrome virus encoded by ORF2b [J].Virology, 2011, 287(1):183-191.
[3] Meng X J. Heterogeneity of porcine reproductive and respiratorysyndrome virus: implications for curent vaccine efficacy and future vaccine development[J]. Vet Microbiol, 2000, 74(4): 309-329.
[4] 韦祖樟, 孙志, 袁世山. 猪繁殖与呼吸综合症病毒分子生物学研究进展[J]. 微生物学通报, 2008, 35(3): 408-413.
[5] Fang Y, Snijderb E J. The PRRSV replicase: Exploring the multifunctionality of an intriguing set of nonstructural proteins[J]. Virus Res, 2010, 154(1-2): 61-76.
[6] Shen S, Kwang J, Liu W,et al.Determination of the complete nucleotide sequence of a vaccine strain of porcine reproductive and respiratory syndrome virus and identification of the nsp2 gene with a unique insertion[J]. Arch Virol, 2000, 145(5): 871-883.
[7] Snijider E J, Wassenaa A L, Spaan W J,et al.The arterivirus nsp2 protease. An unusual cysteine protease with primary structure similarities to both papain-like and chymotrypsin-like proteases[J]. J Biol Chem, 1995, 270(28): 16671-16676.
[8] de Lima M, Pattnaik A K, Flores E F,et al.Serologic marker candidates identified among B-cell linear epitopes of nsp2 and structural proteins of a North American strain of porcine reproductive and respiratory syndrome virus[J]. Virology, 2006, 353(2), 410-421.
[9] Tian K, Yu X, Zhao T,et al.Emergence of fatal PRRSV variants: unparalleled outbreaks of atypical PRRS in China and molecular dissection of the unique Hallmark[J]. PLoS One, 2007, 2(6): e526.
[10] Zhou L, Zhang J L, Zeng J W,et al.The 30-aminoacid deletion in the nsp2 of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China is not related to its virulence[J]. J Virol, 2009, 83(10): 5156-5167.
ISOLATION AND IDENTIFICATION OF A PORCINE REPRODUCTIVE AND RESPIRATORY SYNDROME VIRUS FIELD STRAIN WITH NATURAL DELETION OF PARTIAL NSP2 PROTEIN
HONG Shao-feng, WEI Ying, WEI Miao-miao, QIN Lu-ying, QIN Yan-ran, CHEN Ying, HUANG Wei-jian, WEI Zu-zhang
(College of Animal Science and Technology ,Guangxi University, Nanning 530005,China)
The lung tissues with Porcine reproductive and respiratory syndrome virus(PRRSV) RT-PCR positive results were homogenized and inoculated into MARC-145 cells for virus isolation. PRRSV typical CPE was observed at the second passage. PRRSV RNA was detected in supernatants using RT-PCR. PRRSV N protein was conf rmed in inoculate MARC-145 cells. The obtained PRRSV was designated as NN1396. Hypervariable region of GP5 protein was sequenced and analyzed. The GP5 of NN1396 strain shared 62.9% - 98.2% identities with other PRRSV strains. Phylogenetic analysis showed that NN1396 strain belonged to North American genotype and HP-PRRSV. Sequence analysis of partial nsp2 gene showed that NN1396 strain had 81.4% amino acid identity with VR-2332 strain and 96.5% with JXA1. More importantly, NN1396 strain contained a deletion of 19 aa in nsp2 coding region , which was located upstream of29 aa deletion. Characterization of PRRSV NN1396 strain was useful to study the effect of partial gene deletion of nsp2 on its biological activities.
Highly pathogenic Porcine reproductive and respiratory syndrome virus(HP-PRRSV); isolation; GP5; natural deletion of partial Nsp2 protein
S852.659.6
A
1674-6422(2014)06-0006-07
2014-04-29
广西自然科学基金(桂科自0728015);国家自然科学基金(31372444);广西大学大学生实验技能和科技创新能力训练基金项目(SYJN20131705)
洪绍锋,女,硕士研究生,预防兽医学专业
黄伟坚,E-mail:huangweijian-1@163.com;韦祖樟,E-mail:zuzhangwei@163.com
