盐蒿内生耐(嗜)盐真菌的分离及鉴定
2014-03-23丁小维
丁小维
(陕西理工学院 生物科学与工程学院, 陕西 汉中 723000)
0 引 言
耐(嗜)盐真菌是能够在盐浓度为0.2 mol/L以上环境中生长的真菌,主要分布在盐湖、盐田、海洋等高盐环境中。目前,对耐(嗜)盐真菌的研究主要集中在海洋环境耐(嗜)盐真菌物种多样性及其代谢产活性物质方面[1-3],而对植物内生耐(嗜)盐真菌的研究相对较少。钮旭光等[4]从辽宁双台子国家自然保护区内陆盐碱地及潮间带生长的翅碱蓬(Suaedasalsa)中分离到13个属的内生真菌。在高盐环境中生长的耐盐植物组织中可能蕴藏着丰富的耐盐内生真菌。此外,研究证实高盐地区植物内生真菌对宿主适应盐逆境方面发挥重要作用[5-7]。因此,研究内陆极端盐湖区植物内生耐(嗜)盐真菌可为耐盐植物的培育及促进盐碱化土壤的综合利用提供丰富的菌种资源。
花马盐湖属于碱性碳酸盐型内陆高盐湖泊,位于陕西省榆林定边县城西北10 km处的盐场堡乡。花马盐湖蕴藏着非常丰富的碳酸钠、氯化钠、氯化钾及氯化钙等资源。该盐湖区寒暑变化剧烈,年降水量小(年降水约为316.9 mm),日照强烈,蒸发量大(年蒸发量高达2 490 mm),干燥及辐射强烈,因此具有高盐碱、大温差、高寒、高热、干燥及强辐射等极端环境特征。盐湖区沉积物Na+浓度从46.7 g/kg到饱和Na+浓度,生长于湖岸的盐蒿其组织中Na+含量为普通植物6倍多,该地区无疑是发掘极端耐(嗜)盐真菌多样性的理想区域之一。
本文利用不同含盐培养基分离纯化花马盐湖区耐盐植物盐蒿的耐(嗜)盐真菌,基于形态学和ITS序列的系统发育关系认识真菌物种多样性,丰富我国盐湖真菌菌种资源,为耐(嗜)盐真菌的进一步开发利用奠定基础。
1 材料与方法
1.1 材料
1.1.1 实验材料
盐蒿(A.halodendron)采自陕北榆林定边县北盐堡乡花马盐湖区(海拔1 301 m,纬度37°40′478N,经度107°30′724E)。新鲜的植物材料收集于塑料袋中,置于冰盒中带回实验室,用于分离耐(嗜)盐真菌。
1.1.2 分离培养基
分离培养基:(1)改良马铃薯琼脂培养基(cPDA):去皮马铃薯200 g/L,葡萄糖20 g/L,KH2PO43 g/L,MgSO41.5 g/L,维生素B110 mg/L,琼脂粉15 g/L,pH自然;(2)麦芽汁琼脂培养基(MEA):麦芽提取物20 g/L,蛋白胨1 g/L,葡萄糖15 g/L,琼脂15 g/L,pH 6;(3)酵母膏葡萄糖蛋白胨(YPD):酵母膏5 g/L,蛋白胨10 g/L,葡萄糖20 g/L,琼脂粉15 g/L,pH自然。在上述3种分离培养基中分别添加浓度为2.5%,5%,10%和15%的NaCl。
1.1.3 主要仪器和设备
主要仪器设备:光学显微镜(E600型,日本尼康公司),荧光倒置显微成像系统(奥林巴斯IX71/IX81),冷冻超速离心机(Beckman),PCR扩增仪(Bio-Rad),电泳仪(Bio-Rad),紫外凝胶成像分析系统(Bio-Rad)及DNA测序仪(ABI PRISM 377)等。
1.2 方法
1.2.1 耐(嗜)盐真菌的分离纯化
将盐蒿茎、叶组织置于自来水下冲洗4~5 h后,在无菌条件下依次用有效氯含量为4.5%~5%的次氯酸钠(含0.01% Tween 80)处理3 min,用75%乙醇处理15 s,最后用无菌水清洗6次。将表面消毒彻底的植物材料切成0.5 cm2小块,并置于分离培养基上,28 ℃恒温培养箱倒置培养1~3 w,挑取菌丝接入含5% NaCl的YPD斜面纯化培养,纯化斜面于4 ℃保藏备用。
1.2.2 耐(嗜)盐真菌的鉴定
采用形态学并结合ITS系统发育分析对内生真菌进行鉴定。菌落培养特征、菌丝、产孢结构及孢子特征参照魏景超主编的《真菌鉴定手册》进行鉴定[8]。采用CTAB法[9]提取盐蒿内生真菌基因组DNA。PCR反应体系(25 μL):超纯无菌水9 μL,2×PCR Mix 11 μL,10 μmol/L的ITS1 (5′-TCCGTAGGTGAACCTGCGG-3′)和ITS2(5′-TCCTCCGCTTATTGATATGC-3′)各2 μL,基因组DNA约50 ng;PCR反应条件:95 ℃预变性5 min;94 ℃ 变性1 min,55 ℃退火40 s,72 ℃ 扩增1 min,35个循环;72 ℃再保持10 min。PCR产物由上海生工生物技术服务有限公司进行测序。序列提交GenBank,并下载相似性高的数据,用Clustal-X[10]软件对所得序列文件进行比对分析,通过Mega 4.0软件构建N-J系统发育树[11]。
2 结果与分析
2.1 耐(嗜)盐真菌的分离
利用cPDA、YPG及MEA培养基分离盐蒿内生真菌,从表面消毒的茎、叶组织块中共分离到1 215株真菌,以YPG上获得的内生真菌菌株数最多,占35.5%,而在cPDA和MEA上分离到的菌株数较YPG的少。培养基不同盐浓度对内生真菌分离研究表明,以含盐浓度为15%的NaCl培养基上出菌数最多(404株),含5%NaCl的培养基次之(340株),分别占其出菌总数的33.3%和28%。
2.2 耐(嗜)盐真菌的鉴定
2.2.1 耐(嗜)盐真菌的形态学
形态学研究表明,不同形态种的菌落特征正反面形态及颜色差异很大(图1,表1),如AH-1菌落较圆整平铺,正面粉红色,反面内侧浅灰黑色,外侧粉红色,菌丝较粗壮、发达,分枝有隔(图1A,C);AH-18菌落正面乳白色菌丝上有土黄色孢子分生孢子头,反面黄色,菌丝分枝有隔,分生孢子梗上产生球形孢子,大小2.0 μm×2.0 μm(图1B,D);AH-35菌落正面黄绿色,反面黄褐色,分生孢子球形或近球形,大小2.5 μm×2.5 μm,(2.0~2.5)μm×(2.5~4.75)μm。基于菌落和显微形态特征,从盐蒿中获得的内生真菌属于9个不同的形态种,以AH-1为优势种,其分离频率为78.8%,其余菌株分离频率在0.09%~2.1%之间(表1)。
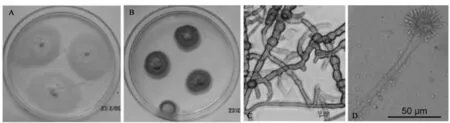
图1 盐蒿部分耐(嗜)盐真菌菌落及显微形态特征

菌株编号分离频率/%形态学特征(菌丝、孢子大小/μm)ITS/%GenBank中最相似种及其登录号分类地位AH-178.8菌落较圆整,正面粉红色,反面产脂溶性黑色素;菌丝(5.0~8.75)粗壮、发达,分枝有隔,且局部膨大;菌丝内生孢子呈圆形,大小(2.25~2.5)×(2.25~2.5)99/97Pleosporales sp.HQ914837Leptosphaeria sp.AH-20.2菌落圆整,正面内侧浅灰色,外侧青绿色,反面浅黄色;菌丝分枝有隔;产生帚状枝,分生孢子呈球形99Penicillium chrysogenum,JQ015265PenicilliumchrysogenumAH-40.7菌落正面黄绿色,反面黄褐色;分生孢子球形或近球形99Trichoderma longibrachiatum,HQ882796Trichoderma sp.AH-180.09菌落正面乳白色菌丝上有土黄色孢子分生孢子头,反面黄色;菌丝分枝有隔,分生孢子梗上产生球形孢子,大小2.0×2.0100Aspergillus westerdijkiae,JN793950Aspergillus westerdijkiaeAH-200.09菌落较圆整平铺,正面粉红色,反面内侧浅灰黑色,外侧粉红色;菌丝较粗壮、发达,分枝有隔;未见产孢结构100Alternaria tenuissima,JQ417902Alternaria sp.AH-222.1菌落呈褐色绒状,菌丝分枝有隔;分生孢子榴弹形,有纵横隔100Alternaria tenuissima,JQ417902Alternaria tenuissimaAH-290.09菌落呈灰褐色绒状,菌丝分枝有隔;分生孢子榴弹形,有3~5个纵横隔,孢子大小(10~15)×(30~36)100Alternaria alternata,JQ070079Alternaria alternataAH-340.2菌落正反面浅粉色,棉絮状;菌丝有隔分枝;未见分生孢子99Fusarium chlamydosporum,HQ671187Fusarium sp.AH-350.09菌落正面黄绿色,反面黄褐色;分生孢子球形或近球形,大小2.5×2.5,(2.0~2.5)×(2.5~4.75)99/100Trichoderma longibrachiatum,JQ653055Trichoderma longibrachiatum
2.2.2 ITS的系统发育分析
盐蒿内生真菌ITS的系统发育树如图2所示,8个代表菌株(AH-2,AH-4,AH-18,AH-20,AH-22,AH-29,AH-34及AH-35)的ITS序列同GenBank数据库中序列相似性≥99%,聚于系统发育树分枝上的步长值在74%~100%之间。结合形态学特征,上述菌株属于Penicillium、Trichoderma、Aspergillus、Alternaria及Fusarium,被鉴定为Penicilliumchrysogenum、Aspergilluswesterdijkiae、Alternariatenuissima和Trichodermalongibrachiatum等。菌株AH-1,AH-32,AH-58,AH-64和AH-77同GenBank数据库中格孢菌目(Pleosporales)一真菌的ITS序列相似性为97%,上述菌株内聚于一个分枝上,步长值分别为99%。基于形态学特征,上述5个菌株为Leptosphaeria属的一潜在新物种。Leptosphaeria是花马盐湖盐蒿内生真菌的优势属,其次为Alternaria,分别占78.8%和2.28%。研究结果表明,陕北花马盐湖碱蓬中蕴藏着较丰富的耐(嗜)盐真菌。上述真菌的ITS序列提交GenBank登录号为:KC460798,KC460799,KC460801,KC460814,KC460816,KC460818,KC460825,KC460828,KC460830,KC460831,KC460858和KC460871。
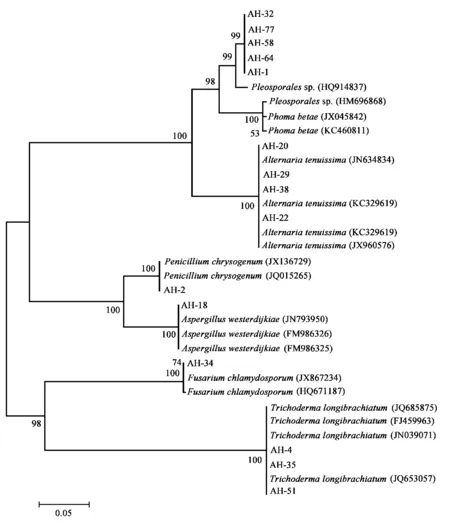
图2 盐蒿内生真菌ITS邻接系统发育树分析
3 讨 论
研究了通过含盐浓度不同的cPDA、MEA及YPG培养基分离盐蒿内生耐(嗜)真菌并研究物种多样性。从盐蒿茎、叶组织中共分离到1 215株真菌,在YPG上获得的内生菌株数最多,以含NaCl浓度为15%的培养基出菌率最高。杨丽源等[12]分离耐(嗜)盐真菌时发现,盐矿水样中的真菌在以5%NaCl的YPG培养基上出菌数最多,某些固体样则以含5%或15%的NaCl培养基为最佳。由于样品类型差异,为获得盐蒿中多样的耐(嗜)盐真菌,分离培养基的盐浓度需要多样化。
近年来,形态学和ITS序列分析被广泛应用于真菌物种多样性研究[13-15]。由于ITS序列长度合适,具有较高的种间变异和低种内变异及其在GenBank,EMBL/ENA及DDBJ等国际数据库中有大量参考序列,被认为是真菌鉴定的理想分子标签[16-17]。本研究从花马盐湖盐蒿中获得6个真菌属,Leptosphaeria为优势属,获得9个形态种如P.chrysogenum、A.westerdijkiae、Al.tenuissima和T.longibrachiatum等。钮旭光等[4]从翅碱蓬根、茎及叶组织中获得49株的内生真菌,以Glomerella、Alternaria、Colletotrichum和Cladosporium为优势菌属。虽然不同生境中的盐蒿内生真菌群落丰富度存在一定相似性,但在某些种属分布存在差异性,这可能由于某些内生真菌具有宿主特异性,且其分布受宿主所生长的环境影响。本研究探讨盐湖盐蒿内生耐(嗜)盐真菌物种多样性,将为耐(嗜)盐真菌的开发利用提供菌种资源。
[参考文献]
[1] WANG W,WANG Y,TAO H,et al.Cerebrosides of the halotolerant fungusAlternariaraphaniisolated from a sea salt field[J].J Nat Prod.,2009,72(9):1695-1698.
[2] ZHENG J,XU Z,WANG Y,et al.Cyclic Tripeptides from the halotolerant fungusAspergillussclerotiorumPT06-1[J].J Nat Prod.,2010,73(6):1133-1137.
[3] 黄菁菁,鲁春华,钱晓鸣,等.台湾海峡海洋耐(嗜)盐真菌及其抗菌活性的初步研究[J].厦门大学学报:自然科学版,2008,47(5):723-727.
[4] 钮旭光,宋立超,韩梅,等.盐生植物翅碱蓬的内生真菌多样性分析.微生物学通报[J].2012,39(10):1388-1395.
[5] RODRINGUEZ R J,HENSON J,VOLKENBURGH Van E,et al.Stress tolerance in plants via habitat-adapted symbiosis[J].ISME J.,2008,2(4):404-416.
[6] YOU Y H,YOON H,KANG S M,et al.Fungal diversity and plant growth promotion of endophytic fungi from six halophytes in Suncheon Bay[J].J Microbiol. Biotechnol,2012,22(11):1549-1556.
[7] Maciá-Vicente J G,Ferraro V,Burruano S,et al.Fungal assemblages associated with roots of halophytic and non-halophytic plant species vary differentially along a salinity gradient[J].Microb Ecol.,2012,64(3):668-679.
[8] 魏景超.真菌鉴定手册[M].上海:上海科学出版社,1979.
[9] ZHANG D,YANG Y,CASTLEBURY L A,et al.A method for the large scale isolation of high transformation efficiency fungal genomic DNA[J].FEMS Microbiol Lett.,1996(145):261-265.
[10] THOMPSON J D,GIBSON T J,PLEWNIAK F,et al.The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Res.,1997(24):4876-4882.
[11] TAMURA K,DUDLEY J,NEI M,et al.MEGA4:Molecular evolutionary genetics analysis (MEGA) software version 4.0[J].Mol Biol Evol.,2007,24(8):1596-1599.
[12] 杨丽源,李治滢,李绍兰,等.一平浪盐矿耐盐真菌的种群调查[J].云南大学学报:自然科学版,2002,24(6):465-468.
[13] BLAXTER M L.The promise of a DNA taxonomy[J].Philos Trans R Soc Lond B Biol Sci.,2004,359(1444):669-679.
[14] SCHINDEL D E,MILLER S E.DNA barcoding a useful tool for taxonomists[J].Nature,2005,435(7038):17.
[15] SCHOCH C L,SEIFERT K A,HUHNDORF S,et al.Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for fungi[J].Proc Natl Acad Sci USA,2012,109(16):6241-6246.
[16] BEGEROW D,NILSSON H,UNTERSEHER M,et al.Current state and perspectives of fungal DNA barcoding and rapid identification procedures[J].Appl Microbiol Biotechnol.,2010(87):99-108.
[17] DENTINGER B T,DIDUKH M Y,MONCALVO J M.Comparing COI and ITS as DNA barcode markers for mushrooms and allies (Agaricomycotina)[J].PLoS One,2011,6(9):e25081.
