利用响应面分析法进行氨肽酶发酵培养基的优化
2012-01-28杨玉兰杨萍姜文侠刘琦周贺梅保良
杨玉兰,杨萍,姜文侠,*,刘琦,周贺,梅保良
(1.天津科技大学,天津300457;2.天津市工业生物系统与过程工程重点实验室,中国科学院系统微生物工程重点实验室,中国科学院天津工业生物技术研究所,天津300308)
氨肽酶(aminopeptidase)是蛋白水解酶中外肽酶的一类,它们能从蛋白质和多肽链的N 末端顺序水解氨基酸。甲硫氨酸氨肽酶是氨肽酶中的一种,在蛋白质加工过程中负责切除新生肽链N 端的起始甲硫氨酸[1]。目前氨肽酶制剂主要应用于功能肽的制备和蛋白质水解食品的生产加工过程,用以提高营养质量,改善产品的最终风味[2]。甲硫氨酸氨肽酶特有的水解方式和功能,决定了其广泛应用于食品、医药、发酵、饲料及生物技术等行业[3-6]。我国的微生物氨肽酶研究工作处于实验室阶段,氨肽酶菌种发酵单位低,难以达到工业化生产的要求,而菌种是工业发酵生产外肽酶的重要条件。从自然界筛选并在实验室诱变进化一直是发现和创新工业微生物菌种的重要途径。本实验从传统食品麦酱中筛选得到一株甲硫氨酸氨肽酶生产菌11-11-16-2-4AL,在对其发酵特性研究的基础上,研究了发酵生产氨肽酶的工艺参数。
Plackett-Burman 设计法是一种两水平的试验设计方法,它试图用最少试验次数达到使因素的主效果得到尽可能精确的估计,适用于从众多的考察因素中快速有效地筛选出最为重要的几个因素,供进一步研究[9]。响应面分析法是一种寻找多因素系统中最佳的数学统计方法,可以通过拟合方程的方法得到显著影响因素的最佳水平组合[10]。但是响应面拟合方程只在考察区域的紧邻范围内才充分近似真实情形,所以,要先接近最佳值区域后才能建立有效的响应面拟合方程。最陡爬坡法是寻找最优区域的一个有效的方法,它可以将显著因素的水平逼近最优区域。这种方法可以迅速搜索到关键因子的极值点。快速登高法缩短了实验的路径,能以较少的实验次数快速逼近最优值区域[11]。
本文以甲硫氨酸氨肽酶生产菌的发酵酶活水平作为响应目标,在培养基成分单因素优化的基础上,进一步采用Plackett-Burman、最陡爬坡、响应面的Box-Benhnken 的中心组合实验设计,通过Minitab 软件对实验数据进行分析,拟合数学模型,寻求最优培养基配方,并进行验证。研究中的试验设计、数据分析及模型建立皆由Minitab 软件辅助完成。
1 材料与方法
1.1 菌种和培养基
甲硫氨酸氨肽酶产生菌11-11-16-2-4AL,为本实验室从麦酱中分离。
种子培养基(g/L):麦芽糊精8,酵母粉2,蛋白胨8,磷酸二氢钾1,硫酸镁0.2,pH 7.2,121 ℃灭菌20 min。
发酵培养基(g/L):麦芽糊精15,酵母粉10,蛋白胨5,牛肉膏2,磷酸二氢钾2.7,硫酸镁0.1,氯化钠1,pH 7.2,121 ℃灭菌20 min。
1.2 主要试剂和仪器
甲硫氨酸对硝基苯胺:sigma 公司,分析纯;其它均为市售分析纯。
TU-1810PC 型紫外分光光度计:北京普析通用;IS-RDH1 型卧式恒温振荡器:美国精骐;电热恒温水浴锅:天津泰斯特;Thermo CR3i高速冷冻离心机:美国赛默飞世尔科技。
1.3 培养方法
挑取氨肽酶产生菌11-11-16-2-4A 的单菌落接入种子培养基中,于36 ℃摇床200 r/min 培养6 h~8 h,制备种子液。将种子液按5 %接种量,接种至发酵培养基,于36 ℃摇床200 r/min 培养24 h,为甲硫氨酸氨肽酶的发酵液。将发酵液于4 ℃、10 000 r/min 离心得上清液,即为甲硫氨酸氨肽酶的粗酶液,最后对粗酶液进行酶活测定。
1.4 酶活检测方法
常规对硝基苯胺法[7-8]:向比色管中加入2 mL氨-氯化铵缓冲液和1 mL 底物溶液,再加入l mL 稀释一定倍数的粗酶液,60 ℃水浴反应10 min,反应结束立即加入终止液6 mL,终止反应。空白对照以蒸馏水代替粗酶液。以紫外分光光度计于380 nm测定吸光度,根据标准曲线计算酶活。酶活定义:60 ℃每分钟水解甲硫氨酸对硝基苯胺产生1 μmol对硝基苯胺所需的酶量,定义为一个酶活单位(U)。
2 结果与分析
2.1 Plackett-Burman 设计试验确定显著因素
在培养基成分单因素优化的基础上,将原发酵培养基的7 种成分麦芽糊精、酵母粉、蛋白胨、牛肉膏、磷酸二氢钾、硫酸镁和氯化钠,分别作为Plackett-Burman 设计的7 个因素A、B、D、E、G、H、K,另设2 个虚拟因素C 和F,以考察试验误差。每个因素取2 个水平,低水平为原发酵培养基水平,高水平取低水平的1.2 倍。并运用Minitab 软件进行试验设计,每个试验重复2 次,对应的响应值取2次试验结果的平均值,试验设计及结果见表1。
从表1 可知,在这7 个因素的主效应中,B、D、E和G 对响应值(酶活)表现为负效应,A、H、J 对响应值(酶活)表现为正效应。其中A、B、D 对响应值(酶活)有显著影响(可信度大于95%,即主效应p<0.05),作为显著因素进行下一步试验。
2.2 最陡爬坡法逼近显著因素最优区域
根据Plackett-Burman 试验结果,确定各因素的水平。由于E 和G 对酶活表现为负效应,所以确定E 和G 的添加量分别为:2、2.5 g/L;H、J 对酶活表现正效应,所以确定H、J 的添加量为6、1.2 g/L。显著因素A 对酶活表现正效应、B 和H 对酶活表现为负效应,故应该适当提高A 的浓度,降低B 和H 的浓度。根据3 个显著影响因素效应的大小确定快速登高试验的方向和变化步长,以最快的速度逼近最大响应值(酶活)的区域,试验设计与结果见表2。
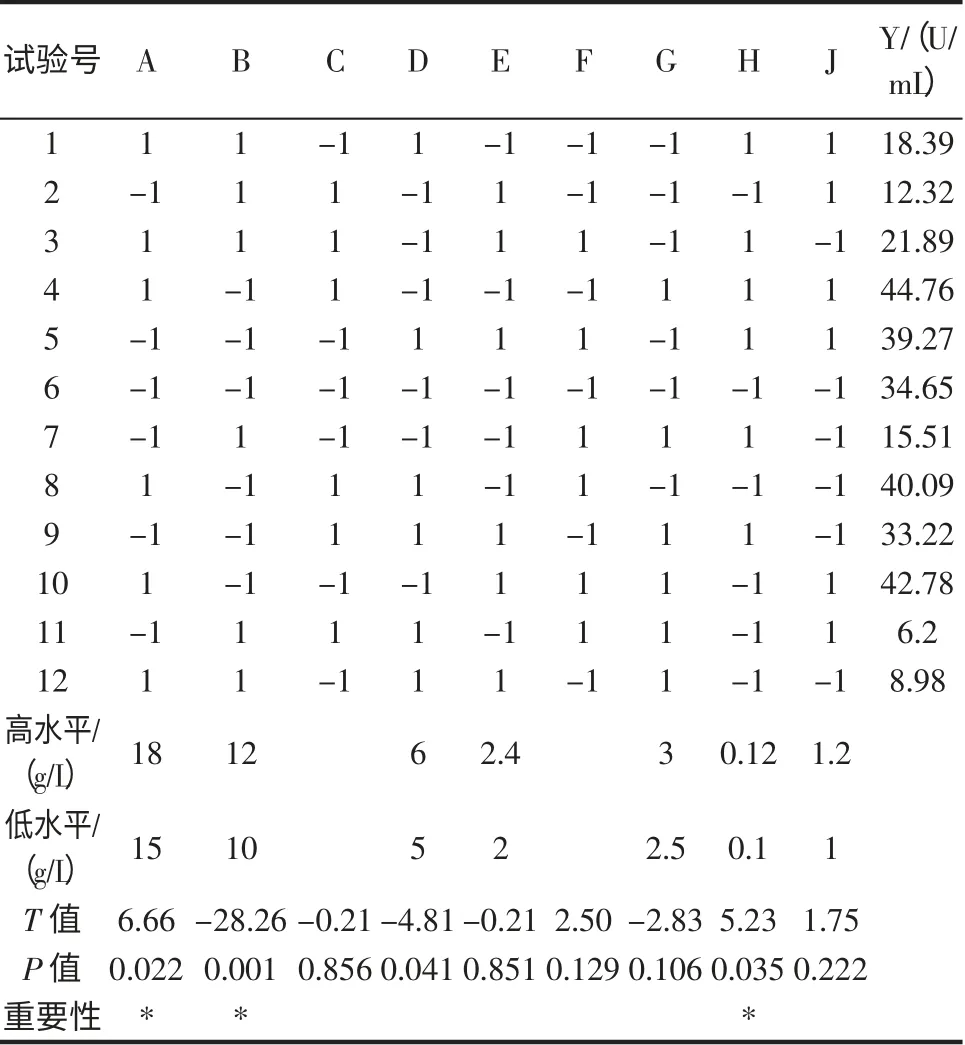
表1 Plackett-Burman 试验设计、相应值及结果分析Table 1 Experimental design,responses and results analysis of PB design
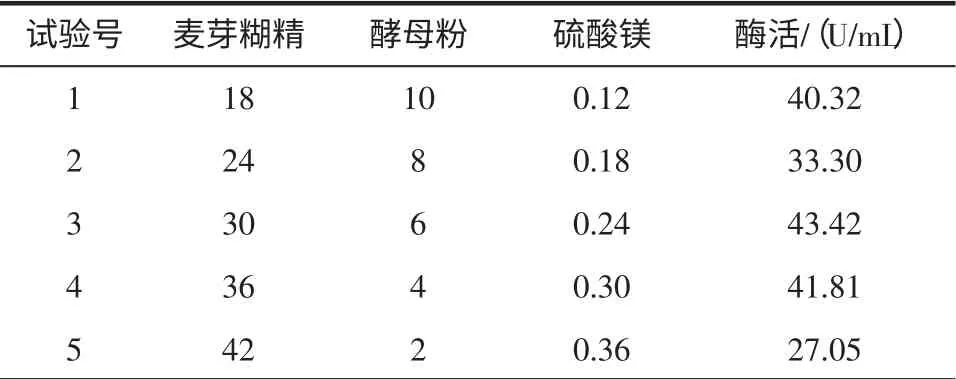
表2 快速登高试验设计与结果Table 2 Design and result of the steepest ascent experiment
由表2 可知,显著因素浓度在编号为3 试验的条件下,酶活最高。故以编号3 的条件作为响应面设计因素水平的中心点。即麦芽糊精、酵母粉和硫酸镁的浓度分别为30、6、0.24 g/L。
2.3 响应面法优化甲硫氨酸氨肽酶的发酵培养基
2.3.1 响应面设计与结果
根据响应面的Box-Benhnken 的中心组合设计原理,以酶活为影响值,设计三因素三水平共15 个试验点的响应面分析试验,其中12 个是析因点,三个位零点重复,零点试验重复三次,用以估计试验误差。响应面设计的因素与水平取值见表3,试验设计与结果见表4。
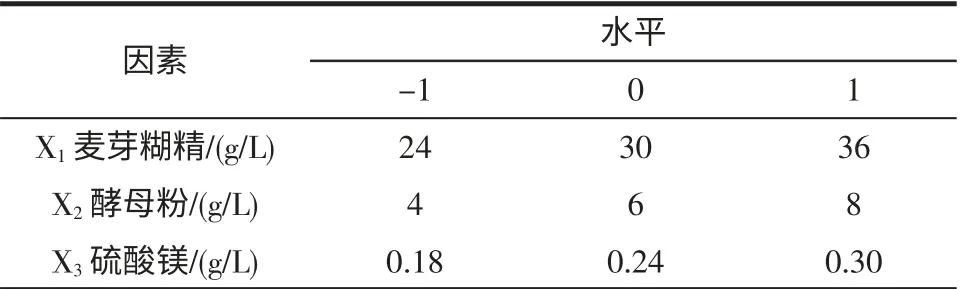
表3 响应面设计的因素编码与水平取值Table 3 The factor codes and levers of experiment of the RSM
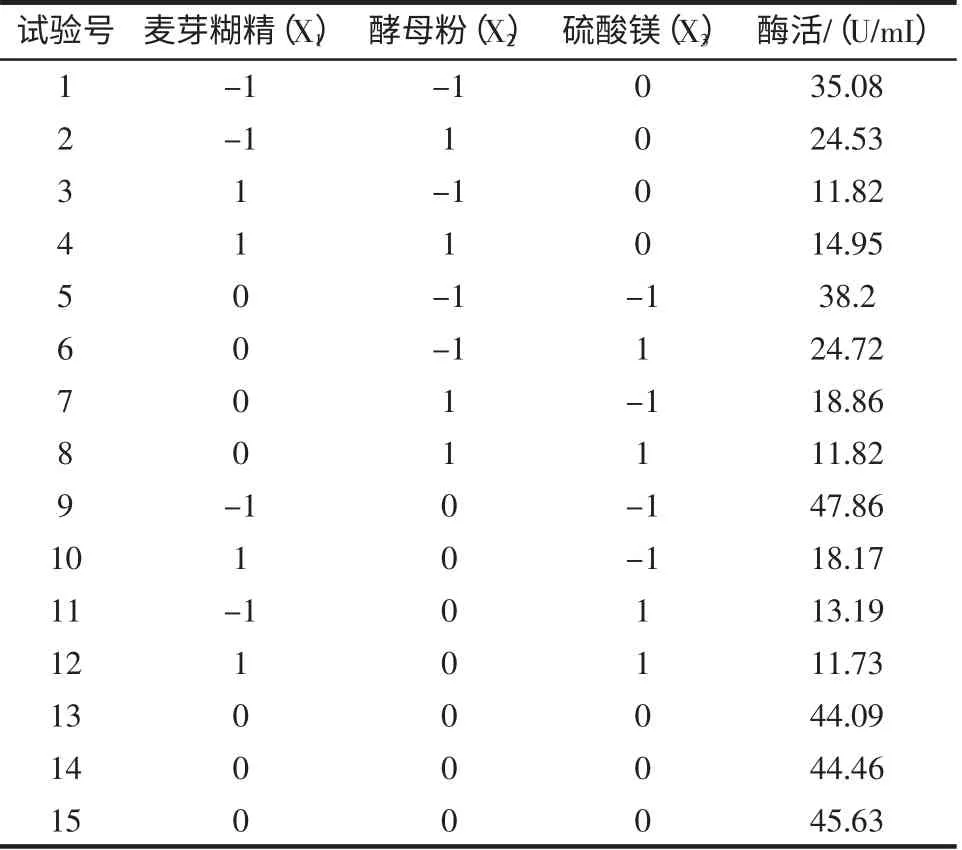
表4 响应面试验设计与结果Table 4 Design and result of the Response Surface Methodology experiment
2.3.2 二次回归拟合及方差分析
以酶活为响应值,运用Minitab 软件进行回归拟合,得到回归方程:Y=44.726 7-7.998 75X1-4.957 5X2-7.703 75X3-11.897 1X1X1-11.234 6X2X2-10.092 1X3X3+3.420X1X2+7.057 5X1X3+1.61X2X3。运用Minitab 软件对回归方程进行方差分析,结果见表5。
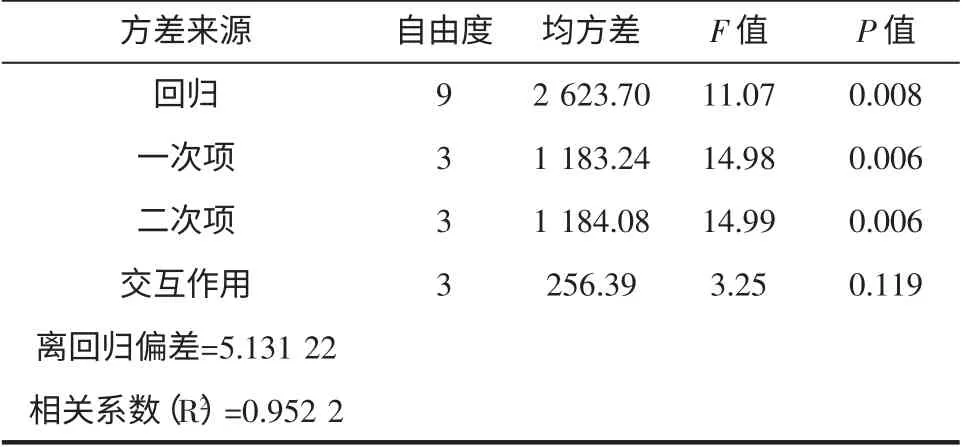
表5 回归方程的方差分析Table 5 Variance analysis of regression equation
由表5 可知,经F 检验,回归方程一次项、二次项显著,回归模型P 值=0.008,离回归偏差S=5.131 22,决定系数R2=0.952 2,说明此模型的可信度较高,关于甲硫氨酸氨肽酶的回归方程的拟合程度较好。
2.3.3 响应面分析及最优培养基成分的确定
对回归模型进行规范分析,结合Matlab 软件作出响应面分析图及等高线图,如图1 所示。
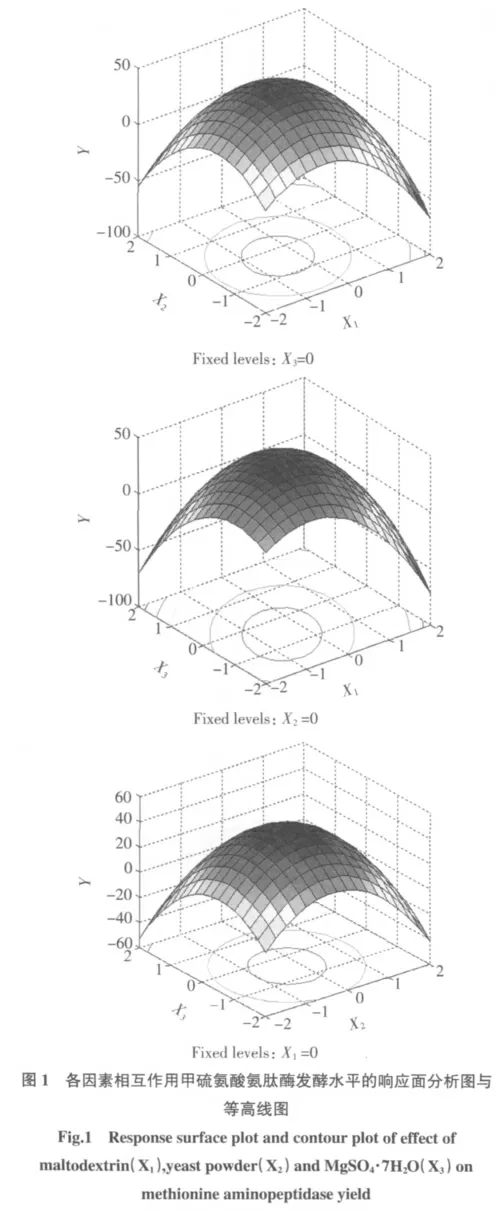
每个响应面分别代表2 个独立变量之间的相互作用。由响应面和相应的等高线可以看出,两两因素之间的相互作用比较明显。经过Matlab 分析,确定三个因素的最优试验点(X1,X2,X3)=(-0.567,-0.350,-0.608),代入回归方程,解得预测的甲硫氨酸氨肽酶的酶活为50.20 U/mL,根据三因素的取值,经过换算,得出三因素的最佳取值分别为X1=26.00 g/L,X2=5.3 g/L,X3=0.204 g/L。
为了证实预测的结果,用所得到的最优培养基配方进行三次重复发酵试验,所测得的甲硫氨酸氨肽酶活分别为49.87、50.89、51.78 U/mL,平均值50.84 U/mL,预测值与试验值相差极小,说明该模型能够较好地预测实际发酵情况。
3 结论
借助Minitab 软件能方便地应用PB 设计和响应面分析法进行合理的试验安排和数据分析,并利用该软件作出响应面分析图及等高线图得到了甲硫氨酸氨肽酶的最佳发酵培养基:麦芽糊精26 g/L,酵母粉5.3 g/L,蛋白胨6 g/L,牛肉膏2 g/L,磷酸二氢钾2.5 g/L,硫酸镁0.204 g/L,氯化钠1.2 g/L,pH 7.2。在此条件下对预测结果进行了验证,得到甲硫氨酸氨肽酶的酶活为50.84 U/mL,与优化前24.06 U/mL 相比,提高了1.11 倍,且预测值与试验值相差极小,表明该模型能够较好地预测实际发酵情况,适合多因素的发酵条件优化。
[1] Gonzales T,Baudouy J R.Bacterial aminopeptidases:properties and functions[J].FEMS Microbiology Reviews,1996,18(4):319-344
[2] Toldrá F,Aristoy M,Flores M. Contribution of muscle amino peptidases to flavor development in dry-cured ham[J].Food research international,2000,33(3/4):181-185
[3] 魏亚娟,田亚平,须瑛敏.枯草芽孢杆菌脱苦氨肽酶在水解大豆分离蛋白中的应用研究[J].食品工业科技,2008(4):149-151
[4] Barry C M,Cuinn G O. Debittering of a tryptic digest of bovine βcasein using porcine kidney general aminopeptidase and X-Prolydiptptidyl aminopeptidase from Lactococcus lactis subsp .Cremoris AM2[J].Journal of food science,2000,65(7):1145-1150
[5] Appelros S,Petersson U. Activation Peptide of Carboxypeptidase B and Anionic Trypsinogen as early Predictors of the Severity of Acute pancreatitis[J].British journal of surgery,2001,88(2):216-221
[6] Toldrá F,Aristoy M,Flores M. Contribution of muscle amino peptidases to flavor development in dry-cured ham[J].Food research international,2000,33(3/4):181-185
[7] Soliman N A,Berekaa M M,Abdel-fattah Y R. Polyglutamic acid(PGA)production by Bacillus sp.SAB-26:application of Plackett-Barman experimental design to evaluate culture requirements[J].Appl Microbiol Biot,2005,69(3):259-267
[8] Ceylan H,Kubilay S,Aktas N,et al.An approach for prediction of optimum reaction conditions for laccase-catalyzed bio-transformation of 1-naphthol by response surface methodology (RSM)[J].Bior-esour Technol,2008,99:2025-2031
[9] 范洪臣,李艳华,梁金钟,等.响应面法优化枯草芽孢杆菌产γ-PGA 的条件[J].生物加工工程,2008(5):17-23
[10] 田亚平, 须瑛敏.一种枯草芽孢杆菌氨肽酶的纯化及酶学性质[J].食品工业科技,2006,32(3):7-9
[11] 吴庆勋.氨肽酶高产菌的选育及发酵条件优化[D].无锡:江南大学生物工程学院,2010
