SSR-PCR和常规PCR检测不同地区并殖吸虫遗传变异的比较研究*
2011-09-26单小云楼宏强屠平光
单小云,楼宏强,胡 野,楼 景,屠平光,方 萍
并殖吸虫是重要的食源性人兽共患并殖吸虫病的病原体。全球已报告有50多种,我国报告的虫种多达29种,其中有些是同物异名,对于并殖吸虫虫种的独立性长期以来存在着较多争议[1]。近些年国内外学者从形态学、生活史、分子水平等方面探讨我国不同地区的并殖吸虫的遗传变异及分类,其中分子生物学技术的发展为并殖吸虫虫种分类研究提升到新的水平提供了条件。本研究采用简单重复序列锚定 PCR(simple sequence repeat-anchored PCR,SSR-PCR)对我国5个地区的并殖吸虫的遗传变异进行研究,并与常规PCR进行比较,以了解并殖吸虫不同地域株的遗传变异情况,为并殖吸虫的分子生物学技术分类提供依据。
1 材料与方法
1.1 标本及其镜下观察 从浙江金华、永嘉,福建平和、政和、周宁的并殖吸虫病流行区采集淡水蟹,以双筛水洗法分离到5地的溪蟹囊蚴。各地标本随机选取10个囊蚴于解剖镜下进行形态学观察,测量囊蚴大小、内外壁厚度,初步判断并殖吸虫虫种。
1.2 囊蚴基因组DNA的提取 按照Promega公司基因组DNA提取试剂盒(Wizard SV Genomic DNA Purification System)使用说明书提取成虫基因组DNA。
1.3 PCR扩增
1.3.1 引物设计
1.3.1.1 SSR-PCR 引物设计 参照文献[2-3]的方法,选用引物5’-(CA)8RY-3’,即5’-CACACACACACACACARY-3’,共18个碱基。
1.3.1.2 常规PCR引物设计 根据COI和ITS2基因序列[4],自行设计引物,COI基因引物序列为:上游5’-CCT GAT TTT GCC TGG GTT
TG-3’,下游5’-ACG ACG AAC CAC GTA TCA T-3’。ITS2基因引物序列为:上游5’-ATA TTG CGG CCA CGG GTT A-3’,下游5’-GGT ACT CAC GTC TGA TCC G-3’。
上述引物委托上海Invitrogen公司合成。
1.3.2 PCR反应体系和条件
1.3.2.1 SSR-PCR扩增反应体系及条件 参照文献[5-6]的方法,总反应体系:DNA 模板2.5μL、10×PCR缓冲液2.5μL、dNTP混合物(2.5mmol/L)2 μL、Ex Taq酶(5U/μL)0.25μL、引物(10μmol/L)0.5μL,加ddH2O至25μL。PCR反应条件为:95℃4min;94℃30s,54℃45s,72℃90s,共37个循环;72℃7min。
1.3.2.2 常规PCR扩增反应体系及条件 COI反应体系:模板DNA1μL,10×PCR缓冲液2.5μL,25mmol/L MgCl24μL,dNTP混合物(2.5mmol/L)2μL,上、下游引物(50pmol/μL)各0.25μL,Ex Taq酶(5U/μL)0.25μL,加ddH2O至25μL。反应条件为:94℃5min;94℃30s,55℃30s,72℃30s,共35个循环;72℃5min。ITS2基因反应体系和扩增反应条件同COI基因。
1.3.3 结果分析
1.3.3.1 电泳检测 用溴乙锭预染色的1.5%琼脂糖凝胶电泳检测PCR产物,用凝胶成像系统拍照并保存结果。
1.3.3.2 SSR-PCR条带分析 根据SSR-PCR扩增产物电泳结果,比较不同标本的条带,按Nei的方法[7]计算相似系数(Genetic Similary)和遗传距离(Genetic Distance)。遗传相似系统 GS=2Nij/(Ni+Nj),其中Nij指材料i和材料j共同所有的片段数,Ni+Nj指两个材料中出现的片段数目之和。遗传距离GD=1-GS。运用SPSS 13.0软件进行聚类分析。
1.3.3.3 常规PCR结果分析 常规PCR产物委托上海Invitrogen公司测序,COI和ITS2基因测序结果用BoiEdit软件进行同源性分析,用MEGA5.0软件的最小进化法(ME)构建种系发生树[8]。
2 结 果
2.1 囊蚴 除福建平和的并殖吸虫囊蚴从溪蟹的心脏组织分离外,其余均从肌肉组织中检出,形态呈圆形或近圆形。浙江金华、浙江永嘉、福建周宁的囊蚴大小为320±40μm,壁2层,厚约15~24μm;福建政和的囊蚴大小为430±10μm,内外壁厚度分别为13~15μm与3~5μm;福建平和的囊蚴大小420±20μm,囊内蚴体充满或有小空隙,蚴体多蜷曲成U形,排泄囊只达到腹吸盘。
2.2 SSR-PCR扩增和分析结果
2.2.1 SSR-PCR 扩增条带 并殖吸虫的 SSRPCR产物呈多态性,共出现18条不同条带,每个标本条带数量在3~6条之间,长度在200~1 000bp之间,见图1。
2.2.2 遗传距离和聚类分析 SSR-PCR扩增条带分析显示,5地并殖吸虫标本的遗传距离为0.3333-0.8667,见表1。根据树状图将不同地区的并殖吸虫分为3类:浙江金华、浙江永嘉、福建周宁为一类,其中浙江金华、浙江永嘉标本为一小类;福建政和和福建平和标本各为一类,见图2。
2.3 常规PCR扩增和分析结果
2.3.1 常规PCR扩增测序结果 测序显示,浙江金华、浙江永嘉、福建周宁、福建平和标本的COI基因为390碱基对,福建政和标本为388碱基对;浙江金华、浙江永嘉、福建周宁标本的ITS2基因为363碱基对,福建政和标本为361碱基对,福建平和为357碱基对,见图3。
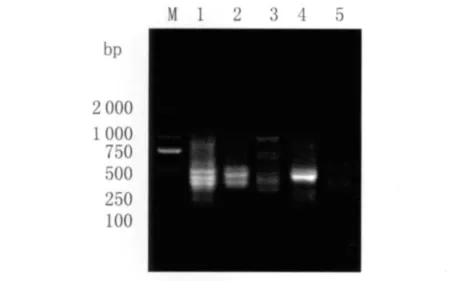
图1 不同地区并殖吸虫标本的SSR-PCR检测结果M:Marker;1:金华;2:永嘉;3:周宁;4:政和;5:平和Fig.1 SSR-PCR detection results of Paragonimus specimen in different areasM:DNA mark;1:Jinhua;2:Yongjia;3:Zhouning;4:Zhenghe;5:Pinghe
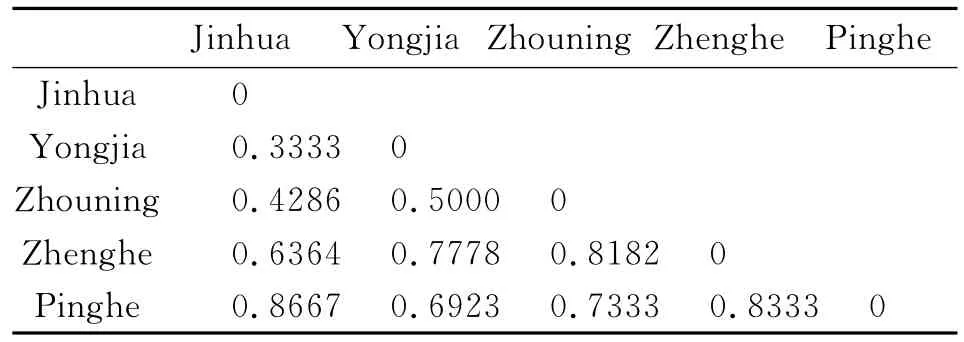
表1 根据SSR-PCR结果计算的不同地区并殖吸虫标本遗传距离Tab.1 The genetic distance of Paragonimusin different areas counted by SSR-PCR
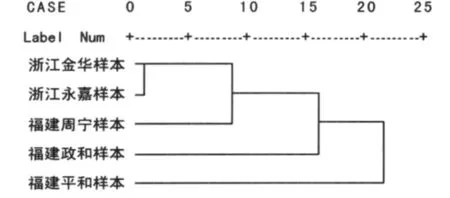
图2 根据SSR-PCR电泳图制作的不同地区并殖吸虫系统树图Fig.2 The dendrogram of Paragonimus in different areas made with the SSR-PCR electrophoretogram
2.3.2 同源性和遗传距离分析 BoiEdit软件分析显示,5个地区并殖吸虫标本的COI基因和ITS2基因的同源性分别为76.9%~98.9%和85.9%~99.4%,见表2和表3。
将MEGA格式排列的COI和ITS2序列分别输入MEGA建树,结果见图4,5。系统进化树将5地并殖吸虫在基因水平上分为3大类:浙江金华、浙江永嘉、福建周宁标本为一类,其中浙江金华、浙江永嘉标本为一小类;福建政和和福建平和标本各为一类。
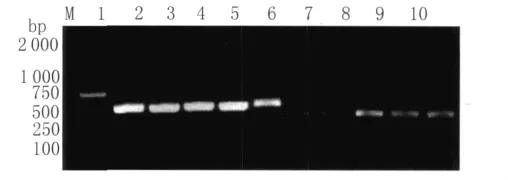
图3 不同地区并殖吸虫标本COI和ITS2基因的PCR检测结果M:Marker;1-5:金华、永嘉、周宁、政和、平和标本 COI基因;6-10:金华、永嘉、周宁、政和、平和标本的ITS2基因Fig.3 PCR detection results of COI and ITS2gene of Paragonimusin different areasM:marker;1-5:COI gene of specimen in Jinhua,Yongjia,Zhouning,Zhenghe,Pinghe;6-10:ITS2gene of specimen in Jinhua,Yongjia,Zhouning,Zhenghe,Pinghe
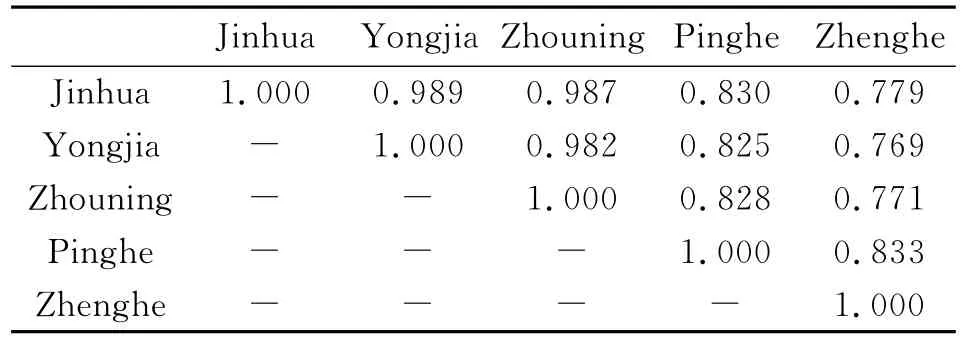
表2 不同地区并殖吸虫标本COI基因同源性比较Tab.2 A comparison of COI gene homology of Paragonimus in different areas
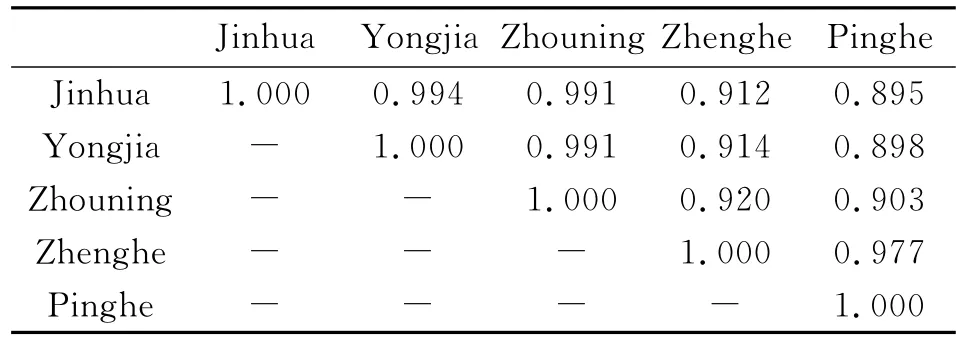
表3 不同地区并殖吸虫标本ITS2基因同源性比较Tab.3 A comparison of ITS2gene homology of Paragonimus in different areas
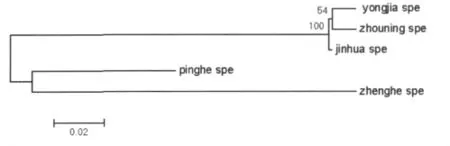
图4 不同地区并殖吸虫标本的COI基因种系发生树Fig.4 COI gene stammbaum of Paragonimusin different areas
3 讨 论
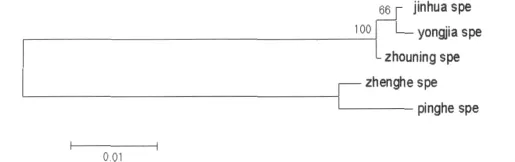
图5 不同地区并殖吸虫标本的ITS2基因种系发生树Fig.5 ITS2gene stammbaum of Paragonimusin different area
并殖吸虫的分类手段从19世纪后半叶以来以形态生活史为主要根据,上个世纪60-80年代出现了染色体、同工酶等技术[1],随着分子生物技术在20世纪90年代的快速发展,SSR-PCR被广泛应用于生物学各方面的研究,如法医鉴定、遗传病的基因诊断、肿瘤、发育生物学、分类学、种群研究等[5],特别是在枯氏锥虫、血吸虫、利什曼原虫等寄生虫分类学方面的研究较多[9],为SSR-PCR在并殖吸虫种群分类研究提供了基础。
本研究首先根据各地并殖吸虫囊蚴的形态特征和大小,结合当地流行病学资料,初步确认浙江金华、浙江永嘉、福建周宁的标本为卫氏并殖吸虫,福建政和的标本为斯氏并殖吸虫,福建平和的标本为三平正并殖吸虫。利用SSR-PCR结果计算出的遗传距离显示,浙江金华、浙江永嘉和福建周宁标本的遗传距离较近,为0.3333和0.4286;浙江金华与福建政和、福建平和遗传距离较远,为0.8333和0.8667,遗传距离的远近与囊蚴形态差异基本一致。作者利用常规PCR扩增了金华市并殖吸虫虫种的COI和ITS2基因,并与GNEBANK中已报道的卫氏并殖吸虫进行同源性和系统进化树的研究,从分子生物学角度证实了浙江金华标本为卫氏并殖吸虫[10]。本研究结果显示,浙江金华、浙江永嘉、福建周宁的标本COI基因和ITS2基因的同源性分别为98.2%~98.9%和99.1%~99.4%,可确定浙江金华、浙江永嘉和福建周宁标本为卫氏并殖吸虫。
虽然浙江金华、浙江永嘉和福建周宁标本为卫氏并殖吸虫,但SSR-PCR和常规PCR构建的系统进化树显示,浙江金华与永嘉标本的遗传距离比浙江金华与福建周宁、浙江永嘉与福建周宁标本之间的遗传距离更近,可能是因为浙江金华(东经119°38′,北纬29°04′)、浙江永嘉(东经120°41′,北纬28°09′)同福建周宁的地理位置相距较远,许多物理因素和生物因素如日照、大气湿度和温度都会影响到物种间的遗传差异。
由于微卫星DNA具有高度多态性、分布广、按孟德尔共显性方式遗传,SSR-PCR所用引物为标准化引物、不需要特殊设计,PCR扩增前也不需要预知模板 DNA 信息等优点[11-12],本研究利用 SSRPCR和常规PCR检测结果为基础绘制的树状图均将不同地区并殖吸虫分3大类:浙江金华、浙江永嘉和福建周宁为一类,福建政和、福建平和各为一类,浙江金华与浙江永嘉标本最近,浙江金华与福建政和最远。说明两种技术用于并殖吸虫分类的结果基本一致,而且与不同疫区囊蚴形态判定的不同虫种之差别相吻合。因此,SSR-PCR是一种比较方便可靠的并殖吸虫遗传变异研究方法,将为寄生虫鉴定与分类方面增加新的手段与方法。
(蒙华东地区肺吸虫病防治研究协作组李友松主任、陈韶红研究员指导,特此致谢。)
[1]沈一平,邵向云,李友松,等.实用肺吸虫病学[M].2版.北京:人民卫生出版社,2008:49-50.
[2]李莉,牛安欧,刘蓉,等.用SSR-PCR和RAPD技术研究中国不同地域旋毛虫的遗传变异[J].中国人兽共患病杂志,2004,20(9):757-761.
[3]Angela Cristina Volpini,Valéia Maria de Azeredo Passos,Alvaro JoséRomanha.Attempt to differentiate Leishmania (Leishmania)amazonensis,L.(L.)chagasi,Leishmania(Viannia)braziliensis and L.(V.)guyanensis using the SSR-PCR technique[J].Parasitol Res,2001,87 :1056-1059.
[4]Blair D,Agatsuma T,Watanobe T,etal.Geographical genetic structure within the human lung fluke,Paragonimus westermanni,detected from DNA sequence[J].Parasitology,1997,115(4):411-418.
[5]熊衍文,年安欧.微卫星锚定PCR及其在寄生虫学中的应用[J].中国寄生虫病防治杂志,2002,15(1):51-54.
[6]Zietkiewicz E,Rafalski A,Labuda D,etal.Genome fingerprinting by simple sequence repeat(SSR )-anchored polymerase chain reaction amplication[J].Genomic,1994,20 (2):176-183.
[7]Nei M,Li WH.Mathematical model for studying genetic variation in terms of restriction endonucleases[J].Proc Natl Acad Sci US A,1979,76(10):5269-5273.
[8]崔爱利,常正山,陈名刚,等.用DNA序列分析我国5种并殖吸虫的分类地位[J].中国寄生虫学与寄生虫病杂志,2003,21(1):27-30.
[9]Oliveira RP.An alternative approach to evaluating the intra specific genetic variability of parasites[J].Parasitol Today,1997,13(5):196-200.
[10]楼宏强,胡野,金耀建,等.浙江省金华地区并殖吸虫自然宿主调查及虫种鉴定[J].中国寄生虫学与寄生虫病杂志,2011,29(5):81-85.
[11]Litt M,Luty JA.A hypervariable microsatellite revealed by in vitro amplification of a dinucleotide repeat within the cardiac muscle actin gene[J].Am J Hum Genet,1989,44(3):397-401.
[12]Weber JL,May PE.Abundant class of human DNA polymorphisms which can be typed using the polymerase chain reaction[J].Am J Hum Genet,1989,44(3):388-396.
