基于苯酚降解的粪产碱杆菌Alcaligenes faecalis JF101的全基因组分析
2023-11-23张傲洁李青云宋文红颜少慧唐爱星刘幽燕
张傲洁 李青云,2 宋文红 颜少慧 唐爱星,2 刘幽燕,2
(1. 广西大学化学化工学院,南宁 530004;2. 广西生物炼制实验室,南宁 530004)
苯酚是一种常见的芳香族污染物,普遍存在于炼油、造纸、印刷、塑料等工业废水中[1-3]。目前处理苯酚废水的主要方法包括物理法、化学法、生物法以及这几种方法相互联用的处理技术,如利用活性炭载体固定微生物去除[4]。相比于其他处理方法,生物法成本较低,更具有可持续性,而且它能使苯酚完全矿化,是最清洁环保的处理方法,在废水处理中发挥着重要作用[5-6]。近年来,越来越多的苯酚降解微生物从污染地区分离出来,目前已知红球菌(Rhodococcus sp.)[7]、假单胞菌(Pseudomonas sp.)[7]、镰刀菌(Fusarium sp.)[8]、芽孢杆菌(Bacillus sp.)[9]和产碱杆菌(Alcaligenes sp.)[10]等微生物都能降解苯酚,其中产碱杆菌属对芳香族化合物表现出广泛的代谢多样性,对苯酚[11-12]、菲[13]、多环芳烃[14-15]、偶氮染料[16]、农药[17-18]等多种物质都具有降解能力。
已知微生物可以通过好氧和厌氧途径降解苯酚,其中好氧途径最为常见。在好氧降解途径中,苯酚首先被羟化生成邻苯二酚,然后经由otho‑(邻位)或者meta‑(对位)降解途径将邻苯二酚脱氢、开环,去除支链最后进入三羧酸循环(tricarboxylic acid cycle,TCA cycle)而被彻底降解[19-20]。苯酚的降解基因通常成簇排列,位于大质粒上或染色体上,编码苯酚降解的第一个酶为苯酚羟化酶(PH),它将苯酚羟基化为邻苯二酚后,由邻苯二酚1,2‑双加氧酶(C12O)和邻苯二酚2,3‑双加氧酶(C23O)将邻苯二酚通过otho‑或者meta‑途径开环裂解为TCA的无毒中间体[21-22]。大量研究表明,编码双加氧酶C12O和C23O的基因在不同的降酚菌株中具有高度的同源性,编码PH的基因一般高度保守,但是存在不同的转录调控机制[23-25]。Teramoto等[26]发现1株来自于降酚菌R5的苯酚羟化酶基因(Phc)中有3个调节蛋白参与转录,使得降酚菌R5表现出相对高的苯酚降解活性。Powlowski等[27]发现菌株Pseudomonas sp. CF600中的苯酚羟化酶与常见的细菌羟基化酚类化合物的黄素蛋白单加氧酶并不相同。同时还发现转录激活因子XylR促进C23O的活性比DmpR作用强,并且XylR可以代替DmpR并控制C23O酶的活性,以此来调节菌株对甲苯和二甲苯的降解[28]。因此,充分开展苯酚降解基因的研究对进一步深入认识苯酚生物降解机制及在此基础上开展定向代谢调控具有重要意义。
本课题组在前期的研究中分离得到1株能够降解苯酚的粪产碱杆菌—A. faecalis JF101,基于产碱菌属的代谢多样性,为了进一步深入了解并挖掘其生物学功能和基因资源,通过基因组测序和生物信息学方法,分析菌株基因组中与苯酚降解相关的功能基因;采用比较基因组学分析,比较菌株JF101与其他近缘菌株的基因组异同,解析JF101基因组中苯酚降解的合成和调控基因,为苯酚污染生物修复的大规模工业应用和高效降解工程菌的构建提供理论基础。
1 材料与方法
1.1 材料
粪产碱杆菌A. faecalis JF101(CMCC17153)由实验室前期分离获得,本实验室保藏。
1.2 方法
1.2.1 菌株JF101的培养及DNA的提取 菌株JF101使用Luria‑Bertani培养基摇瓶培养,30℃、160 r/min培养至静止期。基因组DNA通过参考天根细菌DNA试剂盒(DP302‑02)(天根生物科技(北京)有限公司)说明提取。
1.2.2 基因组测序、组装与注释 粪产碱杆菌JF101的基因组由Pacific Biosciences RS II平台和Illumina MiSeq平台确定。测序数据采用Pbdagcon(https://github.com/PacificBiosciences/pbdagcon)软件对模板序列(Subread)进行自我纠正,基于纠正的Reads采用Celera、Falcon软件组装,选择出最优组装结果。为了提高基因组序列的准确性,利用GATK(https://www.broadinstitute.org/gatk/)和SOap工具包对二代小片段数据进行单碱基纠正,得到高可信的组装序列后对组装结果进行序列成环判断、基因组序列及质粒序列区分。
采用Hidden Markov模型和GLIMER3(http://www.cbcb.umd.edu/software/glimmer/)软件预测JF101的组装结果,同时使用RNAmmer[29]软件、tRNAscan‑SE[30]软件、Infernal[31]软件、Rfam[32]数据库、TRF(Tandem Repeat Finder)[33]软件预测基因组中的rRNA、tRNA、sRNA和重复序列,并根据重复单元长度及数目筛选出其中的微卫星以及小卫星序列。
1.2.3 基因功能注释 各基因功能注释均使用Diamond软件结合各个数据库的特点完成,这些数据库包括COG(https://www.ncbi.nlm.nih.gov/research/cog‑project/)、GO(http://geneontology.org/docs/ontolo gy‑documentation/)、KEGG(https://www.kegg.jp/)等。
1.2.4 比较基因组分析 完成JF101基因组的测序与分析后,进一步将JF101与公布了完整的基因组信息且研究较深入的5株粪产碱杆菌A.faecalis.MUB14、A. faecalis.P156、A. faecalis.ZD02、ASM96730v2、A. faecalis.c16(以下简称MUB14、P156、ZD02、ASM96730v2、c16)进行比较基因组分析,基因组信息来自GenBank。本文通过6株菌株基因组的基本特征比较、基因家族统计及构建系统发育树对6株菌株进行了比较基因组分析。
系统发育树的建立,首先通过蛋白序列BLAST比对、solar去除冗余得到基因家族统计,随后使用Hcluster‑sg(https://anaconda.org/bioconda/hcluster_sg)软件对比对结果进行TreeFam聚类处理以及Muscle[29](http://www.drive5.com/muscle)软件对聚类的基因家族进行多序列比对,并将比对结果转化为CDS区域的氨基酸多序列比对结果,最后使用TreeBeST[34]软件对Muscle多序列比对结果采用NJ法进行单拷贝基因(Gene family模块)的建树分析。
1.2.5 基因功能挖掘 使用antisMASH(https://antismash.secondarymetabolites.org/#!/about)对JF101进行生物合成基因簇进行预测。
2 结果
2.1 粪产碱杆菌JF101基因组基本信息
粪产碱杆菌JF101二代测序共得到1 314 Mb原始数据,Reads长度150 bp,过滤后数据大小为1 310 Mb。三代测序得到的总碱基读数为7 410 174 502 bp,Reads N90为7 978 bp,Reads N50为12 788 bp。经过组装得到完整的基因组,大小为 4 143 816 bp,GC占比为57.44%,组装基因3 804个(图1)。RNA预测结果得知该菌含有55个tRNA,9个rRNA,5个sRNA。目前粪产碱杆菌JF101序列已提交至GenBank数据库,登录号为CP118772。
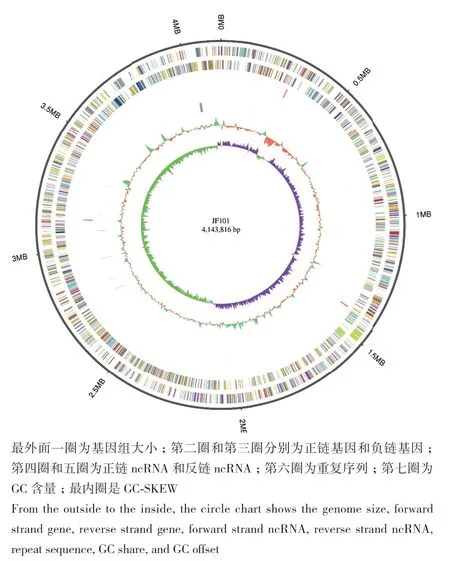
图1 粪产碱杆菌JF101基因组Fig. 1 Circular representation of Alcaligenes faecalis JF101 genome
2.2 基因功能注释分析
2.2.1 COG数据库注释 为了进一步了解JF101基因组的功能,将蛋白序列与COG、GO、KEGG等数据库进行比对获得对应的注释结果,并根据注释结果对蛋白功能归类分析。图2是基于COG数据库差异表达基因的生物学功能注释结果,菌株JF101的全基因组中有3 040个基因得到功能注释,占全基因序列的79.9%,其中cellular processes and signal transduction(细胞过程与信号传导)占21.32%、information processing and storage(信息处理及储存)占28.17%、metabolism(新陈代谢)占33.33%、function unidentified(未知功能)占17.18%。菌株基因组中参与新陈代谢类的基因最多,预示着其对多种污染物具有潜在的降解能力。在细胞过程与信号传导类注释的基因中,细胞壁、细胞膜的生物反应基因占比最大(5.46%),说明菌株JF101在生物膜形成方面具有较强的能力。
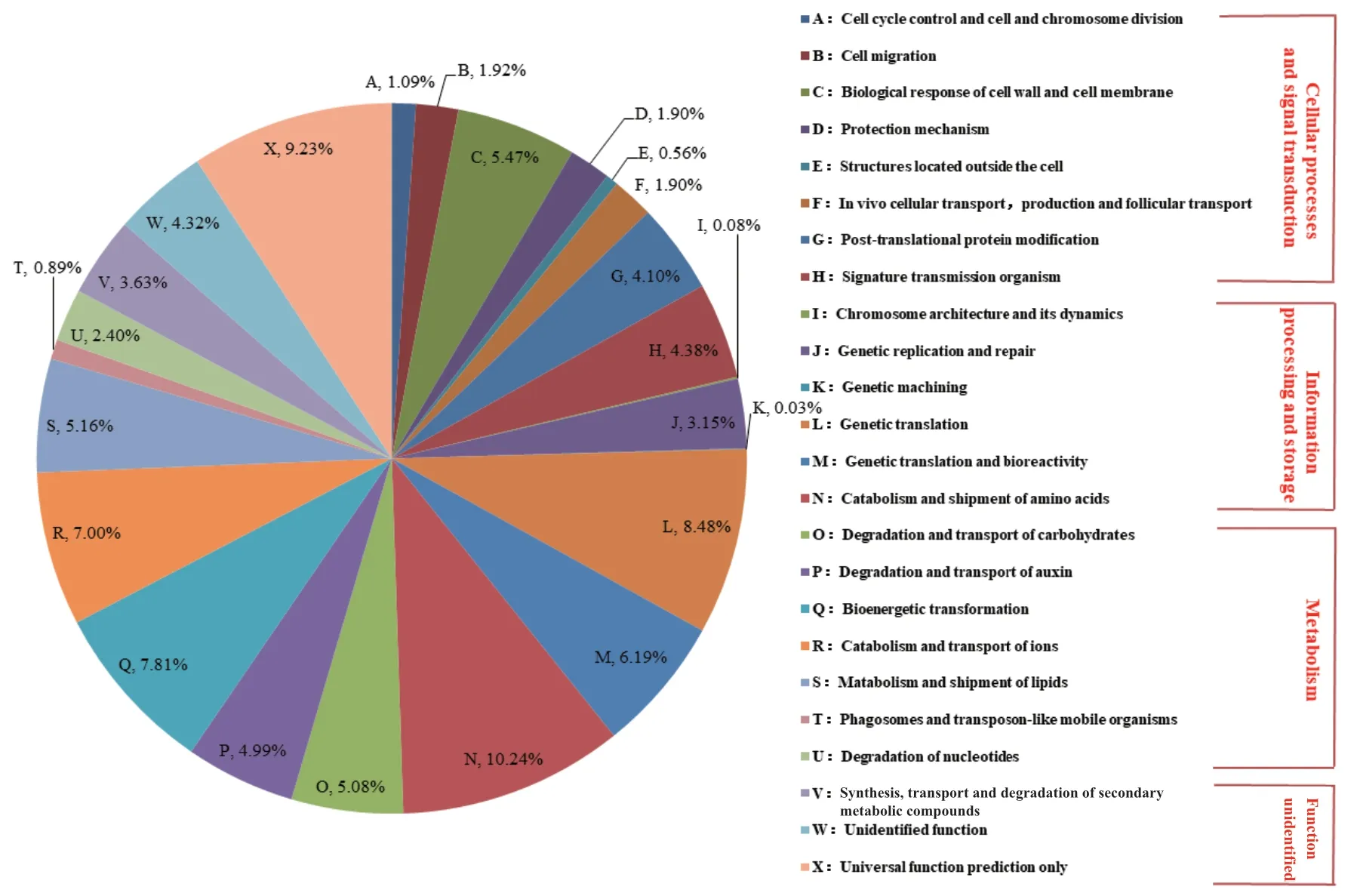
图2 粪产碱杆菌JF101 基因组 COG 功能注释分类Fig. 2 Functional classification of COG of A. faecalis JF101 genome
2.2.2 GO数据库注释 图3为基于GO数据库对基因功能进行cell component(细胞组分)、molecular function(分子功能)和biological process(生物过程)的注释分析,结果表明,菌株JF101有2 529个(占比66.48%)基因得到注释。分子功能注释基因中参与cytological processes(细胞过程)、catabolic processes(代谢流程)、microbial regulation(微生物调节)、positioning(定位)的基因占比较多,其中bioconjugation capacity(生物结合能力)和biocatalyt‑ic viability(生物催化活力)过程的基因在生物过程中的基因注释最多,预示着菌株在催化方面具有良好的潜力。细胞组分注释基因包括cellular anatomi‑cal entities(细胞解剖实体)、cellular intracellular(细胞内)和complex‑containing proteins(含复合体的蛋白质),其中cellular anatomical entities功能注释的基因数量最多。
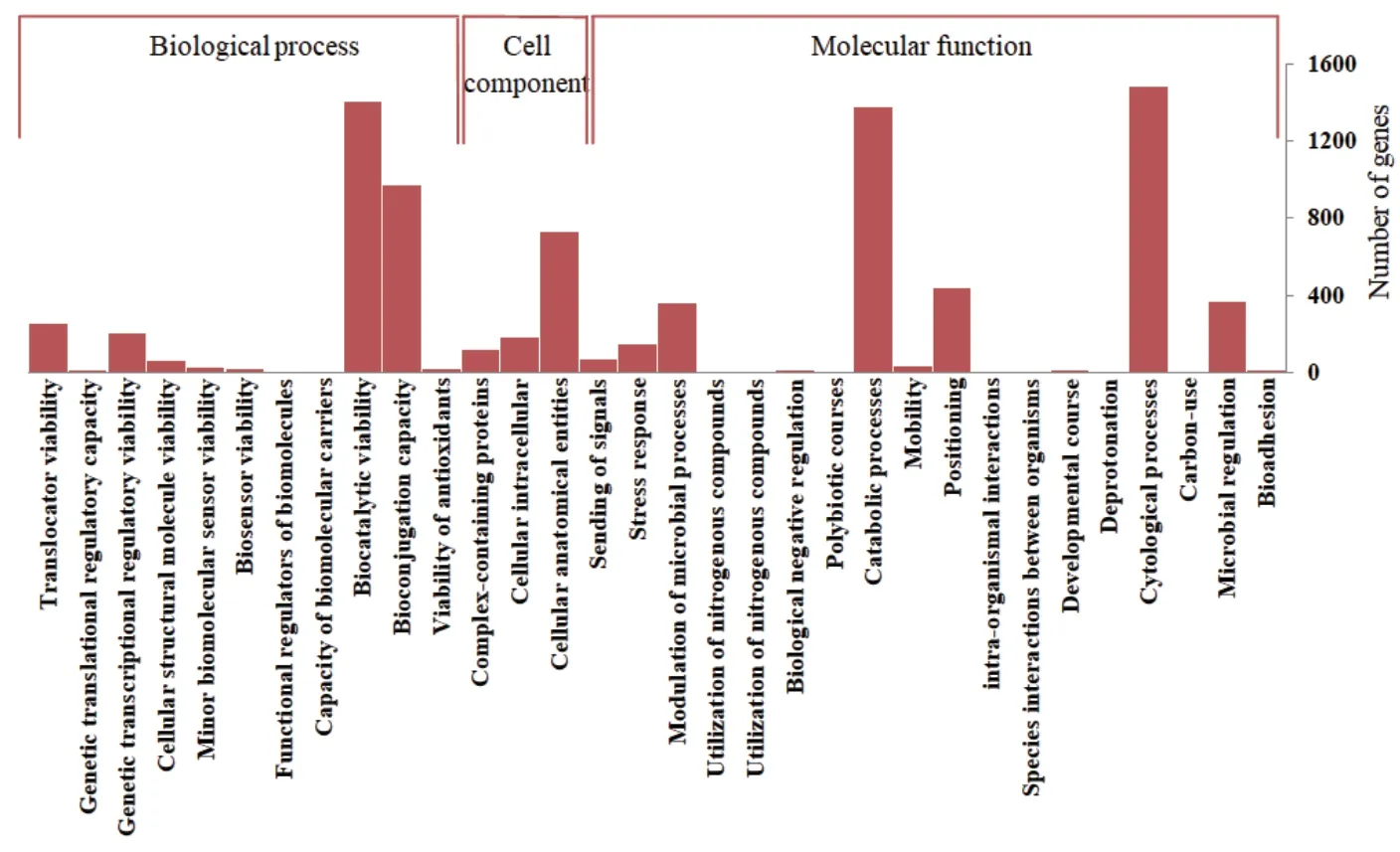
图3 粪产碱杆菌JF101 基因组GO功能注释分类Fig. 3 Functional classification of GO of A. faecalis JF101 genome
2.2.3 KEGG数据库注释 菌株有2 415个基因在KEGG数据库中得到功能注释,图4所示,在6大类的代谢通路中,注释出的基因主要归属于metabo‑lism(新陈代谢)、heredity(遗传)、environment(环境)和cell process(细胞过程)等相关通路。其中,新陈代谢通路中synthesis and metabolism of amino acids(氨基酸合成及代谢)、synthesis and metabolism of carbohydrates(碳水化合物合成及代谢)和general and outlined maps(全部概览)通路的基因得到较多注释,分别有264个、196个和760个。进一步分析发现,新陈代谢通路中有42个基因功能注释与芳香族化合物的降解相关,侧面说明菌株JF101具有潜在的降解苯酚的能力。在环境通路中,cellular transfer(膜转运)的注释基因最多,这与细胞的物质转运有着重要的联系。值得关注的是,在organic system(有机系统)通路中有关于adaptation of bacte‑ria to their environment(细菌对环境的适应性)的注释基因,预示着菌株JF101在酚类等有机化合物的环境中具有良好的适应性。

图4 粪产碱杆菌JF101 基因组KEGG功能注释分类Fig. 4 Functional classification of KEGG of A. faecalis JF101 genome
2.3 苯酚降解基因及其功能分析
对菌株JF101降解苯酚的关键蛋白及其功能进行注释与分析,结果显示菌株JF101有11个潜在的降解苯酚酶:GL‑1751、GL‑1752、GL‑1755、GL‑1756、GL1849、GL‑1850、GL1851、GL1852、GL1853、GL1854、GL‑1855。根据全基因序列的功能注释,GL‑1849为特异性调节蛋白,它对苯酚羟化酶基因的反向转录起到调节作用;GL‑1850、GL1851、GL1852、GL1853、GL1854、GL‑1855位点标记的分别是苯酚羟化酶P0、P1、P2、P3、P4、P5蛋白,其中P2为羟化酶,P4为还原酶,其他3个为调节蛋白,它们共同组成一个多组分PH(苯酚羟化酶),分别由基因dmp‑KLMNOP编码,参与苯酚的羟基化生成邻苯二酚。GL‑1752位点标记为C12O(邻苯二酚1,2‑双加氧酶),催化邻苯二酚转化为顺-顺粘糠酸,启动otho‑通路。粘糠酸环异构酶、粘内脂D‑异构酶和3‑氧代己二酸烯醇内酯酶分别由GL‑1751、GL‑1756、GL‑1755编码,这些基因参与顺-顺粘糠酸转化为(+)‑粘糠酸内脂、3‑氧乙酸烯醇内脂和丁二酸,最后进入三羧酸循环。
将上述11个参与苯酚降解的蛋白通过BLAST软件进行氨基酸序列比对,表1显示同源性最高的相关基因及菌株。在菌株JF101潜在的多组分PH中,GL‑1849由MOPR基因编码,该氨基酸序列与Sediminispirochaeta smaragdinae DSM 11293的MOPR氨基酸序列有47.9%的同源性,而PH(由dmp‑KLMNOP基因编码)与菌株Psychrobacter sp.和Alcaligenes faecalis的PH的同源性高达86.7%-100%。菌株JF101潜在的其他苯酚降解蛋白粘糠酸环异构酶、粘内脂D‑异构酶、3‑氧代己二酸烯醇内酯酶与比对蛋白具有很高的同源性(97%-100%),说明菌株JF101理论上通过otho‑途径降解苯酚。
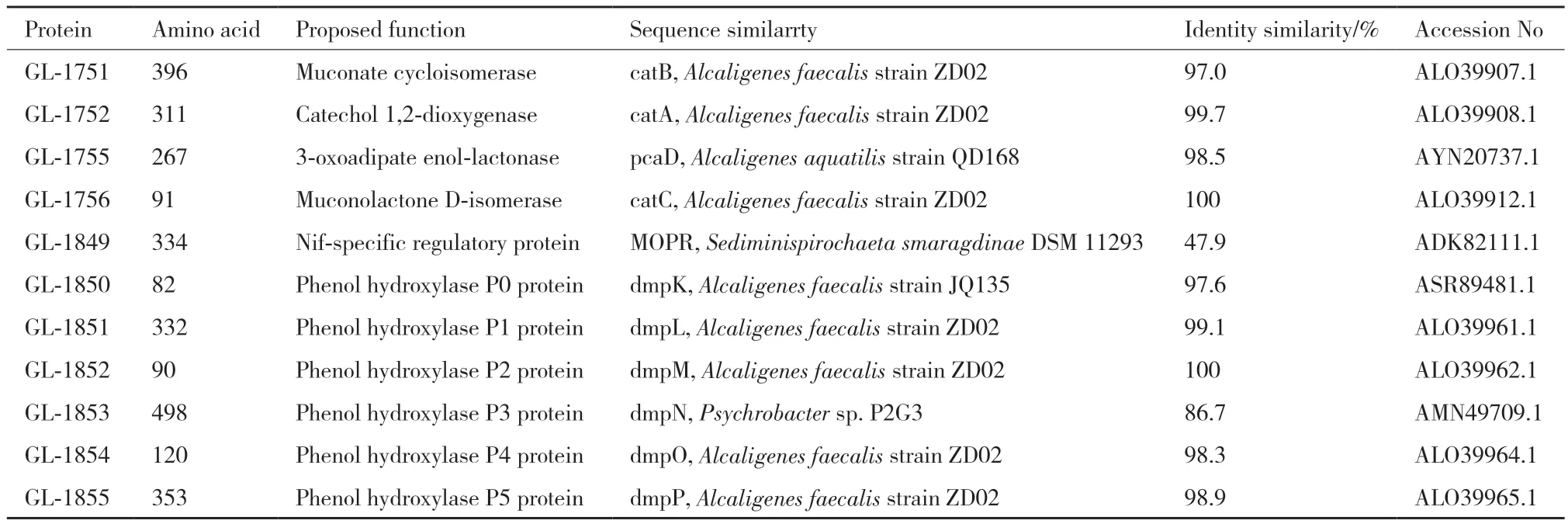
表1 粪产碱杆菌JF101苯酚降解相关基因与其他细菌同源基因的同源性比较Table 1 Homology comparison of phenol degradation-related genes of A. faecalis JF101 and homologous genes in other bacteria
2.4 比较基因组学
为了对比研究相同菌属菌株之间基因组的共性和差异性,选择数据库中公布了全基因组信息的5株粪产碱杆菌MUB14、P156、ZD02、c16和ASM96730v2进行比较分析,结果如表2所示。6株菌的基因组大小为4.04-4.35 Mb,其中基因组最大的为MUB14,最小的为P156,编码蛋白范围为:3 616-3 973个,GC含量范围为:56.3%-57.44%,rRNA与tRNA的数量相差不大。MUB14含有两个分子量分别为0.04 Mb和0.06 Mb的质粒,ZD02和ASM96730v2均含有一个0.01 Mb大小的质粒,而菌株JF101与P156和 c16这2株菌株没有质粒。
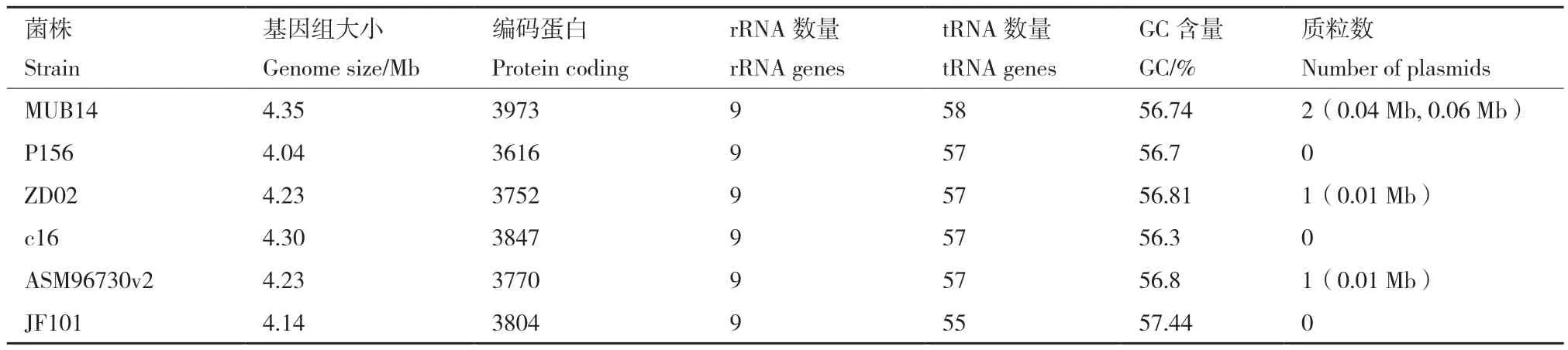
表2 粪产碱杆菌基因组的基本特征Table 2 Basic characteristics of the genome of A. faecalis
基因家族统计结果表明菌株JF101与5株粪产碱杆菌的共有同源基因家族有2 028个(图5),说明这些基因家族成员的功能具有相似性。同时,JF101、MUB14和c16拥有5个特有基因家族,其他3株菌株没有特有基因家族。基于Gene Family分析结果对上述6株粪产碱杆菌构建进化树(图6),粪产碱杆菌JF101与上述5株细菌都聚类到同一分支,说明这6株粪产碱杆菌有着共同的祖先,其中菌株JF101与c16的进化关系最近,它们之间的遗传变异度约为0.006,而与菌株MUB14、ZD02和ASM96730v2的遗传变异度较大,约为0.024。
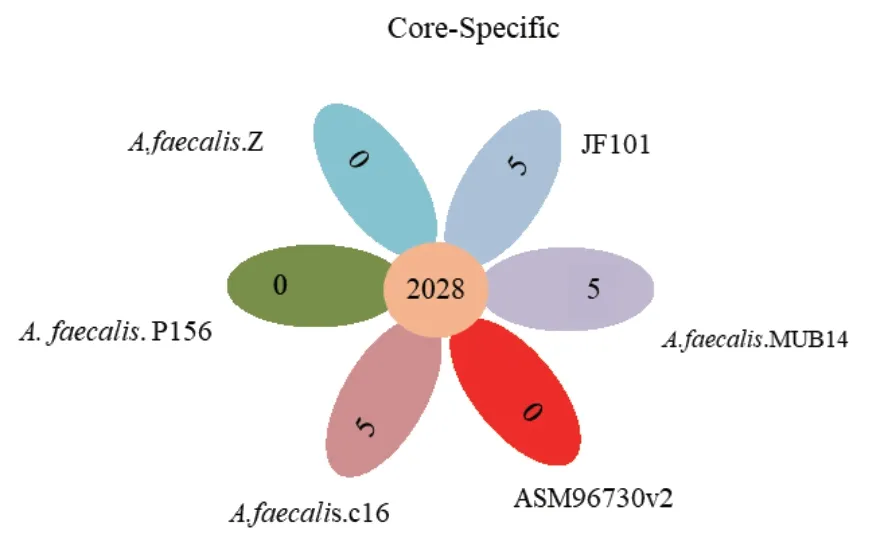
图5 同源基因家族数目Venn图Fig. 5 Venn diagram of the number of homologous gene families
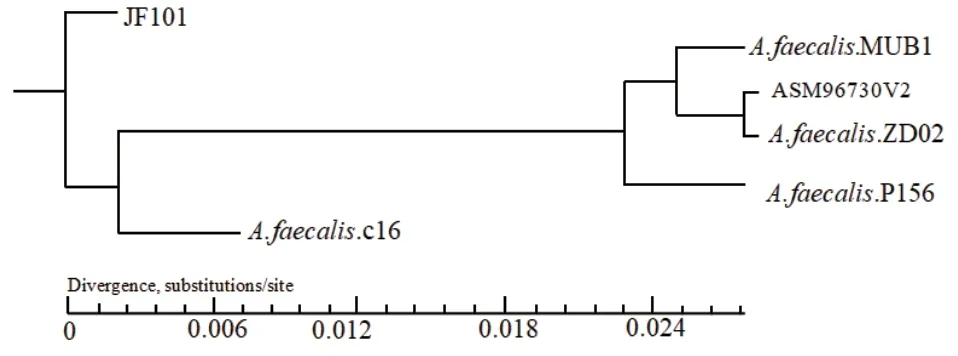
图6 系统发育树Fig. 6 Phylogenetic tree
2.5 菌株JF101潜在功能的挖掘
为了挖掘菌株JF101的耐盐功能,通过基因功能注释进行分析,表3汇总了与细胞内相容性溶质转运相关的基因。其中K+transport system(K+转运系统)包含Trk和Kdp系统,它们分别是组成型表达系统和转录调控型系统;Proline and betaine transport(脯氨酸和甜菜碱转运)系统包含ProP系统,ProP系统在高渗条件下对积累脯氨酸和甜菜碱发挥着重要的作用;choline/glycine/proline betaine transport genes(胆碱/甘氨酸/脯氨酸甜菜碱转运基因)包含betT和betS基因,这些基因表明菌株JF101具有潜在的耐盐能力。基于上述基因分析,本研究初步考察了菌株在盐条件下的降解情况。发现JF101在无盐和3% NaCl条件下,菌株10 h能完全降解50 mg/L苯酚,而在5% NaCl的条件下仍能获得32.4%的降解率,说明菌株JF101具有一定的耐盐性。
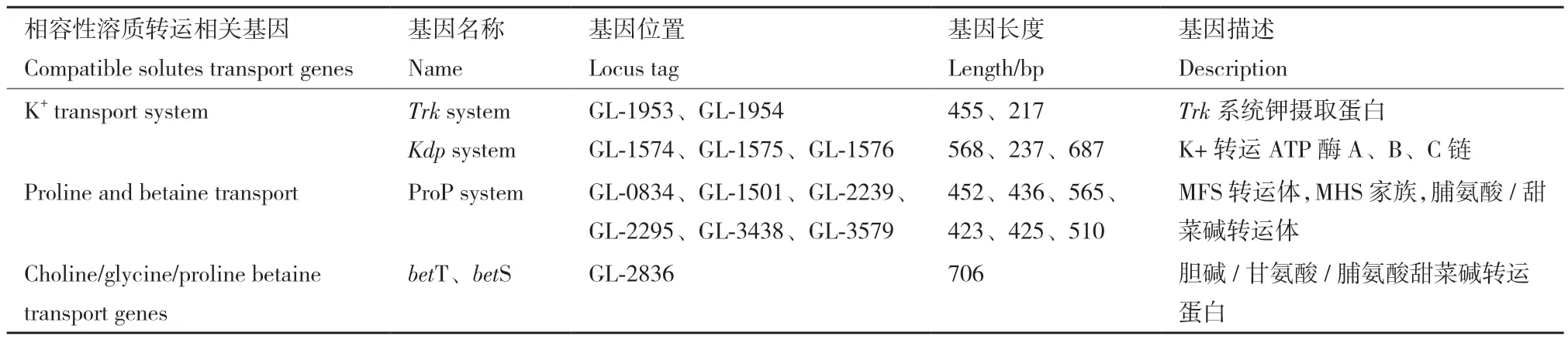
表3 菌株JF101与相容性溶质转运相关的耐盐基因Table 3 Salt-tolerant genes related to compatible solute transport in strain JF101
采用antisMASH分析的结果(图7)表明,菌株JF101潜在有细菌素类基因簇和萜类基因簇。其中JF101中细菌素基因簇与贝氏芽孢杆菌FZB42的杆菌素生物合成基因簇的相似性为60%,与埃尔吉芽孢杆菌B69的嗜铁素生物合成基因簇相似性为50%。另外菌株中萜类基因簇与金链球菌ATCC11523四氢嘧啶生物合成基因簇相似性最高,为75%。说明JF101具有合成杆菌素、嗜铁素和四氢嘧啶等物质的潜力。

图7 粪产碱杆菌JF101次级代谢产物合成基因簇的序列比较Fig. 7 Sequence comparison of the JF101 secondary metabolite synthesis gene cluster in A. faecalis JF101
3 讨论
生物降解是环境污染物转化的根本途径,本研究通过全基因组测序和生物信息学分析的方法,对1株粪产碱杆菌A.faecalis JF101进行了苯酚降解基因解析和比较基因组学分析,为进一步阐释苯酚降解机制以及开展工程应用奠定基础。JF101的基因组大小为4 143 816 bp,GC 含量为57.44%,共编码3 804个编码基因。在GO、COG、KEGG数据库中分别有2 529、3 040、2 415个蛋白序列被注释出了功能,在这些功能蛋白中被注释最多的单元与新陈代谢相关,预示着其对多种污染物具有潜在的降解能力。研究发现,生物体的膜结构会因为芳香族化合物的亲脂性引起疏水作用而受到破坏[35]。细胞过程与信号传导类注释基因中细胞壁和细胞膜的生物反应基因较多,说明菌株JF101在生物膜形成方面具有较强的能力,并且对芳香族化合物的毒害作用具有潜在的耐受能力。同时,KEGG数据库中注释了42个与芳香族化合物的降解相关的基因,表明JF101对芳香族化合物具有降解能力。
通过基因功能注释分析了菌株JF101潜在的苯酚降解酶,包括PH、C12O、粘糠酸环异构酶、粘内脂D‑异构酶和3‑氧代己二酸烯醇内酯酶,预示着菌株JF101通过otho‑途径降解苯酚。其中,由MOPR基因编码的调节蛋白GL‑1849是苯酚降解的正调控基因,此类正调控基因还包括dmpR、mphR、aphR等,它们编码的调控蛋白属于DmpR和XylR调控蛋白家族,能起始基因的转录[36-38]。而在C.testosteroni TA441和R5等苯酚降解菌中存在负调控基因aphS、phcS和phcT[39]。aphS位于正调控基因aphR基因的下游,苯酚存在时会完全抑制aphR蛋白激活的苯酚羟化酶的表达,使得菌株TA441不能以苯酚作为碳源生长[40]。苯酚羟化酶PH是启动苯酚生物降解的第一步关键酶,尽管PH基因一般高度保守,所有多组分PH都由6个亚基组成,分别由KLMNOP基因编码[41],但是不同的菌株可能存在转录调控机制上的差异,从而使得PH的活性产生不同,因此需要进一步开展菌株JF101的代谢调控研究。
JF101与同属的5株粪产碱杆菌基因组的基本特征存在一定的差异,表明不同菌株在进化过程中会发生基因丢失或者获得新的基因和质粒,使得菌株之间在基因水平上逐渐出现差异[42]。基因多样性是物种进化的结果,菌株JF101与5株粪产碱杆菌MUB14、P156、ZD02、c16和ASM96730v2具有2 028个共有同源基因家族,同源基因具有功能相似性[43]。根据文献报道,MUB14具有降解多种物质的基因,如三硝基甲苯、萘、双酚A等[44];P156能以烟酰胺作为唯一碳源生长[45];ZD02可作为根结线虫的生物杀虫剂[46],以及用于有机污染物和化学农药的降解[47];c16具有异养硝化和好氧反硝化能力,能去除高浓度的氨氮[48],由此推测菌株JF101在生物降解方面可能具有相似的功能或者可以作用相似的底物。在菌株JF101的KEGG功能注释分析中,发现新陈代谢通路潜在有15个与氨代谢和6个与萘降解的相关基因。质粒携带的遗传信息能赋予生物体某些生物学性状,有利于菌株在特定的环境条件下生存[49]。菌株JF101与菌株P156和菌株c16不含有质粒,可能不具有某些生物学形状,但是菌株JF101的特有基因数目多于其他5株菌株。在GO数据库分析中,特有基因和非共有基因主要集中于未知功能、转运蛋白活性等过程,预示着菌株JF101可能具有某些潜在的功能,例如对次级代谢产物基因簇的分析发现菌株对萜类、细菌素类物质有潜在的合成能力等。
研究表明,苯酚降解中间产物粘糠酸在高盐条件下易于累积,导致产物抑制使得苯酚降解速率降低[50],而大多数工业废水中都含有可溶性无机盐[51],大部分微生物在盐环境下常因渗透胁迫而受到抑制,甚至完全失活。面向实际废水盐条件而开展的功能挖掘,对进一步发展菌株JF101的工程应用非常有指导意义。通过全基因组分析,菌株JF101潜在有与细胞内相容性溶质转运相关的基因:Trk、Kdp、ProP系统和betT和betS基因,这些基因在高渗透压的刺激下会转运K+、脯氨酸、甜菜碱和胆碱等物质来调节细胞膨胀压力,使细胞能快速适应盐分波动从而抵抗外界的高盐环境。Tanudjaja等[52]发现嗜盐菌株Escherichia coli K‑12具有Trk型钾离子(K+)转运蛋白TrkG和TrkH,其中TrkH比TrkG更支持细胞生长,而TrkG能够补充大肠杆菌中trkh介导的K+吸收的损失。在嗜盐菌Halobacterium salinarum中,K+吸收系统编码的KdpFABC基因表达严格依赖于生长培养基中的K+浓度,当外源K+浓度为250 μmol/L-20 mmol/L时,kdpFABCQ操纵子呈中等水平表达;当胞外K+浓度为20-250 μmol/L时表达显著增加,并且在20 μmol/L时表达最强,说明K+浓度过高会抑制基因转录[53]。Souii等[54]发现甘氨酸甜菜碱可以保护Microbacterium metallidurans TL13免受高渗透胁迫,它主要由胆碱高亲和性转运系统(BetT),胆碱脱氢酶(BetA)和甜菜碱醛脱氢酶(BetB)进行作用合成。另外,试验初步证明了JF101具有一定的耐盐性,目前文献报道的降解苯酚的耐盐菌或者嗜盐菌有Arthrobacter sp.、Brachybacterium sp.、Halomonas sp.和Bacillus sp.[51],这些苯酚降解菌大多数能够在NaCl为1-10%的培养基中降解苯酚,如苯酚降解菌SASS1能够在0-4% NaCl浓度范围内将1 500 mg/L苯酚完全降解[55];Gong等[56]从沿海土壤分离出一株酵母菌SDP‑1能够在0-5% NaCl下有效去除苯酚;Zhang等[57]发现在3% NaCl下耐盐菌株SAS26可以在60 h内完全降解1 500 mg/L的苯酚,但是当盐度升高到4%-6%时苯酚降解率降低。然而,目前关于耐盐并且能够降解苯酚的粪产碱菌属鲜见报道,菌株JF101的耐盐功能为后续系统研究其耐盐特性及作用机制奠定了基础。同时,研究认为苯酚浓度和盐度都会影响相关调控蛋白和代谢基因的表达,因此,菌株JF101的苯酚降解机制和耐盐机制需要进一步深入的研究,以充分开发利用菌株的生物功能价值。
4 结论
本研究对粪产碱杆菌JF101进行了全基因组测序,获得该菌株的完整基因组信息,并对其进行了比较基因组学分析。在功能注释基因中筛选和鉴定了与苯酚降解相关的基因。与耐盐性质相关的相容性溶质转运基因的挖掘和初步试验证明了菌株JF101具有耐盐特性。
