链霉菌Streptomyces sp. FXP04全基因组测序分析
2023-11-23唐碧瑶付学鹏
唐碧瑶 付学鹏
(齐齐哈尔大学生命科学与农林学院,齐齐哈尔 161006)
链霉菌是具有复杂生长周期的有机化能、好氧型革兰氏阳性放线菌(G+),在系统分类上归为放线菌目,是放线菌中最为高等的一类菌群。链霉菌种类繁多,在陆地、海洋、极端环境以及一些生物体内都有广泛分布,其中主要分布在土壤环境中。其孢子抗逆性强、存活时间长,具有良好的分枝菌丝,能产生水溶性或脂溶性的色素。因为其复杂的生长发育周期,产生了多种具有复杂结构的天然代谢产物,其中产生大量的各式各样的抗生素,目前在临床应用的抗生素约 2/3 来源于链霉菌属[1],包括井岗霉素、万古霉素等。除抗生素外还包括抗肿瘤剂、杀虫剂、酶抑制剂、色素及酶类,如几丁质酶、果胶酶、木聚糖酶和蛋白酶等[2]。例如Arasu 等[3]从海洋中分离出Streptomyces sp. AP‑123链霉菌产生的聚酮类化合物对绿猴肾细胞和喉癌细胞有显著毒性。Kim等[4]研究发现沙门氏链霉菌菌株KP1404能产生多烯类抗生素,抑制番茄枯萎病菌菌丝的生长,其拮抗效果与苯菌灵相当。胡磊等[5]发现产水链霉菌和球孢链霉菌可明显抑制由核盘菌引起的油菜菌核病,球孢链霉菌的防治机理可能是产生几丁质酶部分分解油菜菌核。这些产物在医疗、农业、食品、化工、环保等领域都有巨大的发展潜力,对人类的生产生活至关重要。因此,近几十年来,关于链霉菌遗传发育及代谢调控的研究备受重视。
Streptomyces sp. FXP04是本课题组在健康马铃薯根际土壤中分离的链霉菌菌株。该菌对致病疫霉P.infestans有很强的拮抗效果,对于作物生长具有良好的防病和促生[6-7]作用,表现出了较好的应用前景,有作为生物农药的潜力。近年来,对于链霉菌的研究多集中在抗菌、促生、抗肿瘤等生物活性以及分离纯化的表层上,而利用基因组测序技术研究和预测微生物代谢产物的种类、结构和合成途径成为了发掘链霉菌属代谢产物的一种新方向。在自然情况下,链霉菌次级代谢产物的实际表达率并不高,应用效果大大缩减。所以利用全基因组分析有助于人们对这种具有多种次级代谢物生产能力的链霉菌进行更深层次的研究。全基因组测序是针对未知基因组序列的物种进行个体全部基因组测序,具有测序覆盖面广、准确性极高的技术优势[8]。在本研究中,利用Illumina HiSeq高通量测序平台对FXP04的全基因组进行了测序。同时,进行了基因预测和基因序列的功能注释以及次级代谢产物合成基因簇预测,利用生物信息学分析强调了天然产物生物合成在菌株应用中的重要性,比较基因组学为FXP04的进化提供了见解,为揭示FXP04生防作用机制、功能基因的挖掘与利用奠定基础。
1 材料与方法
1.1 Streptomyces sp. FXP04培养和基因组DNA的提取
FXP04菌株接种至Luria‑Bertani(LB)培养基活化,28℃培养 4 d,挑取菌丝转接至三角瓶液体培养基于 28℃、140 r/min 培养 4 d后,12 000 r/min 离心20 min 收集菌体送至华大基因公司进行de novo 测序分析。FXP04已于中国普通微生物菌种保藏管理中心进行保藏,保藏号为CGMCC No.16826。
1.2 全基因组测序
测序采用第三代 PacBio 平台与第二代 Illumina平台相结合的测序技术,得到的测序结果采用Glim‑mer软件对基因组进行编码基因预测;TRF(Tandem Repeat Finder)软件对串联重复序列预测;通过软件PHAST预测前噬菌体(Prophage);利用相关软件完成对非编码RNA的预测,包括RNAmmer软件预测rRNA、tRNAscan软件预测tRNA区域和tRNA的二级结构以及利用Infernal软件与Rfam数据库比对得到sRNA。测序结果再与GO(Gene Ontology)、KEGG(Kyoto Encyclopedia of Genes and Genomes)、COG(Cluster of Orthologous Groups of Proteins)、VFDB(Virulence Factor Database)、ARDB(Antibiotic Resistance Genes Database)、CAZy(Carbohydrate‑Active EnZymes Database)数据库进行比对,完成蛋白序列功能注释[9]。
1.3 比较基因组学分析
先通过FXP04的16S RNA序列比对,从中选取5种与其近缘关系相近且不同类型的模式菌株,从NCBI Genome上下载S. varsoviensis NRRL ISP‑5346(GCF_000718635.1),S. diacarni LHW51701(GCF_003323715.1),S. celluloflavus NRRL B‑2493(GCF_000720995.1),S. kasugaensis BCRC 12349(GCF_002261115.1),S. platensis DSM 40041(GCF_00211 9195.1)共5种模式菌株的全基因组序列,与FXP04全基因组序列一起进行以下比较基因组学分析。其中包括共有基因(Core Gene)和特有基因(Specif‑ic Gene)分析[10]、基因家族(Gene Family)分析[11-13],通过相关软件进行聚类分析,根据基因集分布情况,绘制热图和构建系统进化树。
1.4 次生代谢产物合成基因簇分析
将菌株FXP04的全基因序列采用antiSMASH(V6.1.1)软件(https://antismash.secondarymetabolites.org/#!/ start)在线预测次级代谢产物合成基因簇,并结合 NCBI BLAST 比对分析结果[14]。
2 结果
2.1 菌株FXP04全基因组概况
本研究利用PacBio与Illumina 平台相结合测序技术对FXP04基因组测序,共得到1 367 Mb的原始数据量,对原始数据过滤后得到1 141 Mb的数据量,K‑mer预估其基因组大小为4.89 Mb(K‑mer值为15,深度为37)。FXP04基因组大小为4 535 201 bp(图1)[6],共有5 037个基因,基因的总长度为3 674 928 bp,GC含量为72.95%,基因长度占基因组总长的81.03%。在菌株 FXP04的核基因组中,利用RNAmmer软件[15]和tRNAscan软件[16]对FXP04基因组的ncRNA进行预测分析,其中 rRNA有 3 种类型:3个5S rRNA、3个16S rRNA、3个23S rRNA,33个tRNA以及23个sRNA;利用 TRF软件[17]对FXP04基因组的重复序列进行搜寻,结果显示FXP04共有1 699个串联重复序列,总长度为77 244 bp,大小为4-672 bp,其中含1 284个小卫星 DNA,含188个微卫星DNA;利用 PHAST软件[18]对基因组进行前噬菌体预测的结果仅得到一个可疑的前噬菌体,其长度为34 291 bp。
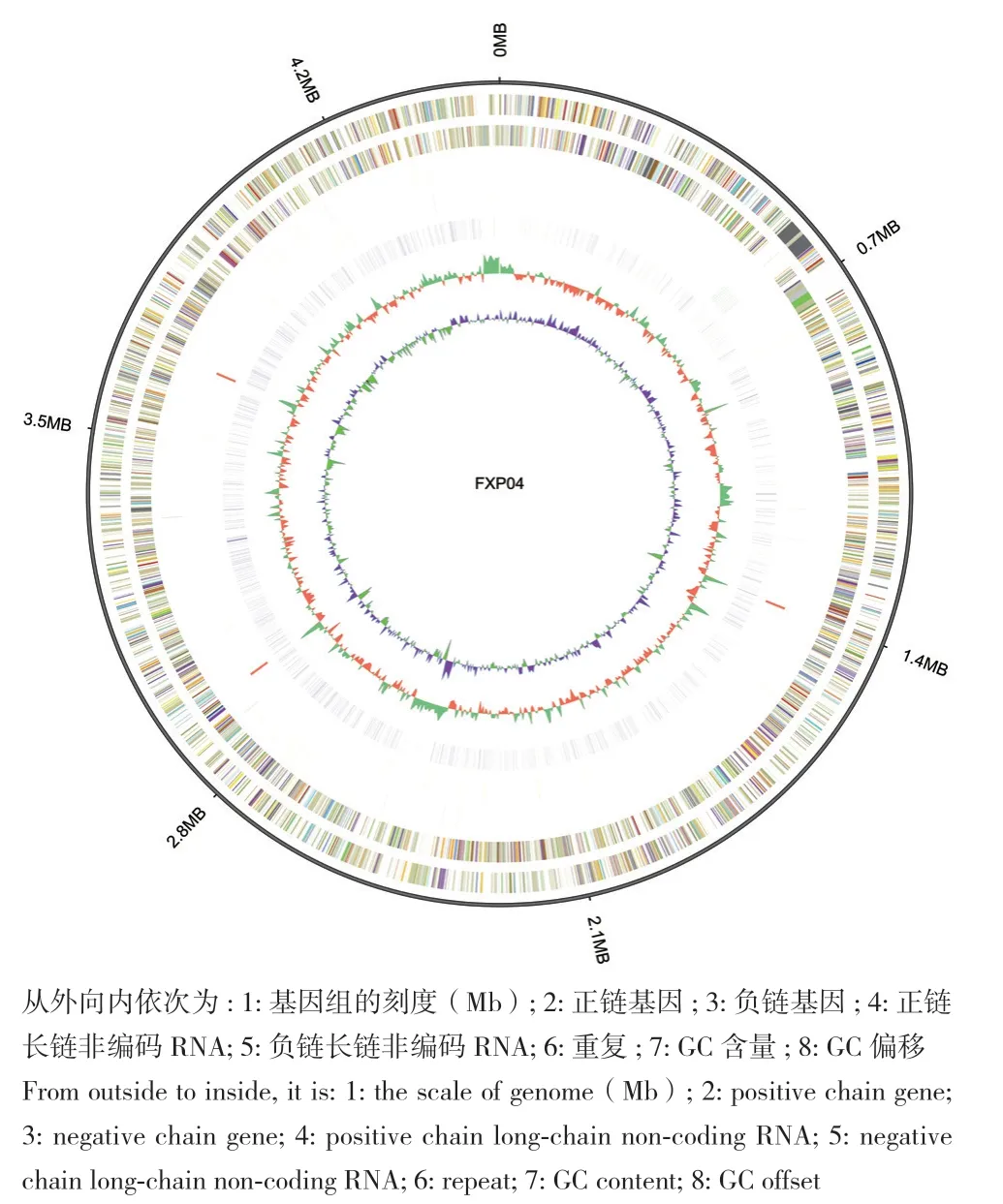
图1 菌株FXP04的全基因组Fig. 1 Whole genome of strain FXP04
2.2 基因功能注释
2.2.1 VFDB数据库注释 将菌株FXP04的基因组与VFDB数据库进行比对分析,在FXP04的全基因组中共发现139个毒力相关基因,其中最大基因序列为1 999 bp,最小基因序列为41 bp(表1)。通过比对发现基因序列主要在PDIM(二霉酚)和PGL(酚类糖脂)的生物合成和运输(ddrA、pks15、ppsC基因)、ABC转运蛋白(irtA、fbpC、fagA/B/C/D基因)、DevR/S双组分系统(devR/dosR基因)、PhoP/R双组分系统(phoP/R基因)4种功能中有较高的覆盖度。ddrA基因编码ABC转运蛋白参与抗肿瘤药物柔红霉素合成,pks15基因编码聚酮合酶,ppsC基因编码β-酮酰基合酶;fbpC、fagA/B/C/D基因与铁转运系统相关,irtA基因编码跨膜ATP结合蛋白帮助药物转运进入细胞;devR/dosR基因和phoP/R基因都为双组分系统调控因子,综上说明菌株FXP04所注释到的139个基因序列并非真正的毒力基因,且identity(一致性)≤70%,所以我们推测FXP04在与病原菌对抗中通过控制某些基因调节自身或共生生物体的生物学过程来发挥作用,也表明了FXP04被用作生物防治时是安全无害的。
2.2.2 ARDB数据库注释 通过ARDB数据库的注释可了解FXP04基因的潜在的抗生素耐受性,通过注释结果找出了14个耐药基因,基因长度介于100-480 bp之间,耐受药物嘌呤霉素(Puromycin)、杆菌肽(Bacitracin)、氨基糖苷(Aminoglycoside)、四烯霉素c(Tetracenomycin_c)、抗菌物(Na_Antimicrobials)、氯霉素(Chloramphenicol)、链霉素(Streptomycin)、万古霉素(Vancomycin)共8种,具体预测结果见表2。
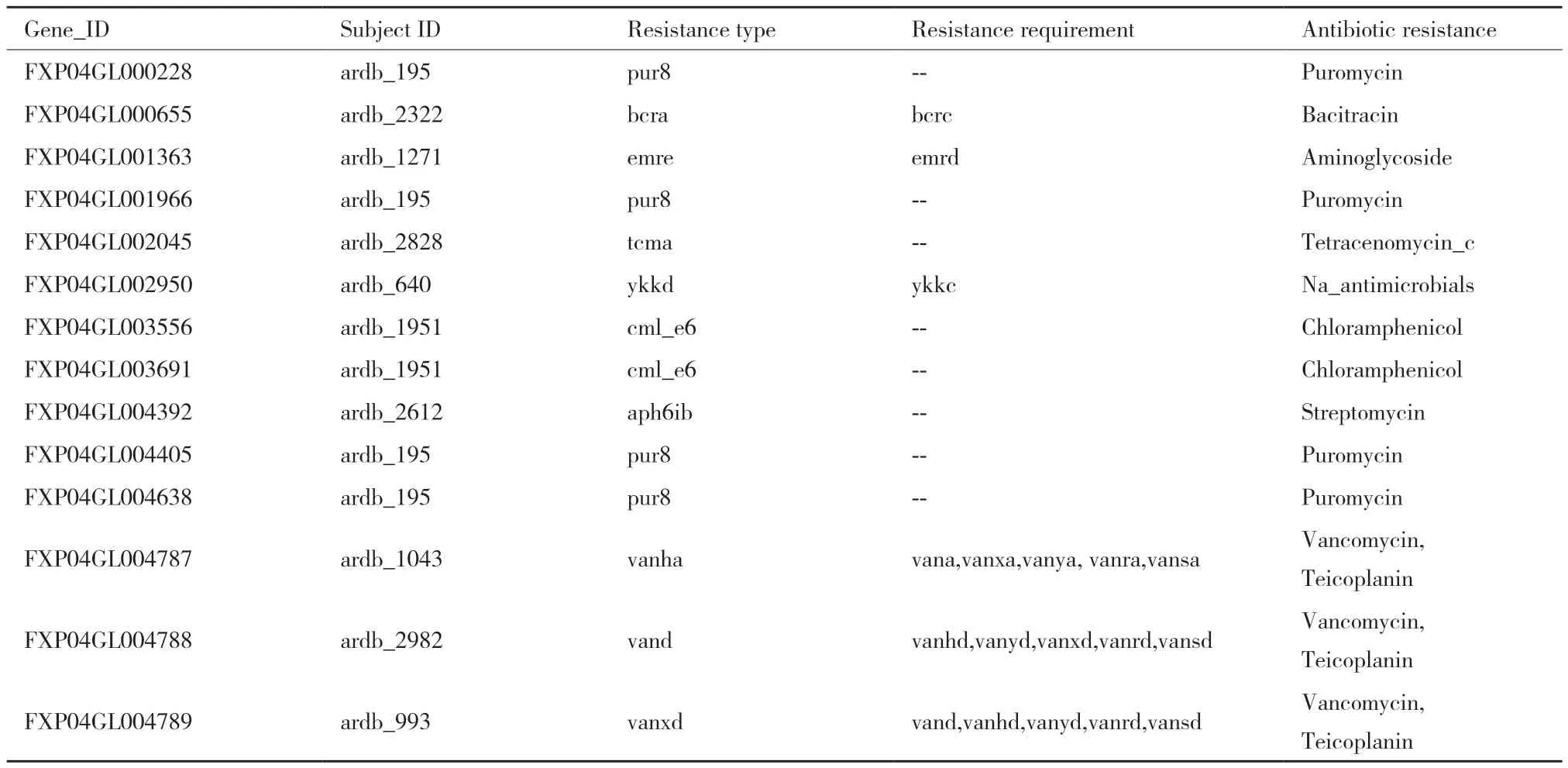
表2 菌株FXP04在ARDB数据库注释结果Table 2 Annotation results of strain FXP04 in ARDB database
2.2.3 CAZy 数据库注释 将基因组序列与 CAZy 数据库进行比对,发现菌株 FXP04的基因组中共有73个编码蛋白结构域归于 CAZy 家族,其蛋白编码基因分布状况如下:糖苷水解酶(glycoside hydrolases,GHs)基因27个,糖类酯解酶(car bohydrate esterases, CEs)基因6个,糖基转移酶(glycosyltransferases, GTs)基因12个,碳水化合物酶相关模块(carbohydrate‑binding modules, CBMs)基因18个,辅助功能酶(auxiliary activities, AAs)基因7个,多糖裂合酶(polysaccharide lyases, PLs)基因3个。在糖苷水解酶中,覆盖度最高的主要是葡聚糖酶(EC 3.2.1.70)。在碳水化合物酶相关模块中,几丁质酶(EC 3.2.1.14)、β-1,4‑木聚糖内切酶(EC 3.2.1.8)等有较高的覆盖度。在糖基转移酶中,磷壁酸酶(EC 2.4.1.129)有着较高覆盖度。在糖类酯解酶中D‑葡萄糖醛酸酶(EC 3.1.1.‑)也有比较高覆盖度。
2.2.4 GO数据库注释 FXP04通过GO注释,共有2 088个基因在GO数据库中被注释到,按照GO数据库的分类方式,FXP04的基因组功能被分为31个分支(图2)。其中有9个跟细胞组分有关,12个与生物学过程有关,10个与分子功能有关。在细胞组分中共有731个基因被注释,其中与细胞膜、细胞膜部分有关的基因表现出最高相关性,各有306、160个;在生物学过程中共3 867个基因被注释,与其细胞过程、代谢过程以及单有机体过程的基因表现出很高的相关性,各有831、1 193、752个;分子功能分支共2 531个注释结果,其中涉及最多的基因与结合(binding)、催化活性(catalytic activity)有关,分别为925、1 261个。
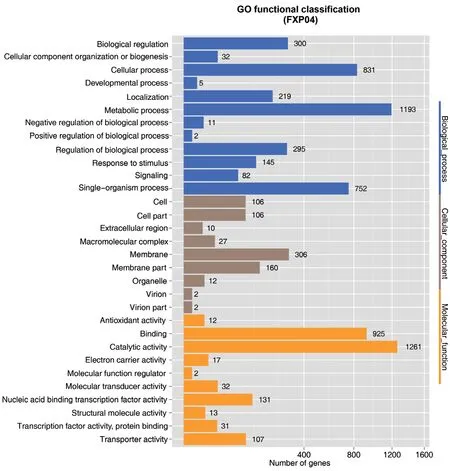
图2 菌株FXP04基因组的GO功能分类图Fig. 2 GO functional classification map of strain FXP04 genome
2.2.5 COG数据库注释 将FXP04基因组中能编码蛋白的基因与COG进行比对,得到FXP04的COG 功能数据分析结果如图3所示。在 FXP04菌株有2 319个可以在 COG 数据库中找到分类信息,包含COG数据库功能的23类。主要涉及 General function prediction only(通用功能预测)345个,占功能注释基因的14.88%;Transcription(转录)371个,占功能注释基因的16%;Amino acid transport and metabolism(氨基酸的运输与代谢)229个,占功能注释基因的9.87%;Carbohydrate transport and metabolism(碳水化合物的运输与代谢)205个,占功能注释基因的8.84%;Signal transduction mechanisms(信号转导结构)198个,占功能注释基因的8.54%,不同类型的基因执行功能有所侧重。
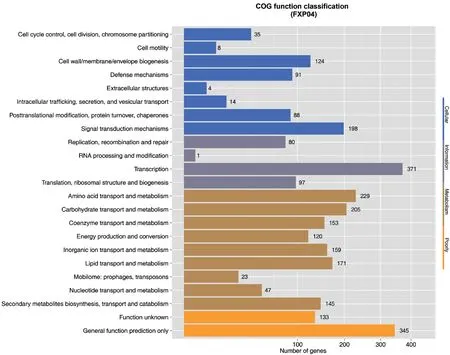
图3 菌株FXP04基因组的COG功能分类图Fig. 3 COG functional classification diagram of the genome of strain FXP04
2.2.6 KEGG数据库注释 FXP04菌株共有1 530个基因在KEGG中被注释,被分为6个分类31个功能条目(图4)。基因数量占比最多的是与新陈代谢相关的。在KEGG注释的代谢中,参与概述的基因有535个,辅因子和维生素的代谢130个,氨基酸代谢182个,碳水化合物代谢175个。
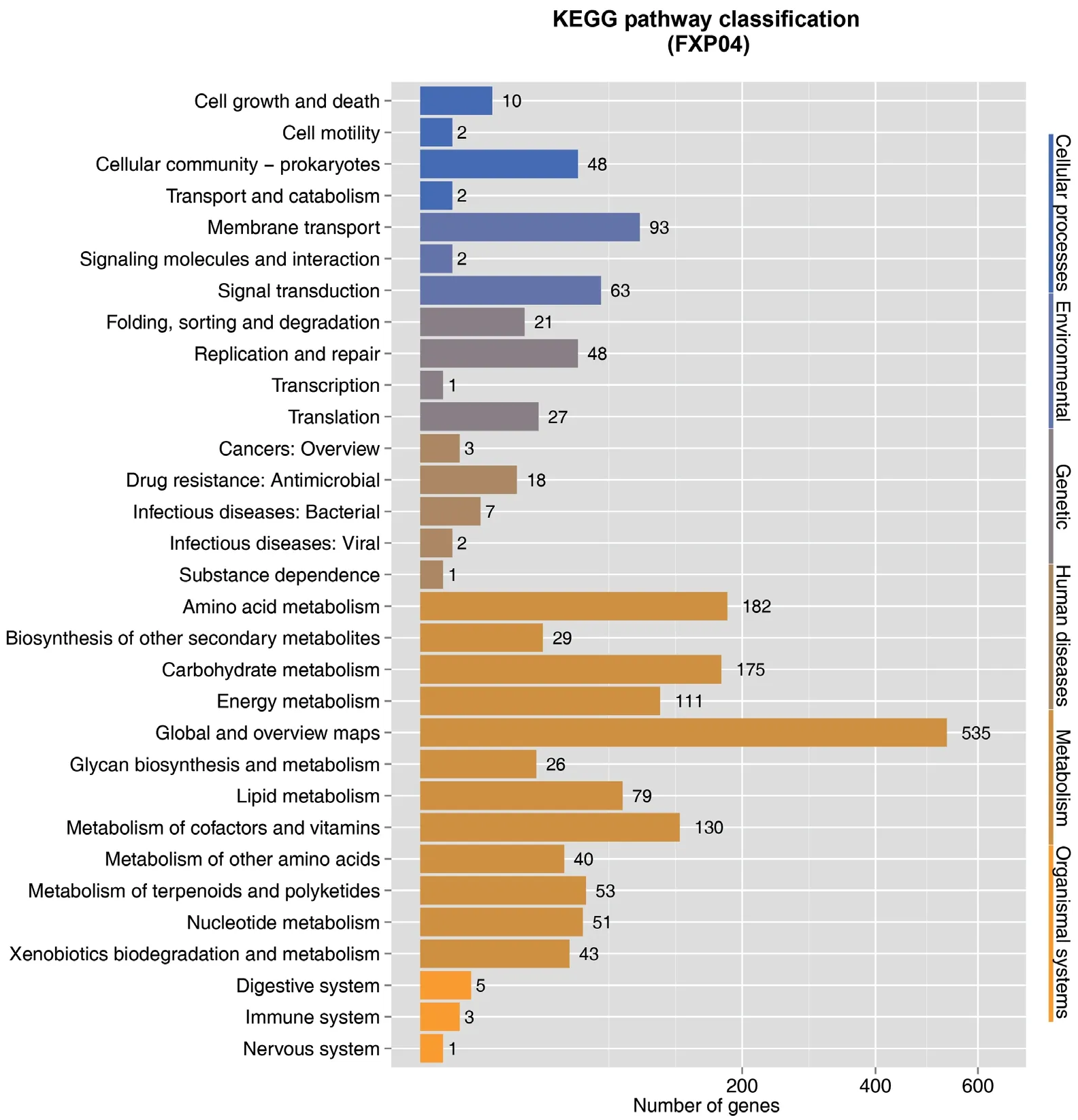
图4 菌株FXP04基因组的KEGG功能分类图Fig. 4 KEGG functional classification diagram of FXP04 genome
2.3 比较基因组学分析
2.3.1 共有基因和特有基因分析 通过 Core/Pan分析发现,FXP04与5个模式菌株的所有非冗余的 Pan Gene 数量为18 057个,长度为 5 710 958 bp;其中共有的Core Gene 数量为532 个,长度为180 719 bp;非共有的Dispensable Gene 数量为6 778个(图5‑B),长度为2 315 398 bp。通过Dispensable Gene同源关系热图(图5‑A)和Core Gene系统发育树(图5‑C)可看出,FXP04的基因组与S. varsoviensis NRRLISP‑5346最为相似,其次是S. diacarni LHW51701和S. platensis DSM40041,与另外2 株模式菌株具有明显的遗传距离。

图5 6个菌株之间基因组Core/Pan 分析结果Fig. 5 Genome Core/Pan analysis results among 6 strains
2.3.2 基因家族分析 基于6个菌株全基因组序列的 Gene Family分析发现,FXP04与S. varsoviensis NRRLISP‑5346、S. diacarni LHW51701、S. platensis DSM40041等 6个菌株能够聚类到Gene Family 的基因数量分别为 3 010 个、5 827个、5 287 个、5 974个、6 692个和6 376个,Gene Family 数量分别为 1 437个、2 469 个、2 294个、2 613 个、3 101个和3 019个;其中共有的 Gene Family 数量为 871个,各菌株特有的 Gene Family 数量分别为 25 个、25 个、24个、40个、9个、3个(图6‑A、图6‑B)。通过 Gene Fam‑ily系统发育树可看出,FXP04 菌株与 S. varsoviensis NRRLISP‑5346聚类到同一分支,其次是S. diacarni LHW51701和S. platensis DSM40041,与另外2株菌株之间的遗传距离约为0.45(图6‑C)。通过Gene Family和Core Gene系统发育树发现FXP04 菌株与S. varsoviensis NRRLISP‑5346模式菌株的相似性最高,选取S. varsoviensis NRRLISP‑5346的基因序列与FXP04进行共线性分析(图7),结果与系统发育树的结果相同,但也存在倒位、缺失和重排等现象。
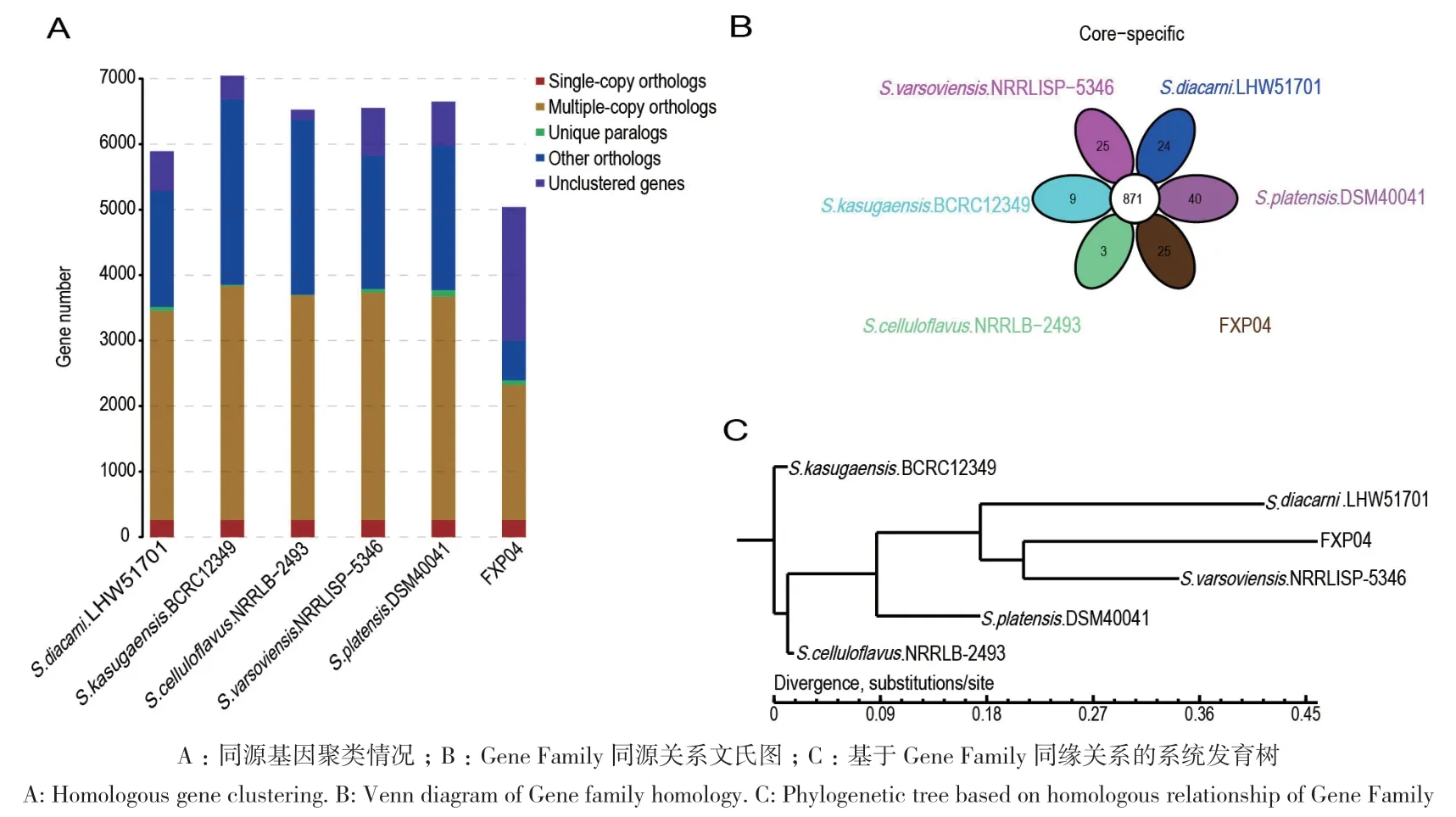
图6 6个菌株之间基因组Gene family分析结果Fig. 6 Genome Gene family analysis results among 6 strains
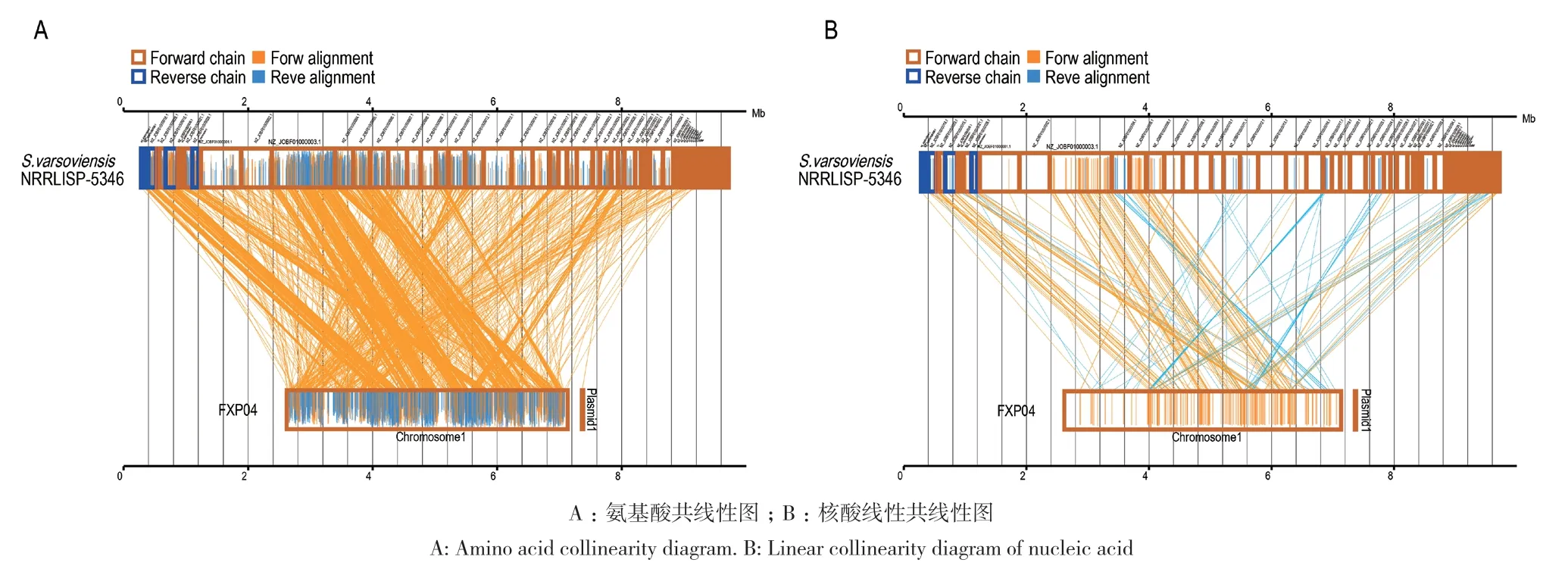
图7 FXP04与S. varsoviensis NRRLISP-5346共线性分析Fig. 7 Collinearity analysis between FXP04 and S.varsoviensis NRRLISP-5346
2.3.3 次生代谢产物基因簇分析 利用 anti SMASH(V6.1.1)[14]对FXP04基因组的次级代谢产物合成基因簇进行预测分析,结果(表3)表明该菌株基因组中预测到了13个次级代谢产物合成相关基因簇,分别为聚酮合酶(polyketide synthase,PKS)基因簇3种、非核糖体多肽合成酶(non‑ribosomal peptide synthase,NRPS)基因簇3种和萜烯类簇(Terpene)2种、芳基聚烯烃(arylpolyene)基因簇2种、套索肽(lassopeptide)基因簇1种、四氢嘧啶(ectoine)基因簇1种和尚未被定义出类型的基因簇1种。进一步将FXP04预测的13个基因簇与其中已知合成基因簇序列进行 BLAST 比对后发现,其中有些与参考基因簇表现为完全相似或高度相似的合成基因簇。Cluster 3与BGC0001742(Candidatus Streptomyces philanthi bv. Triangulum)来源的杀粉蝶菌素(piericidin A1)基因簇相似度达到100%;Cluster 8与BGC0002052(Streptomyces sp. ID38640)来源的四氢嘧啶(ectoine)基因簇相似度也达到100%; Cluster 13与BGC0002547来源的Youssoufene类合成基因簇的相似度达到了88%。据相关的文献报道,基因簇分析中确定已知化合物的标准是基因簇与已报道基因簇之间的相似度≥85%。所以,上述基因簇与已知基因簇序列的相似度≥85% 时,由该基因编码形成的化合物是已知的;即低于85% 时,可以推测出该基因中存在独特的序列。如Cluster 5与BGC0000663(Streptomyces coelicolor A3(2))来源的何帕烯(Hopene)基因簇的相似度达到61%;Cluster 7与BGC0000368来源的Griseobactin基因簇相似度达到53%;Cluster 4与BGC0001110(Uncultured bacterium psy1)来源的羊海绵抑素(Psymberin/Irciniastatin B)基因簇的相似度达到31%;Cluster 1与BGC0002662来源的二聚化Piperazyl环肽Petrichorin A /Petrichorin B基因簇的相似度达到24%;Cluster 6与 BGC0000431来源的Stenothricin基因簇和Cluster 11与BGC0002591来源的Aurachin类基因簇的相似度都达到13%;Cluster 9与BGC0001764来源的s56‑p1基因簇相似度达到11%,这些基因簇可能会合成新的抑菌物质,有待于后续研究。除了上述的这些基因簇和一个尚未被定义出类型的基因簇,还有2个功能未知的基因簇(Cluster2、10)存在,说明菌株FXP04可能存在能合成新型抑菌物质的潜力,对农业的发展需求有着重要意义。
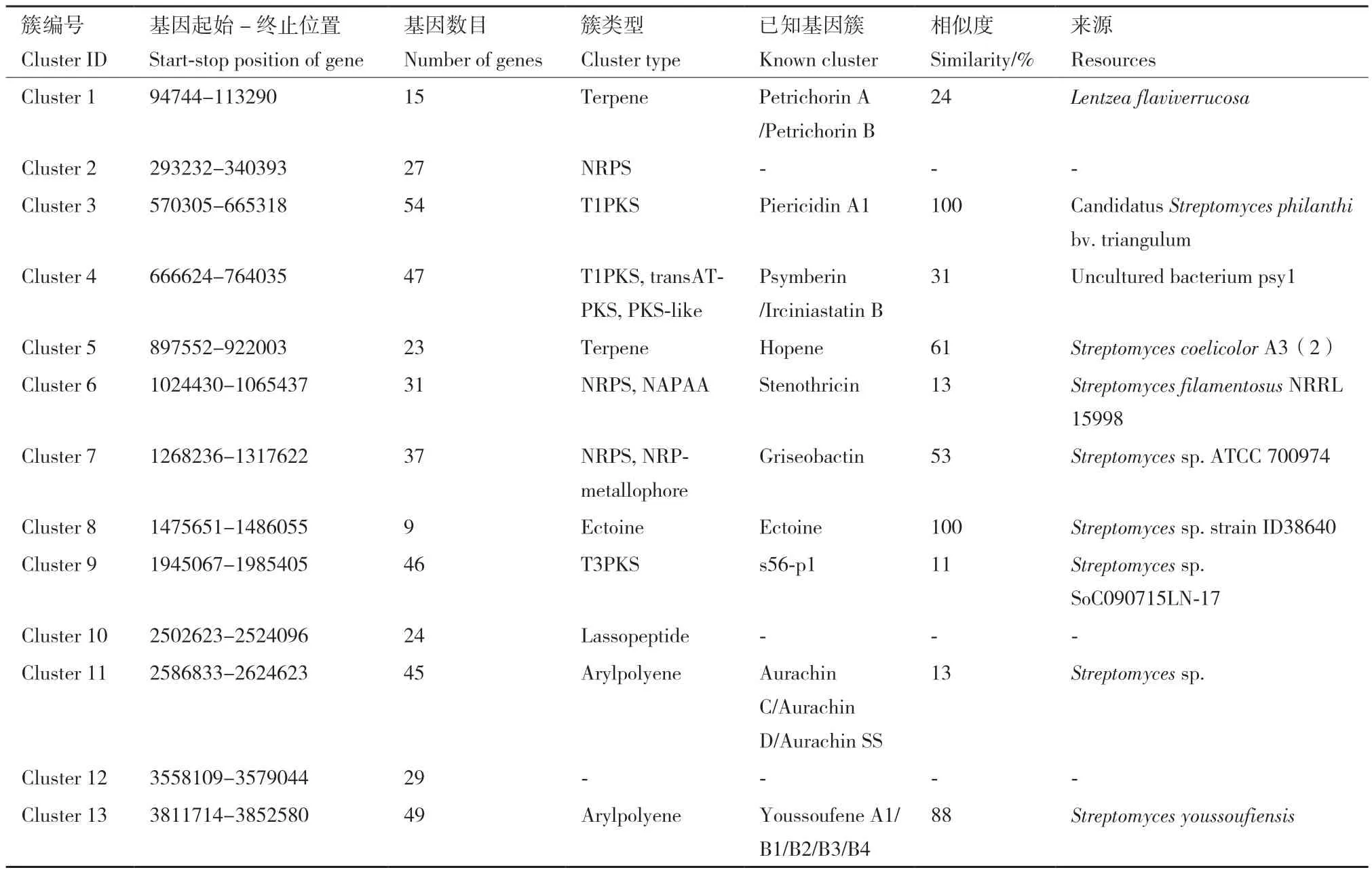
表3 次生代谢产物合成区域鉴定结果Table 3 Identification results of synthetic regions of secondary metabolites
3 讨论
Streptomyces sp. FXP04是从健康马铃薯根际土壤中分离的一株对致病疫霉具有明显拮抗作用的链霉菌菌株[6],且前期研究也发现其对水稻等作物有促生效果[7]。日常生活中常见的抗生素大多来自链霉菌,而传统实验和鉴定方法很难全面地分析链霉菌的作用机制,对此,深入研究FXP04在基因组层面的内在原因有着重要意义。所以我们通过全基因组测序和生物信息学分析得到了FXP04的全基因组序列,确定了FXP04基因组大小为4 535 201 bp,共编码5 037个基因,通过GO、KEGG、COG等数据库比对分析,完成了FXP04基因组的注释结果和数据整理。结果表明无论在GO、COG还是KEGG 数据库注释中,菌株FXP04的基因功能多集中于氨基酸的转运及代谢和碳水化合物的转运及代谢。此外,在KEGG注释中还发现53个基因与萜类和聚酮化合物的代谢相关,可能参与抗菌活性物质的生物合成,例如,ncsB1(Gene636)、ncsB3(Gene2 206)为烯二炔类抗生素(biosynthesis of enediyne antibiotics)的生物合成基因[19];tktA(Gene751)为芳香族氨基酸生物合成中心途径的关键酶基因,糖肽类抗生素(glycopeptide antibiotics)几乎都含有芳香族氨基酸,如万古霉素等[20-21]。
碳水化合物在动物生长生产方面具有重要作用。在碳水化合物功能注释结果中显示,菌株FXP04基因组中含有与葡聚糖酶、几丁质酶、β-1,4‑木聚糖内切酶、D‑葡萄糖醛酸酶等相关的基因。β-1,4‑木聚糖内切酶、D‑葡萄糖醛酸酶属于木聚糖降解酶家族,木聚糖是仅次于纤维素存在于植物细胞壁的一种重要的异源多糖。葡聚糖酶属纤维素酶家族,对纤维素的分解有着重要意义,几丁质广泛存在于甲壳类动物的外壳、真菌的胞壁以及一些高等植物的细胞壁等,而菌株FXP04中存在的几丁质酶可以降解和破坏病原菌的外壁,从而起到防治作用,推测可能为其抑菌机理之一。
利用antiSMASH工具对菌株FXP04的次级代谢产物基因簇进行预测分析,发现了10种已知的抗菌活性物质合成基因簇,Hopene是合成具有巨大商业价值的生物活性物质Hopanoids(何帕烷类)的重要前体,据相关报道何帕烷类化合物具有抗炎、抗菌等生理活性[22-24];Psymberin也称为Irciniastatin A,是一种聚酮酸的生物活性天然产物。Psymberin于2004年由Crews和Pettit的研究小组从海绵Psammocinia sp.中独立发现,是一种有效的抗癌剂[25];Petrichorin A是一种具有抗癌、抗肿瘤活性的二聚化Piperazyl环肽[26];Griseobactin是一种具有儿茶酚肽结构的铁载体,在2010年Patzer等[27]发现由链霉菌属ATCC 700974和几种灰链霉菌株合成的,参与灰霉菌素的合成需求;s56‑p1是一种具有独特的肼单元的二肽天然产物,s56‑p1生物合成基因簇是第一个被确定为负责含肼类天然产物生物合成的基因簇[28];Aurachin是各种细胞色素复合体的抑制剂,具有强大的抗菌、抗真菌和抗疟原虫药物[29-30]。上述7种活性物质与已知基因簇相似度低,推测可能合成新的活性物质,对于链霉菌属的潜在的天然产物开采提供理论,对于农业、医学方面的应用有着重要的研究意义。同时,通过预测得到的已确定的3种活性物质,包括Youssoufene、四氢嘧啶、杀粉蝶菌素,四氢嘧啶(Ectoine)可以帮助微生物适应在高盐、高渗透压和紫外辐射等逆境中正常生长[31],使得促生菌株在恶劣气候下能在根系或根际稳定繁殖。Youssoufene化合物是含有肉桂酰基团的天然产物,对于Youssoufene化合物的报道较少,但肉桂酰基类化合物有着广泛的生理活性,包括抗菌和抗结核活性等[32]。杀粉蝶菌素(Piericidin A1)最早由日本科学家发现,是一种具有良好生物活性的 α-吡啶酮类天然产物,后来在多个链霉菌中都有发现[33-34]。其可能是线粒体呼吸作用抑制剂,对多种生物具有较强的生物活性。首先,杀粉蝶菌素 A1 对蚕、菜青虫有很显著的杀虫效果[35],对多种癌细胞和肿瘤细胞也有很好的生长抑制作用[36-37]。其次,杀粉蝶菌素对多种病原真菌和粘红酵母等也有很强的抑制活性[38]。最后,何亚文等[39]发现杀粉蝶菌素A1 对水稻白叶枯菌的生长有较强的抑制作用,在温室条件下喷施杀粉蝶菌素A1能够减轻白叶枯症状。而且在我们前期研究中已经对FXP04的次生代谢物进行纯化,与杀粉蝶菌素标品对比出的抑菌效果相同[6],进一步说明菌株FXP04有抗菌、抗虫、抗肿瘤以及促生长的潜力。
通过以上的数据结果分析,我们了解了菌株FXP04的全部基因组序列信息,发现其属于天然小基因组,其中存在有3种重要的次级代谢产物基因簇和7种有望合成新型抑菌物质的抗生素基因簇,极可能挖掘出结构新颖、活性更强、靶向性更好的天然药物用于临床研究;而且据相关报道表明链霉菌基因组中含有大量的冗余基因,其中就包括次级代谢合成基因簇、可移动元件等[40],它们是菌株正常生长发育中非必需的,可以通过大规模删除冗余基因用于构建高版本工业底盘细胞,用作天然产物基因的异源表达;其次对于杀粉蝶菌素的抑菌效果有了初步了解,后期将通过预测到的杀粉蝶菌素的合成途径进行超表达和构建缺失突变体,以验证其在FXP04中的地位,同时也为通过遗传技术获取高产杀粉蝶菌素的菌株提供理论基础,有待于对Streptomyces sp. FXP04的次级代谢产物进行后续研究与应用。
4 结论
通过Illumina和PacBio平台相结合的测序技术,对链霉菌Streptomyces sp. FXP04进行全基因组测序,获得了菌株FXP04的全部基因组序列信息。分析结果可知FXP04基因组大小仅为4.5 M,属天然的小基因组,后续可以构建成高版本工业底盘细胞用作天然产物基因的异源表达,满足微生物药物工业化生产需求,发挥其应用潜力。
