小麦矮秆基因Rht-B1b和Rht-D1b的降秆效应及株高相关QTL挖掘
2023-08-31单宝雪刘秀坤肖延军展晓孟黄金鑫刘百川张玉梅李豪圣刘建军曹新有赵振东
单宝雪,刘秀坤,肖延军,展晓孟,黄金鑫,刘百川,张玉梅,李豪圣,刘建军,高 欣,曹新有,赵振东
(1.青岛农业大学 农学院,山东青岛 266109;2.山东省农业科学院作物研究所/小麦玉米国家工程中心/农业部黄淮北部小麦生物学与遗传育种重点实验室/山东省小麦技术创新中心,山东济南 250100)
小麦是世界主要粮食作物,也是中国第三大粮食作物[1],可为世界人口提供20%的卡路里和蛋白质[2],也是人体微量元素的重要来源[3]。小麦株高与抗倒伏能力[4]具有显著的相关性,是影响小麦产量的重要因素之一。据研究,矮化或降低株高与产量潜力的大幅增加有关[5]。自19世纪60年代的绿色革命以来,我国在小麦育种工作中引进了小麦矮秆、半矮秆基因Rht,小麦产量因此上升[6]。合理利用矮秆基因现已成为小麦育种的主要策略之一[7],小麦株高是多基因控制的数量性状,主要受矮秆基因控制[8],还受到大量数量性状位点的影响,同时根据遗传背景和环境条件的不同而表现出差异[9-10]。目前已经被命名的矮秆基因有27个,分布在2AS、2BL、2DS、3BS、4BS、4DS、5AL、5DL、6AS、7AS和7BS等染色体上[11],多由突变产生。
Rht-B1b和Rht-D1b半矮秆基因来源于日本品种农林10号(Norin 10),分别定位于4B和4D染色体短臂,目前存在于70%的全球商品化小麦品种中[12]。Ellis[13]开发的小麦Rht-B1b和Rht-D1b基因特异性引物,利用等位基因特异性标记进行基因分型。1996年,郭保宏等[14]检测我国76个矮秆小麦品种中的矮秆基因分布情况,单独含有Rht-B1b或Rht-D1b基因的品种分别占21%和72%,同时含有Rht-B1b和Rht-D1b基因的品种占7%。含有Rht-B1b基因的小麦品种生产上累计推广面积河南省最大,为524万hm2,含有Rht-D1b基因的品种以山东省累计推广面积最大,为1 575万hm2。2005年慕美财等[15]对150份山东小麦品种的Rht-B1b和Rht-D1b基因进行鉴定,结果表明20.67%含有Rht-B1b基因,有54.00%含有Rht-D1b基因,同时含有Rht-B1b和Rht-D1b矮秆基因的品种仅占3.33%。2017年周晓变等[16]对黄淮麦区246份小麦材料进行矮秆基因检测,结果表明41.46%含有Rht-B1b基因,63.41%含有Rht-D1b基因,同时含有Rht-B1b和Rht-D1b基因的材料占45%。2018年朱浩等[17]检测202份小麦的矮秆基因分布情况,发现10.89%含有Rht-B1b基因,73.76%含有Rht-D1b基因。在过去20多年间大量研究表明,Rht-B1b、Rht4-D1b基因的分布频率逐年提高,黄淮麦区Rht-D1b基因的分布频率高于Rht-B1b基因,表明Rht-D1b基因的利用更广泛,普及率更高。随着分子生物技术的不断发展,DNA芯片技术由于其快捷和精确的特性在QTL定位中得到了广泛的应用。廖思敏等[18]利用55K SNP芯片,在W7268和川育12衍生的RIL群体中检测到两个控制株高的稳定的主效QTL:QPh.cib-4B.1和QPh.cib-2D.1。王盈等[19]在高代分离群体中检测到一个控制株高的新位点:Qph-6A,可以解释8.33%~13.93%的表型变异。前人分别利用芯片、GWAS技术以及分子标记对多个小麦群体进行QTL定位,在不同环境下检测到大量的株高QTL位点,广泛地分布于基因组染色体上,但由于检测到的QTL位点数量多、效应小,导致在生产上应用较少。因此挖掘在多环境下稳定的QTL位点,在矮化育种中加以应用,是目前亟需解决的问题。
为发掘新的株高相关QTL位点,分析矮秆基因Rht-B1b和Rht-D1b在遗传群体内的降秆效应,本研究选用黄淮北片育成的济麦44×济麦229衍生的重组自交系(RIL)群体为材料,结合株高表型数据,分析不同基因型的降秆效应并进行株高QTL定位,以期为发掘新的矮秆基因和矮化育种提供指导。
1 材料与方法
1.1 试验材料
本研究选用黄淮北片育成的小麦新品种济麦44和济麦229衍生的重组自交系(F2∶6,285个株系)群体为材料。其中济麦44含有Rht-D1b基因,济麦229含有Rht-B1b基因。
1.2 田间试验设计
试验材料于2020-2021年种植在济南试验基地(21JN)、2021-2022年种植在济南(22JN)和济阳(22JY)试验基地。试验点采用随机区组设计,1.5 m×6 m小区种植,机播180 g。按常规方法进行大田试验管理。
1.3 表型鉴定及统计分析
小麦自然成熟收获前,对济南、济阳的重组自交群体分别进行株高的数据测定,分别进行3次重复,取平均值用于表型和遗传分析。
1.4 数据分析
利用Execl 2016对测量的株高数据进行整理和分析,在QTL IciMapping V4.0软件(http://www.isbre ed.net)假设基因型的固定效应,使用ANOVA函数跨环境计算最佳线性无偏估计(BLUE)。采用SAS 9.4软件(SAS institute,http://www.sas.com)进行方差分析(ANOVA)。利用Origin 2022b绘制矮秆基因降秆效应图。利用R studio绘制群体株高的相关性热图。
1.5 DNA提取
利用Tiangen快捷植物基因组DNA提取系统来提取小麦苗期的幼嫩叶片DNA,利用NANO DROP 2000检测DNA浓度及质量,将终浓度调至50 ng/μL,并置于-20℃冰箱保存备用。
1.6 PCR扩增
根据Ellis[13]发表的序列设计引物。引物对BF/WR1可扩增Rht-B1a野生型基因,引物对BF/MR1可扩增Rht-B1b突变型基因,引物对DF2/WR2可扩增Rht-D1a野生型基因,引物对DF1/MR2可扩增Rht-D1b突变型基因。
试验分别以小偃6号(含矮秆基因Rht-B1b)、济南17(含矮秆基因Rht-D1b)做阳性对照,以中国春(不含任何矮秆基因)做阴性对照,来验证试验结果的准确性。引物由上海生工生物股份有限公司合成,序列信息见表1。

表1 引物序列Table 1 Primer sequences
PCR反应体系:10 μL体系中,DNA模板(已进行稀释)1 μL,正、反向引物(10 μmol·L-1)各1 μL,Mix 5 μL,ddH2O 2 μL。
PCR反应条件:95 ℃预变性2 min,94 ℃变性15 s,退火15 s,退火温度见表1,72 ℃延伸15 s,35个循环,72 ℃延伸5 min。
PCR扩增产物以2%的琼脂糖凝胶电泳检测,缓冲体系为1×TAE缓冲液,150 V电压电泳30 min,凝胶成像仪下观察条带并照相。
1.7 遗传图谱的构建与QTL定位
利用中玉金标记(北京)生物技术股份公司开发的55K小麦专用芯片进行基因型检测,删除杂合率≥10%和缺失率≥10%的标记,用TASSEL V5.0软件将偏分离标记(Min∶Max=0.3∶0.7)过滤,使用QTL IciMapping V4.2软件整理剩余的标记。利用JionMap4构建全基因组遗传图谱,然后利用Map Chart绘制遗传图谱。利用软件QTL IciMapping 4.1进行QTL分析,选择复合完备区间作图(ICIM-ADD)法,做1 000次重复,临界阈值P=0.05,作为判定QTL存在与否的标准。QTL的置信区间(CI)为LRs峰值±1所在位置。QTL命名以“Q+性状名称缩写+染色体编号+编号”。在超过一半的环境中发现相同性状的QTL认为是稳定的QTL。
2 结果与分析
2.1 亲本及重组自交系的株高表型分析
济南和济阳基地的基本气候和土壤情况见表2,两地块在小麦播种前土壤均进行旋耕处理。在三个环境下对RIL群体的285个株系进行株高的测定,结果(表3、图1)表明,21JN中RIL群体的株高范围为34.33~103.00 cm,平均值为64.53 cm;22JN中RIL群体的株高范围为44.67~100.00 cm,平均值为78.07 cm;22JY中RIL群体的株高范围为45.33~119.00 cm,平均值为91.33 cm。由于济阳试验田的土壤速效磷及速效钾含量整体高于济南试验田,年平均降雨量相近,造成22JN和22JY之间的株高存在差异。济南试验基地2022年小麦生长期间及小麦拔节期降水量(395.7和40.2 mm)高于2021年(324.4和27.8 mm);进而造成2022年群体的平均株高高于2021年。21JN、22JN未发生倒伏情况,22JY由于株高较高、灌浆后期出现大风降雨天气,23.5%的小区发生了不同程度的点片倒伏。因此群体内家系间株高变异幅度较大。三个环境下株高表型数据的偏度与峰度(表3)的绝对值均小于1。
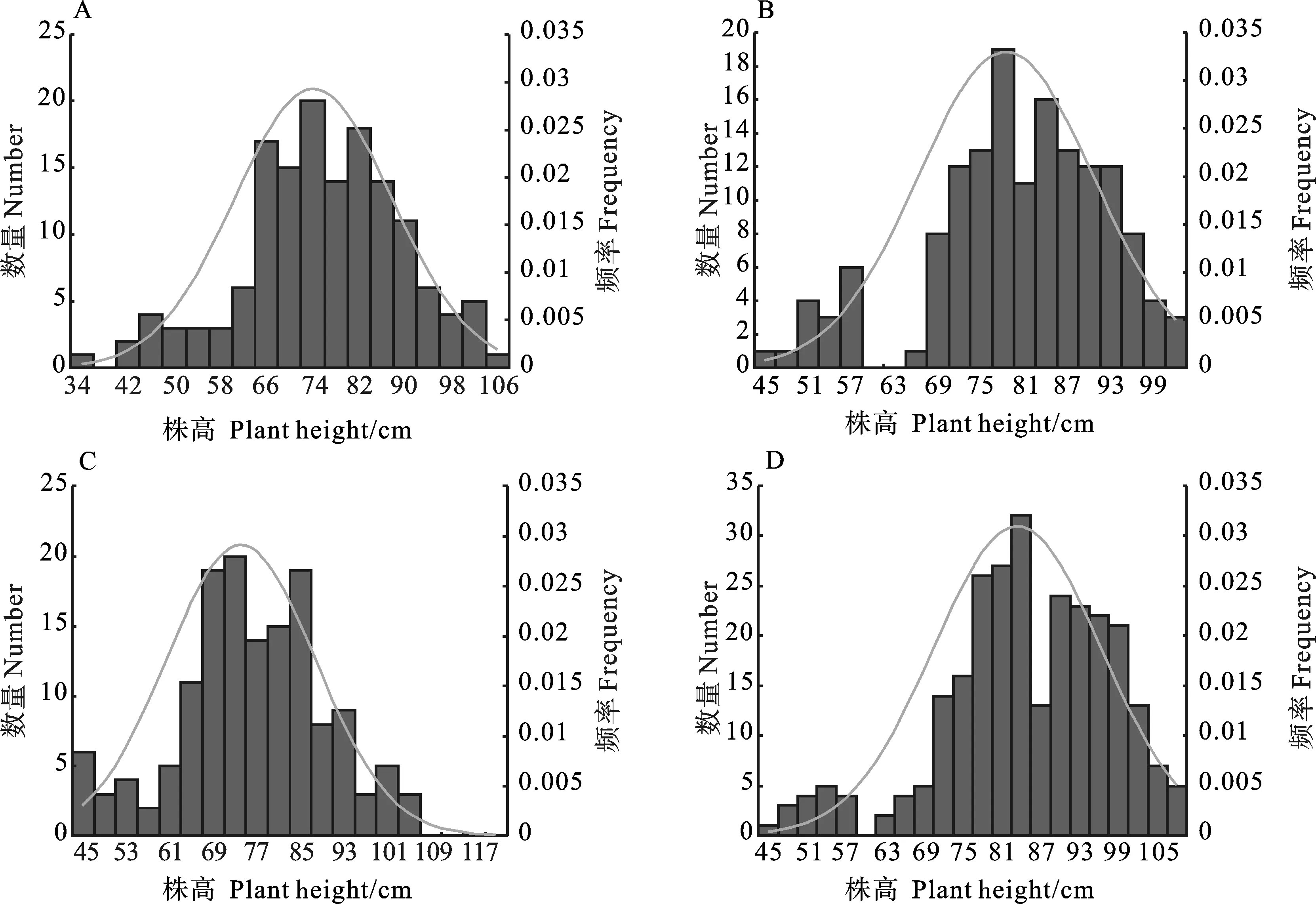
图1 21JN(A)、22JN(B)、22JY(C)和BLUE(D)环境下RIL群体株高的频率分布Fig.1 The frequency distribution of plant height of RIL population in 21JN(A), 22JN(B), 22JY(C), and BLUE(D) environments

表2 济南和济阳基地气候和土壤情况Table 2 Climate and soil conditions of JN and JY locations
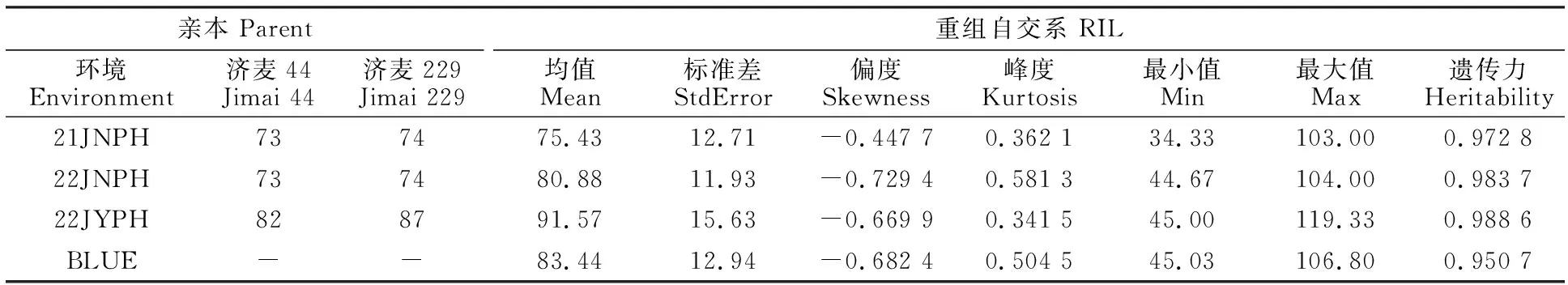
表3 RIL群体的株高表现Table 3 Plant height of RIL population
株高的遗传力在21JN、22JN和22JY三个环境分别为0.9728、0.9837和0.9886,表明株高主要受遗传因子的影响。由图1可知,重组自交系的株高在不同的环境下均表现为近似正态分布,群体中株高出现了超亲分离的现象,符合数量性状遗传的特点,可以进行株高性状的QTL定位。
2.2 重组自交系的矮秆基因Rht-B1b与Rht-D1b的检测
引物对BF/WR1和BF/MR1分别用来检测Rht-B1a和Rht-B1b基因, BF/WR1可以从含有Rht-B1a基因的材料中扩增出一条237 bp的特异性片段,BF/MR1可以从含有Rht-B1b基因的材料中扩增出一条237 bp的特异性片段。结果验证济麦44不含Rht-B1b基因,济麦229含有Rht-B1b基因,在285份重组自交系材料中,113份材料含有Rht-B1b基因,占总数的39.65%;172份材料含有Rht-B1a基因,占总数的60.35%。
引物对DF2/WR2和DF1/MR1分别用来检测Rht-D1a和Rht-D1b基因,DF2/WR2可以从含有Rht-D1a基因的材料中扩增出一条254 bp的特异性片段,DF1/MR1可以从含有Rht-D1b基因的材料中扩增出一条264 bp的特异性片段。结果验证济麦44含有Rht-D1b基因,济麦229不含Rht-D1b基因,在285份重组自交系材料中,102份含有Rht-D1b基因,占全部材料的35.79%;183份含有Rht-D1a基因,占全部材料的64.21%。同时含有Rht-B1b与Rht-D1b两基因的有29份,占全部材料的10.18%。
2.3 Rht-B1b和Rht-D1b对株高的遗传效应
将285个RIL家系的基因检测结果与其对应株高表型进行比较,发现在三个环境下含有Rht-B1b基因的株系株高均大于基因型Rht-B1a的株系(图2A),其中21JN、22JN和22JY三个环境下表现为显著或极显著的差异,相较于Rht-B1a,Rht-B1b可降低株高8.45 cm(10.75%)、6.76 cm(8.10%)和8.83 cm(9.41%)。根据图2B,在三个环境下含有Rht-D1b基因的株系株高均大于基因型Rht-D1a的株系,其中21JN、22JN和22JY三个环境下均表现为显著或极显著的差异,相较于Rht-D1a,Rht-D1b可降低株高11.68 cm(14.68%)、12.74 cm(14.93%)和16.60 cm(17.36%)。
根据图2C,Rht-B1b和Rht-D1b对小麦群体株高的联合效应可以显著降低株高,根据群体中Rht-B1b基因与Rht-D1b基因单独存在的株高性状对比,可以看出Rht-D1b基因的降秆效应要大于Rht-B1b基因。且Rht-B1b和Rht-D1b基因同时存在可显著降低株高8.85~35.80 cm(11.05%~34.82%)。
矮秆基因Rht-B1b和Rht-D1b存在的前提下,重组自交群体内株高仍有较大差异,两基因无法解释全部的株高变异,因此利用已构建的285个家系,利用55K芯片进行分析,期望找到新的控制株高的QTL,可以更合理地解释小麦群体株高的差异分布。
2.4 遗传连锁图谱
经过小麦55K SNP芯片分析,亲本间具有多态性的SNP标记共有53 064个,其中2 344个骨架标记用于遗传图谱的构建。获得的遗传图谱含有29个连锁群,全长3 349.95 cM,覆盖基因组全部21条染色体,其中1A、2B、2A、4B、5D、6B、7A、7B分别有两个连锁群。B基因组标记最多(936个)且长度最短,为1 091.06 cM。A基因组有815个标记,D基因组含593个标记,覆盖染色体长度分别为1 123.57、1 091.06和1 135.32 cM。(表4)。平均标记密度为1.43/cM。
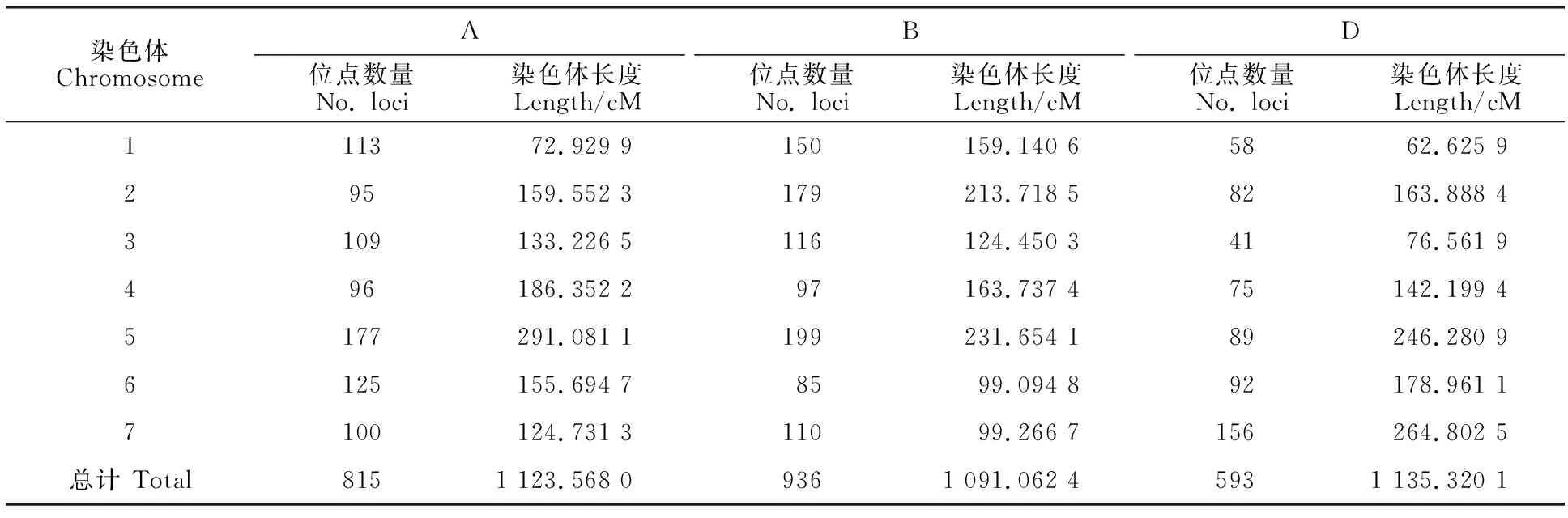
表4 “济麦44×济麦229”RIL群体的高密度遗传图谱Table 4 Distribution and length of the markers in genetic map of the RILs derived from “Jimai 44×Jimai 229”
2.5 株高相关QTL位点
共检测出6个与株高性状相关的QTL(表5,图3),共分布在1A、1B、2B、4B和4D(2)共5条染色体上。单个QTL可以解释0.81%~32.32%的表型变异,其中可以被稳定检测到主效的QTL有2个,分别是位于4B和4D染色体的Qph.saas-4B.1、Qph.saas-4D.2,分别可以解释10.40%~20.12%和22.25%~32.32%的表型变异。Qph.saas-2B.2在两个环境下被检测到,可以解释0.81%~1.52%的表型变异,Qph.saas-4D.1在一个环境和BLUE值被检测到,可以解释21.58%~22.88%的表型变异。还有两个QTL:Qph.saas-1A、Qph.saas-1B在一个环境中被检测到,可以解释1.88%~2.23%的表型变异。
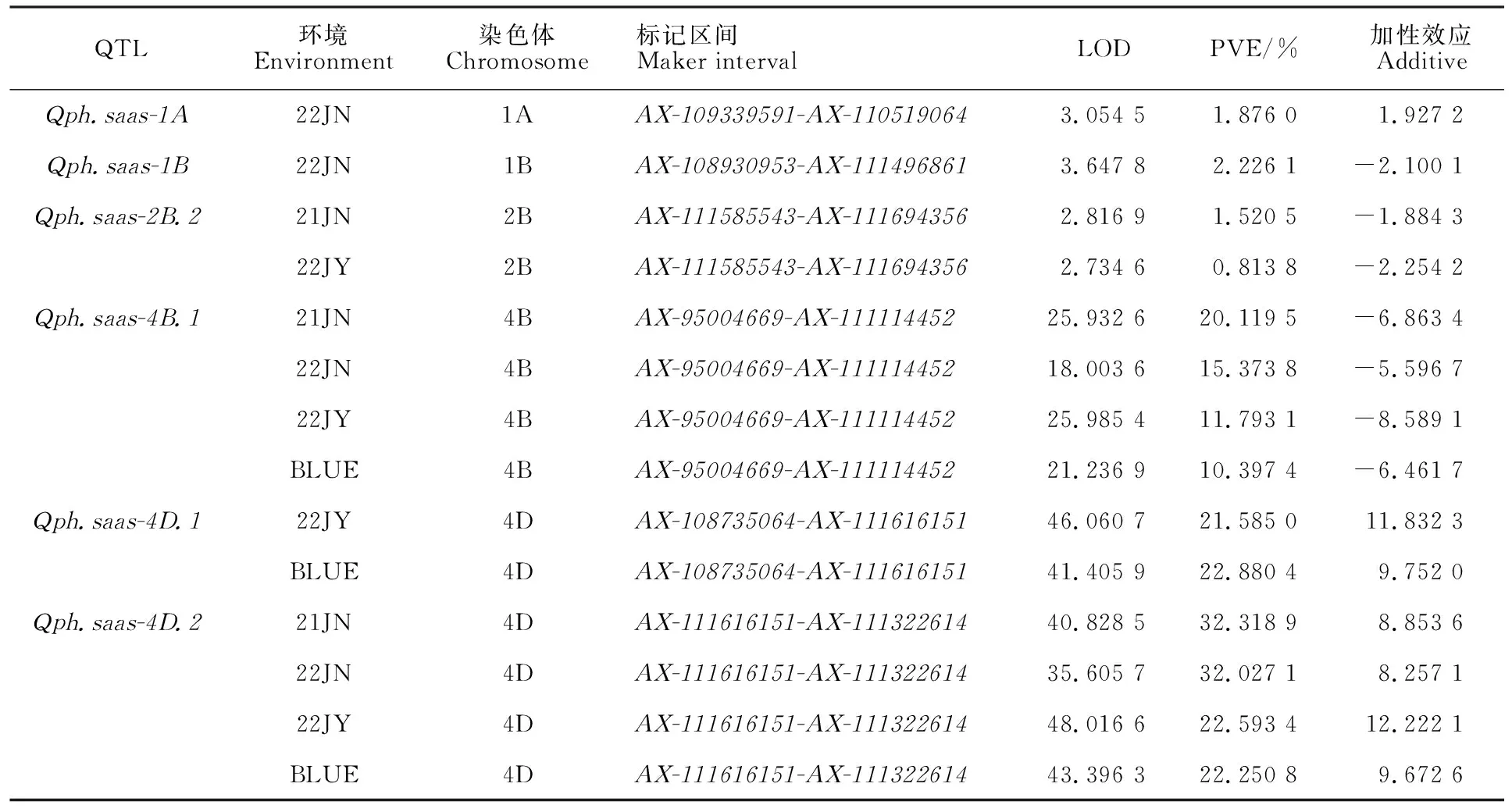
表5 “济麦44×济麦229”RIL群体株高性状的QTLTable 5 QTLs for plant height of the RIL population derived from “Jimai 44×Jimai 229”
3 讨论
2014年许琦等[20]研究发现Rht-B1b基因的降秆效应达到10.4%,Rht-D1b的降秆效应达到16.7%。Wang等[21]和Akman等[22]发现,Rht-D1b能够降低16%~30%的株高。丁梦云等[23]检测Rht-B1b基因的降秆效应为8.2%~8.9%。Flintham[24]检测发现Rht-B1b和Rht-D1b单独存在时的平均降秆效应为15.5%,两基因同时存在时的降秆效应可达到42%。Richards[25]证明Rht-B1b和Rht-D1b单基因的降秆效应在23%,而基因组合的降秆效应可达到47%。Butlerd等[26]检测到单个Rht-B1b和Rht-D1b单基因的降秆效应平均达到14.8%,两基因同时存在的降秆效应达到35.7%。Miralles等[27]和Wurschum等[28]研究发现Rht-B1b和Rht-D1b基因单独存在时的平均降秆效应在24%左右。不同研究中得到的降秆效应的差异表明基因型背景和环境条件对株高也有影响。
本试验利用Ellis[13]发表的4对特异性分子标记对矮秆基因Rht- B1b和Rht-D1b在群体中的分布情况进行检测,由于标记的多态性强且扩增出的目的片段条带清晰,该分子标记被广泛应用于小麦分子标记辅助育种。小麦矮秆基因Rht-B1b和Rht-D1b是隐性基因,不适宜在早代筛选,通过利用分子标记辅助选择可以提前确定含有矮秆基因的个体,进而加速小麦育种的进程[29]。通过对285份RIL材料的Rht-B1b和Rht-D1b基因进行检测,结果表明82份材料含有Rht-B1b基因,占39.65%;78份材料含有和Rht-D1b基因,占35.79%;29份材料同时含有Rht-B1b和Rht-D1b基因,占10.18%。本研究中得到Rht-B1b和Rht-D1b两基因的分布情况略低于前人研究的结论,这可能与本试验选取的材料有关,RIL群体的基因主要来源于两亲本,具有基因组合的局限性,因此与其他群体的基因分布情况有差异。
根据本研究结果,Rht-B1b可降低株高6.76~8.83 cm,降秆效应在8.10%~10.75%;Rht-D1b可降低株高11.68~16.60 cm,降秆效应达到14.68%~17.36%。Rht-B1b和Rht-D1b基因同时存在可显著降低株高8.85~35.80 cm,降秆效应达到11.05%~34.82%。绿色矮秆基因Rht-B1b和Rht-D1b的广泛应用,极大地促进了绿色革命的进程,不仅保证了小麦的稳产,更促进了小麦产量潜力的提高[30]。与1990年前后的品种相比,Rht-B1b的频率从8.6%提高至32.2%,Rht-D1b的频率从36.2%提高至53.4%[31],基因分布频率的显著提高表明矮秆基因Rht-B1b和Rht-D1b在中国不同环境下的小麦生产中都获得了大量的应用。
本研究通过构建高密度遗传图谱定位到两个稳定表达的主效QTL位点,分别是Qph.saas-4B.1和Qph.saas-4D.2。试验前期已验证亲本济麦44含有Rht-D1b,济麦229含有Rht-B1b。Qph.saas-4B.1的物理位置在31.85~150.62 Mb之间,Rht-B1b基因在4B染色体上的33.61 Mb处,表明Qph.saas-4B.1就是Rht-B1b基因。Qph.saas-4D.2在16.15~37.49 Mb之间,Rht-D1b在4D染色体的18.78 Mb处,表明Qph.saas-4D.2就是Rht-D1b基因。Mao等[32]利用Meta分析法,确定了30个与株高相关的QTL,其中P13对应矮秆基因Rht-B1b,定位在Xgwm495-Xfba78侧翼序列之间;P14对应矮秆基因Rht-D1b,定位在Xwmc419-Xwmc457侧翼序列之间,本研究所结果与其定位结果相近。
本研究中定位到的Qph.saas-4D.1位于4D染色体11.87~16.15 Mb之间,可以解释22.23%的表型变异,在多个环境中被检测到。梁子英等[33]定位到多个株高及其相关性状QTL位点,位于Xwmc473-Xwmc285区间内,可以解释13.49%~63.42%的表型变异。刘宾等[34]定位到一个株高QTL:Oph4D-2,位于Xbarc334-Xwmc331之间。本研究定位到的株高相关位点Qph.saas-4D.1与上述多个QTL位点物理位置重叠,可能为同一QTL,是一个新的矮秆基因,目前未被克隆利用。Qph.saas-4D.1可以解释22.23%的表型变异,是“济麦44×济麦229”衍生的重组自交群体在Rht-B1b和Rht-D1b两个主效矮秆基因存在的情况下,群体中仍存在较大的株高分离现象的主要原因之一。本研究中的定位到的QTL位点Qph.saas-2B.2位于2B染色体678.84~680.04 Mb之间,与前人研究报道的QTL位点不重合,可以解释2.73%~2.82%的表型变异,在两个环境下被检测到,可能为一个新的微效株高QTL位点,后续可进一步开展相关研究并在矮秆育种中加以利用。
4 结论
本研究利用济麦44与济麦229构建的285份RIL家系对Rht-B1b与Rht-D1b基因及其降秆效应进行检测,结果表明82份材料含有Rht-B1b基因,降秆效应达到8.10%~10.75%;78份材料含有Rht-D1b基因,降秆效应达到14.68%~17.36%;29份材料同时含有Rht-B1b和Rht-D1b基因,两基因同时存在的降秆效应达到11.05%~34.82%。构建高密度遗传图谱对群体矮秆基因进行检测,共检测到6个QTL,其中两个被稳定检测到的QTL分别为已克隆的Rht-B1b和Rht-D1b基因。此外,Qph.saas-2B.2和Qph.saas-4D.1可在2个环境下被检测到,Qph.saas-2B.2可能为影响株高相关新QTL位点,用于小麦矮化育种实践中,为进一步矮秆基因的精细定位和矮化育种提供理论参考。
