枯草芽孢杆菌脂肪酶LipA 的生物信息学分析
2022-07-29黎菁菁赖昕珏李鑫尧马俊炜余铭怡罗佳伟陈胤熹余洁婷郑少鹏郑敦锦曹诗林陈宛涓
黎菁菁,赖昕珏,李鑫尧,马俊炜,余铭怡,罗佳伟,陈胤熹,余洁婷,郑少鹏,郑敦锦,曹诗林*,陈宛涓
(1.佛山科学技术学院广东省食品智能制造重点实验室,广东佛山 528000)
(2.佛山科学技术学院食品科学与工程学院,广东佛山 528000)
(3.佛山科学技术学院材料科学与氢能学院,广东佛山 528000)
脂肪酶,是一类能特异性地在油水界面上水解三脂酰甘油酯键的酶[1],广泛存在于微生物、人体、动物以及植物中。目前最常见的脂肪酶生产方式为微生物发酵法,其次还有提取法。由于微生物具有繁殖速度快、种类多的特性,其产生的脂肪酶种类多,具有较强的稳定性、底物特异性,水解作用的温度和pH范围更广[2],并且大多数的脂肪酶为胞外酶[3],使得更方便快捷地在工业生产中得到纯度较高的酶制剂,因此微生物发酵产脂肪酶已成为工业生产脂肪酶的主要途径,有关微生物脂肪酶生产的相关研究也越来越多。
枯草芽孢杆菌是革兰氏阳性菌,由于其具有非致病性、分泌能力较强以及发酵工艺成熟等优势,已成为进行基因研究及其表达的一种重要的工具系统,在基因工程中也有深入的研究[4,5]。枯草芽孢杆菌能够产生的使脂类水解的酶主要有三种,分别是脂肪酶(LipA)、磷脂酶以及酯酶(LipB),其中脂肪酶(LipA)是由lipA 基因编码的胞外水解酶,能够较好地水解中等长度链长脂肪酸形成的酯键,在食品、医药、化妆品等多个领域具有广泛的应用前景[6,7]。枯草芽孢杆菌脂肪酶LipA 结构基因全长为636 bp,能够编码212个氨基酸,并组装成分子量为22644 u 的脂肪酶前体,枯草芽孢杆菌所产生的脂肪酶(LipA)中缺乏半胱氨酸残基,难以形成二硫键稳定结构,并缺乏多数脂肪酶普遍存在的盖子结构[8]。此外,LipA 序列中含有A-X-S-X-G 保守序列。目前,对LipA 的研究主要集中于分泌途径、催化机理、酶学性质、固定化提高酶活力以及通过基因工程改变其立体选择性、热稳定性与提高酶产量[9,10]。本研究应用生物信息学方法对枯草芽孢杆菌脂肪酶LipA 的基本结构、基本特性、模体、表面电位、可及性等进行系统分析,为后续的LipA的分子设计和改造工作提供一定的参考。
1 材料与方法
1.1 数据来源
LipA 的氨基酸序列来源于美国国家生物信息中心NCBI,GenBank 登录号:ACA60974.1。
1.2 LipA 性质预测
LipA 的亲/疏水性分析:使用ProtScale 在线程序[11]对亲/疏水性预测,并对疏水性最强的区域及其亲水性最强的区域进行分析。
LipA 的基本特性预测分析:使用ProtParam 在线程序[12]对LipA 的不稳定指数、消光系数、脂溶指数、平均亲水性进行预测。
LipA 表面电位预测:使用软件Swiss-Pdb-viewer 4.1.0[13]对LipA 蛋白质表面静电构象进行建模。
LipA 的可及性分析:使用软件Swiss-Pdb-viewer 4.1.0 对LipA 蛋白质的可及性模型进行建模。
1.3 LipA 结构预测
LipA 一级结构分析:使用ProtParam 在线程序对碱性氨基酸、酸性氨基酸个数、相对质量及其等电点、氨基酸含量进行预测。
LipA 二级结构的预测:使用NPSA 在线网站所提供的二级结构预测程序[14],用SOPM 的预测方法分析二级结构中的α螺旋、β转角和无规则卷曲等结构。
LipA 的特殊卷曲螺旋预测:使用COILS 在线程序[15]对LipA 的卷曲螺旋区域进行分析,通过在线网址Multicoil Scoring Form[16]对LipA 的二聚体、三聚体卷曲螺旋进行预测。
LipA 的模体预测:使用MotifScan 在线网站[17]对脂肪酶LipA 蛋白序列进行模体预测。
LipA 的PEST 序列预测:使用epestfind 程序[18]预测LipA 的PEST 序列。
LipA 的三级结构分析:通过在线工具trRosetta在线程序[19,20]对脂肪酶LipA 进行建模,选取最合理的模型,用Pymol 软件对LipA 进行可视化分析,使用PSIPRED 在线程序[21]分析α-螺旋、β-折叠、无规则卷曲3 种结构的数目。
LipA 的 Ramachandran 图分析:使用软件Swiss-Pdbviewer 4.1.0 获得Ramachan-dran Plot(拉氏构象图),并使用PROCHECK 在线网址[22]分析得到其区域数据。
1.4 数据处理
使用Excel 2019 对模体预测数据进行归纳处理,使用PowerPoint 2019 对二级结构、三级结构、表面电位、可及性和Ramachandran 图分析的图片进行处理。
2 结果与讨论
2.1 LipA 一级结构分析
使用在线程序ProtParam 进行预测,得出其相对分子质量为22664.02,预测其等电点pI=9.57。20 种氨基酸中的含量排序依次是甘氨酸Gly(11.3%)、缬氨酸Val(9.9%)、亮氨酸Leu(9.9%)、天冬酰胺Asn(8%)、丝氨酸Ser(8%)、丙氨酸Ala(7.5%)、赖氨酸Lys(6.6%)、异亮氨酸Ile(5.7%)、苏氨酸Thr(5.7%)、天冬氨酸Asp(4.2%)、酪氨酸Tyr(4.2%)、谷氨酰胺Gln(3.3%)、精氨酸Arg(2.8%)、甲硫氨酸Met(2.8%)、组氨酸His(2.4%)、脯氨酸Pro(2.4%)、苯丙氨酸Phe(2.4%)、谷氨酸Glu(1.4%)、色氨酸Trp(0.9%)、半胱氨酸Cys(0.5%)。
LipA 的本质是由氨基酸为基本单位构成的蛋白质,蛋白质的酸碱性是其重要的理化性质之一。蛋白质的一级结构是研究蛋白质的结构、生理功能和作用机制的基础。蛋白质的一级结构是根据遗传密码编码的氨基酸通过肽键结合形成的多肽链,蛋白质侧链上含有的酸性氨基酸和碱性氨基酸上带有可解离的基团,这些可解离基团可以决定蛋白质的酸碱性,因此蛋白质的酸碱性可依赖于蛋白质一级结构中酸性氨基酸和碱性氨基酸的数量之比[23]。蛋白质的等电点与氨基酸的组成、属性和分子量的分布有关。蛋白质侧链可电离氨基酸的pKR 值可影响蛋白质的等电点,即酸性氨基酸和碱性氨基酸侧链上的氨基和羧基的电离常数与蛋白质的等电点有关[24]。根据ProtParam 的预测结果显示,脂肪酶LipA 呈碱性,由212 个氨基酸构成,其中包含25 个碱性氨基酸和12 个酸性氨基酸,等电点pI=9.57,而微生物脂肪酶的等电点多为酸性,如疏棉状嗜热丝孢菌的脂肪酶等电点为4.4、白地霉脂肪酶的等电点为5.39[25,26]。
2.2 LipA 的亲/疏水性分析
用在线程序ProtScale 对脂肪酶LipA 进行亲/疏水性预测,确定最大亲水值和最大疏水值及其相对应的位置。根据负值越大表示蛋白亲水性越强、正值越大表示蛋白疏水性越强,可知第12 位的缬氨酸正值最大为3.111,在12 个区域具有疏水性,LipA 疏水性最强的区域在Val9~Leu17 区间;第78 位的苏氨酸负值最大为-2.333,LipA 亲水性最强的区域在Lys75~Asn81区间,如图1 所示。
氨基酸的亲、疏水性分布对蛋白质的结构和生理功能有着重要意义。亲水性和疏水性氨基酸可以预测跨膜蛋白的位置,构成疏水的二级结构,便于蛋白质跨膜、形成更高级结构,同时疏水氨基酸的疏水基团还可以促使蛋白质二级结构的折叠[27]。根据预测结果可知疏水性最强区域与其他天然脂肪酶一样在N 端,这可能与蛋白质的定位与分泌有关。
2.3 LipA 的基本特性预测分析
使用在线程序ProtParam对LipA的基本特性预测分析。结果表明LipA 的不稳定指数为19.12,可知其属于稳定蛋白质;在280 nm 波长下消光系数为24410M-1cm-1;脂溶指数为96.98;平均亲水性是0.009。
蛋白质的稳定性受到许多因素的影响,若要预测蛋白质的稳定性可以用蛋白质的不稳定指数,蛋白质的不稳定指数低于40 时,则为稳定性蛋白质,相反,不稳定指数高于40 则为不稳定性蛋白质[28]。通过在线程序ProtParam 预测得到LipA 的不稳定指数为19.12,因此LipA 属于稳定蛋白质,具有一定的稳定性。蛋白质的消光系数是指蛋白质对某波长的光的吸收能力的量度,与半胱氨酸的组成比例有一定的关系[29],由于LipA 仅有一个半胱氨酸残基,蛋白中不存在二硫键,因此LipA 在280 nm 波长下消光系数只有24410 M-1cm-1。脂溶指数为96.98(<100),并且平均亲水性是0.009,可知LipA 是属于脂溶性蛋白质[30]。
2.4 LipA 二级结构的预测
蛋白质的二级结构主要是指蛋白质的多肽链主链上规则卷曲或折叠的构象,利用NPSA 在线软件对脂肪酶LipA 的二级结构进行预测,统计其中α-螺旋、β-转角、延伸链和无规则卷曲分别对应的百分比,结果表明,二级结构中组分含量从大到小排序均为α-螺旋(29.72%)>无规则卷曲(29.25%)>β-延伸链(26.42%)>β-转角(14.62%)。(图2)
LipA 的二级结构预测得知LipA 含有α-螺旋、无规则卷曲、延伸链和β-转角等四种结构,其中α-螺旋和无规则卷曲的占比较大,分别为29.72%和29.25%,推测其空间结构较小,且受到侧链间相互作用较大的影响,而β-延伸链能够进一步形成β-折叠结构且占比达26.42%,其二级结构比例符合α/β类蛋白的特征,与绝大多数的微生物脂肪酶相似[31,32]。同时,LipA 的催化三联体结构Ser77、Asp133 和His156 与其他的第一家族脂肪酶相似,例如铜绿假单胞菌PA01 脂肪酶的催化三联体结构为Ser82、Asp229 和His251[33]。已有研究表明LipA 具有耐碱性强,无界面活性等特点,且由于缺乏盖子结构,其活性中心暴露,因此不需经过界面活化即能进行催化作用,但同时野生型的LipA也存在着热稳定性差、表达量低等问题[9]。与之相比,大多数的微生物脂肪酶如米根霉脂肪酶、白地霉脂肪酶等都具有盖子结构以及界面活化现象,盖子结构中的α-螺旋的亲水/亲油性的强弱会影响底物与盖子结构的结合,影响活性部位的打开,进而影响脂肪酶的催化活性,而界面活化作用则是通过提高活性部位附近的疏水性使活性部位暴露,影响脂肪酶的催化作用[34,35],后续对LipA 的改造可以从提高表达量及热稳定性等方面进行。
2.5 LipA 的特殊卷曲螺旋预测
通过COILS 算法将LipA 的序列与已知的卷曲螺旋数据库中的数据进行比较,得出相似性得分,并计算出相应的卷曲概率,结果LipA 的卷曲概率均小于0.5,不存在卷曲螺旋区域;同时LipA 的二聚体、三聚体卷曲螺旋预测分值皆为0,不能形成稳定的二聚体、三聚体卷曲。
2.6 LipA 的模体预测
使用MotifScan软件对LipA蛋白序列进行模体预测,结果表明,该蛋白可能存在的翻译后修饰位点有3 种,其中包括2 个蛋白激酶C 磷酸化位点、2 个酪蛋白激酶Ⅱ磷酸化位点、8 个N-肉豆蔻酰基化位点;可参与磷酸化、酰基化等生物化学反应。LipA 的模体名称、序列号、类型及其氨基酸序列的位置信息如表1 所示。
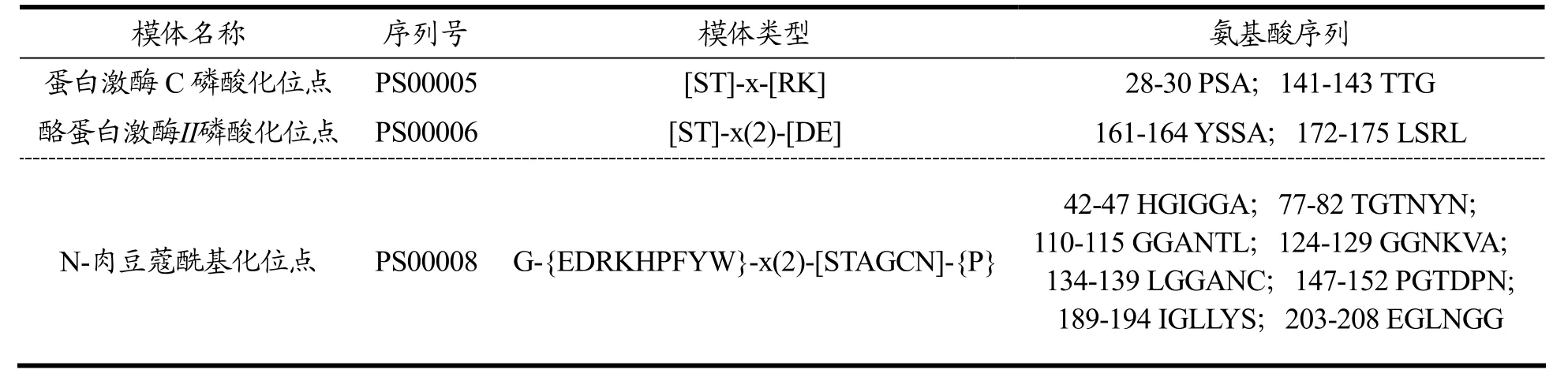
表1 LipA 的模体预测Table 1 The motif prediction of LipA
2.7 LipA 的PEST 序列预测
使用在线网址EMBOSS 的epestfind 程序进行预测LipA 的PEST 序列,无得分低于阈值5.0 的序列,可知在脂肪酶LipA 上不存在非PEST 序列。PEST 序列是富含脯氨酸(P)、谷氨酸(E)、丝氨酸(S)和苏氨酸(T)的相对保守的特征序列[36],并且PEST序列可以通过泛素蛋白酶体途径加速蛋白的降解[37],PEST 序列可能还存在其他的生理功能,但具体的作用机制尚未明确,而脂肪酶LipA 上不存在非PEST序列,可能具有较强的降解蛋白的能力。
2.8 LipA 的三级结构分析
通过在线工具trRosetta 对脂肪酶LipA 进行建模,使用Pymol 视图软件进行图像显示,得到LipA 的三级结构图(图3a),其中能清晰的看见其结构中的α-螺旋、β-折叠、无规则卷曲3 种结构,图中红色、黄色、绿色分别代表α-螺旋、β-折叠、无规则卷曲。结果显示其结构中包含了5 段α-螺旋、6 段β-折叠,因此脂肪酶LipA 属于典型的α/β类蛋白,其中无规则卷曲(包括β-转角)的作用是连接α-螺旋或β-折叠。
蛋白质的三级结构是在蛋白质的二级结构的基础上进一步盘旋、折叠形成的,是蛋白质具有生物活性的基础,其常用的结构表征的方法包括近紫外圆二色谱、X-射线衍射、荧光光谱和核磁共振等[38]。LipA的三级结构预测结构显示LipA 三级结构中包含5 段α-螺旋、6 段β-折叠和丰富的无规则卷曲,而自然界中的脂肪酶一般为α/β类蛋白[39],因此LipA 的预测结果与二级结构的预测结果基本一致,与同类脂肪酶的结构一致。
2.9 LipA 表面电位预测
使用软件Swiss-Pdb-viewer 4.1.0 对LipA 蛋白质表面静电构象进行建模的结果(见图3b)。图中分布着蓝色和红色两种结构,前者代表正电势,后者代表负电势。由图可知,蓝色的正电势集团分散分布,占据了大部分的位置,红色的负电势集团相对集中,占据较少的位置但基团较大,使得LipA 整体呈弱正电势。
蛋白质的电荷密度目前大多通过分子帽分割法进行研究[40],而蛋白质的电势分布能够影响蛋白质在带电表面的吸附行为[41],通过LipA 的表面电位预测构象可知LipA 的正电势基团的分布相对分散,而负电势基团分布相对集中,LipA 整体上呈弱正电势,这结果对于后续研究LipA 的分子表面吸附和电位差异有一定的意义。
2.10 LipA 的可及性分析
LipA 可及性模型的建模结果(图3c),一般情况下,处于分子表面的氨基酸残基(尤其是带正电荷的赖氨酸和精氨酸)的可及性较大,深埋于分子内部的残基可及性很小。判断氨基酸残基与溶剂的接触程度的强弱可以通过观察其颜色的强弱程度(黄色>绿色>蓝色)。
蛋白质的可及性反映了蛋白质表面的氨基酸残基与水或其他溶剂接触的可能性[42],通过建模发现LipA的表面氨基酸残基与水分子接触的可及性大,而LipA内部的氨基酸残基可及性小,这说明可及性的大小与蛋白质的结构有密切关系,对于研究蛋白质的结构有重要意义。
2.11 LipA 的Ramachandran 图分析
为了判断LipA 的构像合理性,本研究利用软件Swiss-Pdbviewer 4.1.0 进行拉氏分析得到Ramachandran Plot(拉氏构象图)见(图3d)。如坐标图中蓝线内区域内为一般允许区,蓝线外的区域为不允许区。一般而言,蛋白质结构模型中如果有超过90%的二面角位于一般允许区,则其空间结构具有稳定性。LipA 的结果(图3d)表明:拉式图中最合理区(黄线内)和一般允许区(蓝线内)以及不允许区(蓝线外)的残基数占比分别为91.7%、7.2%和1.1%,在允许区内总计占98.9%(大于90%),由此可知,利用trRosetta 进行建模得到的LipA 的模型是合理和可靠的。
Ramachandran 图可以用于描述蛋白质的主链碳的ψ角与Ф角所产生的低能构象,如果没有任何非键合原子间距离小于短接触准则的限制距离,则(φ,ψ)值被认为是允许的或无空间冲突的,Ramachandran 图还定义了蛋白质骨架的可访问构象空间的限制[43],因此Ramachandran 图可用于判断蛋白质构象的合理性。对LipA 的Ramachandran 图进行分析,得到LipA 的ψ角与Ф角的98.9%位于Ramachandran 的允许区,这说明通过建模得到的LipA 的三维构象具有科学性、合理性和可靠性。
3 结论
使用生物信息学方法分析LipA 的结构特性与蛋白质特性,得出枯草芽孢杆菌脂肪酶LipA 由212 个氨基酸组成,等电点为9.57,呈碱性;亲、疏水性最强的区域分别为Lys75~Asn81 和Val9~Leu17,该区域发生突变时可能会影响蛋白质的折叠,进而影响其结构与生理功能;其不稳定指数、脂溶指数和平均亲水性表明LipA 属于稳定的脂溶性蛋白质;但不存在稳定的二聚体、三聚体卷曲螺旋;有3 个不同的模体,可能参与了不同的生化反应;不存在非PEST 序列,可能具有较强的降解蛋白的能力;有5 段α-螺旋、6段β-折叠,属于典型的α/β类蛋白;整体呈弱正电势;LipA 的可及性和Ramachandran 图表明建模得到的三维结构是合理、可靠的。LipA 与其他微生物脂肪酶的不同之处主要在于其缺乏盖子结构,活性中心暴露,除此之外,LipA 的二、三级结构和催化活性基团均与其他微生物脂肪酶相似。本研究系统地分析了LipA的结构与蛋白质特性,为后续LipA 在提高表达量和热稳定性等方面的改造研究提供一定的科学依据和理论基础。
