抗SARS-CoV-2药物研究进展
2022-06-11白晓明王丽非洪斌
白晓明,王丽非,洪斌
·新型冠状病毒专栏·
抗SARS-CoV-2药物研究进展
白晓明,王丽非,洪斌
过去二十年全球曾经发生了两次重大的冠状病毒(coronavirus,CoVs)感染事件,分别是 2003 年暴发的严重急性呼吸窘迫综合征(severe acute respiratory syndromes,SARS)和 2012 年暴发的中东呼吸综合征(middle east respiratory syndrome,MERS)。2019 年,第三次冠状病毒感染事件暴发,新型冠状病毒 SARS-CoV-2 在全球大流行并造成了巨大的社会危害。最新数据显示,SARS-CoV-2 已导致全球超过 5.2 亿人感染,628 万人死亡[1]。鉴于疫情的严重形势,多个抗 SARS-CoV-2 药物已被各国政府紧急批准临床应用,如莫努匹韦、瑞德西韦、法匹拉韦、洛匹那韦/利托那韦等,世界卫生组织(WHO)也将 7 种 SARS-CoV-2 疫苗纳入紧急使用清单。遗憾的是,上述药物的特异性和临床治疗效果并不理想,而新冠病毒不断产生新的变异,导致目前广泛使用的多种疫苗防治作用减弱,成为全球抗疫最为棘手的问题。为此,本文针对目前新冠病毒的研究现状,综述了 SARS-CoV-2 生物学特征、基于宿主/病毒的治疗靶点和抗 SARS-CoV-2 药物,希望能为抗 SARS-CoV-2 新型药物的设计和开发提供新的思路。
1 SARS-CoV-2 的生物学特征
SARS-CoV-2 是一种正链 RNA 病毒,基因组从 5' 至 3' 分别为蛋白酶和复制酶编码区(ORF1ab)、结构蛋白(S、E、M 和 N)编码区、3' 非翻译区(UTRs)和多聚(A)尾。基因组包含 14 个开放阅读框(open reading frames,ORFs)编码 29 种蛋白质(图 1)。蛋白酶和复制酶编码区编码两种多聚蛋白 pp1a 和 pp1ab,多聚蛋白被病毒 nsp5 基因编码的3C样蛋白酶(3C-like protease,3CLpro)白(或称主蛋白酶)和nsp3 基因编码的木瓜蛋白酶样蛋酶(papain-like protease,PLpro)消化为 16 种非结构蛋白nsps1-16。非结构蛋白主要负责病毒基因组复制和转录,其中nsp12 是病毒RNA 依赖的 RNA 聚合酶(RNA-dependent RNA polymerase,RdRp),与辅因子 nsp7 和 nsp8 共同组装成复制转录复合体(replication and transcription complex,RTC),在病毒基因组的转录和复制中发挥至关重要的作用。而结构蛋白编码区主要编码刺突蛋白(S)、膜蛋白(M)、包膜蛋白(E)和核衣壳蛋白(N)[2]。S 蛋白是同源三聚体蛋白,在病毒表面形成独特的刺突结构类似皇冠[3],介导病毒膜与宿主细胞膜的融合,是病毒侵入宿主的关键蛋白。S 蛋白可被宿主蛋白酶裂解为两个功能域 S1 和 S2[4]。S1 亚基包含信号肽段(SP)和受体结合结构域(RBD),负责病毒与宿主细胞受体的结合;S2 包含两个七肽重复区(heptad region,HR1/2)、一个中心螺旋和跨膜区,介导病毒包膜和细胞膜融合[5]。
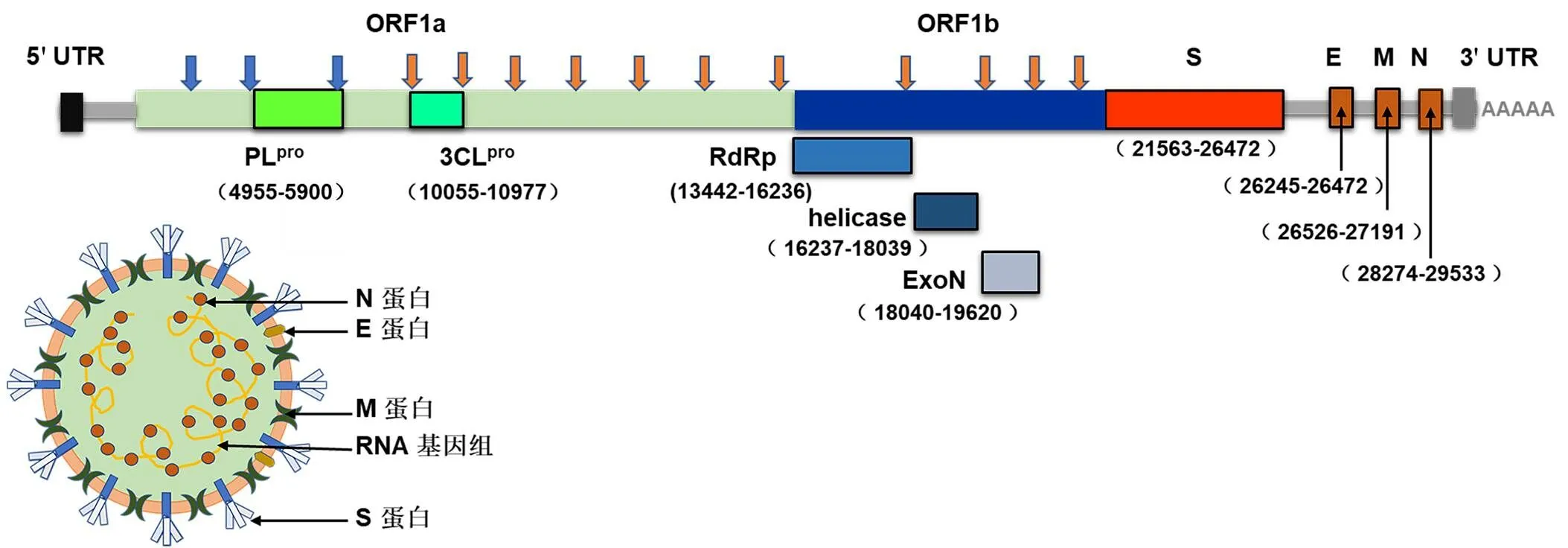
图1 SARS-CoV-2 基因组结构及其结构蛋白组成
2 依赖于宿主/病毒的治疗靶点和抗 SARS-CoV-2 药物
2.1 病毒侵袭宿主细胞的相关靶点
SARS-CoV-2 通过 S 蛋白与宿主细胞受体 ACE2 结合启动入侵过程,S 蛋白的水解活化需要跨膜丝氨酸蛋白酶(TMPRSS2)和弗林蛋白酶(furin)[6]参与。因此,S 蛋白、TMPRSS2、弗林蛋白酶和 ACE2 都是限制 SARS-CoV-2 侵入细胞的药物作用靶点。
2.1.1 S 蛋白
2.1.1.1 靶向 S 蛋白的中和抗体药物 截至 2021 年12 月,至少有 27 个国家的 274 家公司/机构正在开发抗体疗法[7],已有 25 个S 蛋白靶向性单克隆抗体进入临床试验,其中有 7 种单抗(巴尼韦单抗、埃特司韦单抗、卡斯瑞韦单抗、依米得韦单抗、索托韦单抗、西加韦单抗和替沙格韦单抗)获得美国FDA批准使用或者紧急使用(表 1)。巴尼韦单抗(LY-CoV555)是全球首个治疗新冠的单克隆抗体,以 S 蛋白的 RBD 为靶点[8]。II 期临床结果显示巴尼韦单抗减少病毒载量,降低新冠患者症状和住院率,FDA 批准用于治疗中度症状的成人或儿童[9]。虽然巴尼韦单抗对突变毒株 B.1.1.7 有效,但对 B.1.351、P.1 和 B.1.617.2 无效[10-13]。索托韦单抗是 S309 单抗的衍生物,III 期临床结果显示索托韦单抗治疗的患者(n = 291)住院(超过 24 h)或死亡的主要终点降低 85%。FDA 批准紧急使用 0.5 g 单剂量的索托韦单抗治疗高风险发展为重症的 12 岁或以上的儿童患者。REGN-COV2 是一种双抗体鸡尾酒疗法,其中包含针对 S 蛋白 RBD 的单抗卡斯瑞韦单抗和依米得韦单抗。单独使用两种单抗的抑制d值大约为 0.04 nmol/L,PRNT50值为0.0374 nmol/L 和 0.0421 nmol/L[14]。临床试验数据显示,REGN-COV2 可降低 70 % 死亡率和住院风险,非住院患者症状持续时间中位数从 14 d 缩短到 10 d。REGN-COV2 对 B.1.1.7(Alpha)、B.1.351(Beta)、P.1(Gamma)、B.1.429(Epsilon)和 B.1.526(Iota)均有效[15]。另一双抗组合策略 AZD7442 是利用分离自新冠感染者的抗体替沙格韦单抗和西加韦单抗[16-17]。预防和暴露后预防的III 期临床数据显示 AZD7442 降低了 83% 感染风险,感染开始 3 d 内给药降低了 88% 的重症或死亡风险[18]。
2.1.1.2 S 蛋白与宿主细胞 ACE2 融合抑制剂 由于 S 蛋白的HR1 和 HR2 结构域介导病毒与细胞膜融合,根据这两个七肽重复序列可以设计抗病毒肽类抑制剂,阻断膜融合。Xia 等[19]参考泛冠状病毒融合抑制剂(EK1 肽)分别制备了 HR1 和 HR2 的类似肽(HR1P 和 HR2P),HR2P 抑制 IC50为 0.18 μmol/L。为了增加抗病毒效力并改善药物动力学,Xia 等[20]通过聚乙二醇(polyethylene glycol,PEG)和(或)氨基酸将胆固醇连接到 EK1 的 C 端合成了一系列脂肽,其中 EK1C4 能有效抑制 SARS-CoV-2 S 蛋白介导的细胞融合和假病毒感染,并在小鼠模型中可预防 HCoV-OC43 感染。Zhu 等[21]基于 HR2 序列开发了一种脂肽融合抑制剂 IPB02,抑制 S 蛋白 IC50为 0.025 μmol/L,单周期感染试验中对 SARS-CoV-2 感染的抑制 IC50为 0.08 μmol/L。
阿比多尔最初用于治疗甲型流感和乙型流感病毒引起的上呼吸道感染。近年来的研究证实了其对 SARS-CoV 和 MERS-CoV 的抑制作用[22]。阿比多尔可有效阻断 SARS-CoV-2 S 蛋白的三聚体化[23],导致宿主细胞中的 S 蛋白稳定性显著下降,最终被溶酶体降解。一项临床研究发现给药 14 d 后,洛匹那韦/利托那韦对照组有 15 例(44.1%)检测到病毒,阿比多尔组新冠患者全部病毒转阴,并且 RNA 检测阳性持续时间短(< 0.01)[24],表明阿比多尔具有发展为抗新冠药物的潜力。
2.1.2 蛋白酶 S 蛋白的激活过程依赖宿主蛋白酶 TMPRSS2 和弗林蛋白酶,它们分别在 S2 位点和 S1/S2 位点裂解 S 蛋白,因此可以根据 TMPRSS2 和弗林蛋白酶切割位点序列设计蛋白酶短肽抑制剂,并组合两种序列来协同抑制。Bestle 等[25]发现 TMPRSS2 抑制剂 MI-432 和 MI-1900 显著抑制 SARS-CoV-2 在 Calu-3 细胞中增殖导致的细胞病变效应(cytopathic effect,CPE),20 μmol/L 的 MI-432 和 50 μmol/L 的 MI-1900 处理细胞后感染病灶变小。TMPRSS2 抑制剂甲磺酸卡莫司他可阻断 SARS-CoV 和HCoV-NL63 对 HeLa 细胞的感染[4]。Hoffmann 等[26]在细胞试验中证实甲磺酸卡莫司他和其类似物甲磺酸萘莫司他可有效阻断 SARS-CoV-2 进入 Calu-3 细胞,EC50分别为 87 nmol/L 和 5 nmol/L,目前有 5 个临床试验正在评估甲磺酸卡莫司他的疗效。此外,溶酶体中的组织蛋白酶对病毒通过胞吞作用进入宿主至关重要,阿洛司他丁是组织蛋白酶 L(cathepsin L,CatL)的抑制剂,可阻断 SARS-CoV 和 SARS-CoV- 2 假病毒的感染[27]。
表1 FDA 批准或紧急批准使用的抗体药物
名称公司剂量批准日期 巴尼韦单抗Eli Lilly/AbCellera700 mg2020.11.09 巴尼韦单抗/埃特司韦单抗Eli Lilly/AbCellera/Junshi700 mg/1400 mg2021.09.16 卡斯瑞韦单抗/依米得韦单抗Regeneron600 mg/600 mg2021.08.10 索托韦单抗Vir biotechnology/GlaxoSmithKline500 mg2021.05.26 西加韦单抗/替沙格韦单抗AstraZeneca/Vanderbilt University Medical Center150 mg/150 mg2020.12.08
弗林蛋白酶是一种前蛋白转化酶(PC),可被酸性 pH 环境激活[28]。Ivanova 等[29]发现弗林蛋白酶抑制剂在细胞实验中表现出对西尼罗病毒和登革病毒的抗病毒活性。一种弗林蛋白酶抑制剂(MI-1851)可以强烈抑制新冠病毒在 Calu-3 细胞中的复制,10 μmol/L 处理可以使病毒滴度降低96.67% ~ 99.47%。此外,MI-1851 与 TMPRSS2 抑制剂 MI-432 具有协同效应,联合用药使病毒滴度又降低 90% ~ 96.67%[25]。Cheng 等[30]发现弗林蛋白酶抑制剂 decanoyl-RVKR-chloromethylketone(CMK)和萘荧光素(图 2)可以抑制蛋白裂解和合胞体形成。空斑法测定 IC50值分别为 0.057 μmol/L 和 9.025 μmol/L。CMK 的选择性指数(selectivity index,SI)为 5567,萘荧光素的 SI 为 6.36。
2.1.3 血管紧张素转换酶 2 重组人血管紧张素转换酶 2(recombinant human angiotensin-converting enzyme 2,rhACE2)又称 APN01,通过干扰 S 蛋白与宿主细胞 ACE2 受体的相互作用,阻断 SARS-CoV-2 进入宿主细胞[31]。在 Vero E6 细胞中,rhACE2 可以将病毒载量降低 99.9% ~ 99.98%。人肾脏类器官和血管类器官经 rhACE2 处理后,显著抑制 SARS-CoV-2 的入侵[32]。一项临床研究表明 rhACE2(GSK2586881)治疗可以改善急性呼吸窘迫综合征患者的临床指标,并且具有良好的耐受性[33]。氯喹和羟氯喹(图 2)是传统的抗疟药,它们对内溶酶体和高尔基体的脱酸作用可能阻止 SARS-CoV-2 整合到宿主细胞中。氯喹还可以通过减少宿主细胞表面 ACE2 末端糖基化,干扰 S 蛋白与 ACE2 的结合来抑制病毒侵入[34]。氯喹和羟氯喹都被证明可阻断 SARS-CoV-2 从初级内体到内溶酶体(endolysosomes,Els)的运输[35]。氯喹能在低剂量下有效阻断SARS-CoV-2 对Vero E6 细胞的感染,且 SI 值较高(EC50= 1.13 μmol/L,CC50> 100 μmol/L,SI > 88.50)。
2.2 病毒复制相关靶点
2.2.1 RdRp 目前用于新冠治疗的 RdRp 抑制剂有莫努匹韦、瑞德西韦、法匹拉韦、利巴韦林和喷昔洛韦[36-37]等,均属于 RdRp 核苷类抑制剂。在 E6 细胞模型中发现瑞德西韦的抗 SARS-CoV-2 IC50为 0.99 μmol/L[38]。早期的一项随机临床试验结果表明接受瑞德西韦 5 d 治疗的患者与标准治疗相比,其临床症状减轻。但是世卫组织和美国国家过敏症和传染病研究所赞助的临床研究最终结果都表明,瑞德西韦治疗新冠患者并无明显效果。因此,世卫组织建议不对 SARS-CoV-2 患者使用瑞德西韦[39]。
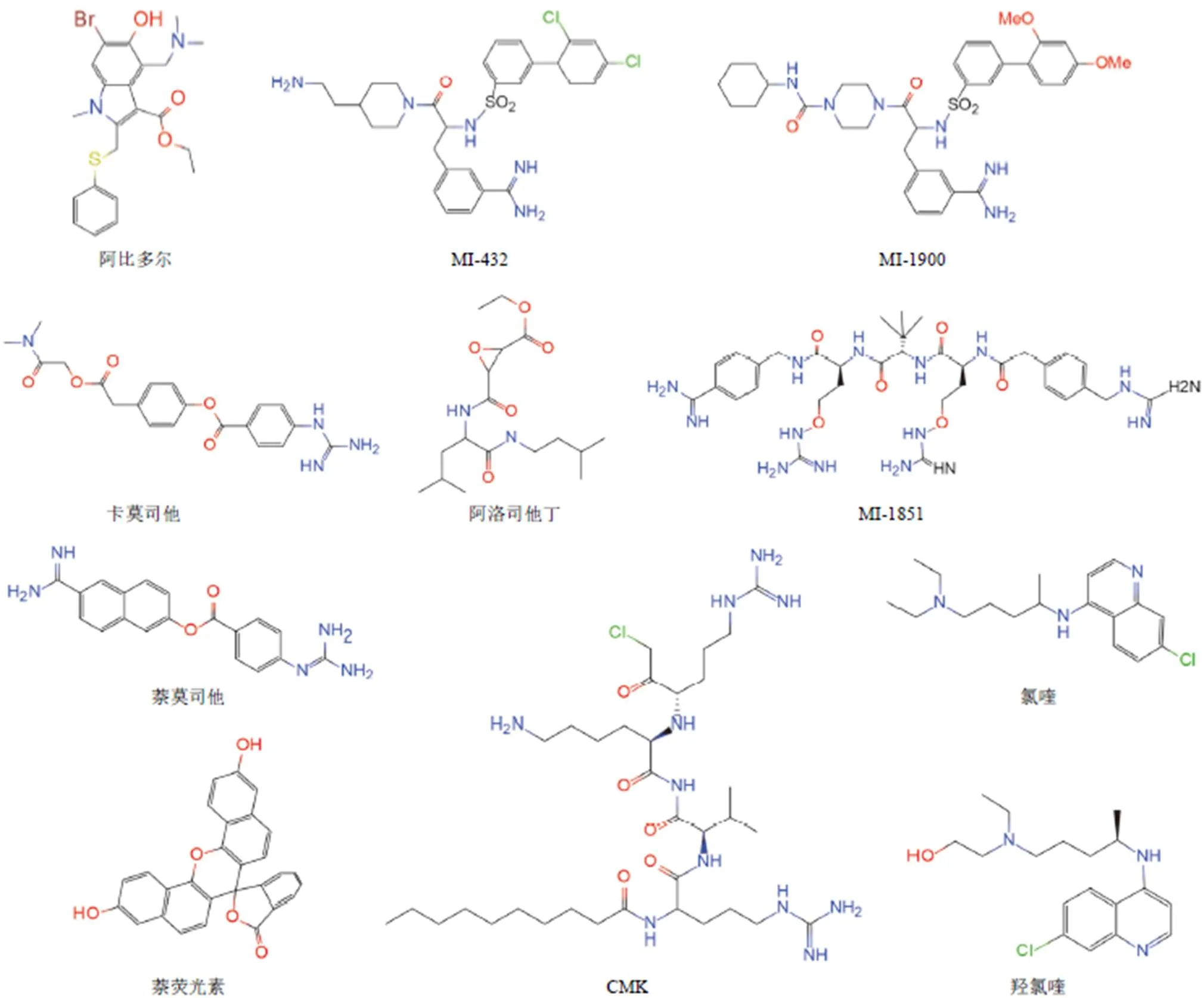
图2 SARS-CoV-2 进入宿主相关靶点的抑制剂
法匹拉韦是一种经过修饰的吡嗪类似物(也称为 6-氟-3-羟基-2-吡嗪羧酰胺,T-705),在日本被批准用于抗病毒的治疗。法匹拉韦对感染新冠病毒的 Vero E6 细胞的抑制 EC50值为 61.88 μmol/L[40]。在临床试验中,法匹拉韦治疗组的病毒清除中位时间更短(内距 2.5 ~ 9.0 d),与对照组(利托那韦/洛匹那韦治疗)(内距 8.0 ~ 13.0 d)相比具有显著差异(< 0.01),胸部 CT 改善率为 91.43%,显著高于对照组62.22%[41]。另一项新冠肺炎的临床试验中,法匹拉韦和奥司他韦联合治疗,可以在 14 d 后显著改善患者的临床症状(62.5% vs 42.2%,= 0.0247)[42]。最新的研究还发现法匹拉韦可能通过诱导分子间或分子内模板转换,引发 RNA 合成缺陷发挥抗病毒作用,此机制给药物设计提供了新的方向[43],但法匹拉韦在动物实验中显现出的血液毒性、肝脏毒性还有致畸性是不容忽视的[44]。
最新开发的抗病毒口服药物莫努匹韦是一个 β-D-N4-羟基胞苷(NHC)前药,酯键水解后得到 NHC。在 Vero细胞中,NHC 抗新冠病毒 IC50为 0.3 μmol/L,CC50>10 μmol/L,在 Calu-3 细胞中 IC50为 0.08 μmol/L[45]。在一项对轻度或中度新冠患者开展的 III 期临床研究中,服用莫努匹韦患者中病重/病死率为 7.3%(28/385),相比于安慰剂组的 14.1%(53/377),莫努匹韦将病重/病死率降低了 50%(= 0.0012)。目前,莫努匹韦已经在英国批准上市,用于治疗重症和住院风险较高的轻至中度成人新冠患者。
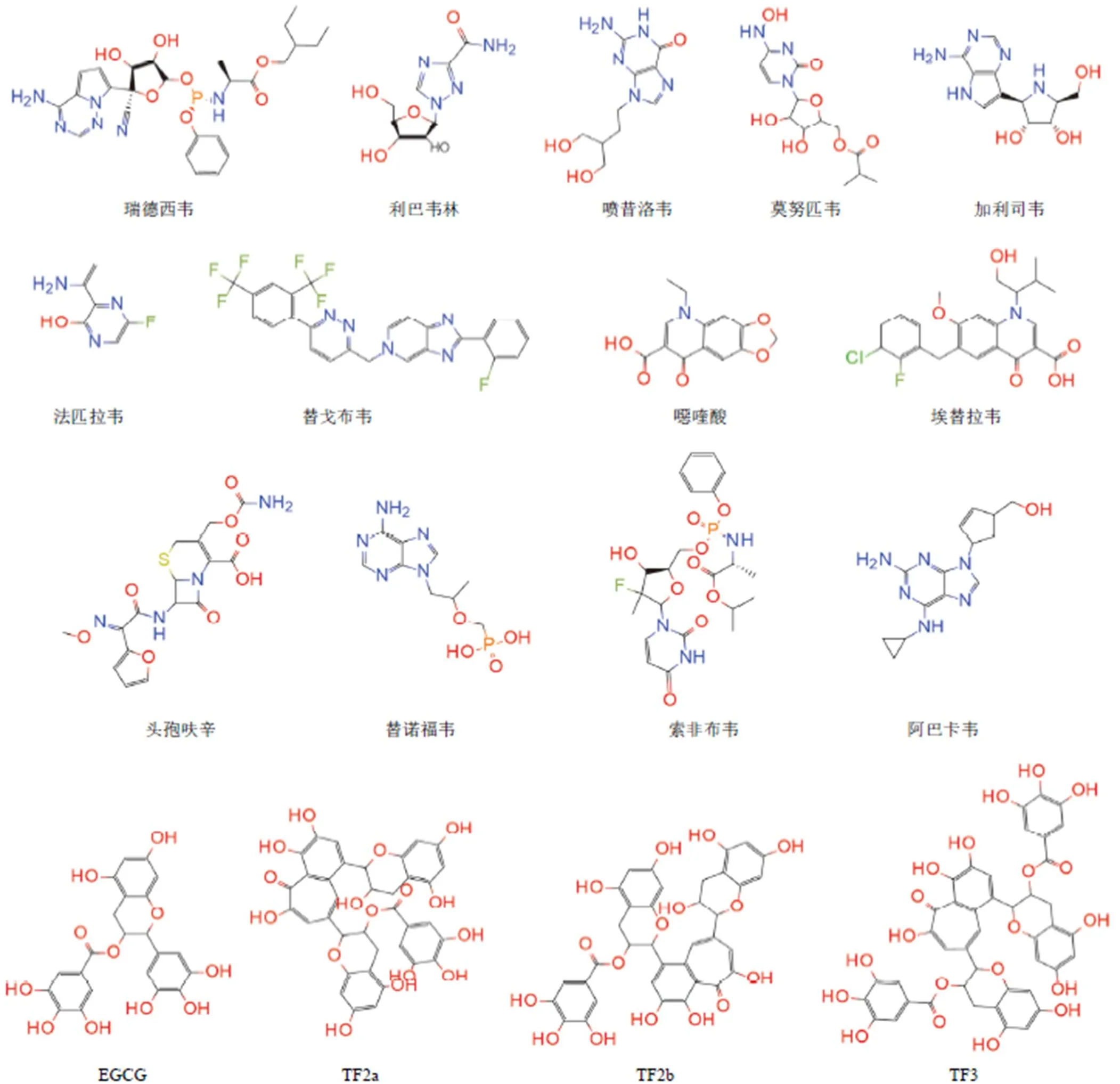
图3 SARS-CoV-2 RdRp 抑制剂
在虚拟对接实验中也发现了一些 RdRp 小分子抑制剂,如加利司韦、头孢呋辛、替诺福韦、羟氯喹、替戈布韦、索非布韦和阿巴卡韦,均可作为潜在的 RdRp 酶抑制剂[46-47]。喹啉类药物中,埃替拉韦和噁喹酸能够干扰 RdRp 的聚合酶活性[48]。植物来源的多酚类化合物 EGCG、TF2a、TF2b 和 TF3 对 RdRp 具有稳定的结合亲和力和良好的药代动力学特性[49],显示出一定的开发价值。细胞实验中,索非布韦可抑制 Huh-2(人肝癌来源)和 Calu-3(II 型肺细胞来源)细胞中 SARS-CoV-2 的复制[50]。RdRp 各抑制剂的化学结构见图 3。
2.2.2 3CLpro3CLpro作为阻碍 SARS-CoV-2 复制的治疗靶点广受关注[51],目前已开发出一系列 3CLpro抑制剂,根据其结构可分为拟肽类抑制剂和非肽类抑制剂。Konno 等[52]设计合成了以苯并噻唑为弹头基团的拟肽化合物 1 和 2(图 4),对SARS-CoV-2 3CLpro的抑制常数(i)分别为 4.1 和 3.0 nmol/L。Dai 等[53]开发了一类新的醛基类拟肽抑制剂,其中化合物 3 和 4(图 4)对 3CLpro的抑制活性 IC50分别为 0.053 和 0.04 μmol/L,并且在细胞实验 EC50值分别为 0.42 和 0.33 μmol/L,有潜力成为新的抗 SARS-CoV-2 临床候选药物。Zhang 等[54-55]优化 α-酮酰胺抑制剂得到化合物 5 对 SARS-CoV-2 3CLpro具有较强的抑制作用,并且药代动力学特性良好。Hoffman 等[56]发现了一种潜在的小分子蛋白酶抑制剂 PF-00835231,在 Vero 细胞中病毒 CPE EC50值分别为 39.7 μmol/L(SARS-CoV-2 Washington strain 1)和 88.9 μmol/L(Belgium/GHB-03021/2020 strain),细胞试验中 PF-00835231 与瑞德西韦联用具有协同作用[57]。辉瑞公司开发了一种口服抗病毒候选药物 PF-07321332,显示出广谱抗冠状病毒活性并且具有良好的药代动力学特性[58]。由 PF-07321332 和利托那韦组成的药物Paxlovid,是首款获 FDA 批准可紧急用于治疗新冠病毒感染的口服药物。临床试验数据显示,该药物可以降低 89% 的病重或死亡率。
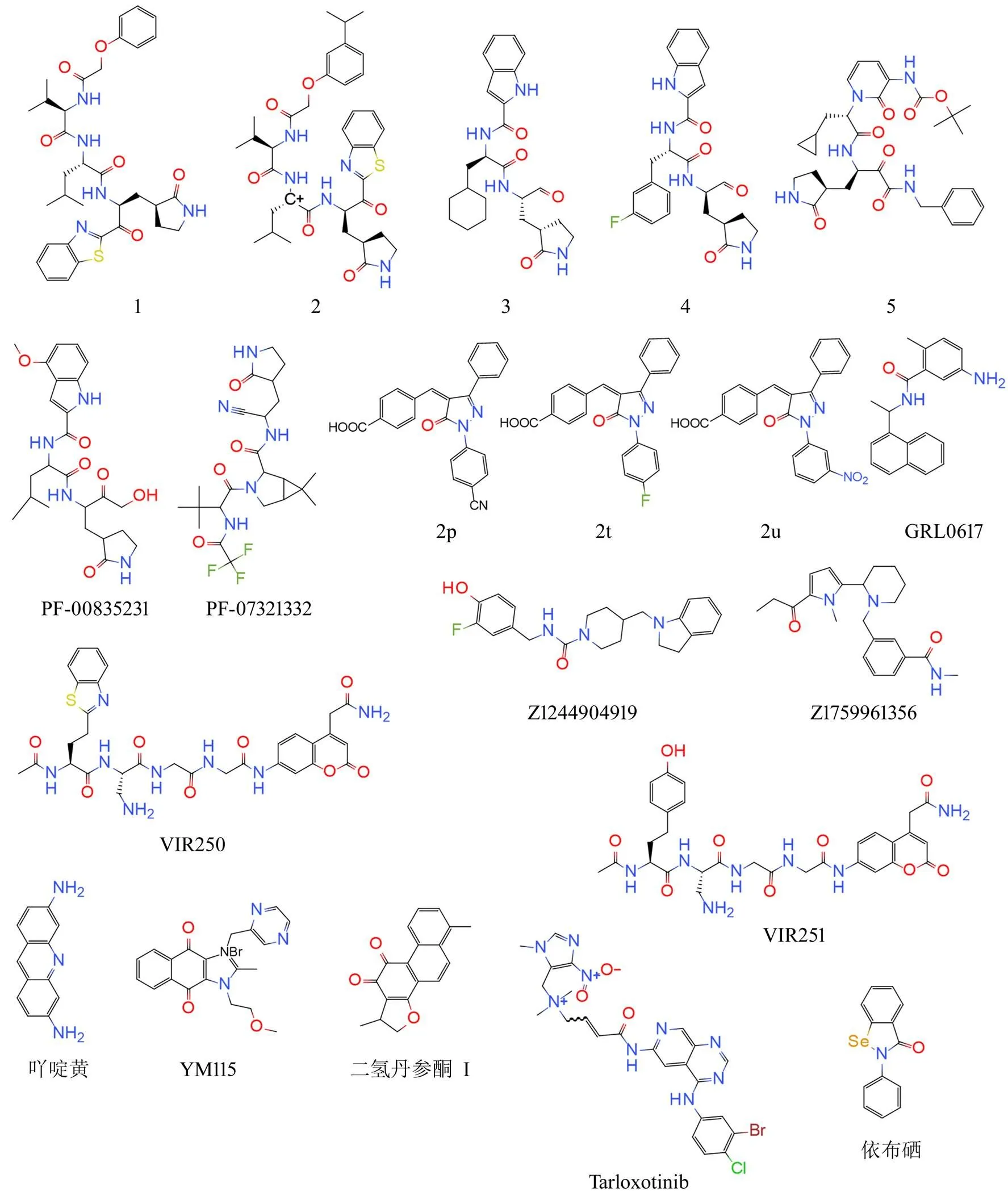
图4 SARS-CoV-2 蛋白酶抑制剂
3CLpro非肽类小分子抑制剂主要通过氢键、疏水键和范德华力与活性位点的残基相互作用,发挥抑制作用[59]。Ramajayam 等[60]设计了一系列吡唑啉酮核心化合物,化合物 2p 对 SARS-CoV 3CLpro的 IC50为 5.5 μmol/L,2t 的 IC50为 6.8 μmol/L。Yang 等[61]通过虚拟筛选发现 Z1244904919 和 Z1759961356 均能抑制 SARS-CoV-2 导致的 Vero E6 细胞病变效应,EC50分别为(4.98 ± 1.83)和(8.52 ± 0.92)μmol/L。另有学者利用双胺支架合成了一系列 SARS-CoV 3CLpro抑制剂,构效关系研究表明,吡啶或三唑是抑制活性所必需的[61-62],这一发现为非肽抑制剂的设计提供了新的思路。十氢异喹啉、八氢异色烯、不对称芳二硫化物还有一些天然产物也表现出良好的抑制活性,可以进一步研究和修饰[59]。
2.2.3 PLproPLpro除了切割病毒多聚蛋白,还通过去除细胞蛋白中的泛素(Ub)和泛素样蛋白干扰素诱导基因 15(ISG15)参与调节免疫反应,其抑制剂可以通过抑制病毒复制和促进抗病毒免疫双重方式发挥作用[63],因此 PLpro被公认为是具有潜力的抗病毒药物靶点[64]。Fu 等[65]在体外实验中证实了 GRL0617 对 PLpro的抑制 IC50为 2.1 μmol/L。在 Vero 细胞中,GRL0617 浓度在100 μmol/L 时病毒复制抑制率超过 50%,CPE 分析显示 EC50为(21 ± 2)μmol/L。Rut 等[66]检测了 SARS-CoV-2 PLpro在 P4-P2 位置的底物偏好,并开发出 VIR250 和 VIR251 选择性地抑制 SARS-CoV 和 SARS-CoV-2 PLpro。Napolitano等[67]发现吖啶黄在 A549 ACE2+细胞和 Vero 细胞中抑制病毒复制的 IC50值分别为 86和 64 nmol/L。在 K18-ACE2 小鼠感染模型中,每天口服 100 mg/kg ACF 或肌肉注射5 mg/kg 都可以完全阻断 SARS-CoV-2 对小鼠大脑的感染。一种以咪唑为基础的化合物 YM-115[68],可抑制 SARS CoV-2 PLpro蛋白酶活性,IC50为 2.47 μmol/L。二氢丹参酮 I[69]、tarloxotinib[70]、依布硒[71]抑制 PLpro的 IC50分别为8 μmol/L、300 nmol/L、2 μmol/L。而具有羟基或甲氧基的依布硒衍生物的 IC50值在纳摩尔范围内[71]。在 Vero E6 细胞中,依布硒以 4 μmol/L的 EC50抑制 SARS-Co-V2 复制[72]。
3 总结和展望
自 2019 年新冠疫情暴发以来,新冠的大流行已经对人类的健康造成了极大的威胁,同时也对全球的经济发展造成了巨大的影响。两年来持续的新冠疫情时刻提醒我们,目前针对冠状病毒感染的治疗手段仍然非常有限。在疫情早期,研究者通过已有药物的再利用、计算模拟设计和改造抑制剂等手段,缩短了药物开发的时间并降低了成本,但获得的药物往往在临床试验中表现不佳,例如洛匹那韦、利托那韦、瑞德西韦[73]等。随着我们对SARS-CoV-2 结构特征、生物学性质、感染过程以及与宿主的关系的认识不断深入,病毒感染周期中各个阶段重要的靶点逐渐显露,研究者们开始针对特定靶点进行药物的全新设计,不断有单克隆抗体、抗病毒融合肽、小分子酶抑制剂、免疫抑制剂等抗新冠药物进入临床试验阶段。值得注意的是,与 SARS-CoV 相比,SARS-CoV-2 具有更高的突变频率[74],新的毒株在全世界不断涌现,现有的药物和疫苗都不能确保针对 SARS-CoV-2 突变体有效[75]。人类与 SARS-CoV-2 的斗争短期之内无法结束,未来的药物研发需要密切关注 SARS-CoV-2 基因突变,不断根据新的靶点设计新的疫苗和抗病毒药物,同时结合临床诊断分析,注重对病毒造成的宿主免疫系统的变化所引起的逃逸、耐药性和发病机制的研究。
[1] World Health Organization. WHO coronavirus (COVID-19) dashboard. [2022-05-26]. https://covid19.who.int/.
[2] Naqvi AAT, Fatima K, Mohammad T, et al. Insights into SARS-CoV-2 genome, structure, evolution, pathogenesis and therapies: Structural genomics approach. Biochim Biophys Acta Mol Basis Dis, 2020, 1866(10):165878.
[3] Delmas B, Laude H. Assembly of coronavirus spike protein into trimers and its role in epitope expression. J Virol, 1990, 64(11): 5367-5375.
[4] Tang T, Bidon M, Jaimes JA, et al. Coronavirus membrane fusion mechanism offers a potential target for antiviral development. Antiviral Res, 2020, 178:104792.
[5] Coutard B, Valle C, De Lamballerie X, et al. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Res, 2020, 176:104742.
[6] Hoffmann M, Kleine-Weber H, Schroeder S, et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor. Cell, 2020, 181(2):271-280, e8.
[7] Yang L, Liu W, Yu X, et al. COVID-19 antibody therapeutics tracker: a global online database of antibody therapeutics for the prevention and treatment of COVID-19. Antib Ther, 2020, 3(3):205-212.
[8] Kaplon H, Reichert JM. Antibodies to watch in 2021. mAbs, 2021, 13(1):1860476.
[9] Chen P, Nirula A, Heller B, et al. SARS-CoV-2 neutralizing antibody LY-CoV555 in outpatients with Covid-19. N Engl J Med, 2021, 384(3): 229-237.
[10] Wang P, Nair MS, Liu L, et al. Antibody resistance of SARS-CoV-2 variants B.1.351 and B.1.1.7. Nature, 2021, 593(7857):130-135.
[11] Wang P, Casner RG, Nair MS, et al. Increased resistance of SARS-CoV-2 variant P.1 to antibody neutralization. Cell Host Microbe, 2021, 29(5):747-751, e4.
[12] Chen RE, Winkler ES, Case JB, et al. In vivo monoclonal antibody efficacy against SARS-CoV-2 variant strains. Nature, 2021, 596(7870): 103-108.
[13] Hoffmann M, Hofmann-Winkler H, Krüger N, et al. SARS-CoV-2 variant B.1.617 is resistant to Bamlanivimab and evades antibodies induced by infection and vaccination. Cell Rep, 2021, 36(3):109415.
[14] Baum A, Fulton BO, Wloga E, et al. Antibody cocktail to SARS-CoV-2 spike protein prevents rapid mutational escape seen with individual antibodies. Science, 2020, 369(6506):1014-1018.
[15] Weinreich DM, Sivapalasingam S, Norton T, et al. REGN-COV2, a neutralizing antibody cocktail, in outpatients with Covid-19. N Engl J Med, 2021, 384(3):238-251.
[16] Zost SJ, Gilchuk P, Case JB, et al. Potently neutralizing and protective human antibodies against SARS-CoV-2. Nature, 2020, 584(7821): 443-449.
[17] Dong J, Zost SJ, Greaney AJ, et al. Genetic and structural basis for SARS-CoV-2 variant neutralization by a two-antibody cocktail. Nat Microbiol, 2021, 6(10):1233-1244.
[18] Mahase E. Covid-19: AstraZeneca says its antibody drug AZD7442 is effective for preventing and reducing severe illness. BMJ, 2021, 375: n2860.
[19] Xia S, Yan L, Xu W, et al. A pan-coronavirus fusion inhibitor targeting the HR1 domain of human coronavirus spike. Sci Adv, 2019, 5(4): eaav4580.
[20] Xia S, Liu M, Wang C, et al. Inhibition of SARS-CoV-2 (previously 2019-nCoV) infection by a highly potent pan-coronavirus fusion inhibitor targeting its spike protein that harbors a high capacity to mediate membrane fusion. Cell Res, 2020, 30(4):343-355.
[21] Zhu Y, Yu D, Yan H, et al. Design of potent membrane fusion inhibitors against SARS-CoV-2, an emerging coronavirus with high fusogenic activity. J Virol, 2020, 94(14):e00635-20.
[22] Rosa SGV, Santos WC. Clinical trials on drug repositioning for COVID-19 treatment. Rev Panam Salud Publica, 2020, 44:e40.
[23] Vankadari N. Arbidol: A potential antiviral drug for the treatment of SARS-CoV-2 by blocking trimerization of the spike glycoprotein. Int J Antimicrob Agents, 2020, 56(2):105998.
[24] Zhu Z, Lu Z, Xu T, et al. Arbidol monotherapy is superior to lopinavir/ritonavir in treating COVID-19. J Infect, 2020, 81(1):e21- e23.
[25] Bestle D, Heindl MR, Limburg H, et al. TMPRSS2 and furin are both essential for proteolytic activation of SARS-CoV-2 in human airway cells. Life Sci Alliance, 2020, 3(9):e202000786.
[26] Hoffmann M, Schroeder S, Kleine-Weber H, et al. Nafamostat mesylate blocks activation of SARS-CoV-2: New treatment option for COVID-19. Antimicrob Agents Chemother, 2020, 64(6):e00754-20.
[27] Zhou N, Pan T, Zhang J, et al. Glycopeptide antibiotics potently inhibit cathepsin L in the late endosome/lysosome and block the entry of Ebola virus, middle east respiratory syndrome coronavirus (MERS-CoV), and severe acute respiratory syndrome coronavirus (SARS-CoV). J Biol Chem, 2016, 291(17):9218-9232.
[28] Jin X, Xu K, Jiang P, et al. Virus strain from a mild COVID-19 patient in Hangzhou represents a new trend in SARS-CoV-2 evolution potentially related to Furin cleavage site. Emerg Microbes Infect, 2020, 9(1):1474-1488.
[29] Ivanova T, Hardes K, Kallis S, et al. Optimization of substrate-analogue furin inhibitors. ChemMedChem, 2017, 12(23): 1953-1968.
[30] Cheng YW, Chao TL, Li CL, et al. Furin inhibitors block SARS-CoV-2 spike protein cleavage to suppress virus production and cytopathic effects. Cell Rep, 2020, 33(2):108254.
[31] Shah JN, Guo GQ, Krishnan A, et al. Peptides-based therapeutics: Emerging potential therapeutic agents for COVID-19. Therapie, 2021. [Online ahead of print]
[32] Monteil V, Kwon H, Prado P, et al. Inhibition of SARS-CoV-2 infections in engineered human tissues using clinical-grade soluble human ACE2. Cell, 2020, 181(4):905-913, e7.
[33] Khan A, Benthin C, Zeno B, et al. A pilot clinical trial of recombinant human angiotensin-converting enzyme 2 in acute respiratory distress syndrome. Crit Care, 2017, 21(1):234.
[34] Liu J, Cao R, Xu M, et al. Hydroxychloroquine, a less toxic derivative of chloroquine, is effective in inhibiting SARS-CoV-2 infection in vitro. Cell Discov, 2020, 6:16.
[35] Chen X, Geiger JD. Janus sword actions of chloroquine and hydroxychloroquine against COVID-19. Cell Signal, 2020, 73: 109706.
[36] Gordon CJ, Tchesnokov EP, Woolner E, et al. Remdesivir is a direct-acting antiviral that inhibits RNA-dependent RNA polymerase from severe acute respiratory syndrome coronavirus 2 with high potency. J Biol Chem, 2020, 295(20):6785-6797.
[37] Tchesnokov EP, Feng JY, Porter DP, et al. Mechanism of inhibition of Ebola virus RNA-dependent RNA polymerase by remdesivir. Viruses, 2019, 11(4):326.
[38] Pizzorno A, Padey B, Dubois J, et al. In vitro evaluation of antiviral activity of single and combined repurposable drugs against SARS-CoV-2. Antiviral Res, 2020, 181:104878.
[39] World Health Organization. WHO recommends against the use of remdesivir in COVID-19 patients. [2022-11-20]. https://www. who.int/news-room/feature-stories/detail/who-recommends-against-the-use-of-remdesivir-in-covid-19-patients.
[40] Wang M, Cao R, Zhang L, et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro. Cell Res, 2020, 30(3):269-271.
[41] Cai Q, Yang M, Liu D, et al. Experimental treatment with favipiravir for COVID-19: An open-label control study. Engineering (Beijing), 2020, 6(10):1192-1198.
[42] Wang Y, Fan G, Salam A, et al. Comparative effectiveness of combined favipiravir and oseltamivir therapy versus oseltamivir monotherapy in critically ill patients with influenza virus infection.J Infect Dis, 2020, 221(10):1688-1698.
[43] Janissen R, Woodman A, Shengjuler D, et al. Induced intra- and intermolecular template switching as a therapeutic mechanism against RNA viruses. Mol Cell, 2021, 81(21):4467-4480, e7.
[44] Delang L, Abdelnabi R, Neyts J. Favipiravir as a potential countermeasure against neglected and emerging RNA viruses. Antiviral Res, 2018, 153:85-94.
[45] Sheahan TP, Sims AC, Zhou S, et al. An orally bioavailable broad-spectrum antiviral inhibits SARS-CoV-2 in human airway epithelial cell cultures and multiple coronaviruses in mice. Sci Transl Med, 2020, 12(541):eabb5883.
[46] Mosayebnia M, Hajiagha Bozorgi A, Rezaeianpour M, et al. In silico prediction of SARS-CoV-2 main protease and polymerase inhibitors:3D-Pharmacophore modelling. J Biomol Struct Dyn, 2021:1-18.
[47] Elfiky AA. SARS-CoV-2 RNA dependent RNA polymerase (RdRp) targeting: an in silico perspective. J Biomol Struct Dyn, 2021, 39(9): 3204-3212.
[48] Alexpandi R, De Mesquita JF, Pandian SK, et al. Quinolines-based SARS-CoV-2 3CLpro and RdRp inhibitors and Spike-RBD-ACE2 inhibitor for drug-repurposing against COVID-19: an in silico analysis. Front Microbiol, 2020, 11:1796.
[49] Singh S, Sk MF, Sonawane A, et al. Plant-derived natural polyphenols as potential antiviral drugs against SARS-CoV-2 via RNA-dependent RNA polymerase (RdRp) inhibition: an in-silico analysis. J Biomol Struct Dyn, 2021, 39(16):6249-6264.
[50] Sacramento CQ, Fintelman-Rodrigues N, Temerozo JR, et al. In vitro antiviral activity of the anti-HCV drugs daclatasvir and sofosbuvir against SARS-CoV-2, the aetiological agent of COVID-19. J Antimicrob Chemother, 2021, 76(7):1874-1885.
[51] Morse JS, Lalonde T, Xu S, et al. Learning from the past: possible urgent prevention and treatment options for severe acute respiratory infections caused by 2019-nCoV. Chembiochem, 2020, 21(5):730- 738.
[52] Konno S, Thanigaimalai P, Yamamoto T, et al. Design and synthesis of new tripeptide-type SARS-CoV 3CL protease inhibitors containing an electrophilic arylketone moiety. Bioorg Med Chem, 2013, 21(2): 412-424.
[53] Dai W, Zhang B, Jiang XM, et al. Structure-based design of antiviral drug candidates targeting the SARS-CoV-2 main protease. Science, 2020, 368(6497):1331-1335.
[54] Zhang L, Lin D, Kusov Y, et al. α-Ketoamides as broad-spectrum inhibitors of coronavirus and enterovirus replication: structure-based design, synthesis, and activity assessment. J Med Chem, 2020, 63(9):4562-4578.
[55] Zhang L, Lin D, Sun X, et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors. Science, 2020, 368(6489):409-412.
[56] Hoffman RL, Kania RS, Brothers MA, et al. Discovery of ketone-based covalent inhibitors of coronavirus 3CL proteases for the potential therapeutic treatment of COVID-19. J Med Chem, 2020, 63(21):12725-12747.
[57] Boras B, Jones RM, Anson BJ, et al. Preclinical characterization of an intravenous coronavirus 3CL protease inhibitor for the potential treatment of COVID19. Nat Commun, 2021, 12(1):6055.
[58] Owen DR, Allerton CMN, Anderson AS, et al. An oral SARS-CoV-2 Mproinhibitor clinical candidate for the treatment of COVID-19. Science, 2021, 374(6575):1586-1593.
[59] Liu Y, Liang C, Xin L, et al. The development of coronavirus 3C-like protease (3CL(pro)) inhibitors from 2010 to 2020. Eur J Med Chem, 2020, 206:112711.
[60] Ramajayam R, Tan KP, Liu HG, et al. Synthesis and evaluation of pyrazolone compounds as SARS-coronavirus 3C-like protease inhibitors. Bioorg Med Chem, 2010, 18(22):7849-7854.
[61] Yang J, Lin X, Xing N, et al. Structure-based discovery of novel nonpeptide inhibitors targeting SARS-CoV-2 M(pro). J Chem Inf Model, 2021, 61(8):3917-3926.
[62] Turlington M, Chun A, Tomar S, et al. Discovery of N-(benzo[1,2,3]triazol-1-yl)-N-(benzyl)acetamido)phenyl) carboxamides as severe acute respiratory syndrome coronavirus (SARS-CoV) 3CLpro inhibitors: identification of ML300 and noncovalent nanomolar inhibitors with an induced-fit binding. Bioorg Med Chem Lett, 2013, 23(22):6172-6177.
[63] Chen Z, Cui Q, Cooper L, et al. Ginkgolic acid and anacardic acid are specific covalent inhibitors of SARS-CoV-2 cysteine proteases. Cell Biosci, 2021, 11(1):45.
[64] Shin D, Mukherjee R, Grewe D, et al. Papain-like protease regulates SARS-CoV-2 viral spread and innate immunity. Nature, 2020, 587(7835):657-662.
[65] Fu Z, Huang B, Tang J, et al. The complex structure of GRL0617 and SARS-CoV-2 PLpro reveals a hot spot for antiviral drug discovery. Nat Commun, 2021, 12(1):488.
[66] Rut W, Lv Z, Zmudzinski M, et al. Activity profiling and crystal structures of inhibitor-bound SARS-CoV-2 papain-like protease: A framework for anti-COVID-19 drug design. Sci Adv, 2020, 6(42):eabd4596.
[67] Napolitano V, Dabrowska A, Schorpp K, et al. Acriflavine, a clinically approved drug, inhibits SARS-CoV-2 and other betacoronaviruses. Cell Chem Biol, 2022. [Online ahead of print]
[68] Zhao Y, Du X, Duan Y, et al. High-throughput screening identifies established drugs as SARS-CoV-2 PLpro inhibitors. Protein Cell, 2021, 12(11):877-888.
[69] Lim CT, Tan KW, Wu M, et al. Identifying SARS-CoV-2 antiviral compounds by screening for small molecule inhibitors of Nsp3 papain-like protease. Biochem J, 2021, 478(13):2517-2531.
[70] Redhead MA, Owen CD, Brewitz L, et al. Bispecific repurposed medicines targeting the viral and immunological arms of COVID-19. Sci Rep, 2021, 11(1):13208.
[71] Weglarz-Tomczak E, Tomczak JM, Talma M, et al. Identification of ebselen and its analogues as potent covalent inhibitors of papain-like protease from SARS-CoV-2. Sci Rep, 2021, 11(1):3640.
[72] Jin Z, Du X, Xu Y, et al. Structure of Mprofrom SARS-CoV-2 and discovery of its inhibitors. Nature, 2020, 582(7811):289-293.
[73] Magro P, Zanella I, Pescarolo M, et al. Lopinavir/ritonavir: Repurposing an old drug for HIV infection in COVID-19 treatment. Biomed J, 2021, 44(1):43-53.
[74] Shang J, Wan Y, Liu C, et al. Structure of mouse coronavirus spike protein complexed with receptor reveals mechanism for viral entry. PLoS Pathog, 2020, 16(3):e1008392.
[75] Lesté-Lasserre C. Mutant coronaviruses found in mink spark massive culls and doom a Danish group’s research. (2020-11-11) [2022-05-26]. https://www.science.org/content/article/mutant-coronaviruses-found-mink-spark-massive-culls-and-doom-danish-group-s-research.
10.3969/j.issn.1673-713X.2022.03.001
中国医学科学院医学与健康科技创新工程重大协同创新项目(2020-I2M-2-010)
100050 北京,中国医学科学院北京协和医学院医药生物技术研究所国家卫生健康委员会抗生素生物工程重点实验室/中国医学科学院药物合成生物学重点实验室
王丽非:Email:lifeiwang2002@hotmail.com;洪斌:Email:binhong69@hotmail.com
2021-12-03
