NanoLuc的发展及其在药物研发中的应用
2022-06-11张志恒刘建喜
张志恒,刘建喜
·综述·
NanoLuc的发展及其在药物研发中的应用
张志恒,刘建喜
生物发光是生物产生光的一种自然现象。当小分子萤光素被萤光素酶催化氧化,形成发光激发态时,就会出现生物发光的现象。不同的萤光素-萤光素酶具有不同的发光波长,因此适用于不同的需求。在过去十年左右的时间里,蛋白质工程、合成化学和物理学取得了巨大的进步,使得萤光素和萤光素酶的应用得到不断拓展。生物发光反应现在广泛用于基因分析、蛋白质-蛋白质相互作用的检测、药物发现中的高通量筛选(HTS)、卫生控制、生态环境污染分析和小型哺乳动物的活体成像等。
萤光素酶和相应的底物已被开发并应用了十余年,如萤火虫萤光素酶(firefly luciferase,FLuc)等。NanoLuc 萤光素酶(NLuc)作为最新商业化的萤光素酶[1]已被用于生物医学研究,包括研究蛋白质-蛋白质相互作用、研究遗传调控和细胞信号转导、监测蛋白质表达的稳定性、基于生物发光共振能量转移(BRET)原理的传感器和分子成像。NLuc 萤光素酶具有显著的优点,但与传统萤光素酶系统一样也有其局限性。本文将介绍萤光素酶的种类,重点讨论 NLuc 在药物研发中的应用。
1 萤光素酶的种类
1.1 萤火虫萤光素酶
1985 年,Kricka 和Leach[2]报道了在大肠杆菌中克隆萤火虫萤光素酶,其底物是 D-萤光素,FLuc 与其底物的反应式如图1A 所示。与其他萤光素-萤光素酶系统相比,其优势在于 D-萤光素在氧化前需要被 ATP 激活,它对自动氧化的敏感性更低,在溶液中更稳定,背景化学发光更少。FLuc 作为报告基因的特点是其分子量为 62 kD,蛋白表达在细胞内,发光颜色为黄绿色,半衰期为 3 h,其他特点如表1 所示。
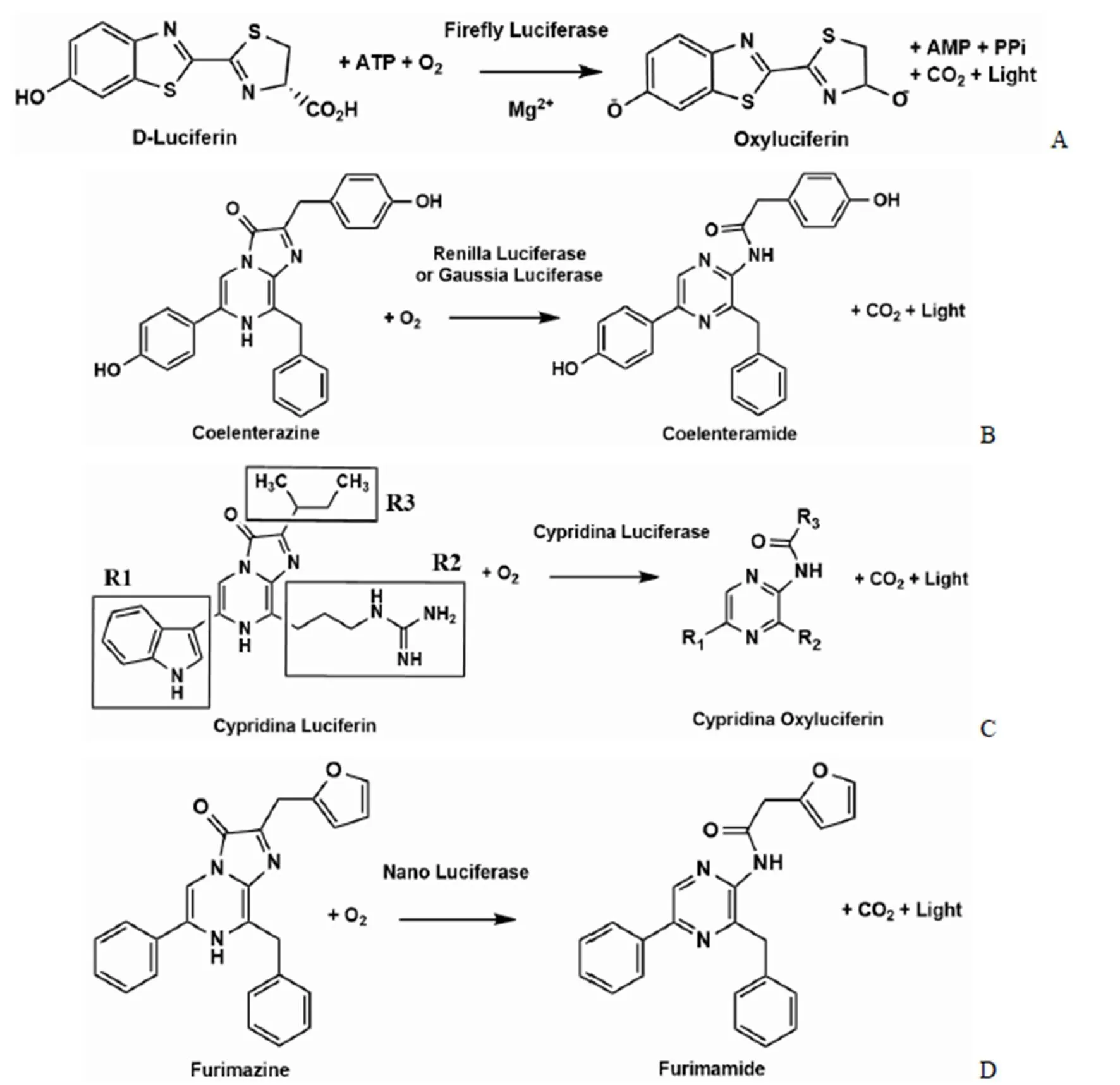
图1 萤光素酶催化其底物的反应式(A:FLuc 催化其底物的反应式;B:RLuc 和 GLuc 催化其底物的反应式;C:CLuc 催化其底物的反应式;D:NLuc 催化其底物的反应式)
表1 萤光素酶的种类与特点
名称底物稳定性可否互补灵敏度多色检测参考文献 萤火虫萤光素酶(firefly luciferase)D-萤光素(D-Luciferin)(C11H8N2O3S2)++可互补+可用于多色检测[3] 海肾萤光素酶(renilla luciferase)腔肠素(Coelenterazine)(C26H21N3O3)++可互补++可用于多色检测[4-5] 高斯萤光素酶(gaussia luciferase)腔肠素(Coelenterazine)(C26H21N3O3)++可互补+++可用于多色检测[6] 海萤萤光素酶(cypridina luciferase)海萤萤光素(Cypridina luciferin)+ +++可用于多色检测[7] NanoLucFurimazine(C24H19N3O2)+++可互补++++可用于多色检测[1, 8]
由于 D-萤光素-FLuc 系统产生 ATP 依赖的光输出,该方法已被广泛应用于各种系统中 < 100 nm 的 ATP 浓度的测量,通常用于确定细菌污染的程度[9]。它还用于监测 ATP 形成反应和 ATP 降解反应[10]。随着更灵敏的光度计的出现和分析技术的改进,现在可以使用市售的 ATP 生物发光试剂和试剂盒常规检测低至阿托摩尔水平的 ATP(对应于单个细菌细胞)。D-萤光素具有最高的量子产率(41.0 ± 7.4)%,而大多数萤光素-萤光素酶对的量子产率仅为 15% ~ 30%;FLuc 系统具有最长的发射波长(λmax= 558 nm),更适合于血液的成像应用[11]。哺乳动物细胞和生物实体,如细菌、真菌、原生动物和病毒,可以用合适的萤光素酶基因进行基因编码[12],并导入小型哺乳动物体内进行增殖、跟踪和成像。
D-萤光素的细胞通透性并不是很好,因此在活体实验中必须大量使用[13]。使用14C标记的放射性 D-萤光素底物的研究也表明底物在啮齿动物中的分布不均匀[14]。此外,在一些器官,如大脑,对探针的摄取很差[15]。D-萤光素与不同的辅助剂突变体相互作用能发出不同波长的光,发射波长的范围不适合活体多参数成像,特别是在深层组织成像,因为获得的 D-萤光素和 FLuc 突变体的红移最大为620 nm[16],这个波长的光在组织中会被吸收、衰减和散射,使得不同萤光素酶的光谱分离更具挑战性[17]。
1.2 海肾萤光素酶
在 20 世纪 70 年代,Matthews 等[18]从海参中纯化了海肾萤光素酶(renilla luciferase,RLuc),其底物是腔肠素。RLuc(34 kD)分子量小于 FLuc,这使得它更适合于小载体和蛋白质的应用。RLuc 的蛋白表达在细胞内,发光颜色为蓝色,半衰期为 5 h(表1)。与 FLuc 相比,RLuc 的量子产率较低,而且酶效率也较低[18]。
为了鉴定 HSP90/CDC37 PPI 的抑制剂,Siddiqui 等[19]建立了一种基于哺乳动物细胞的中等通量筛选方法,将海肾萤光素酶分为了两个亚基,再把人类 HSP90 和 CDC37 与海肾萤光素酶的 N 端和 C 端片段融合而构成一个筛选系统。萤光素酶的生物发光传感器在分子生物学和成像系统中有着广泛的应用。Mokhtar-Ahmadabadi 等[20]使用循环置换策略设计并构建了一种用于检测和测量caspase-9 活性的基于海肾萤光素酶的生物传感器。Dou 等[21]使用 mKATE-RLuc评估了在勃起功能障碍动物模型中追踪植入的人胎盘基质细胞的功效。
腔肠素是比 D-萤光素更大的分子,水溶性差,由于不需要以腺苷酸化的形式激活,因此也容易发生自氧化,导致溶液中的化学发光较多。此外,腔肠素还可以通过其他机制转运到细胞中。例如,多药耐药性 P-糖蛋白(MDR1 Pgp)介导腔肠素转运到细胞系中,导致更多的腔肠素被转运到表达更多 MDR1 Pgp 的癌细胞中。从而导致肿瘤在小型哺乳动物体内成像不准确,即无法检测到不表达 MDR1 Pgp 的肿瘤[22]。
1.3 高斯萤光素酶
高斯萤光素酶(Gaussia luciferase,GLuc)是一种分泌型的蓝色萤光素酶,其底物是腔肠素,GLuc 与底物的催化反应式如图1B。该萤光素酶最初是从海洋桡足类动物中提取的[23]。其蛋白分子量更小,只有22 kD,含有天然的分泌型信号肽,可以引导萤光素酶分泌到细胞培养上清中,从而无需裂解细胞即可检测报告基因活性,并可用于活细胞实时动力学分析。而体内试验也不需要处死动物,直接取微量的动物血液或尿液,就能用于定量检测肿瘤细胞生长增殖情况及抗癌药物药效评价[24]。GLuc 没有细胞毒性[24],当作为报告基因转染细胞后,对细胞实验没有影响。它的半衰期比较短,在体内只有 20 min,萤光性质稳定,含有 GLuc 的细胞培养基在 4 ℃可以保存6 d,而在–80 ℃可以保存几个月。
在监测肿瘤发生发展情况和肿瘤模型药物治疗评价方面,Chung 等[25]应用 GLuc 进行了乳腺癌脑转移、骨转移、肺转移及其他器官的研究。Shresthai 等[26]应用 GLuc 联合感光试剂原卟啉 IX 治疗小鼠黑色素瘤。在高通量药物筛选方面,Capul 和 de la Torre[27]应用 GLuc 开发高通量筛选平台筛选抗沙粒病毒芽孢的候选抑制剂,但其荧光信号衰减较快,影响信号采集。在其他应用方面,Wille 等[28]用 GLuc 作为报告基因,用蛋白互补分析法研究沙门菌中 SipA 与 InvB 的相互作用。Suzuki 等[29]将表达 GLuc 的质粒转染 CHO 细胞,发现分子内的二硫键是 GLuc 沿内质网-高尔基体分泌到胞外的必需结构。
1.4 海萤萤光素酶
大约 80% 的发光生物生活在海洋中[30],也被称为海萤火虫,是最简单的海洋生物之一,身长只有 2 ~ 3 mm[31]。海萤萤光素酶(cypridina luciferase,CLuc)是从中提取的,它也是一种分泌型萤光素酶,呈蓝紫色,蛋白分子量大小有 62 kD。同 GLuc 一样,大部分萤光素酶产物会分泌到胞外,可对活细胞实施连续检测,非常方便。由于信号很强,细胞内残留的萤光素酶还足以进行常规的裂解细胞检测,可实现多重检测。
如图1C 所示,该反应只需要分子氧、海萤萤光素酶和海萤萤光素,随着反应的进行可发出最大发射波长为 465 nm 的生物发光[31]。与其他生物发光反应不同,该系统不需要任何辅因子,从而简化了其在生物分析和生物成像中的使用。该系统另一个值得注意的特点是它的量子产率为 28%[32]。由于这些特点,海萤萤光素酶在酶免疫分析[33]、成像[34]、双光子荧光探针、免疫组织学[35]、能量转移分析[36]和作为基因报告[37]等方面是一个不可或缺的工具。
1.5 NLuc 萤光素酶
NLuc 是萤光素酶系统家族的最新成员。该萤光素酶来源于深海虾,经过三轮诱变优化酶的发光输出而获得[1]。NLuc 比 FLuc 或 RLuc 的发光量高两个数量级,在难转染的细胞中也能获得较好的结果。NLuc 简单易用,且信号稳定,可以不经任何改良,实现从实验台操作到高通量筛选的规模放大应用。使用 Nano-Glo 萤光素酶检测试剂,发光量在跨越 1 000 000 倍的浓度范围内呈线性关系,且信号半衰期≥ 2 h。
NLuc 使用的底物为 Furimazine,是一种新型的底物,其反应机制如图1D 所示。它发光更亮,比现有的生物发光酶用途更加广泛,是目前性能最好的生物发光报告基因之一。它具有无可比拟的小巧体积,可以用于增强病毒传递和蛋白融合。
2 NLuc 萤光素酶在药物研发中的应用
NLuc 系统凭借灵敏度高、稳定性高、体积小等优势,使得这一独特技术的应用越来越广泛,特别是在药物研发的领域。
2.1 基于萤光素酶的传感器
Griss 等[38]开发的基于萤光素酶的药物指示剂(LUCID),是半合成的生物发光传感器蛋白,由三个部分组成,分别是目标药物的受体蛋白、萤光素酶(NLuc)和自标记蛋白,如 SNAP-Tag。另一个合成分子由 BRET 萤光团和受体蛋白的配基组成,蛋白质与配体结合,导致 NLuc 与萤光团紧密结合,并由于 BRET 而红移发射。在药物分子存在的情况下,这种相互作用会受到干扰,因此测量蓝光/红光的比率就可以得到药物浓度的测量结果。后来针对靶向药物,还用抗体取代了生物传感器中的结合蛋白质[39]。研究表明,LUCID 既能检测小分子药物,也能检测较大的肽类和大环类药物浓度。它可以帮助医生评价疗效或确定给药方案,使得给药方案更加个体化。
2.2 基因检测
NLuc最常见的应用之一是用作报告基因,用于研究原核和真核细胞和系统中的基因表达[40]。这种分析可用于确定SARS-CoV-2 冠状病毒和在蝙蝠中发现的RaTG13 冠状病毒的感染[41]。冠状病毒中刺突糖蛋白与人类细胞中的细胞受体血管紧张素转换酶 2(ACE2)结合被认为是病毒进入人类宿主细胞的关键[42]。将包含 FLuc 基因和目标冠状病毒株的刺突蛋白基因的质粒共转染宿主细胞并孵育 72 h。收集这段时间后形成的假病毒,这些假病毒的表面具有所需的刺突蛋白,并在其内部具有编码 FLuc 的遗传物质。然后将这些假病毒与表达 ACE2 的人类细胞孵育 60 h。如果病毒能够成功感染人类细胞,就会产生子病毒和 FLuc,用于跟踪病毒感染过程和候选药物对病毒的作用。
2.3 蛋白质-蛋白质相互作用
裂解萤光素酶分析也被广泛地用于检测和评价蛋白质间的相互作用(PPI)[43]。萤光素酶被分成两个部分,其中一个部分由 N 端结构域组成,另一个由 C 端结构域组成。这两部分分别被标记到两个目标的蛋白质上。若两个蛋白之间有相互作用,萤光素酶的两个末端变得非常接近,因此重建了萤光素酶报告功能。
基于 BRET 的分析已经很好地研究了各种目标蛋白质-蛋白质相互作用,例如基于肿瘤学的靶标,包括p53/hDM2 以及体外和纤维素中的 G 偶联蛋白受体[44]。最近,使用 NanoBRET 技术成功检测到其他 PPI 的抑制剂或稳定剂,包括病毒 HCV NS5A 蛋白的定位和构象、PRAS40 和 Hippo 通路之间的相互作用分析[45]、细胞中 CD26-ADA-A2AR 三聚体复合物的分析、使用 miniG 蛋白作为 GPCR 的探针[46]以及分析 H2 松弛素蛋白和 RXFP1 蛋白之间的相互作用[47]。随着发出红光的 BRET 受体的发展,NanoBRET 技术也被用于测量乳腺癌小鼠模型体内的蛋白质-配体相互作用,以确定靶点[48]。NLuc在这一方面的应用可以帮助研究者确定药物靶点并进行潜在抑制剂的筛选。
2.4 活体成像
萤光素酶在分子水平上的生物发光成像所提供的灵敏度和特异性使其特别适用于小型哺乳动物的活体成像[49]。
为了进行活体生物发光成像,要对目标细胞进行基因改造,使其包含产生萤光素酶的基因。这些细胞被注射到小型哺乳动物的体内,当加入相应的萤光素时,就可以进行追踪。这样可以实时监测体内过程,而不需要牺牲动物。但是在对某些目标的成像上还具有一定的挑战性,例如在活体动物的大脑中对新的红移和明亮的萤光素-萤光素酶对进行成像。此外,还可用于感染性疾病的体外和活体发展成像[50]。基因修饰的生物发光病原体,如细菌、寄生虫、病毒和真菌,可在体内和体外进行监测,生物发光光输出已被证明与感染负荷相关。萤光素酶在该方面的应用使得成像变得更加方便。
2.5 在蛋白质稳定性监测中的应用
蛋白质聚集已被发现是导致某些人类疾病的关键因素,如亨廷顿病和阿尔茨海默病。虽然蛋白质聚集的作用已经有了很详细的文献记载,但在体外和体内研究蛋白质聚集的方法有限。
考虑到这些限制,Zhao 等[51]将 NLuc 系统分成两个部分监测活细胞中的蛋白质聚集。一部分 NLuc(N65)与已知聚集的蛋白质融合,而另一部分 NLuc 用于测量蛋白质聚集水平。如果蛋白质-NLuc 融合聚集,则第二部分NLuc的加入不会导致底物的生物发光;反之,在加入 Furimazine 时产生生物发光信号。NLuc 的两个片段在体外进行了鉴定,以确保这两个片段(N65 和 66C)都是产生发光信号所必需的。最后,将 NLuc 系统从细菌转移到哺乳动物细胞上,表明只有当 NIH-3T3 细胞同时转染两个 NLuc 片段时才能观察到发光,进一步验证了 NLuc 可用于监测体外蛋白质聚集。NLuc在体外和体内研究蛋白质聚集方面提供了新的方向。
2.6 高通量筛选
基于生物发光的分析方法通常用于药物发现,特别是针对细菌、病毒或寄生虫等传染性病原体的筛选。这是因为在活细胞测定中,高通量筛选的优点在于可以选择分析规模,如96 孔或384 孔板,以及快速、灵敏和直接的光输出读数。将目标基因、蛋白质、酶或病原体与萤光素酶基因一起进行基因编码,根据样品在加入底物时的光输出,可以确定萤光素酶的表达,同时也反映了病原体的复制情况。
用于高通量筛选的标签和报告基因必须满足所需的灵敏度、速度和经济性,迄今为止,没有标签或报告基因能同时满足所有的参数。基于 NLuc 新的优势,Boute 等[52]检测了 NLuc 是否适用于高通量筛选,首先构建了几个载体,可以克隆单链抗体 C 末端的 NLuc 或完整 IgG 分子的重链。将多克隆产物亚克隆到新的 NLuc 融合载体后,通过 scFv 文库筛选对新的报告系统进行应用和检验,对敏感性和对高通量筛选的适应性进行了评估。此外,NLuc 还展示了它在均质分析设置、竞争 ELISA 和 Western blot 中的多功能性。
Alport 综合征是一种遗传性肾小球疾病,由 IV 型胶原 a3-a5 链(a3-a5(IV))突变引起,如破坏三聚化,会导致肾小球基底膜变性。而纠正 A3/A4/A5 链的三聚化是一种可行的治疗方法,但由于缺乏关于细胞内 a(IV) 链调节的信息,以及缺乏高通量筛选(HTS)平台来评估 A345(IV) 三聚体的形成,导致在开发可行的治疗方面没那么顺利。Omachi 等[53]开发了一组分裂的 NLuc 融合 A345(IV) 蛋白来监测野生型和临床相关突变株 A5(IV) 的 A345(IV) 三聚化。筛选确定了几个有可能促进 A345(IV) 三聚体形成的化学伴侣。这种基于分裂 NLuc 的三聚体形成试验是一个功能性 HTS 平台,实现了靶向 A345(IV) 三聚体治疗 Alport 综合征的可行性。
目前还没有一种简单、快速和定量的检测方法,能够对这些分泌系统的生物学作用进行分析以及针对这些系统的抑制剂进行鉴定。Westerhausen 等[54]提出了一种通用的解决方案,利用小而非常明亮的 NLuc 来评估沙门氏菌致病岛 1 编码的 III 型分泌系统的功能。III 型分泌底物-NLuc 融合很容易分泌到培养上清中,去除细菌后通过发光法进行定量。基于 NLuc 的分泌物检测具有非常高的信噪比和低至纳升的灵敏度,该检测方法适用于高通量评估分泌动力学。
NLuc 在监测细胞膜上受体内化方面也有较为广泛的应用。2015 年,Liu 等[55]利用 NLuc 成功地监测了 G 蛋白偶联松弛素家族多肽受体(RXFP3)的内化,获得了一个稳定的 HEK293T 细胞系,该细胞系共表达 NLuc 标记的RXFP3 和增强型绿色荧光蛋白(EGFP)标记的 RXFP3。EGFP 用于通过显微镜观察受体内化过程,NLuc 用于通过生物发光对测量结果进行量化。若生物发光减少,则表明NLuc 标记的 RXFP3 已被内化到酸性溶酶体中。NLuc 除了能够监测 G 蛋白偶联受体内化,还可以用于发现该类受体的抑制剂或激动剂。在一项类似的研究中,Wu 等[56]研究了人松弛素与 RXFP1 的相互作用,他们发现 NLuc 结合的松弛素与 RXFP1 保持了高亲和力(d= 1.11 ± 0.08 nmol/L)。因此,NLuc 在监测 G 蛋白家族受体内化方面以及在高通量筛选 G 蛋白家族的激动剂和抑制剂方面起到了重要的作用。
除了以上的应用,图2 显示了基于 NLuc 开发的不同的高通量筛选方法。其中图2A 使用 NLuc 作为报告基因来检测细胞膜上的受体内化的情况[57],图2B 利用 NLuc 高通量筛选抗体[52]。图2C 和 2D 是将 NLuc 分成 HiBiT 和 LgBiT 两个亚基[58],图2E 和 2F 是将 NLuc 分成 SmBiT 和LgBiT 两个亚基[59]。HiBiT 和 SmBiT 标签是一种仅由11 个氨基酸组成的肽标签,当插入到目标蛋白中时,通过与 LgBiT 形成二聚体而起到 NLuc 的作用[42, 58]。而两者的区别是,HiBiT 与 LgBiT 之间的亲和力比 SmBiT 与LgBiT 之间的亲和力高[58],前两者能自发形成 NLuc 功能性复合物,而后两者因为较低的亲和力,不易自发形成完整的 NLuc。SmBiT 与 LgBiT 的结合需要借助外力,如图2F 中,当两个蛋白相互作用时,两个 NLuc 亚基距离足够近,此时才能互补形成完整的 NLuc[42]。
3 NLuc 萤光素酶在药物研发应用过程中的优势及局限
NLuc 凭借其独特的优势在药物研发中得到了广泛的应用,然而与其他萤光素酶技术一样,NLuc 也有局限性,这可能会导致它在分子生物学的某些领域受到限制。
3.1 优势
NLuc 平台的优点包括其增强的亮度、热稳定性、pH稳定性和在细胞中的无偏分布。此外,与绿色荧光蛋白(GFP)等其他报告基因不同,NLuc 不需要在哺乳动物细胞中进行翻译后修饰[60]。在这种条件下,报告基因可以低水平表达[61-62]。此外,NLuc 还改进了以前的类似技术,NanoBiT 和NanoBRET 可以用于体外和活体可逆地检测蛋白质相互作用[63]。NanoBRET 避免了 Forster 共振能量转移(FRET)与荧光团的问题,例如需要外部光源、供体荧光蛋白的光漂白、供体和受体分子的同时激发以及细胞或动物模型中的自发荧光。绝大多数 BRET 系统使用 NanoLuc/Furimazine 组合作为能量供体,因为它比 D-萤光素/FLuc 发光明显更亮,信号更强,并且分子量比 FLuc 小得多,这使得将其标记到目标蛋白质上不会影响蛋白质的功能。由于这些系统是可逆的,因此当它们与竞争性抑制剂孵育时,也可以用来监测相互作用的蛋白质的位移。在活体成像中使用荧光探针存在光漂白和背景自发光,放射性示踪剂保质期很短等问题,需要同步加速器才能生产,而生物发光探针不存在这些缺点。萤光素酶催化底物产生光,因此背景极低,信号/背景比极高。采用荧光测量的方法必须处理自发荧光背景的问题,使用红移染料必须要有良好的组织渗透性,而使用萤光素酶进行活体成像能避免这些问题。NLuc 平台与其他萤光素酶系统的多路复用能力,允许双萤光素酶分析,光谱发射曲线充分分离,以获得更大的动态范围和更高的灵敏度。
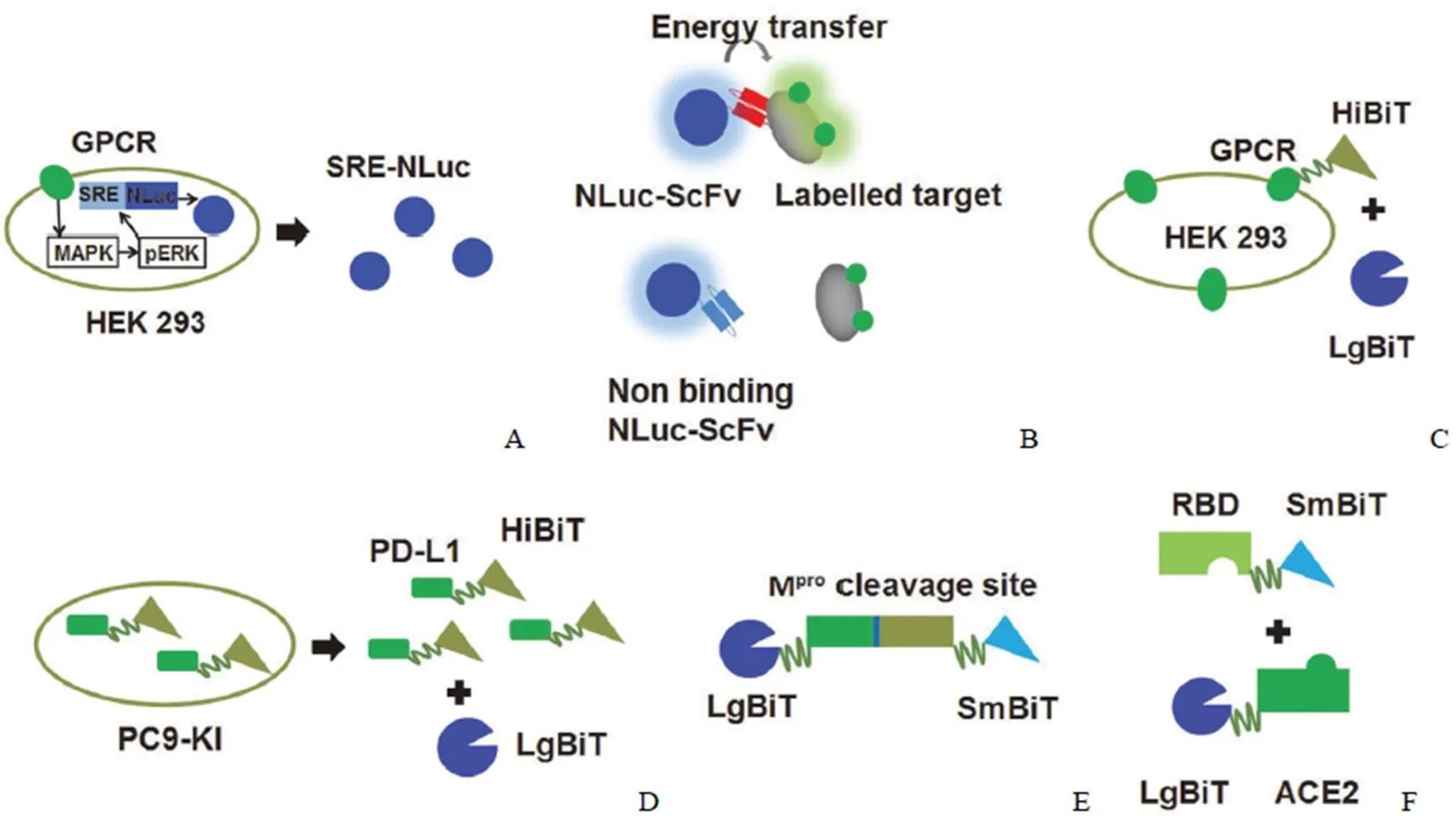
图2 NLuc 萤光素酶在高通量筛选中的应用[A:利用 NLuc 萤光素酶作为报告基因用于细胞膜受体内化的测定;B:利用 NLuc 萤光素酶高通量抗体筛选;C:利用 NLuc 萤光素酶(HiBiT + LgBiT)筛选 GPCR 的激动剂和抑制剂;D:利用 NLuc 萤光素酶(HiBiT + LgBiT)筛选调节 PD-L1 表达的化合物;E:利用 NLuc 萤光素酶(SmBiT + LgBiT)筛选 SARS-CoV-2 主蛋白酶的抑制剂;F:利用 NLuc 萤光素酶(SmBiT + LgBiT)筛选 SARS-CoV-2 RBD 和人 ACE2 相互作用的抑制剂]
3.2 局限性及解决方案
NLuc 在药物筛选应用过程中的第一个局限是它的发射波长。NLuc 的最大发射波长为 460 nm,相对于RLuc 和 GLuc 分别蓝移了约 20 nm 和 25 nm,这对于活体研究来说并不是最理想的。第二个潜在局限是其底物 Furimazine 仅针对 NLuc,且不易获得。因此,NLuc 平台的成本高于传统的萤光素酶系统,这可能会限制其在实验室的可及性。另外还有一个局限在于在高通量筛选中,使用萤光素酶作为报告基因可能导致某些化合物在抑制试验中出现假阳性或假阴性。FLuc、RLuc 和NLuc 都可以被各种小分子抑制,例如,FLuc 尤其受到与其底物 D-萤光素具有相似化学结构的化合物的竞争抑制,这些化合物包括苯并噻唑、苯并咪唑、苯并噁二唑和联芳基噁二唑。还有些化合物可能是结合在目标蛋白上从而影响了萤光素酶的构象,导致萤光素酶的活性发生了变化,从而导致假阳性结果。此外,一些化合物还可能导致报告酶转录或翻译增加,从而导致假阴性结果[64]。
对于第一个局限,由于 NLuc 不是活体成像的最佳候选者,但是利用 NLuc 的 BRET 能力可以有效地克服这一限制。对于第二个局限成本问题,随着该系统变得成熟,并在科学界得到更广泛的应用,成本问题也将不再是其局限了。第三点,为了克服 NLuc 在高通量筛选中出现的假阳性的不足,可以采用平行药物筛选靶点。通过两个甚至多个药物靶点的筛选相互作对照,比对同一个化合物对于不同药物靶点的作用,把共同的阳性小分子药物挑选出来,这可能就是 NLuc 的抑制剂。对该化合物进行进一步的确认,用非萤光素酶的方法来排除假阳性的结果,增加实验结果的可靠性。
还可以通过建立一个能够在给定筛选数据集中识别假阳性分子的化学信息学模型,来剔除药物筛选中出现的可能是假阳性的结果。Ghosh 等[65]探索了在基于萤光素酶的 HTS 检测中筛选和检测萤光素酶抑制剂的各种方法,如分子对接、SMARTS 筛选、药效团和机器学习方法等。了解抑制剂是如何与萤光素酶结合的,并进行了结构分析,以更好地了解此类抑制剂的化学性质。在基于结构的方法中,显示出良好的结果,模型准确率为 74.2%。然而,使用关联神经网络的机器学习方法的表现优于其他探索的方法,模型的准确率为 89.7%。该模型的高预测准确度有望用于鉴别哪些化合物是高通量筛选中潜在的萤光素酶抑制剂。在这项工作中开发的模型可以在 ochem 平台(http://ochem.eu)免费获得。
4 小结
随着时间的推移,生物发光机制的研究和各式发光生物的开发都取得了惊人的进展。早期发现并开发的萤光素酶如FLuc、RLuc、GLuc 等在生物医学领域得到了广泛的使用,最新商业化的 NLuc 在临床前和临床阶段具有广阔的前景。
[1] Hall MP, Unch J, Binkowski BF, et al. Engineered luciferase reporter from a deep sea shrimp utilizing a novel imidazopyrazinone substrate. ACS Chem Biol, 2012, 7(11):1848-1857.
[2] Kricka LJ, Leach FR. In memoriam Dr Marlene DeLuca. 1987 O. M. Smith Lecture. Firefly luciferase: mechanism of action, cloning and expression of the active enzyme. J Biolumin Chemilumin, 1989, 3(1):1-5.
[3] Villalobos V, Naik S, Piwnica-Worms D. Detection of protein-protein interactions in live cells and animals with split firefly luciferase protein fragment complementation. Methods Mol Biol, 2008, 439:339-352.
[4] Kato N, Jones J. The split luciferase complementation assay. Methods Mol Biol, 2010, 655:359-376.
[5] Lorenz WW, McCann RO, Longiaru M, et al. Isolation and expression of a cDNA encoding Renilla reniformis luciferase. Proc Natl Acad Sci U S A, 1991, 88(10):4438-4442.
[6] Remy I, Michnick SW. A highly sensitive protein-protein interaction assay based on Gaussia luciferase. Nat Methods, 2006, 3(12):977-979.
[7] Nakajima Y, Kobayashi K, Yamagishi K, et al. cDNA cloning and characterization of a secreted luciferase from the luminous Japanese ostracod, Cypridina noctiluca. Biosci Biotechnol Biochem, 2004, 68(3):565-570.
[8] Wang FZ, Zhang N, Guo YJ, et al. Split Nano luciferase complementation for probing protein-protein interactions in plant cells. J Integr Plant Biol, 2020, 62(8):1065-1079.
[9] Branchini BR, Southworth TL, Fontaine DM, et al. An enhanced chimeric firefly luciferase-inspired enzyme for ATP detection and bioluminescence reporter and imaging applications. Anal Biochem, 2015, 484:148-153.
[10] Lundin A. Optimization of the firefly luciferase reaction for analytical purposes. Adv Biochem Eng Biotechnol, 2014, 145:31-62.
[11] Weissleder R, Ntziachristos V. Shedding light onto live moleculartargets. Nat Med, 2003, 9(1):123-128.
[12] Contag CH, Spilman SD, Contag PR, et al. Visualizing gene expression in living mammals using a bioluminescent reporter. Photochem Photobiol, 1997, 66(4):523-531.
[13] Evans MS, Chaurette JP, Adams ST, et al. A synthetic luciferin improves bioluminescence imaging in live mice. Nat Methods, 2014, 11(4):393-395.
[14] Berger F, Paulmurugan R, Bhaumik S, et al. Uptake kinetics and biodistribution of 14C-D-luciferin--a radiolabeled substrate for the firefly luciferase catalyzed bioluminescence reaction: impact on bioluminescence based reporter gene imaging. Eur J Nucl Med Mol Imaging, 2008, 35(12):2275-2285.
[15] Iwano S, Sugiyama M, Hama H, et al. Single-cell bioluminescence imaging of deep tissue in freely moving animals. Science, 2018, 359(6378):935-939.
[16] Jathoul AP, Grounds H, Anderson JC, et al. A dual-color far-red to near-infrared firefly luciferin analogue designed for multiparametric bioluminescence imaging. Angew Chem Int Ed Engl, 2014, 53(48): 13059-13063.
[17] Stowe CL, Burley TA, Allan H, et al. Near-infrared dual bioluminescence imaging in mouse models of cancer using infraluciferin. Elife, 2019, 8:e45801.
[18] Matthews JC, Hori K, Cormier MJ. Purification and properties of Renilla reniformis luciferase. Biochemistry, 1977, 16(1):85-91.
[19] Siddiqui FA, Parkkola H, Manoharan GB, et al. Medium-throughput detection of Hsp90/Cdc37 protein-protein interaction inhibitors using a split renilla luciferase-based assay. SLAS Discov, 2020, 25(2):195- 206.
[20] Mokhtar-Ahmadabadi R, Madadi Z, Akbari-Birgani S, et al. Developing a circularly permuted variant of Renilla luciferase as a bioluminescent sensor for measuring Caspase-9 activity in the cell-free and cell-based systems. Biochem Biophys Res Commun, 2018, 506(4):1032-1039.
[21] Dou L, Matz EL, Gu X, et al. Non-invasive cell tracking with brighter and red-transferred luciferase for potential application in stem cell therapy. Cell Transplant, 2019, 28(12):1542-1551.
[22] Pichler A, Prior JL, Piwnica-Worms D. Imaging reversal of multidrug resistance in living mice with bioluminescence: MDR1 P-glycoprotein transports coelenterazine. Proc Natl Acad Sci U S A, 2004, 101(6): 1702-1707.
[23] Fraga H. Firefly luminescence: a historical perspective and recent developments. Photochem Photobiol Sci, 2008, 7(2):146-158.
[24] Tannous BA, Kim DE, Fernandez JL, et al. Codon-optimized Gaussia luciferase cDNA for mammalian gene expression in culture and in vivo. Mol Ther, 2005, 11(3):435-443.
[25] Chung E, Yamashita H, Au P, et al. Secreted Gaussia luciferase as a biomarker for monitoring tumor progression and treatment response of systemic metastases. PLoS One, 2009, 4(12):e8316.
[26] Shrestha TB, Seo GM, Basel MT, et al. Stem cell-based photodynamic therapy. Photochem Photobiol Sci, 2012, 11(7):1251-1258.
[27] Capul AA, de la Torre JC. A cell-based luciferase assay amenable to high-throughput screening of inhibitors of arenavirus budding. Virology, 2008, 382(1):107-114.
[28] Wille T, Blank K, Schmidt C, et al. Gaussia princeps luciferase as a reporter for transcriptional activity, protein secretion, and protein-protein interactions in Salmonella enterica serovar typhimurium. Appl Environ Microbiol, 2012, 78(1):250-257.
[29] Suzuki T, Usuda S, Ichinose H, et al. Real-time bioluminescence imaging of a protein secretory pathway in living mammalian cells using Gaussia luciferase. FEBS Lett, 2007, 581(24):4551-4556.
[30] Haddock SH, Moline MA, Case JF. Bioluminescence in the sea. Ann Rev Mar Sci, 2010, 2:443-493.
[31] Shimomura O, Johnson FH, Masugi T. Cypridina bioluminescence: light-emitting oxyluciferin-luciferase complex. Science, 1969, 164(3885):1299-1300.
[32] Shimomura O, Johnson FH. Mechanisms in the quantum yield of cypridina bioluminescence. Photochem Photobiol, 1970, 12(4):291- 295.
[33] Wu C, Kawasaki K, Ogawa Y, et al. Preparation of biotinylated cypridina luciferase and its use in bioluminescent enzyme immunoassay. Anal Chem, 2007, 79(4):1634-1638.
[34] Wu C, Mino K, Akimoto H, et al. In vivo far-red luminescence imaging of a biomarker based on BRET from Cypridina bioluminescence to an organic dye. Proc Natl Acad Sci U S A, 2009, 106(37):15599-15603.
[35] Wu C, Wang KY, Guo X, et al. Rapid methods of detecting the target molecule in immunohistology using a bioluminescence probe. Luminescence, 2013, 28(1):38-43.
[36] Wu C, Kawasaki K, Ohgiya S, et al. Chemical studies on the BRET system between the bioluminescence of Cypridina and quantum dots. Photochem Photobiol Sci, 2011, 10(10):1531-1534.
[37] Yamada Y, Nishide SY, Nakajima Y, et al. Monitoring circadian time in rat plasma using a secreted Cypridina luciferase reporter. Anal Biochem, 2013, 439(2):80-87.
[38] Griss R, Schena A, Reymond L, et al. Bioluminescent sensor proteins for point-of-care therapeutic drug monitoring. Nat Chem Biol, 2014, 10(7):598-603.
[39] Xue L, Yu Q, Griss R, et al. Bioluminescent antibodies for point-of-care diagnostics. Angew Chem Int Ed Engl, 2017, 56(25): 7112-7116.
[40] Iannotti FA, Pagano E, Guardiola O, et al. Genetic and pharmacological regulation of the endocannabinoid CB1 receptor in Duchenne muscular dystrophy. Nat Commun, 2018, 9(1):3950.
[41] Yang Y, Du L, Liu C, et al. Receptor usage and cell entry of bat coronavirus HKU4 provide insight into bat-to-human transmission of MERS coronavirus. Proc Natl Acad Sci U S A, 2014, 111(34):12516- 12521.
[42] Azad T, Singaravelu R, Taha Z, et al. Nanoluciferase complementation-based bioreporter reveals the importance of N-linked glycosylation of SARS-CoV-2 S for viral entry. Mol Ther, 2021, 29(6):1984-2000.
[43] Wehr MC, Rossner MJ. Split protein biosensor assays in molecular pharmacological studies. Drug Discov Today, 2016, 21(3):415-429.
[44] Dudgeon DD, Shinde SN, Shun TY, et al. Characterization and optimization of a novel protein-protein interaction biosensor high-content screening assay to identify disruptors of the interactions between p53 and hDM2. Assay Drug Dev Technol, 2010, 8(4): 437-458.
[45] Mo XL, Luo Y, Ivanov AA, et al. Enabling systematic interrogation of protein-protein interactions in live cells with a versatile ultra-high- throughput biosensor platform. J Mol Cell Biol, 2016, 8(3):271-281.
[46] Wan Q, Okashah N, Inoue A, et al. Mini G protein probes for active G protein-coupled receptors (GPCRs) in live cells. J Biol Chem, 2018, 293(19):7466-7473.
[47] Hoare BL, Bruell S, Sethi A, et al. Multi-component mechanism of H2 relaxin binding to RXFP1 through NanoBRET kinetic analysis. iScience, 2019, 11:93-113.
[48] Alcobia DC, Ziegler AI, Kondrashov A, et al. Visualizing ligand binding to a GPCR in vivo using nanoBRET. iScience, 2018, 6:280-288.
[49] Mezzanotte L, van't Root M, Karatas H, et al. In vivo molecular bioluminescence imaging: New tools and applications. Trends Biotechnol, 2017, 35(7):640-652.
[50] Hutchens M, Luker GD. Applications of bioluminescence imaging to the study of infectious diseases. Cell Microbiol, 2007, 9(10):2315- 2322.
[51] Zhao J, Nelson TJ, Vu Q, et al. Self-assembling nanoLuc luciferase fragments as probes for protein aggregation in living cells. ACS Chem Biol, 2016, 11(1):132-138.
[52] Boute N, Lowe P, Berger S, et al. NanoLuc luciferase - a multifunctional tool for high throughput antibody screening. Front Pharmacol, 2016, 7:27.
[53] Omachi K, Kamura M, Teramoto K, et al. A split-luciferase-based trimer formation assay as a high-throughput screening platform for therapeutics in alport syndrome. Cell Chem Biol, 2018, 25(5):634-643, e4.
[54] Westerhausen S, Nowak M, Torres-Vargas CE, et al. A NanoLuc luciferase-based assay enabling the real-time analysis of protein secretion and injection by bacterial type III secretion systems. Mol Microbiol, 2020, 113(6):1240-1254.
[55] Liu Y, Song G, Shao XX, et al. Quantitative measurement of cell membrane receptor internalization by the nanoluciferase reporter: Using the G protein-coupled receptor RXFP3 as a model. Biochim Biophys Acta, 2015, 1848(2):688-694.
[56] Wu QP, Zhang L, Shao XX, et al. Application of the novel bioluminescent ligand-receptor binding assay to relaxin-RXFP1 system for interaction studies. Amino Acids, 2016, 48(4):1099-1107.
[57] Robers MB, Binkowski BF, Cong M, et al. A luminescent assay for real-time measurements of receptor endocytosis in living cells. Anal Biochem, 2015, 489:1-8.
[58] Boursier ME, Levin S, Zimmerman K, et al. The luminescent HiBiT peptide enables selective quantitation of G protein-coupled receptor ligand engagement and internalization in living cells. J Biol Chem, 2020, 295(15):5124-5135.
[59] Zhang ZH, Meng QY, Liu JX, et al. Development of high throughput screening to discover SARS-CoV-2 main protease inhibitors with nanoBit complementation. Open Access J Biomed Sci, 2021, 3(6): 1369-1375.
[60] Coralli C, Cemazar M, Kanthou C, et al. Limitations of the reporter green fluorescent protein under simulated tumor conditions. Cancer Res, 2001, 61(12):4784-4790.
[61] Hasson SA, Fogel AI, Wang C, et al. Chemogenomic profiling of endogenous PARK2 expression using a genome-edited coincidence reporter. ACS Chem Biol, 2015, 10(5):1188-1197.
[62] Lackner DH, Carré A, Guzzardo PM, et al. A generic strategy for CRISPR-Cas9-mediated gene tagging. Nat Commun, 2015, 6:10237.
[63] Demont EH, Bamborough P, Chung CW, et al. 1,3-Dimethyl benzimidazolones are potent, selective inhibitors of the BRPF1 bromodomain. ACS Med Chem Lett, 2014, 5(11):1190-1195.
[64] Thorne N, Auld DS, Inglese J. Apparent activity in high-throughput screening: origins of compound-dependent assay interference. Curr Opin Chem Biol, 2010, 14(3):315-324.
[65] Ghosh D, Koch U, Hadian K, et al. Luciferase advisor: High-accuracy model to flag false positive hits in luciferase HTS assays. J Chem Inf Model, 2018, 58(5):933-942.
10.3969/j.issn.1673-713X.2022.03.012
226019 南通大学特种医学研究院
刘建喜,Email:liujianxi01@yahoo.com
2021-11-15
