成都市人源沙门菌基因组特征分析
2022-06-08武雅婷赵婉妤崔学文
肖 颖,武雅婷,赵婉妤,2,崔学文,黎 明,许 欣
沙门菌(Salmonella)是革兰阴性人兽共患病原菌,与人类食源性疾病密切相关。近年来,由沙门菌引起的食源性疾病暴发事件与日俱增。2010-2018年,四川省食源性疾病暴发事件中,44.01%由沙门菌感染引起,其中成都市的发病人数居四川省首位[1]。了解成都市沙门菌的基因组特征对于监测与防控沙门菌引发的食源性疾病具有重要意义,但目前未见成都市人源沙门菌全基因组分析及报道。
沙门菌的血清型种类复杂,截止目前,世界范围内已经发现了2 659种不同的沙门菌血清型[2]。然而传统的血清学分型方法工作量大、耗时长,全基因组测序(whole genome sequencing,WGS)作为一种快速且分辨率更高的方法,可通过测序数据预测血清型、分析耐药基因与毒力因子等,国外已有学者将其运用在沙门菌的监测与疫情调查中[3-5]。研究表明[6],WGS与常规血清学分型方法的符合率为97.6%,具有广阔的应用前景。
因此,本研究对成都市35株人源沙门菌分离株进行全基因组测序,预测其血清型、耐药基因及可移动遗传元件(mobile genetic elements,MGE),注释并分析其毒力因子及分布,以探究成都市人源沙门菌耐药与毒力分子特征,为深入研究沙门菌致病机理、监测与预防沙门菌感染提供参考依据。
1 材料与方法
1.1 菌株来源 35株临床沙门菌,分离自2020年5-6月的腹泻患者粪便,菌株来自成都市青羊区(14株)、郫都区(7株)、成华区(4株)、金牛区(3株)、武侯区(2株)、天府新区(2株)、新都区(2株)、锦江区(1株)8个行政区,菌株分离与收集由成都市疾病预防控制中心完成。利用VITEK 2 COMPACT 全自动微生物分析鉴定仪(法国BioMerienx公司)对菌株进行沙门菌鉴定。
1.2 全基因组测序 收集到的沙门菌分离株送往中国科学院微生物研究所进行全基因组测序,流程如下:提取菌株DNA,使用Nanodrop检测DNA基因组浓度与质量,进行文库构建;使用IlluminaXten测序系统进行双端150 bp测序;利用Fastqc、Trimmomatic对下机原始数据进行质量检测、统计;使用SPAdes的Unicycler短序列组装软件进行组装,并根据reads中paired-end、overlap关系对组装结果进行局部优化;使用GapClose 1.1.2软件对组装结果进行修补,除去冗余序列获得最终组装结果。
1.3 血清学预测与MLST分型 基于ZOOM TECH 微生物数据分析平台(http://39.107.235.93:8080/#/login),使用SeqSero根据特定抗原的多态性完成血清型预测,使用PubMLST对分离株进行MLST分型。
1.4 基因预测、注释与分析
1.4.1 基因组组分分析 使用 GeneMarkS 分析工具(http://topaz.gatech.edu/GeneMark/genemarks.cgi)预测沙门菌分离株编码基因。
1.4.2 耐药基因预测、注释与MGE分析 基于CARD(Comprehensive Antibiotic Research Database)数据库对沙门菌分离株耐药基因进行预测及注释。利用Center for Genomic Epidemiology平台的Mobile Element Finder分析工具(https://cge.cbs.dtu.dk/services/MobileElementFinder)预测菌株的可移动遗传元件,包括质粒与插入片段。
1.4.3 毒力因子预测、注释与分析 基于VFDB(Virulence Factor Database)数据库对沙门菌分离株毒力因子进行预测及注释。使用R 4.1.0软件构建毒力因子数目分析热图,对每个基因组的毒力因子数目进行Scale参数标准化。
2 结 果
2.1 血清学预测与MLST分型 35株人源沙门菌分离株共预测出5种血清型(表1),其中I 4,[5],12:i:-血清型沙门菌最多(15株,42.9%),其次为鼠伤寒血清型(Typhimurium)沙门菌(12株,34.3%)。

表1 35株沙门菌分离株血清型预测结果Tab.1 Serotype prediction results for 35 Salmonella isolates
MLST分析共鉴定出5种ST型。如图1所示,血清型预测与ST型结果对应良好,I 4,[5],12:i:-血清型预测菌株除2株未在数据库中查得所对应的ST型,其余13株均为ST34型,鼠伤寒血清型(Typhimurium)预测菌株均为ST19型,肠炎血清型(Enteritidis)预测菌株为ST11型,德比血清型(Derby)预测菌株为ST40型,火鸡血清型(Meleagridis)预测菌株为ST463型。
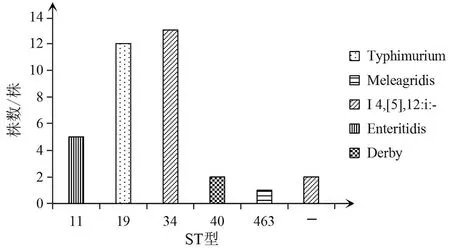
“-”表示未在数据库中查得所对应的ST型。图1 35株沙门菌分离株血清型预测与MLST分型结果对比Fig.1 Comparison between serotype prediction and MLST typing of 35 Salmonella isolates
2.2 基因组组分分析 35株沙门菌分离株基于测序数据的基因组组分结果见表2,基因组范围为4.7~5.2 Mb;GC含量为52%左右;编码基因数量4 600~5 200 个,平均长度约 900 bp,总长度约占基因组的 88%。I 4,[5],12:i:-血清型沙门菌的基因组大小和编码基因数目高于其他血清型菌株。
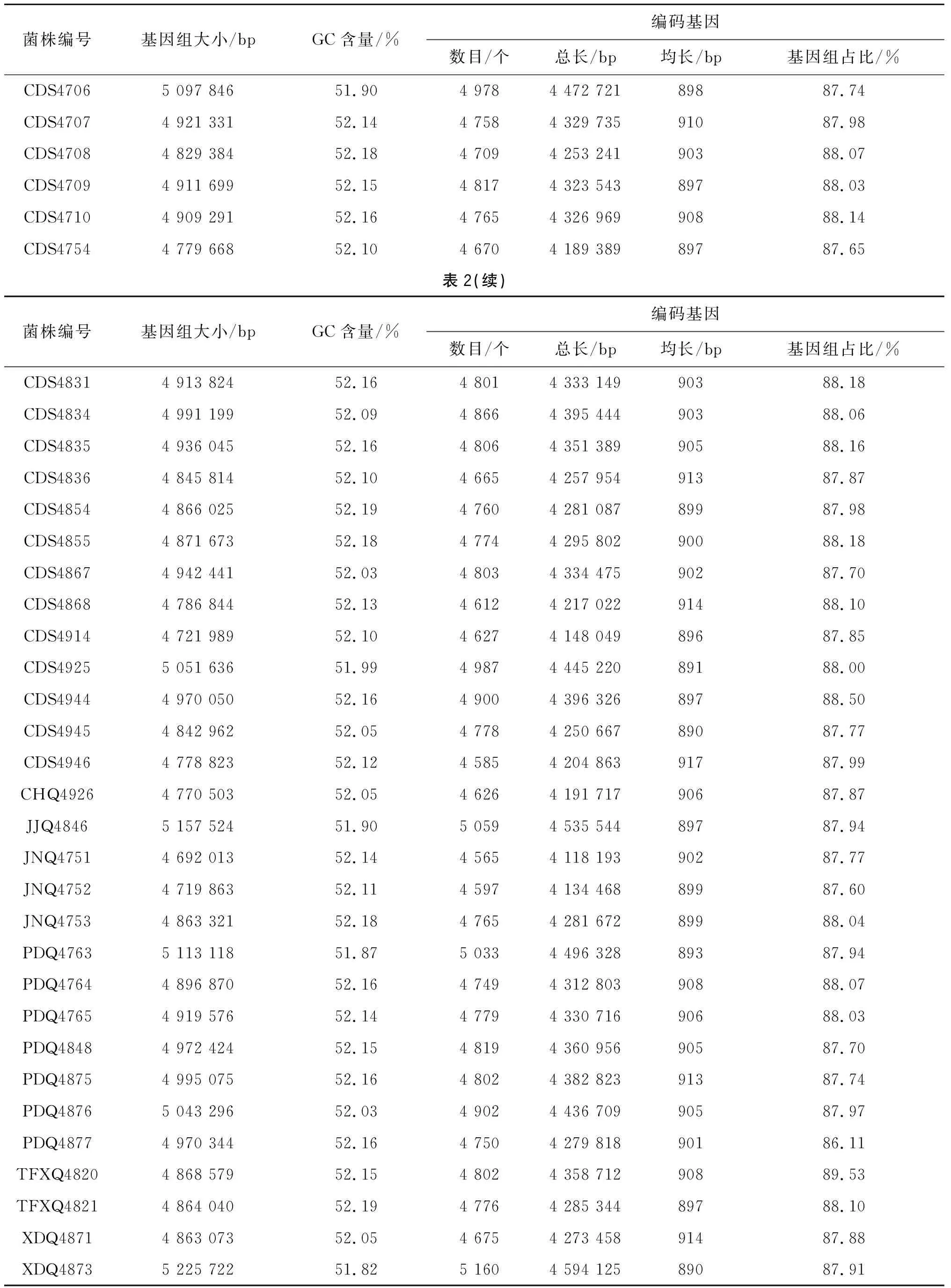
表2 35株沙门菌分离株基因组组分预测Tab.2 Genome component prediction for 35 Salmonella isolates
2.3 耐药基因与MGE分析 35株沙门菌分离株基于CARD数据库共预测到10类42种不同的耐药基因(图2),以氨基糖苷类耐药基因(42.68%,102/239)最多,其中aac(6′)-Iaa携带率为100%(35/35)。不同血清型沙门菌的耐药基因携带情况存在差异。I 4,[5],12:i:-沙门菌携带耐药基因数目更多,且硫胺类sul2、四环素类tet(B)携带率为100%(15/15)。
35株沙门菌分离株质粒携带率为97.14%(34/35),单个菌株最多同时携带5个质粒。共预测出17种不同的质粒,包括IncQ1(40.00%,14/35)、IncFII(S)与IncFIB(S)(37.14%,13/35)、Col440I(22.86%,8/35)等。其中IncQ1质粒只在I 4,[5],12:i:-沙门菌中携带,肠炎沙门菌和部分鼠伤寒(8株)均同时携带IncFII(S)和IncFIB(S)质粒。MGE分析结果如表3,部分质粒与耐药基因关系密切,且存在插入序列。I 4,[5],12:i:-沙门菌携带更多的质粒与插入序列,其携带的IncQ1质粒(93.33%,14/15)与多种耐药基因相关。
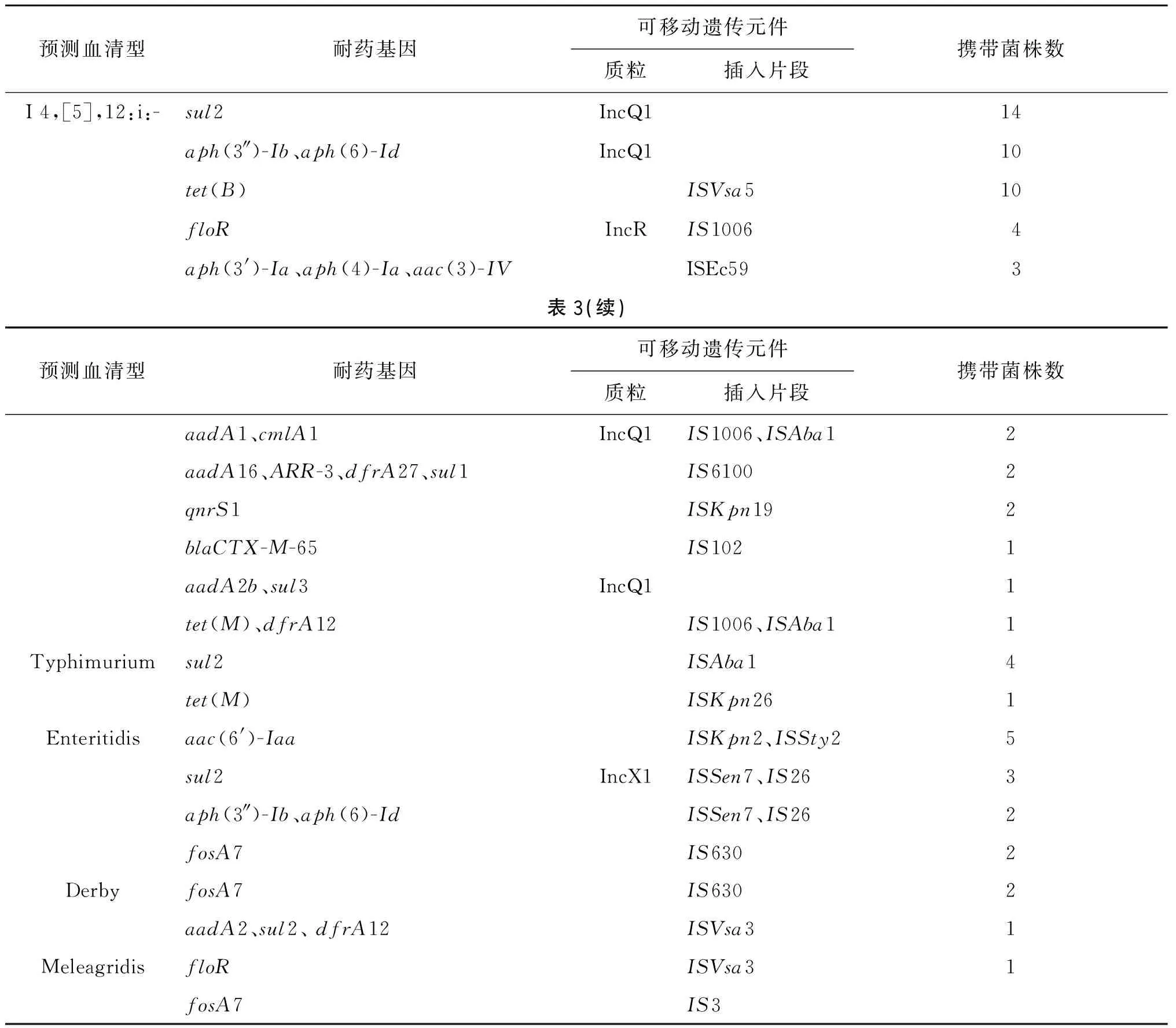
表3 35株沙门菌分离株耐药基因及相关可移动遗传元件的携带情况Tab.3 Drug resistance genes and mobile genetic elements carried by 35 Salmonella isolates
2.4 毒力因子分析 基于VFDB数据库对35株沙门菌分离株的毒力因子注释(Identity≥80),共检出120种3 998个毒力基因,菌株毒力因子数目为103~123个(表4)。分泌系统相关的毒力因子数目最多,为76~79个。不同血清型的菌株之间毒力因子数目不同,I 4,[5],12:i:-、鼠伤寒和肠炎沙门菌的毒力因子数目大于其他血清型沙门菌。Spv、Pef、Rck毒力因子均只在13株沙门菌中存在,即全部的肠炎沙门菌和8株鼠伤寒沙门菌,且仅该8株鼠伤寒沙门菌保留GrvA毒力因子。仅德比和火鸡沙门菌缺失SodCI毒力因子。Fim、Agf、Csg、Mgt、Inv、misL、avrA、Org、Ssa、ompA、mig-14、Ste、Sop等毒力因子在所有菌株中数目相等。

表4 35株沙门菌分离株基因组VFDB毒力因子注释个数Tab.4 Genome annotation numbers of virulence factors in VFDB for 35 Salmonella isolates
毒力因子分布热图(图3)所示,35株沙门菌基于毒力因子分布分成3簇,其中德比沙门菌单独成簇,肠炎沙门菌和部分鼠伤寒沙门菌(8株)共同成簇,剩余鼠伤寒沙门菌(4株)与火鸡、I 4,[5],12:i:-沙门菌共同成簇。鼠伤寒沙门菌均具有更高的鞭毛毒力因子,且呈现出两种毒力因子分布,其中,部分鼠伤寒沙门菌(8株)呈现出更高的毒力质粒、质粒编码菌毛、血清抗性3种毒力因子分布。肠炎沙门菌的毒力质粒、质粒编码菌毛、血清抗性毒力因子分布与部分鼠伤寒沙门菌(8株)类似,但鞭毛毒力因子分布更少。德比沙门菌纤维粘附素相关毒力因子Lpf(Long polar fimbriae)分布低于其他血清型。火鸡、I 4,[5],12:i:-沙门菌毒力因子分布较为相似。

图3 35株沙门菌分离株毒力因子分布热图Fig.3 Virulence factor annotation heatmap for 35 Salmonella isolates
3 讨 论
I 4,[5],12:i:-沙门菌是过去20年里世界范围内迅速出现的新亚型,被认为是鼠伤寒沙门菌第Ⅱ相鞭毛蛋白不表达的变种[7]。国内已有多地报道[8-10]I 4,[5],12:i:-为临床沙门菌分离株的优势血清型,且在多种动物中发现该血清型沙门菌的流行率逐年增高[11-12],提示I 4,[5],12:i:-已逐渐成为人与动物沙门菌感染的主要血清型之一。本研究I 4,[5],12:i:-沙门菌主要为ST34型,这与国内外学者的研究结果一致[3,13]。
本研究I 4,[5],12:i:-沙门菌耐药基因以氨基糖苷类aac(6′)-Iaa、硫胺类sul2、四环素类tet(B)为主,这与目前国内研究结果一致[13]。本研究几乎全部I 4,[5],12:i:-沙门菌携带lncQ1质粒,已有研究发现[14]lncQ1质粒是sul2、aph(6)-Id耐药基因的载体,可在不同来源的多种细菌中稳定维持和转移,这提示I 4,[5],12:i:-血清型可能存在该质粒基因介导的获得性耐药。此外,本研究MGE分析发现aadA1、cmlA1等耐药基因与lncQ1质粒相关,且存在插入片段,其具体关系目前尚未得到充分研究与阐述,有待于进一步探明。
Spv毒力质粒(SalmonellaPlasmid Virulence)是沙门菌重要的毒力因子,其基因包括spvR、spvA、spvB、spvC和spvD[15],其中,spvB、spvC和spvR是沙门菌的毒力表型所必需的[16]。spvB具有细胞毒性作用,与沙门菌感染期间的宿主细胞晚期凋亡有关[17];spvC具有苏氨酸裂解酶活性,可以抑制宿主细胞的MAP激酶信号[18-19];spvR对促进下游spvA、spvB、spvC和spvD基因的转录具有核心作用[16,20]。本研究中,spvB、spvC、spvR均只在肠炎血清型菌株和部分鼠伤寒血清型菌株中发现(37.1%,13/35)。这与目前研究结论一致[19,21],即只有肠炎和鼠伤寒沙门菌携带Spv毒力质粒。研究表明Spv操纵子通常位于IncFII质粒或IncFII-InFIB杂交质粒上[22],本研究同样发现仅预测出Spv毒力基因的沙门菌携带IncFII(S)和IncFIB(S)质粒。此外,本研究发现,pefA、pefB、pefC、pefD、rck毒力因子也只在上述肠炎和鼠伤寒沙门菌中保留。Pef为质粒编码菌毛(Plasmid-encoded fimbriae)相关基因,可调节沙门菌对肠上皮细胞的粘附[23];Rck为血清抗性相关基因,与沙门菌抵抗补体杀伤作用有关[24]。一项体外实验显示沙门菌毒力与Spv、Pef、Rck毒力基因的携带呈正相关[21]。Spv与Pef、Rck毒力因子可能存在协同致病机制,值得在转录组和蛋白组进一步研究。
SodCI毒力因子是Cu-Zn 超氧化物歧化酶,被认为有助于沙门菌在巨噬细胞中的生存[25],且是鼠伤寒沙门菌发挥毒力所必需的[26]。本研究中,SodCI毒力因子在鼠伤寒、肠炎、I 4,[5],12:i:-沙门菌中保留,德比和火鸡沙门菌缺失,这与Kuijpers等[21]的结果一致,SodCI毒力因子可能与这些沙门菌的较强致病力有关。
本研究系首次采用WGS技术对成都市人源沙门菌进行分型、耐药基因与毒力因子注释及分析,描述其基因组特征,丰富了该地沙门菌监测数据库,在基因水平为研究沙门菌耐药、毒力机制提供了资料,其致病机理还需在基因组、转录组与蛋白组不断探究及验证。
利益冲突:无
引用本文格式:肖颖,武雅婷,赵婉妤,等. 成都市人源沙门菌基因组特征分析[J].中国人兽共患病学报,2022,38(5):433-440,446. DOI:10.3969/j.issn.1002-2694.2022.00.059
