小麦DEAD-box基因家族鉴定及其低温下的表达模式分析
2022-03-01彭瞰看王多佳王政委任治鹏王晓楠
齐 越,于 晶,苍 晶,田 宇,彭瞰看,王多佳,王政委,任治鹏,梁 伊,王晓楠,孟 婧
(1.东北农业大学生命科学学院,黑龙江哈尔滨 150030; 2.东北农业大学农学院,黑龙江哈尔滨 150030)
RNA分子在RNA代谢过程中会经历一系列修饰[1],其自身的不稳定性容易引起RNA代谢紊乱[2],进而会在不同程度上影响植物的生长发育和对逆境胁迫的抵抗能力[3-4]。
DEAD-box RNA解旋酶属于解旋酶家族的SF2家族,作为最大的解旋酶家族,DEAD-box RNA解旋酶参与转录、翻译、mRNA前体剪接、核糖体生物发生、mRNA核-质输出以及RNA无义衰变等RNA代谢过程,调控植株的生长发育与抗逆性[5-25]。部分DEAD-box RNA解旋酶缺失可能抑制植物生长发育,甚至导致植株死亡。不同非生物胁迫会引起不同DEAD-box解旋酶基因的表达量发生变化,从而提高植株的胁迫耐受能力[5]。拟南芥中,DEAD-box家族基因AtRH3、AtRH7、AtRH9、AtRH25、AtHELP、LOS4和RCF1均响应低温胁迫[6-8],其中,AtRH3和AtRH7完全缺失会导致植株死亡[7-8]。水稻中,DEAD-box家族基因TCD33可提高水稻耐寒能力,缺失突变体tcd33表现出子叶白化表型,说明TCD33是叶绿素合成的必需基因[9];另外,OsRH42也可提高水稻的抗寒能力[10]。甘蓝中,DEAD-box家族基因BrRH22除响应低温胁迫外,还响应干旱胁迫和盐胁迫[11]。除此之外,前人还发现,多个物种的DEAD-box基因家族成员均响应逆境胁迫,如龙眼DlDDX、小立碗藓eIF4A1、番茄SlDEAD39、拟南芥AtRH17和大豆P68均响应盐胁迫[12-16];水稻OsRH58、小立碗藓eIF4A1和拟南芥AtRH8均响应干旱胁迫[17-19]。但目前关于小麦DEAD-box基因家族成员参与植物非生物胁迫抵抗的研究尚未见报道。
为了解小麦DEAD-box基因的功能,本研究对小麦DEAD-box基因家族成员进行全基因组鉴定,并对本课题组前期在转录组与蛋白磷酸化数据库中筛选获得的7个差异表达的小麦DEAD-box基因(未发表)进行表达模式分析,以期为小麦DEAD-box基因的功能研究奠定 基础。
1 材料与方法
1.1 小麦DEAD-box基因家族成员的鉴定及其特征分析
从Ensembl Plants数据库(http://plants.ensembl.org/index.html)下载小麦全基因组序列文件及其基因注释文件,从UniProtKB/Swiss-Prot数据库(https://www.uniprot.org/uniprot/? query=reviewed:yes)下载已确认的60个水稻DEAD-box基因,并以此为参照(基因提取比例为1∶5,并去除重复基因)从小麦全基因组数据库中检索小麦DEAD-boxRNA解旋酶家族基因。在Pfam数据库(http://pfam.xfam.org/)中进行结构域预测,人工筛选掉预测错误的DEAD-box基因模型,确认小麦DEAD-box家族基因成员。
用ExPASy网站的Compute pI/MW tool软件(https://web.expasy.org/ compute_pi/)分析小麦DEAD-box基因家族蛋白的相对分子质量和等电点。用Softberry网站的ProtComp 9.0软件(http://linux1.softberry.com/berry.phtml?topic=protcomppl& group=programs&subgroup=proloc)对小麦DEAD-box基因家族蛋白进行亚细胞定位预测。
1.2 TaDEAD-box蛋白系统发育树的构建及其保守结构域和基因结构分析
用MEGAX(https://www.megasoftware.net/)软件中的Clustal W对小麦、拟南芥和水稻DEAD-box基因家族蛋白序列进行序列比对,用最大似然法(maximum Likelihood,ML)和最优进化模型LG+G构建系统发育树,bootstrap值设置为1 000。用MEME软件(https://meme-suite.org/meme/tools/meme)分析小麦DEAD-box基因家族蛋白保守基序,位点分布方式为Zero or One Occurrence per Sequence(zoops),检索基序数量为9。用Pfam数据库对小麦DEAD-box基因家族进行蛋白保守结构域分析,E-value≤1.0。用GSDS 2.0软件(http://gsds.gao-lab.org/)对小麦DEAD-box基因家族进行基因内含子和外显子结构分析。
1.3 DEAD-box基因家族的共线性分析
从Phytozome数据库(https://data.jgi.doe.gov/refine-download/phytozome)下载水稻和拟南芥的全基因组序列文件及其基因注释文件。用Circos软件从小麦基因组数据库中提取物理位置信息,将所有小麦DEAD-box基因定位到染色体上。使用小麦与水稻、拟南芥全基因组文件相互对照提取,每个基因提取数≤30。用多重共线性扫描工具MCScanX对基因重复事件进行分析,参数为默认值。使用KaKs_Calculator 2.0计算每个重复DEAD-box基因的非同义替换(ka)和同义替换(ks),构建小麦种内共线性图谱和小麦与水稻、拟南芥的种间共线性图谱。
1.4 小麦DEAD-box基因家族在低温下的表达模式分析
对本课题组前期转录组数据库分析发现,Dn1分蘖节含有大量与低温胁迫相关的DEAD-box RNA解旋酶基因,对其进行筛选整合,获得7个低温胁迫下表达量变化较为明显的DEAD-box基因(TaRH7、TaRH30、TaRH20、TaRH10-1、TaRH10-2、TaRH35和TaRH5)(未发表),本研究对这7个DEAD-box基因进行表达模式分析。供试材料为强抗寒冬小麦品种Dn1和Dn2。
Dn1和Dn2种子于2020年9月12日播种于东北农业大学试验田(行长4 m,行距0.2 m,播种深度5 cm),田间常规管理。在大田自然降温连续10 d平均最低温度分别为5、0、-10和 -25 ℃时,剪取分蘖节和叶片,置于-80 ℃冰箱保存。
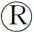

表1 组织特异表达检测所需的引物Table 1 Primers for tissue specific expression detection
2 结果与分析
2.1 小麦DEAD-box基因家族鉴定结果、蛋白质理化性质及亚细胞定位
根据60个水稻DEAD-box基因的蛋白序列,在Ensembl Plants小麦全基因组数据库中共鉴定到134个小麦DEAD-box基因(表2)。理化性质分析发现,小麦DEAD-box家族基因编码的蛋白序列长度为408~1 276 aa,等电点为 4.99~10.17。109个小麦DEAD-box蛋白等电点大于7,为弱碱性蛋白;25个小麦DEAD-box蛋白等电点小于7,为弱酸性蛋白。亚细胞定位分析发现,78个小麦DEAD-box蛋白定位于细胞核中,19个定位于细胞外基质中,9个定位于原生质膜中,21个定位于细胞质中,6个定位于线粒体中,1个定位于叶绿体中。

表2 小麦中鉴定出的DEAD-box家族基因Table 2 DEAD-box family genes identified in wheat

(续表2 Continued table 2)
2.2 小麦、水稻和拟南芥DEAD-box家族蛋白系统发育树分析
利用MEGAX软件对小麦、水稻和拟南芥DEAD-box基因家族进行系统进化分析。结果(图1)发现,通过拓扑结构域分区可将DEAD-box基因分为12组。其中,L组中DEAD-box基因最多,共有60个DEAD-box基因,包含30个小麦DEAD-box基因、16个水稻DEAD-box基因和14个拟南芥DEAD-box基因。F组、G组和H组中DEAD-box基因较少,均有5个DEAD-box基因,包含3个小麦DEAD-box基因、1个水稻DEAD-box基因和1个拟南芥DEAD-box基因。各组中小麦DEAD-box基因数量均最多,可能与其基因组较大有关,各组中水稻和拟南芥的DEAD-box基因数量相近。

红色、蓝色和绿色字体分别表示冬小麦、拟南芥和水稻DEAD-box家族基因。Red,blue and green fonts represent the DEAD-box family genes of winter wheat,Arabidopsis and rice,respectively.图1 小麦、水稻和拟南芥DEAD-box基因的系统进化树Fig.1 Phylogenetic tree of DEAD-box genes in wheat,rice and Arabidopsis
小麦、水稻和拟南芥三个物种中DEAD-box基因保守性较高。进化树分析发现,同物种的部分同源DEAD-box基因均位于同一组,如TaRH7、TaRH13、TaRH14等。同一组中小麦与水稻DEAD-box基因家族成员聚类关系较近,而与拟南芥较远,原因可能是水稻和小麦均为禾本科单子叶植物。
2.3 小麦DEAD-box基因结构及蛋白保守结构域分析
为深入了解小麦DEAD-box基因家族的进化过程,构建小麦DEAD-box蛋白进化树,根据拓扑结构分区可将鉴定到的134个DEAD-box蛋白划分为11组(图2a)。核心基序分析发现,大部分小麦DEAD-box家族基因均具有排列相似的9个核心基序,部分DEAD-box家族基因只有7或8个基序,如TaEif4A3缺乏motif I和motif IV,TaRH38缺乏motif Ib和motif VI,TaRH39缺乏motif IV,TaRH50缺乏motif VI。不同组中部分DEAD-box蛋白DEAD结构域(一般由motif Q、motif I、motif Ia、motif Ib、motif II和motif III组成)的核心基序排列顺序存在差异;而Helicase C结构域(一般由motif IV、motif V和motif VI组成)中的核心基序排列顺序均相同(图2b和图3)。基因结构分析发现,小麦DEAD-box基因家族成员的内含子分布不均匀,数量为1~15个。保守结构域分析发现,小麦DEAD-box基因家族蛋白均含有DEAD与Helicase C保守结构域,其中DEAD结构域含有的内含子数为0~6个,Helicase C结构域含有的内含子数为0~3个。同一组大部分小麦DEAD-box基因均具有相似的结构,可能存在串联重复基因。

A:小麦DEAD-box进化树。通过拓扑结构分析将小麦DEAD-box蛋白分成11组,不同颜色代表不同组。B:小麦DEAD-box核心基序分布分析。C:小麦DEAD-box结构域与基因结构图。A:Phylogenetic tree of wheat DEAD-box proteins.The TaDEAD-box proteins were divided into 11 regions by topology analysis,and different colors represent different groups.B:Distribution analysis of core motif of wheat DEAD-box.C:Dead-box domain and gene structure of wheat.图2 小麦DEAD-box进化树、核心基序、结构域和基因结构分析Fig.2 Phylogenetic tree,core motifs,domain and gene structure analysis of the DEAD-box gene family

图3 小麦TaDEAD-box蛋白家族核心基序示意图Fig.3 Schematic diagram of the core motifs of the wheat DEAD-box gene family
2.4 小麦DEAD-box基因染色体分布与共线性分析
从基因在染色体上的分布来看,小麦各染色体上均有DEAD-box基因分布,但分布不均匀。
其中5A染色体上分布数量最多,含有13个DEAD-box基因;7D染色体上分布数量最少,仅有1个DEAD-box基因(图4)。
从共线性分析结果来看,分布于同一基因组不同染色体上的小麦DEAD-box基因普遍存在共线性,说明其大多数基因为同源染色体上的等位基因,在进化上具有高度保守性。而值得注意的是,5A染色体上的TaRH29-1基因与4D染色体上的TaRH29-3基因之间存在共线性关系;5A染色体上的TaRH50-1、TaRH50-2和TaRH50-3基因与4B染色体上的TaRH29-2基因之间存在共线性关系;6A染色体上的TaRH8-3基因与2A、2B和2D染色体上的TaRH13-1、TaRH13-2和TaRH13-3基因之间存在共线性关系;2D染色体上的TaRH13-2基因与6A、6B和6D上的TaRH8-1、TaRH8-2和TaRH8-3基因之间存在共线性关系(图4)。这些结果表明,小麦中DEAD-box基因可能是由基因复制产生的,节段性复制事件对小麦DEAD-box蛋白的进化起着重要的推动作用。

图4 小麦DEAD-box基因在染色体上的分布Fig.4 Distribution of wheat DEAD-box gene on chromosomes
种间共线性分析发现,小麦与水稻的DEAD-box基因共线性连线数量远多于小麦与拟南芥的共线性连线数量(图5)。小麦和拟南芥基因组中,仅有6个小麦DEAD-box基因存在共线性关系,分别位于2A、2B、2D、4A、4B、4D和6B染色体上,与拟南芥2号、3号和5号染色体上的3个基因存在共线性关系。小麦和水稻基因组中,除水稻的11号染色体外,其他染色体上共有38个水稻DEAD-box基因与小麦基因组中的83个小麦DEAD-box基因存在共线性关系。其中,水稻1号染色体的7个DEAD-box基因与小麦3A、3B和3D染色体上15个DEAD-box基因发生有序配对。这说明水稻1号染色体和小麦3号染色体在DEAD-box基因进化的线性关系上存在高度的保守性和一致性。值得注意的是,有3个水稻DEAD-box基因与小麦不同基因组上的DEAD-box基因发生了共线性配对,如水稻2号染色体上的OsEif4A3与小麦的6B、7A和7B染色体上的TaEif4A3C、TaEif4A3D、TaEif4A3B发生共线性配对;水稻2号染色体上的OsRH8和4号染色体上的OsRH6均与小麦1号染色体上的TaRH12-2(1A)和TaRH12-3(1B)以及6号染色体上的TaRH8-1(6A)、TaRH8-2(6B)和TaRH8-3(6D)发生共线性配对,推测这些基因所在的染色体可能在进化上发生过异位突变。

图5 小麦、水稻和拟南芥DEAD-box基因的共线性关系Fig.5 Collinearity of DEAD-box genes among wheat,rice and Arabidopsis
2.5 低温胁迫下差异表达的小麦DEAD-box基因的表达模式
Dn1分蘖节中基因的表达量分析结果(图6)显示,随着温度的降低,TaRH7的表达量呈先降后升的变化趋势,在-25 ℃时达到最大值,显著高于5 ℃和0 ℃时的表达量,但与 -10 ℃时的表达量无显著差异。TaRH30的表达量呈升-降-升的变化趋势,在-25 ℃时达到最大值,显著高于5 ℃和-10 ℃的表达量,但与0 ℃时的表达量无显著差异。TaRH20、TaRH10-1、TaRH10-2、TaRH35和TaRH5的表达量均呈先升后降的变化趋势,TaRH20与TaRH5的表达量在0 ℃时达到最大值,均显著高于-10 ℃和-25 ℃时的表达量,但与5 ℃时的表达量无显著差异;TaRH10-1、TaRH10-2和TaRH35的表达量均在-10 ℃时达到最大值,其中TaRH10-1和TaRH35显著高于5 ℃、0 ℃和-25 ℃的表达量,TaRH10-2显著高于5 ℃时的表达量。7个小麦DEAD-box基因中,TaRH10-1和TaRH35对低温胁迫的响应较为明显,在-10 ℃时表达量最高,约为5 ℃时表达量的5倍。
Dn1叶片中基因的表达量分析结果(图6)显示,随着温度的降低,TaRH7、TaRH10-1和TaRH35的表达量呈缓慢上升趋势,-25 ℃时达到最大值,显著高于5 ℃、0 ℃和-25 ℃的表达量;TaRH30、TaRH20、TaRH10-2和TaRH5的表达量呈先升后降的变化趋势,在-10 ℃达到最大值,且TaRH20、TaRH10-2和TaRH5的表达量显著高于5 ℃、0 ℃和-25 ℃的表达量,而TaRH30在各温度间的表达量无显著差异。7个小麦DEAD-box基因中,TaRH10-2对低温胁迫的响应较为明显,在-10 ℃时的表达量最高,约为5 ℃时表达量的85倍。
Dn2分蘖节中基因的表达量分析结果(图6)显示,随着温度的降低,TaRH7、TaRH30、TaRH20、TaRH10-2和TaRH5的表达量呈升-降-升的变化趋势,其中,TaRH7、TaRH30和TaRH10-2的表达量在-25 ℃达到最大值,显著高于5 ℃、0 ℃和-25 ℃的表达量;而TaRH20和TaRH5的表达量在0 ℃时达到最大值,均显著高于5 ℃和-10 ℃的表达量。TaRH10-1的表达量呈先升后降的变化趋势,在-10 ℃达到最大值,显著高于5 ℃、0 ℃和-25 ℃的表达量。TaRH35的表达量呈降-升-降的变化趋势,在-10 ℃达到最大值,显著高于0 ℃和-25 ℃的表达量,但与5 ℃时的表达量无显著差异。7个小麦DEAD-box基因中,TaRH7、TaRH30、TaRH10-1和TaRH10-2对低温胁迫的响应较为明显,TaRH7和TaRH30在-25 ℃时的表达量约为5 ℃表达量的10倍,TaRH10-1和TaRH10-2在-25 ℃时的表达量约为5 ℃时表达量的5倍。
Dn2叶片中基因的表达量分析结果(图6)显示,随着温度的降低,TaRH7、TaRH30、TaRH20和TaRH10-2的表达量呈升-降-升的变化趋势,在-25 ℃时达到最大值,均显著高于5 ℃、0 ℃和-25 ℃的表达量。TaRH5的表达量呈持续升高趋势,均在-25 ℃时达到最大值,显著高5 ℃、0 ℃和-10 ℃的表达量。TaRH10-1和TaRH35的表达量在各温度间无显著差异。7个小麦DEAD-box基因中,TaRH10-2对低温胁迫的响应较为明显,在-25 ℃时的表达量最高,约为5 ℃时表达量的125倍。

*、**、***和****分别表示基因间的表达量在 0.05、0.01、 0.001和0.0001水平上差异显著。*,**,*** and **** indicate significant differences between genes at 0.05,0.01,0.001 and 0.0001 levels.图6 低温胁迫下Dn1和Dn2的叶片和分蘖节中DEAD-box基因的表达模式分析Fig.6 Expression patterns of DEAD-box in leaves and tillering nodes of Dn1 and Dn2 under cold stress
3 讨 论
3.1 DEAD-box解旋酶进化关系分析
从种间进化树分析看,小麦与水稻DEAD-box解旋酶的亲缘性高于拟南芥,且种间共线性分析发现,小麦与水稻DEAD-box基因存在共线性的数量远多于小麦与拟南芥DEAD-box基因存在共线性的数量。原因可能是单子叶与双子叶植物在DEAD-box解旋酶进化过程中存在进化差异。种间进化树同一组内均存在三物种的DEAD-box解旋酶,说明DEAD-box解旋酶在单子叶与双子叶分化之前就已经存在[20]。从种内进化树分析看,TaRH9-1、TaRH9-2和TaRH2基因在进化分支上相近,TaRH47B和TaRH3-4基因在进化分支上相近,推测它们具有相似的分子 功能。
3.2 小麦DEAD-box解旋酶结构分析与功能 预测
DEAD-box RNA解旋酶几乎参与所有RNA代谢活动[21],根据其参与RNA代谢的种类和行使功能的空间定位可以大致判断其分子功能。另外,DEAD-box RNA解旋酶在进化中保持高度的保守性[1]。DEAD-box RNA解旋酶的功能依赖于其高度保守的核心基序组成与排列,在不同物种间DEAD-box RNA解旋酶的功能具有较高的相似性,在同源物种间该相似性更为明显。
从结构域分析发现,本研究中所有小麦DEAD-box蛋白均有其特征性结构域DEAD和Helicase C,部分DEAD-box解旋酶含有特殊结构域,如TaEIF4A2C和TaRH35含有C2H2锌指结构域,TaRH41含有HIT锌指结构域。而锌指结构域能够提高解旋酶RNA结合活性[22],并提高植株非生物胁迫抗性[23]。推测这3个DEAD-box蛋白也参与逆境胁迫。本研究还发现,TaRH3与TaRH7均含有GUCT结构域。而小立碗藓中含有GUCT结构域的DEAD-box解旋酶可调控孢子体和配子体发育[24],此外,拟南芥中TaRH7基因缺失会导致拟南芥配子体发育畸形,抑制种子萌发[8]。由此推测,小麦中TaRH3与TaRH7基因也可能具有相似功能。
从核心基序分析发现,本研究中大多数小麦DEAD-box解旋酶主要由motif Q、motif I、motif Ia、motif Ib、motif II、motif III、motif IV、motif V、motif VI九个核心基序组成,部分DEAD-box解旋酶存在部分核心基序缺失的现象。如TaEIF4A3缺乏motif I和motif IV,TaRH38缺乏motif Ib和motif VI,TaRH39缺乏motif IV,TaRH50缺乏motif VI。DEAD结构域中的motif Q和motif I、motif II可能参与ATP结合和水解;motif III可能与ATP水解和解旋酶活性的偶联有关;motif V可能通过糖-磷酸基骨架与底物结合,并与NTP相互作用;motif VI可能与ATP磷酸盐的结合有关,而这种基序功能的丧失与某些DEAD-box蛋白的ATP水解功能受损有关;motif Ia、motif Ib、motif IV可能参与核酸结合诱导的ATP水解[25-26]。由此猜测,部分DEAD-box解旋酶缺失的基序可能引起相应的分子功能缺失,或由其他基序弥补其分子功能。
3.3 小麦DEAD-box RNA解旋酶参与冬小麦抵抗低温胁迫
对冬小麦分蘖节和叶片的7个DEAD-box基因进行qRT-PCR定量分析,发现不同品种和不同组织中DEAD-box基因的表达模式随温度的变化差异较大,其中叶片中DEAD-box基因对低温的响应比分蘖节更为强烈,如Dn1叶片中TaRH10-2基因在-10 ℃时的表达量约为5 ℃时表达量的85倍,Dn2叶片中TaRH10-2基因在-25 ℃时的表达量约为5 ℃时表达量的125倍,远高于分蘖节中TaRH10-2基因的表达量变化幅度,这可能是由于叶片组织在地上部,会受到低温和风干缺水的双重影响[11,17-19],而分蘖节在地下,受土壤层的保护,在一定程度上地下温度会略高于地上温度,受风干失水的影响较小,因此,叶片对低温胁迫的敏感性比分蘖节更强。不同品种中DEAD-box基因家族成员对冷胁迫的响应速度不同。如Dn1中DEAD-box基因家族表达量峰值普遍在-10 ℃,而Dn2的DEAD-box基因家族表达量峰值普遍在-25 ℃,响应冷胁迫基因激活时间的差异可能会对不同冬小麦品种抗寒能力与植株生理长势产生影响。
总之,鉴于小麦DEAD-box基因的多样性和重要功能,对小麦DEAD-box基因家族进行系统的功能研究十分必要。本研究为小麦DEAD-box基因功能的解析奠定了基础,对进一步了解多倍体植物DEAD-box基因的特点,挖掘优异抗低温基因资源,培育作物抗低温新品种,具有重要的理论和实践意义。
