单核苷酸多态性微阵列芯片在分析流产或死胎染色体中的应用*
2022-01-28钱芳波司雯淼袁文博
沈 晔,钱芳波,司雯淼,袁文博,张 怡,徐 晔
(1.南京医科大学附属无锡妇幼保健院计划生育科,无锡 214000;2.浙江博圣生物技术股份有限公司,杭州 310012;3.新西兰奥克兰大学,奥克兰 1142)
研究报道,女性发生1次自然流产的风险约为10%,复发性流产的发生率为1%~5%[1]。流产与染色体异常、免疫、易栓症(血栓前状态)、内分泌、感染、母体及环境等因素相关,其中约50%流产与染色体异常有关[2-3]。染色体异常主要包括数目异常(非整倍体、多倍体异常)、结构畸变、嵌合体以及杂合性缺失等。拷贝数变异(copy number variations,CNVs)是指染色体上大于1kb的DNA片段的增加或减少[4],致病性CNVs可导致基因组病,从而引起流产、死胎、发育滞后、多发畸形等[5]。染色体微阵列分析技术(chromosomal microarray analysis,CMA)在全基因组水平检测染色体数目异常、CNVs、大多数的杂合性缺失(loss of heterozygosity,LOH)和一定水平的嵌合体,检测范围较广。本研究对673例流产样本进行CMA检测,探讨CMA技术在流产或死胎遗传学诊断的应用价值及流产或死胎频率和母体年龄与染色体异常的关系。
1 资料与方法
1.1 研究对象 2016年10月至2020年5月无锡市妇幼保健院就诊的673例患者的流产样本(568例流产绒毛、55例流产组织、49例引产羊水和1例引产脐血),患者年龄22~45岁,孕周6~28周。本研究经无锡市妇幼保健院伦理委员会审批通过,患者全部知情同意并签署知情同意书。
1.2 方法 采用组织提取试剂盒(德国QIAGEN公司)提取流产标本基因组DNA,用CytoScan 750k(美国Affymetrix公司)单核苷酸多态性微阵列芯片(single nucleotide polymorphism array,SNP array)进行检测,通过对基因组DNA酶切消化、PCR扩增、磁珠纯化、片段化、添加生物素标记、芯片杂交、洗脱、染色和扫描,利用Chromosome Analysis Suite软件及相关生物信息学方法比对GRCH37(hg19)分析原始数据,报告阈值为200kb以上的重复和100kb以上的缺失,不报告已知属于正常多态的拷贝数变化。
2 结 果
2.1 染色体异常类型分布 673例样本中有9例样本存在严重的母血污染或DNA降解导致质控不达标,其余664例样本检测成功,检测成功率为98.7%(664/673)。染色体正常276例(41.6%,276/664),其中男性占53.6%(148/276),女性占46.4%(128/276),男女比例为1.16。染色体异常388例(41.6%,388/664),包括非整倍体变异、整倍体变异、结构异常、杂合性缺失(LOH)。非整倍体变异(含嵌合体)占比最大,共271例(40.8%,271/664);整倍体变异48例(7.2%,48/664);结构变异75例(11.3%,75/664);LOH共11例(1.7%,11/664)。详细的CMA结果汇总如图1。

图1 染色体微阵列分析的结果汇总
2.2 染色体数目异常 本研究共发现271例(40.8%,271/664)非整倍体,包括260例单一染色体非整倍体和11例合并多个染色体的非整倍体。除染色体1、Y外,所有染色体都鉴定出了非整倍体。单一染色体非整倍体多为染色体三体(77.7%,202/260),其余为染色体单体(22.3%,58/260),其中1例21号染色体单体,1例嵌合型14号染色体单体,其余全部为X单体,即特纳综合症(45,X)患者。在11例多重非整倍体的病例中,包括5例(2.2%)发生了两个染色体三体,5例(1.8%)发生了三个染色体三体和1例49,XXXXY。染色体非整倍体异常分布见图2。整倍体变异共检出48例(7.2%,48/664),全部为三倍体。
2.3 染色体结构异常 本研究共检出染色体结构异常75例(11.3%,75/664),片段大小在106.4kb至105.2Mb之间,其中25例(3.8%,25/664)存在≥10Mb的缺失或重复,50例(7.5%,50/664)存在<10Mb的微缺失或微重复。在染色体结构异常的病例中,有31例CNV偏致病性的可能性大,占结构异常的41.3%(31/75)。根据CNVs的大小和位置,建议对夫妇进行进一步相应的遗传学检查。其中有24例病例做了相应的父母验证(表1),其中病例199的父亲被确认为相互平衡易位携带者,FISH验证见图3,该患者为1号染色体长臂末端片段与19号染色体短臂末端片段易位的携带者,另外6例CNV被确定遗传自父母一人,其余病例的CNV来源均被认为是新发。

图2 首次流产和复发性(≥2)流产患者非整倍体中各染色体占比

图3 病例199父源的FISH验证结果

表1 染色体结构异常做父母验证的结果
2.4 纯合区域 664例样本中有11例(1.7%,11/664)出现了LOH(表2),其中3例为全基因组UPD(uniparental disomy),3例为单条染色体UPD,5例为区域性UPD。
2.5 流产次数和年龄与染色体异常的关系 将单核苷酸多态性微阵列芯片结果分为首次流产(spontaneous abortion,SA)和复发性流产(recurrent miscarriage,RM)。本研究的复发性流产定义为两次或两次以上的流产或胎停。SA组和RM组微阵列芯片的检出率比较见表3。根据母亲的年龄对结果进行分析,染色体异常在高龄产妇(≥35岁)与低龄产妇(<35岁)的分布情况见表4。

表2 11例涉及杂合性缺失的结果
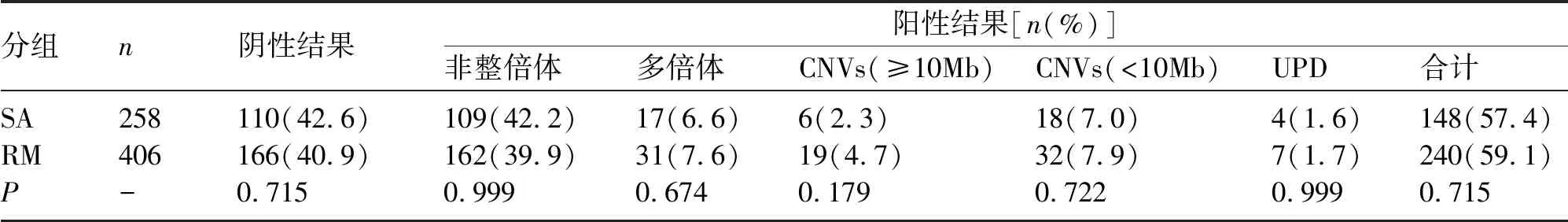
表3 流产次数与流产物染色体异常的关系

表4 母亲年龄与流产物染色体异常的关系
3 讨 论
本研究检测成功的664例标本中,染色体异常总检出率为58.4%(388/664),与之前研究报道的57.9%基本相符[6]。本研究发现,染色体异常占比最大的是非整倍体(40.8%),证实了染色体非整倍体变异是引起临床流产或死胎的最主要遗传因素[7]。其次是染色体结构异常(11.3%,75/664),多倍体异常(7.2%,48/664)占染色体异常的第三大比例,染色体异常比例最小的为杂合性缺失(1.7%,11/664)。
本次研究的非整倍体和多倍体的频率(40.8%和7.2%)与其他大规模研究的频率(40%和7.5%)[6]相似。非整倍体异常中,16号染色体三体和特纳综合症(45,X)数量最多,均为56例,分别占染色体数目异常的20.7%(56/271),是最常见的非整倍体数目异常。其余异常主要集中在13、14、21、22三体,这与肖艳华等[8]报道基本一致。除染色体1、Y外,所有染色体都鉴定出了非整倍体。其中1号染色体占人类基因组全长的8%,发生数目异常将导致严重的胚胎畸形,基本在妊娠4周内流产,故很难在流产物遗传学检测中发现[9]。染色体非整倍体胎儿形成主要原因是精子或卵子在减数分裂时,同源染色体或姐妹染色单体不分离,当这种异常的配子形成受精卵就会导致非整倍体异常的发生。对于流产物遗传学检测报告结果为13、14、15、21、22号染色体三体或单体时,一般建议其父母进行染色体核型分析,以排除亲本存在染色体罗氏易位的可能性[10]。据报道罗氏易位的人群携带率为1.23‰[11],本次研究暂未发现罗氏易位的携带者。
CNVs是产前超声异常、多发畸形、智力落后、发育迟缓的重要原因[12],现也被认为可能导致流产、死胎。CNVs常表现为染色体的缺失和重复,通过基因剂量改变、染色体断裂等方式影响基因的表达,从而产生致病性变异,严重可导致流产或胎停。对于CNVs致病性的判读,常需考虑CNVs所包含基因的数目、功能及非编码区重要的调控元件等来判断致病性等级,同时还需参考数据库、正常人群中出现类似CNVs的频率、变异来源等进行综合分析。根据2019年ClinGen/ACMG拷贝数变异解读标准,将CNVs按五级分类系统进行分类,分别为致病变异、疑似致病变异、临床意义未明变异、疑似良性变异、良性变异。
本研究共检出75例CNVs,片段大小106.4kb~105.2Mb,通过致病性判读发现31例(4.7%,31/664)CNVs涉及大量功能基因,致病性可能性较高,19例(2.9%,19/664)疑似致病变异,23例(3.5%,23/664)临床意义不明变异,2例(0.3%,2/664)疑似良性变异。不平衡易位被定义为一个染色体的末端存在缺失,同时另一个染色体的末端存在重复,本研究中有4例发生了不平衡易位。本研究中,可能来源于亲本染色体平衡重排的染色体不平衡重排的频率(0.6%,4/664)与之前的研究(0.6%,12/1861)[13]相似,其中1例经FISH验证明确来源于父源的平衡易位。因此CNVs发生在染色体末端时,父母之一可能是该变异片段平衡易位的携带者,建议对夫妇进行核型或FISH等相应的遗传学检查;而对于非染色体末端的微缺失微重复,大多数为新发变异,少数遗传自父母。
在这些CNVs中值得注意的是,1p36微缺失出现了5次(0.75%,5/664),大小1.75Mb~9.21Mb,4例确定为新发变异,1例未知。1p36缺失综合征缺失片段介于1p36.13~1p36.33,其发生率为1/5000~1/10000,此类患者一般具有不同程度的智力障碍、结构性心脏缺陷、颜面部异常(如眼睛凹陷)及其他并发症[14-15]。已有多篇文章对于产前和产后的1p36缺失进行了报道[14,16],但对于流产物遗传学检测发现1p36缺失的报道不多。本研究流产物遗传学研究中1p36缺失出现了5次,因此,1p36缺失可能与流产或胎停有关。导致胚胎早期死亡的可能潜在机制是由1p36缺失导致的畸形胎儿心脏异常、脑发育异常有关,具体致病机理有待进一步研究。
此外,本次研究还检测出11例(1.7%,11/664)LOH,与Wang等[6]研究的1.9%基本一致,高于用其他平台检测LOH检出率为0.7%[17],表明SNP-array可更好地检出LOH。研究表明,接近3%染色体核型无异常的妊娠失败与全基因组UPD相关[18]。由于技术的局限性,CMA只能检测出等单亲二倍体。目前已知母源的7、14和15号染色体及父源的6、11、14和15号染色体的UPD可导致临床的异常表型,也有少数涉及母源的2、16和20号染色体及父源的20号染色体UPD表征异常的病例报道[19],CMA可以通过父母的SNP微阵列结果对比确定亲本起源。本研究中单一染色体UPD与区域性UPD是否携带有与遗传印记(imprinting)相关的基因尚不完全清楚。该区域相关隐性致病基因变异的检测有助于确认是否有导致胎儿严重结构畸形甚至致死性基因。
对于LOH的解读原则大致有四种:(1)血源同一,这是由于父母是远亲关系,在基因组中常表现为小的LOH分散在少数几条染色体上面;(2)近亲关系,这是由于父母亲缘关系较近,在基因组中表现为许多染色体上面有较大的LOH片段,这样会增加隐性遗传病的发生风险[20];(3)LOH发生在整条染色体或某一染色体部分区域,主要产生机制有受精后分裂错误、配子互补、三体拯救及单体拯救等。依据不同的产生机制,可以形成完全性或嵌合性的整条或区段单亲二倍体;(4)全基因组UPD,一般是由孤雌生殖或孤雄生殖引起的,全基因组UPD是无法正常发育,在早孕期自然流产,本研究检出3例全基因组单亲二倍体。
常规核型分析、基于微阵列的比较基因组杂交(array-based comparative genomic hybridization,aCGH)[21]、低深度高通量测序(copy number variation sequencing,CNV-seq)[10]等都无法检测单亲二倍体,而单核苷酸多态性微阵列芯片不但可进行染色体剂量的检测,还能对单核苷酸多态性位点进行分型检测,故其能检出单亲二倍体,体现了单核苷酸多态性微阵列芯片的独特优势。
目前,在国内复发性流产仍定义为同一夫妻发生3次或3次以上在妊娠28周之前的流产或胎停[22]。由于我国现阶段生育年龄延迟、计划生育政策改变,高龄孕妇越来越多,流产再发风险也相应增加。同时有研究发现自然流产2次和3次者的病因构成比相似[23]。近年来大多数专家认为将连续两次的自然流产定义为复发性流产。本研究中,比较了首次流产和2次及2次以上流产者的染色体异常发生频率和分布,发现两组间没有显著差异,这与Wang等[6]研究结果一致,提示染色体异常的频率和分布与流产的频率无关。因此,建议在临床实践中,无论是首次还是复发性流产,如需要查明流产原因,应对妊娠产物标本做CMA分析。
此外,本结果还显示,高龄(≥35岁)产妇组染色体总异常的频率明显高于低龄(<35岁)产妇组,这与Wang等[6]研究结果一致。非整倍体发生频率在高龄产妇组明显高于年轻产妇组,而其他染色体异常的频率在两组间无显著差异。表明胚胎非整倍体的发生率随母体年龄的增加而增加,而多倍体、染色体结构异常和UPD的发生率似乎与母体年龄无关。
综上所述,SNP微阵列芯片是一种可靠、快速、高分辨率、全基因组水平的临床妊娠流产的染色体检测方法。SNP微阵列芯片可以同时检测非整倍体、多倍体、亚显微观结构的染色体拷贝数变异以及LOH,提高了流产物遗传学检测中染色体异常的检出率。总之,SNP微阵列分析是确定流产遗传病因的一种有价值的方法。
