上海凡纳滨对虾源副溶血弧菌耐药性及其耐药基因检测分析
2021-08-02高晓华安伟张明辉
高晓华 安伟 张明辉


摘要:【目的】掌握上海地區养殖凡纳滨对虾源副溶血弧菌的耐药性及耐药基因携带情况,并明确耐药表型与耐药基因间的相关性,为科学防控凡纳滨对虾副溶血弧菌病及揭示耐药机制提供理论依据。【方法】通过K-B纸片扩散法检测21株上海凡纳滨对虾源副溶血弧菌对14种常见抗生素的敏感性,采用二倍稀释法测定高度敏感抗生素对副溶血弧菌的最小抑菌浓度(MIC)和最小杀菌浓度(MBC),并以PCR检测其耐药基因的携带情况,包括β-内酰胺类耐药基因blaTEM和blaCARB、磺胺类耐药基因 Sul II和Sul III、氨基糖苷类耐药基因strA和strB、四环素类耐药基因tetA及 I类整合子inT1基因。【结果】21株上海凡纳滨对虾源副溶血弧菌对14种常见抗生素表现出不同程度的多重耐药性,六重耐药1株(4.76%),五重耐药3株(14.29%),四重耐药5株(23.81%),三重耐药8株(38.10%),二重耐药4株(19.05%);AMP/PG/SMX和PG/SMX为优势耐药菌谱。上海凡纳滨对虾源副溶血弧菌对恩诺沙星的敏感率最高(90.48%),对应的MIC为0.10~1.60 μg/mL、MBC为3.20~12.80 μg/mL。blaTEM、blaCARB、Sul II、Sul III、strB和inT1等耐药基因的检出率分别为23.81%、71.43%、33.33%、19.05%、9.52%和23.81%,未检测出strA和tetA基因。从21株上海凡纳滨对虾源副溶血弧菌中共检出11种耐药基因型,其优势耐药基因型有blaCARB(28.57%)、blaCARB-blaTEM(14.29%)、blaCARB-Sul II(14.29%)和Sul III-inT1(9.52%)。除strA基因和tetA基因外,其余耐药基因与其对应抗生素耐药表型间均存在相关性,但其相关性不显著(P>0.05)。【结论】上海凡纳滨对虾源副溶血弧菌对14种常见抗生素表现出不同程度的敏感性,对恩诺沙星的敏感性最高,故可考虑将恩诺沙星作为上海地区凡纳滨对虾副溶血弧菌病防控的备选药物。此外,针对对虾源副溶血弧菌日趋严重的多重耐药问题,应同时加强副溶血弧菌耐药基因及整合子携带情况的监测,掌握其流行趋势,以提高对虾副溶血弧菌病的防控策略。
关键词: 凡纳滨对虾;副溶血弧菌;耐药性;耐药基因;上海
中图分类号: S945.49 文献标志码: A 文章编号:2095-1191(2021)03-0827-10
Resistance of Litopenaeus vannamei from Shanghai to Vibrio parahaemolyticus and detection and analysis of resistance genes
GAO Xiao-hua, AN Wei, ZHANG Ming-hui*
(Shanghai Fisheries Research Institute/Shanghai Fisheries Technical Extension Station, Shanghai 200433, China)
Abstract:【Objective】In order to provide theoretical basis for scientific control of Vibrio parahaemolyticus disease in Litopenaeus vannamei and further reveal the antibiotics resistance mechanism of V. parahaemolyticus, the antibiotics resistance and the carrying status of related resistance genes were detected in V. parahaemolyticus isolated from L. vannamei in Shanghai, the correlation between resistance phenotype and resistance genes was also studied. 【Method】The susceptibility of 21 strains of V. parahaemolyticus (isolated from L. vannamei in Shanghai) against 14 common antibiotics were detected by K-B disc diffusion test, and the minimum inhibitory concentration(MIC) and minimum bactericidal concentration(MBC) of more sensitive antibiotics were determined by double dilution method. The carrying statuses of blaTEM and blaCARB of β-lactam resistance genes, Sul II and Sul III of sulfanilamide resistance genes, strA and strB of aminoglycoside resistance genes, tetA of tetracycline resistance genes and inT1 of integrons of class I genes in V. parahaemolyticus were detected by PCR method. 【Result】The 21 strains of V. parahaemolyticus showed multi-drug resistance to 14 antibiotics, which were one strain of resistance manifested to 6(4.76%), three strains of resistance manifested to 5(14.29%), five strains of resistance manifested to 4(23.81%), eight strains of resistance manifested to 3(38.10%) and four strains of resistance manifested to 2(19.05%). The dominant antibiotic resistance phenotypes were AMP/PG/SMX and PG/SMX. For the sensitivity rates of the tested antibiotics in the strains of V. parahaemolyticus,enrofloxacin was the most sensitive antibiotic(90.48%). MIC and MBC of enrofloxacin were 0.10-1.60 μg/mL and 3.20-12.80 μg/mL. PCR results of resistance genes showed that the detection rates of gene blaTEM, blaCARB, Sul II, Sul III, strB and inT1 were 23.81%,71.43%,33.33%,19.05%,9.52% and 23.81%, respectively. strA and tetA were not detected . A total of 11 drug-resistant genotypes were detected from 21 strains of V. parahaemolyticus from L. vannamei in Shanghai, the dominant genotypes including blaCARB(28.57%), blaCARB - blaTEM(14.29%), blaCARB-Sul II(14.29%) and Sul III-inT1(9.52%). Except strA and tetA, other resistance genes were correlated with their antibiotic resistance phenotypes, but the correlation was not significant(P>0.05). 【Conclusion】The strains of V. parahaemolyticus isolated from L. vannamei in Shanghai showed different sensitivity levels to 14 antibiotics, enrofloxacin with the most sensibility. So, enrofloxacin can be considered as an alternative drug for the prevention and control of V. parahaemolyticus in L. vannamei in Shanghai. Besides, to solve the problem of multiple drug resistance of V. parahaemolyticus, detecting drug resistance genes and integrons in V. parahaemolyticus frequently and studying the trends are recognized as the effective strategies in V. parahaemolyticus prevention and control in L. vannamei.
Key words: Litopenaeus vannamei; Vibrio parahaemolyticus; antibiotics resistance; resistance genes; Shanghai
Foundation item:Youth Talent Growth Plan Project of Shanghai Municipal Agricultural System(Hunongqingzi〔2017〕3-9)
0 引言
【研究意义】副溶血弧菌(Vibrio parahaemolyticus)为革兰氏阴性菌,广泛分布于海洋、河海交汇处及鱼虾贝类等水生动物体内,是多种水生动物的条件致病菌(李毅财等,2012;茆丹,2014)。近年来的研究表明,携带有特异质粒的副溶血弧菌是引起对虾急性肝胰腺坏死病(Acute hepatopancreatic necrosis disease,AHPND)暴发的主要原因(Lee et al.,2015)。AHPND可造成养殖对虾大批量死亡,严重制约对虾养殖业的可持续健康发展(文国樑等,2015)。目前,抗生素治疗仍是防治对虾副溶血弧菌病的主要手段,但长期使用抗生素极易造成菌株多重耐药现象加重及环境污染(Rodríguez-Blanco et al.,2012;Lopatek et al.,2018),已引起从业者们的高度重视。此外,副溶血弧菌能通过食物链传播给人类,而引起食源性中毒,危及人体健康(陈志芸等,2015)。因此,加强副溶血弧菌的耐药性检测及其耐药机制研究,对指导人工养殖对虾副溶血弧菌病科学防控和维护人类健康均具有重要意义。【前人研究进展】至今,针对人工养殖对虾及鱼源副溶血弧菌的耐药性问题国外已有较多研究报道(de Melo et al.,2011;Mohamad et al.,2019);国内也有学者对江苏(张晓君等,2009)、广西(黄伟德等,2018)及广东(刘欢等,2018)等地的虾源副溶血弧菌分离株进行耐药性研究,并证实不同来源分离株的耐药程度存在一定差异,部分菌株已出现多重耐药现象。细菌多重耐药性的形成与菌株携带相应耐药基因及耐药基因可随质粒或整合子等可移动遗传元件在细菌间水平传播的机制密切相关(Ceccarelli et al.,2006)。blaTEM和blaCARB是β-内酰胺类常见耐药基因,分别位于质粒和染色体上(Colomer-Lluch et al.,2011;Chiou et al.,2015);Sul II和Sul III基因编码二氢叶酸合成酶,介导细菌对磺胺类药物的耐药性,在质粒和染色体上均有检出(乔毅等,2018);strA和strB是常见的氨基糖苷类耐药基因,编码氨基糖苷类磷酸转移酶(Kitiyodom et al.,2010;刘旭,2016);tetA基因常位于质粒或转座子上,介导四环素类抗生素的耐药性(Ng et al.,2001);I类整合子(inT1)在细菌中最常见,能整合、捕获外源耐药基因,且可在细菌间进行水平转移(Hall and Collis,1995)。黄伟德等(2018)通过检测广西凡纳滨对虾源副溶血弧菌对常见抗生素的敏感性及其耐药基因携带率,结果显示,广西沿海凡纳滨对虾源副溶血弧菌对氟苯尼考最敏感,对磺胺二甲嘧啶和青霉素G耐药性最强,其优势耐药基因型是ant(3")-I ?Sul1+tet(A)?TEM ?和ant(3")-I ?Sul1?tet(A)?TEM ?。赵姝等(2019)通过分析海南、江苏及山东等地分离自海水患病水产动物源副溶血弧菌喹诺酮类药物耐药基因的分布状况,发现副溶血弧菌对喹诺酮类药物的耐药表型与基因型存在明显差异,其中外排泵抑制剂对副溶血弧菌喹诺酮类药物的耐药性具有显著影响。【本研究切入点】目前,有关凡纳滨对虾源副溶血弧菌的研究主要集中在分离株耐药性与耐药基因相关性方面(黄伟德等,2018;赵姝等,2019;贺晓晨等,2020),但鲜见针对上海地区养殖凡纳滨对虾源副溶血弧菌耐药性及其耐药基因检测的相关研究。【拟解决的关键问题】通过K-B纸片扩散法检测分离自上海地区凡纳滨对虾源副溶血弧菌对常见抗生素的敏感性,运用PCR检测副溶血弧菌耐药基因的携带情况,分析耐药表型与耐药基因间的相关性,并测定敏感抗生素对凡纳滨对虾源副溶血弧菌的最小抑菌浓度(MIC)和最小杀菌浓度(MBC),以期为科学防控凡纳滨对虾副溶血弧菌病及揭示其耐药机制提供理论依据。
1 材料与方法
1. 1 菌株来源
2017—2020年从上海地区低盐度淡化对虾养殖场凡纳滨对虾肝胰腺部位分离得到21株副溶血弧菌,由上海市水产研究所病害防治科鉴定并采用甘油冷冻保存法保存于-80 ℃冰箱中。菌株按分离时间顺序分别编号为1~21,菌株来源信息详见表1。菌株药敏试验采用大肠埃希菌(Escherichia coli)ATCC25922为质控菌株。
1. 2 主要试剂
胰蛋白胨大豆肉汤培养基(TSB)和胰蛋白胨大豆琼脂培养基(TSA)购自北京陆桥技术股份有限公司;Muller-Hinton琼脂培养基(MHA)购自英国Oxoid公司,琼脂糖、4S Red Plus核酸染色剂及4种抗生素(恩诺沙星、氟苯尼考、硫酸新霉素和盐酸多西环素)购自生工生物工程(上海)股份有限公司;PCR Premix Taq、DL2000 DNA Marker及TaKaRa MiniBEST细菌基因组DNA提取试剂盒购自宝生物工程(大连)有限公司;14种常见抗生素药敏纸片均购自英国Oxoid公司,包括青霉素G(PG,10 μg/片)、氨苄西林(AMP,10 μg/片)、磺胺甲噁唑(SMX,100 μg/片)、多西环素(DO,30 μg/片)、土霉素(OT,30 μg/片)、新霉素(N,30 μg/片)、链霉素(S,10 μg/片)、庆大霉素(CN,10 μg/片)、氯霉素(C,30 μg/片)、氟苯尼考(FFC,30 μg/片)、恩諾沙星(ENR,5 μg/片)、诺氟沙星(NOR,10 μg/片)、环丙沙星(CIP,5 μg/片)、红霉素(E,15 μg/片),所有药敏纸片置于4 ℃冰箱保存备用。
1. 3 菌株活化
无菌条件下,将-80 ℃保存的副溶血弧菌在室温解冻后,接种至TSA培养基上,置于30 ℃恒温培养箱培养24 h,挑取单个菌落接种至TSB液体培养基中,再置于30 ℃恒温摇床(200 r/min)过夜培养,获得的菌悬液用于药敏試验及细菌基因组DNA提取。
1. 4 药敏试验
采用K-B纸片扩散法检测21株上海凡纳滨对虾源副溶血弧菌对14种常见抗生素的药敏性,参照美国临床和实验室标准协会(CLSL)抗生素敏感试验标准进行判定,药敏结果分别以S(敏感)、I(中介)和R(耐药)进行记录。
1. 5 最小抑菌浓度(MIC)和最小杀菌浓度(MBC)测定
根据药敏试验结果选取高度敏感的抗生素,采用试管二倍稀释法,将抗生素分别加入含2.00 mL无菌TSB培养基的试管中,使其终质量浓度分别为0、0.05、0.10、0.20、0.40、0.80、1.60、3.20、6.40、12.80、25.60、51.20、102.40和204.80 μg/mL,并设阴性对照组(空白无菌TSB培养基),然后接种菌悬液至终浓度为1.0×108 CFU/mL,置于30 ℃恒温摇床(200 r/min)培养24 h,无菌生长的最高抗生素质量浓度即为MIC。从所有无细菌生长的各试管中分别取100 μL菌悬液,涂布于无菌TSA培养基上,置于30 ℃生化培养箱培养24 h,无菌生长的最低抗生素质量浓度即为MBC。
1. 6 耐药基因检测
1. 6. 1 基因组DNA提取 取适量活化菌悬液置于离心管内中,12000 r/min离心2 min,收集菌体,根据TaKaRa细菌基因组DNA提取试剂盒说明提取各菌株基因组DNA,利用酶标仪进行DNA浓度测定, -20 ℃保存备用。
1. 6. 2 引物设计与合成 参照Colomer-Lluch等(2011)、Chiou等(2015)的方法设计β-内酰胺类耐药基因blaTEM和blaCARB的扩增引物,参照Pei等(2006)的方法设计磺胺类耐药基因Sul II和Sul III的扩增引物,参照Kitiyodom等(2010)、刘旭(2016)的方法设计氨基糖苷类耐药基因strA和strB的扩增引物,参照Ng等(2001)的方法设计四环素类耐药基因tetA的扩增引物,参照左志晗等(2018)的方法设计整合子inT1基因的扩增引物,上述所有扩增引物(表2)均委托生工生物工程(上海)股份有限公司合成。
1. 6. 3 耐药基因PCR扩增 PCR反应体系25.0 μL:PCR Premix 12.5 μL,上、下游引物各1.0 μL,细菌基因组DNA模板2.0 μL,无菌双蒸水补足至25.0 μL。设无菌双蒸水为阴性对照模板。扩增程序:95 ℃预变性5 min;95 ℃ 30 s,退火30 s(具体退火温度见表2),72 ℃ 60 s,进行35个循环;72 ℃ 延伸10 min。PCR扩增产物经1.0%琼脂糖凝胶电泳(130 V,30 min)检测后,送至生工生物工程(上海)股份有限公司测序,测序结果输入GenBank中进行BLAST比对分析。
1. 7 数据分析
耐药表型与耐药基因间符合率(%)=携带耐药基因且具有相应耐药表型菌株数/相应耐药表型菌株总数×100。菌株耐药表型和耐药基因的相关性采用SPSS 13.0进行费尔希精确检验。
2 结果与分析
2. 1 药敏试验结果
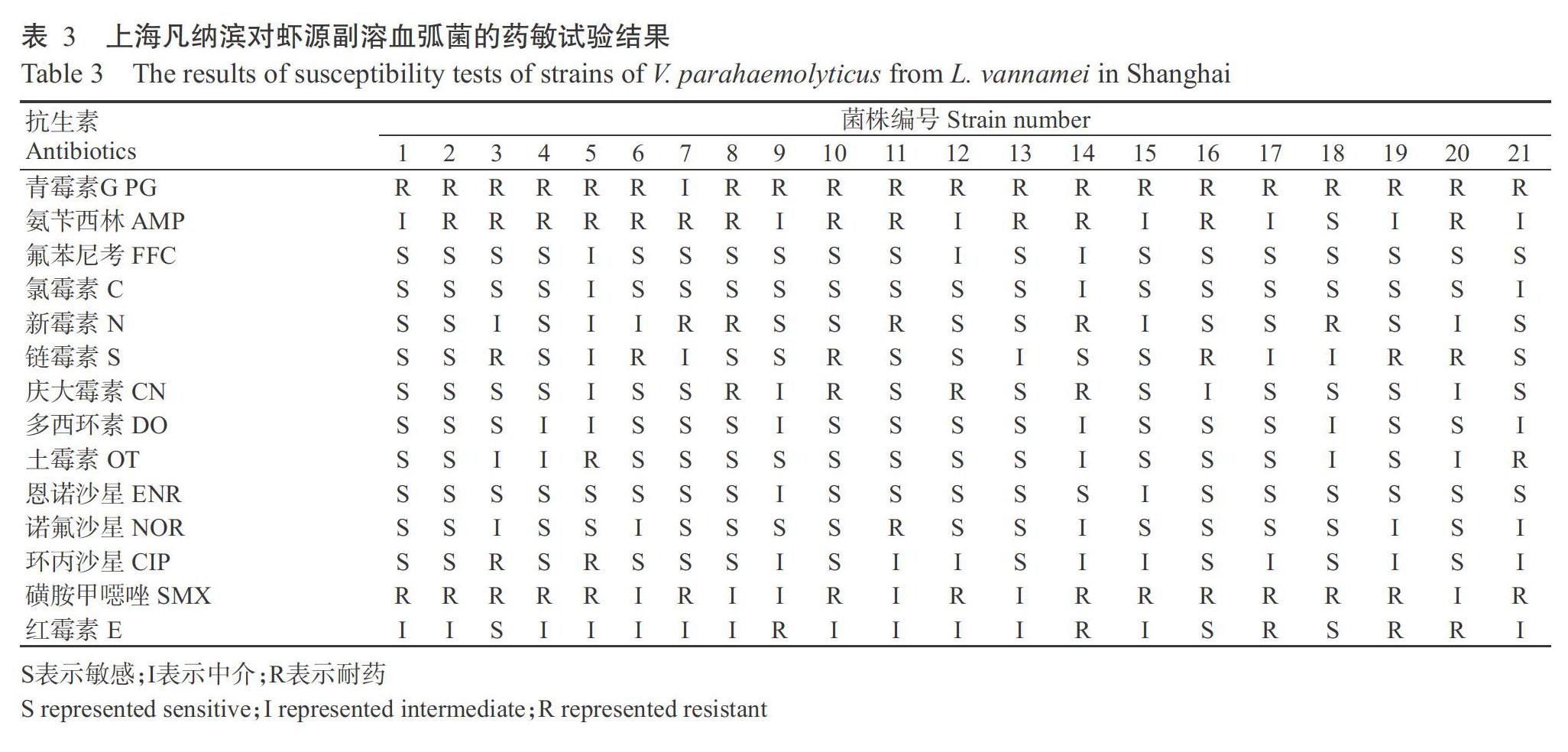
由表3可知,21株上海凡纳滨对虾源副溶血弧菌对14种常见抗生素表现出不同程度的多重耐药性,其中,六重耐药1株(4.76%),五重耐药3株(14.29%),四重耐药5株(23.81%),三重耐药8株(38.10%),二重耐药4株(19.05%)。21株副溶血弧菌的耐药菌谱分别是:AMP/PG/SMX占被检测菌株总数的14.29%(3/21),PG/SMX占被检测菌株总数的9.52%(2/21),其余依次为AMP/PG/SMX/CIP/S、AMP/PG/SMX/OT/CIP、PG/SMX/S、AMP/PG/CN、PG/E、AMP/PG/S/CN/SMX、AMP/PG/NOR、PG/SMX/CN、PG/AMP、AMP/PG/CN/SMX/E、AMP/PG/S/SMX、PG/SMX/E、PG/SMX/N、PG/SMX/E/S、AMP/PG/S/E和PG/SMX/OT,均占被检测菌株总数的4.76%(1/21)。
21株上海凡纳滨对虾源副溶血弧菌对14种常见抗生素的耐药程度也存在一定差异(图1),对青霉素G、磺胺甲噁唑、氨苄西林的耐药率分别95.24%、71.43%和61.91%。21株副溶血弧菌对恩诺沙星、氟苯尼考、氯霉素和多西环素均呈敏感或中介,其中对恩诺沙星的敏感率最高(90.48%),其次为氟苯尼考(85.71%)和氯霉素(85.71%);对新霉素、土霉素、庆大霉素、链霉素、环丙沙星和诺氟沙星的敏感率介于45.00%~70.00%。大部分上海凡纳滨对虾源副溶血弧菌对红霉素的敏感性为中介,其中介率为61.91%。
2. 2 MIC和MBC测定结果
根据药敏试验结果,选取高度敏感且水产养殖中允许使用的4种抗生素(恩诺沙星、氟苯尼考、多西环素和新霉素)进行MIC和MBC测定,结果表明,恩诺沙星对21株上海凡纳滨对虾源副溶血弧菌的MIC为0.10~1.60 μg/mL,MBC为3.20~12.80 μg/mL;氟苯尼考对上海凡纳滨对虾源副溶血弧菌的MIC为0.80~25.60 μg/mL,MBC为6.40~51.20 μg/mL;多西环素对上海凡纳滨对虾源副溶血弧菌的MIC为0.20~12.80 μg/mL,MBC为3.20~25.60 μg/mL;新霉素对上海凡纳滨对虾源副溶血弧菌的MIC为0.80~51.20 μg/mL,MBC为6.40~51.20 μg/mL。这4种抗生素对上海凡纳滨对虾源副溶血弧菌的抑菌效果和杀菌效果排序为恩诺沙星>多西环素>氟苯尼考>新霉素。
2. 3 耐药基因检测结果
7种常见耐药基因及I类整合子inT1基因的PCR扩增电泳结果见图2。其中,β-内酰胺类耐药基因blaTEM、blaCARB检出率分别为23.81%(5/21)和71.43%(15/21);磺胺类耐药基因Sul II、Sul III检出率分别为33.33%(7/21)和19.05%(4/21);氨基糖苷类耐药基因strB检出率为9.52%(2/21),但未检出strA基因;四环素类耐药基因tetA也未检出;整合子inT1基因检出率为23.81%(5/21),且inT1阳性菌株均表现为多重耐药表型,部分副溶血弧菌可同时对青霉素G、氨苄西林、环丙沙星、磺胺甲噁唑及链霉素等5种抗生素表现出耐药表型。从21株上海凡纳滨对虾源副溶血弧菌中共检出11种耐药基因型,分别为:blaCARB(28.57%)、blaCARB-blaTEM(14.29%)、blaCARB-Sul II(14.29%)、Sul III-inT1(9.52%)、bla-CARB-strB-Sul II-inT1(4.76%)、strB-Sul II-Sul III-inT1(4.76%)、blaCARB-inT1(4.76%)、blaTEM-Sul II(4.76%)、blaCARB-Sul III(4.76%)、blaTEM(4.76%)和Sul II(4.76%)。
2. 4 耐药表型与耐药基因型的相关性
由表4可知,在21株上海凡纳滨对虾源副溶血弧菌中,青霉素G耐药表型与blaCARB基因间的符合率为75.00%,与blaTEM基因间的符合率为20.00%;氨苄西林耐药表型与blaCARB基因间的符合率为69.23%,与blaTEM基因间的符合率为23.08%;土霉素耐药表型与tetA基因间的符合率为0,由于未检测出对多西环素的耐药菌株,故无法计算多西环素耐药表型与tetA基因间的符合率;磺胺甲噁唑耐药表型与Sul II基因间符合率为40.00%,磺胺甲噁唑耐药表型与Sul III基因间符合率为20.00%;新霉素、链霉素、庆大霉素耐药表型与strA基因、strB基因间符合率均为0;由于未检测出strA基因和tetA基因,故无法计算新霉素、链霉素和庆大霉素耐药表型与strA基因间的相关性,以及土霉素和多西环素耐药表型与tetA基因间的相关性。除strA基因和tetA基因外,其余耐药基因与其对应抗生素耐药表型间均存在相关性,但其相关性不显著(P>0.05)。
3 讨论
近年来,我国学者针对不同地区虾源副溶血弧菌耐药性问题已开展了系列相关研究。魏文娟等(2020)对2017—2018年分离自江苏和福建等地的虾源副溶血弧菌进行耐药性检测,结果发现这些分离株对磺胺甲噁唑的耐药率为66.70%。本研究结果表明,21株上海凡纳滨对虾源副溶血弧菌对磺胺甲噁唑的耐药率高达71.43%,究其原因是磺胺甲噁唑因具有广谱抗菌作用、价格低廉等特点,被长期应用于水产养殖动物细菌性疾病防治,随着时间积累在水体环境中的残留问题日渐凸显。汪涛等(2017)研究认为,养殖水环境中残留的磺胺类药物能诱导副溶血弧菌对其产生耐药性。本研究中,上海凡纳滨对虾源副溶血弧菌对青霉素G和氨苄西林均表现出较高耐药性,与张晓君等(2009)对江苏省凡纳滨对虾源副溶血弧菌耐药性的研究结果相似,可能与菌株携带能水解β-内酰胺类抗生素的耐药基因有关(Chiou et al.,2015)。此外,上海凡纳滨对虾源副溶血弧菌对红霉素、土霉素和新霉素等表现出不同程度的耐药性,且多重耐药比例为80.85%,明显高于刘旭(2016)检测2014—2015年分离自上海郊区弧菌分离株的多重耐药比例(66.67%),表明副溶血弧菌多重耐药问题已经较严重,且呈逐年持续增长的趋势。上海凡纳滨对虾源副溶血弧菌对恩诺沙星的敏感率为90.48%,对应的MIC、MBC分别为0.10~1.60和3.20~12.80 μg/mL,即恩诺沙星的抑菌和杀菌效果明显优于其他抗生素,因此可考虑将其作为上海凡纳滨对虾副溶血弧菌病防控的备选药物,但需谨慎选择使用浓度,避免大剂量滥用,已减少抗生素残留诱发水体细菌产生多重耐药的风险。
在细菌耐药表型与耐药基因型相关性的研究中,乔毅等(2018)研究认为Sul基因通过编码产生二氢叶酸合成酶,促使细菌失去对磺胺类药物的敏感性,而介导细菌对磺胺类药物耐药。本研究结果表明,磺胺甲噁唑耐药表型与Sul II和Sul III基因间的符合率分别为40.00%和20.00%,但在携带Sul基因(Sul II和Sul III)的10株副溶血弧菌中仅有2株对磺胺甲噁唑中介,说明Sul基因与副溶血弧菌对磺胺类耐药表型间并非完全对应,可能与不同来源副溶血弧菌体内耐药基因的拷贝量及耐药基因有效表达程度存在差异有关。在本研究中,上海凡纳滨对虾源副溶血弧菌的blaTEM基因检出率为23.81%,高于广西凡纳滨对虾源副溶血弧菌的检出率(13.30%)(黄伟德等,2018),但低于宁波地区副溶血弧菌的检出率(91.47%)(吴蓓蓓等,2011);blaCARB基因检出率高达71.43%,青霉素G、氨苄西林耐药表型与bla-CARB基因间的符合率分别为75.00%和69.23%,青霉素G、氨苄西林耐药表型与blaTEM基因间的符合率分别为20.00%和23.08%,说明上海凡纳滨对虾源副溶血弧菌blaCARB基因产生β-内酰胺酶(CARB),可能是介导副溶血弧菌对青霉素G和氨苄西林耐药的主要原因,也提示blaCARB基因在采样区的养殖虾塘内已广泛流行,而养殖环境中的细菌是加速耐药基因传播的主要媒介。tetA基因是四环素类耐药基因,其编码产生的膜蛋白tetA可特异性将细菌胞内的四环素类药物泵到胞外,以降低胞内药物浓度,介导细菌对四环素类的耐药性(Miranda et al.,2003)。在21株上海凡纳滨对虾源副溶血弧菌中均未检测到tetA基因,但仍有9.25%的试验菌株对四环素类抗生素耐药,与黄伟德等(2018)对广西虾源副溶血弧菌的研究结果相似。四环素类药物耐药机理除了tetA基因介导的外排泵耐药机制外,还存在tetM和tetX等基因介导的四环素类钝化及核糖体保护等其他作用机制(梁静真等,2018;黄雪龙等,2019)。因此,上海凡纳滨对虾源副溶血弧菌对四环素类的耐药表型是否由其他耐药基因介导产生尚有待进一步探究。strA和strB基因编码产生链霉素钝化酶,介导细菌对链霉素的耐药性(杨元斌等,2011)。strB基因在上海凡納滨对虾源副溶血弧菌中的检出率为9.52%,对链霉素、新霉素、庆大霉素的耐药率分别为28.57%、23.81%和19.05%,耐药基因与耐药表型间的符合率为0。杨明伟等(2018)研究发现,在鱼源链球菌中链霉素耐药表型与ant(3')-I基因间的符合率高达75.00%,氨基糖苷类的耐药基因还包括aac(3')-Ib、aph(3')-Ia及aac(6')-Ib等。本研究仅检测strA基因和strB基因有一定局限性,今后应增加耐药基因不同类型的检测,而更有利于揭示菌株耐药基因与耐药表型间的相关性。inT1基因在上海凡纳滨对虾源副溶血弧菌中的检出率为23.81%,与李林桂(2013)、姚小娟(2014)的研究结果存在明显差异,且携带inT1基因的菌株均出现多重耐药表型,可能与整合子具有整合、捕获外源耐药基因的特性相关(Hall and Collis,1995)。因此,针对对虾源副溶血弧菌日趋严重的多重耐药问题,应同时加强副溶血弧菌耐药基因及整合子携带情况的监测,掌握其流行趋势,以提高对虾副溶血弧菌病的防控策略。
本研究还发现,上海凡纳滨对虾源副溶血弧菌对抗生素的耐药表型与耐药基因间的相关性并不显著,究其原因可能是:①不同来源菌株的基因拷贝量存在明显差异,以致部分低拷贝量的耐药基因不能被检出(石优章等,2007)。②细菌外膜蛋白参与启动菌体的主动药物外排系统,减少胞内药物浓度而导致细菌耐药(Matsuo et al.,2007)。③不同菌株耐药基因转录与翻译受其启动子类别、强弱及距启动子距离等多种因素的影响。如blaTEM基因位于质粒上,其上游启动子有P3、P4及Pa/Pb等不同类别,尤其是P4启动子能有效促进blaTEM基因转录与表达(Lartigue et al.,2002)。④菌株耐药表型还由其他耐药基因介导产生,当菌株所处环境不利于其生长时,可通过整合外源耐药基因,调节下游基因的转录和表达以适应环境变化(袁璐等,2014)。⑤抗生素能诱导细菌耐药形成,如嗜水气单胞菌在多次低抑菌浓度的盐酸沙拉沙星刺激下,可诱导产生对盐酸沙拉沙星高耐药性的菌株(黄新财等,2013)。综上所述,菌株对抗生素的耐药表型受诸多因素影响,且大部分耐药基因可在菌株间水平传播,势必增加凡纳滨对虾副溶血弧菌病的防控难度和风险。因此,加强副溶血弧菌耐药性及其耐药基因的监测,可为科学防控凡纳滨对虾副溶血弧菌病及揭示其耐药机制提供理论依据。
4 结论
上海凡纳滨对虾源副溶血弧菌对14种抗生素表现出不同程度的敏感性,对恩诺沙星的敏感性最高,故可考虑将恩诺沙星作为上海凡纳滨对虾副溶血弧菌病防控的备选药物。此外,针对对虾源副溶血弧菌日趋严重的多重耐药问题,应同时加强副溶血弧菌耐药基因及整合子携带情况的监测,掌握其流行趋势,以提高对虾副溶血弧菌病的防控策略。
参考文献:
陈志芸,施春雷,周秀娟,张茜,周敏,王大鹏,史贤明. 2015. 上海市售海产品中副溶血性弧菌的分布状况[J]. 中国食品学报,15(8):196-202. doi:10.16429/j.1009-7848.2015. 08.028. [Chen Z Y,Shi C L,Zhou X J,Zhang Q,Zhou M,Wang D P,Shi X M. 2015. Distribution status of Vi-brio parahaemolyticus in seafood markets of Shanghai[J]. Journal of Chinese Institute of Food Science and Technology,15(8):196-202.]
贺晓晨,韩书煜,黎姗梅,胡大胜,黄德生,吴伟锋,黄钧,梁静真,胡庭俊. 2020. 广西凡纳滨对虾源副溶血弧菌耐药性及其整合子—基因盒检测[J]. 西南农业学报,33(4):893-900. doi:10.16213/j.cnki.scjas.2020.4.032. [He X C,Han S Y,Li S M,Hu D S,Huang D S,Wu W F,Huang J,Liang J Z,Hu T J. 2020. Detection of antibiotic resi-stance and integron-gene cassettes in Vibrio parahaemolyticus from Litopenaeus vannamei in Guangxi[J]. Southwest China Journal of Agricultural Sciences,33(4):893-900.]
黄伟德,肖双燕,黎姗梅,黄德生,张振豪,钟昌艳,陈福彩,梁毅,梁静真,黄钧. 2018. 广西凡纳滨对虾源副溶血弧菌耐药性和耐药基因的检测[J]. 西南农业学报,31(9):1979-1988. doi:10.16213/j.cnki.scjas.2018.9.034. [Huang W D,Xiao S Y,Li S M,Huang D S,Zhang Z H,Zhong C Y,Chen F C,Liang Y,Liang J Z,Huang J. 2018. Detection of antibiotics resistance and resistance genes in Vibrio parahaemolyticus from Litopenaeus vannamei of Guangxi[J]. Southwest China Journal of Agricultural Scien-ces,31(9):1979-1988.]
黃新财,彭民毅,黄钧,胡大胜,彭亚,黄艳华,施金谷,温华成,韦贵花. 2013. 黄颡鱼源嗜水气单胞菌对盐酸沙拉沙星耐药性获得与消失速率的测定[J]. 南方农业学报,44(10):1731-1734. doi:10.3969/j:issn.2095-1191.2013. 10.1731. [Huang X C,Peng M Y,Huang J,Hu D S,Peng Y,Huang Y H,Shi J G,Wen H C,Wei G H. 2013. Acquisition and vanishing rate of drug resistance of Aeromonas hydrophila in Pelteobagrus fulvidraco against sarafloxacin hydrochloride[J]. Journal of Southern Agriculture,44(10):1731-1734.]
黄雪龙,单敏,陈烨,刘玲俐,何晶,王小兰,于圣青,罗廷荣,涂剑,李涛. 2019. 鸭疫里默氏杆菌Tet(X)基因原核表达及其功能鉴定[J]. 南方农业学报,50(4):844-850. doi:10.3969/j.issn.2095-1191.2019.04.23. [Huang X L,Shan M,Chen Y,Liu L L,He J,Wang X L,Yu S Q,Luo T R,Tu J,Li T. 2019. The prokaryotic expression and function identification of Tet(X) gene in Riemerella anatipestifer[J]. Journal of Southern Agriculture,50(4):844-850.]
李林桂. 2013. 海水弧菌耐药性I类整合子分析与副溶血弧菌分子分型研究[D]. 雅安:四川农业大学. [Li L G. 2013. Study on class I antibiotic resistance integrons of Vibrios and multilocus sequence typing of Vibrio parahaemolyticus[D]. Yaan:Sichuan Agricultural University.]
李毅财,赵峰,朱兰兰,赵晓君,周德庆. 2012. 黄渤海区域贝类中副溶血弧菌污染调查及血清学分型[J]. 南方农业学报,43(2):241-244. doi:10.3969/j:issn.2095-1191.2012. 02.241. [Li Y C,Zhao F,Zhu L L,Zhao X J,Zhou D Q. 2012. Investigations on contamination and serotype of Vibrio parahaemolyticus in shellfish obtained from Ye-llow Sea and Bohai Sea areas[J]. Journal of Southern Agriculture,43(2):241-244.]
梁静真,黄立春,韦慕兰,马沙,黎姗梅,文衍红,蒙兰丽,黄维,黄松,黄钧. 2018. 广西罗非鱼源无乳链球菌耐药性及其四环素耐药基因检测[J]. 南方农业学报,49(10): 2077-2086. doi:10.3969/j.issn.2095-1191.2018.10.26. [Liang J Z,Huang L C,Wei M L,Ma S,Li S M,Wen Y H,Meng L L,Huang W,Huang S,Huang J. 2018. Detection of antibiotics resistance and tetracycline resistance genes in Streptococcus agalactiae from tilapia in Guangxi[J]. Journal of Southern Agriculture,49(10):2077-2086.]
劉欢,谢婷,何美珊,丁楠,钟青萍. 2018. 广州市售海产品中副溶血弧菌分离菌株的鉴定及其耐药性分析[J]. 食品与发酵工程,44(11):28-34. doi:10.13995/j.cnki.11-1802/ts.017408. [Liu H,Xie T,He M S,Ding N,Zhong Q P. 2018. Isolation, identification and antibiotic resistance analysis of Vibrio parahaemolyticus in seafood from Guangzhou[J]. Food and Fermentation Industries,44(11):28-34.]
刘旭. 2016. 海水养殖源弧菌耐药性调查及qnrVC基因在弧菌中的流行情况研究[D]. 上海:上海海洋大学. [Liu X. 2016. Investigation on antimicrobial resistance and the prevalence of qnrVC gene in Vibrios from mariculture sources[D]. Shanghai:Shanghai Ocean University.]
茆丹. 2014. 不同来源副溶血弧菌分子分型及其毒力基因筛查[D]. 上海:上海交通大学. [Mao D. 2014. Molecular typing and virulence genes screening of Vibrio parahaemolyticus from different sources[D]. Shanghai:Shanghai Jiao Tong University.]
乔毅,沈辉,万夕,王李宝,史文军,黎慧,蒋葛,范贤平,张建明. 2018. 异育银鲫源嗜水气单胞菌对磺胺类耐药性分析[J]. 水产科学,37(4):456-463. doi:10.16378/j.cnki. 1003-1111.2018.04.004. [Qiao Y,Shen H,Wang X,Wang L B,Shi W J,Li H,Jiang G,Fan X P,Zhang J M. 2018. Drug resistance of Aermonas hydrophila isolate from allogyogenetic silver crucian carp Carassius auratus gibelio to sulfonamides[J]. Fisheries Science,37(4):456-463.]
石优章,宋启发,徐景野,金春光,杨元斌. 2007. O139群霍乱弧菌β-内酰胺酶相关耐药基因检测及分析[J]. 中国卫生检验杂志,17(8):1390-1391. doi:10.3969/j.issn.1004-8685.2007.08.017. [Shi Y Z,Song Q F,Xu J Y,Jin C G,Yang Y B. 2007. Study of β-lactamase related resistance genes in Vibrio cholerae O139 serogroup[J]. Chinese Journal of Health Laboratory Technology,17(8):1390-1391.]
汪涛,杨再福,陈勇航,孙冉冉,张姚姚,王亚楠. 2017. 磺胺类抗性基因的产生及演变研究进展[J]. 环境污染与防治,39(11):1251-1255. doi:10.15985/j.cnki.1001-3865.2017. 11.019. [Wang T,Yang Z F,Chen Y H,Sun R R,Zhang Y Y,Wang Y N. 2017. Review on the production and evolution of sulfonamide resistance genes[J]. Environmental Pollution & Control,39(11):1251-1255.]
魏文娟,赵姝,王元,周俊芳,李新苍,房文红. 2020. 副溶血弧菌养殖对虾分离株耐药性及耐药基因分析[J]. 南方水产科学,16(1):9-16. doi:10.12131/20190165. [Wei W J,Zhao S,Wang Y,Zhou J F,Li X C,Fang W H. 2020. Detection of antibiotics resistance and distribution of resistance genes in Vibrio parahaemolyticus from cultured shrimp[J]. South China Fisheries Science,16(1):9-16.]
文国樑,曹煜成,徐煜,胡晓娟,徐武杰,李卓佳. 2015. 养殖对虾肝胰腺坏死综合症研究进展[J]. 广东农业科学,42(11):118-123. doi:10.16768/j.issn.1004-874x.2015.11. 022. [Wen G L,Cao Y C,Xu Y,Hu X J,Xu W J,Li Z J. 2015. Review on hepatopancreas necrosis syndrome of shrimp[J]. Guangdong Agricultural Science,42(11):118-123.]
吴蓓蓓,俞盈,金培婕,方维焕. 2011. 宁波地区海产品及环境中副溶血弧菌主要毒力及耐药性分析[J]. 中国人兽共患病学报,27(5):381-385. [Wu B B,Yu Y,Jin P J,Fang W H. 2011. Analysis of major virulence and antibiotics resistance in Vibrio parahaemolyticus isolates from environment and seafood in Ningbo of Zhejiang Province[J]. Chinese Journal of Zoonoses,27(5):381-385.]
杨明伟,韦慕兰,罗福广,黎姗梅,黄立春,李明,吕小丽,王强,米强,梁静真,黄钧. 2018. 罗非鱼源无乳链球菌对氨基糖苷类耐药性及耐药基因检测[J]. 西南农业学报,31(11):2438-2444. doi:1016213/j.cnki.scjas.2018.11.035. [Yang M W,Wei M L,Luo F G,Li S M,Huang L C,Li M,Lü X L,Wang Q,Mi Q,Liang J Z,Huang J. 2018. Resistance and related genes detection of tilapia Streptococcus agalactiae against aminoglycosides[J]. Southwest China Journal of Agricultural Sciences,31(11):2438-2444.]
杨元斌,宋启发,徐景野,金春光,闫鹏. 2011. O139群霍乱弧菌耐药谱和相关耐药基因研究[J]. 中国卫生检验杂志,21(5):1070-1072. [Yang Y B,Song Q F,Xu J Y,Jin C G,Yan P. 2011. Study of resistance patterns and related resistance genes in O139-Serogroup Vibrio cholrae[J]. Chinese Journal of Health Laboratory Technology,21(5):1070-1072.]
姚小娟. 2014. 海水养殖源弧菌耐药性检测与整合子分析[D]. 上海:上海海洋大学. [Yao X J. 2014. Drug resistance detection and integrons analysis of Vibrios from mariculture source[D]. Shanghai:Shanghai Ocean University.]
袁璐,刘红波,邹雅如,许欢,陈体,漆涌,伍勇. 2014. 细菌整合子调控机制中启动子的作用研究进展[J]. 臨床检验杂志,32(8):603-605. doi:10.13602/j.cnki.jcls.2014.08. 13. [Yuan L,Liu H B,Zou Y R,Xu H,Chen T,Qi Y,Wu Y. 2014. Research progress on the role of promoters in the regulation mechanism of bacterial integrons[J]. Chinese Journal of Clinical Laboratory Science,32(8):603-605.]
张晓君,陈翠珍,阎斌伦,房海,秦国民,徐静. 2009. 凡纳滨对虾(Litopenaeus vannamei)病原副溶血弧菌(Vibrio parahaemolyticus)的表型及分子特征[J]. 海洋与湖沼,40(5):654-662. [Zhang X J,Chen C Z,Yan B L,Fang H,Qin G M,Xu J. 2009. Phenotypic and molecular characterization of pathogenic Vibrio parahaemolyticus isolated from Litopenaeus vannamei[J]. Oceanologia et Limnologia Sinica,40(5):654-662.]
趙姝,李健,马立才,刘旭,王元,房文红. 2019. 海水养殖动物源弧菌喹诺酮类药物耐药表型与基因型分析[J]. 海洋渔业,41(4):463-471. doi:10.13233/j.cnki.mar.fish.2019. 04.008. [Zhao S,Li J,Ma L C,Liu X,Wang Y,Fang W H. 2019. Analysis of phenotype and genetype in quinolone resistance in Vibrio from mairculture[J]. Marine Fishe-ries,41(4):463-471.]
左志晗,李艳红,邵迎春,耿绪云,董学旺,孙金生. 2018. 天津市周边地区养殖凡纳滨对虾肠道菌株的分离鉴定及耐药性分析[J]. 水产学报,42(5):797-807. doi:10.11964/jfc.20170410809. [Zuo Z H,Li Y H,Shao Y C,Geng X Y,Dong X W,Sun J S. 2018. Isolation,identification and antibiotic resistance analysis of intestinal strains of aquaculture Litopenaeus vannamei in surrounding areas of Tianjin[J]. Journal of Fisherier of China,42(5):797-807.]
Ceccarelli D,Salvia A M,Sami J,Cappuccinelli P,Colombo M M. 2006. New cluster of plasmid-located class 1 integrons in Vibrio cholerae O1 and a dfrA15 cassette-containing integron in Vibrio parahaemolyticus isolated in Angola[J]. Antimicrobial Agents and Chemotherapy,50(7):2493-2499. doi:10.1128/AAC.01310-05.
Chiou J C,Li R C,Chen S. 2015. CARB-17 family of β-lactamases mediates intrinsic resistance to penicillins in Vibrio parahaemolyticu[J]. Antimicrobial Agents and Chemotherapy,59(6):3593-3595. doi:10.1128/AAC.00047-15.
Colomer-Lluch M,Jofre J,Muniesa M. 2011. Antibiotic resistance genes in the bacteriophage DNA fraction of environmental samples[J]. PLoS One,6(3):e17549. doi:10. 1371/journal.pone.0017549.
de Melo L M R,Almeida D,Hofer E,Reis C M F D,Theophilo G N D,Santos A F M,Vieira R H S D F. 2011. Antibiotic resistance of Vibrio parahaemolyticus isolated from pond-reared Litopenaeus vannamei marke-ted in natal,brazil[J]. Brazilian Journal of Microbiology,42(4):1463-1469. doi:10.1590/S1517-83822011000400 0032.
Hall R M,Collis C M. 1995. Mobile gene cassettes and integrons:Capture and spread of genes by site-pecific recombination[J]. Molecular Microbiology,15(4):593-600. doi:10.1111/j.1365-2958.1995.tb02368.x.
Kitiyodom S,Khemtong S,Wongtavatchai J,Chuanchuen R. 2010. Characterization of antibiotic resistance in Vibrio spp. isolated from farmed marine shrimps(Penaeus mono-don)[J]. FEMS Microbiology Ecology,72(2):219-227. doi:10.1111/j.1574-6941.2010.00846.x.
Lartigue M F,Leflon-Guibout V,Poirel L,Nordmann P,Nicolas-Chanoine M H. 2002. Promoters P3,Pa/Pb,P4,and P5 upstream from bla(TEM) genes and their relationship to beta-lactam resistance[J]. Antimicrobial Agents and Chemotherapy,46(12):4035-4037. doi:10.1128/aac.46.12. 4035-4037.2002.
Lee C T,Chen I T,Yang Y T,Ko T P,Huang Y T,Huang J Y,Huang M F,Lin S J,Chen C Y,Lin S S,Lightner D V,Wang H C,Wang A H J,Wang H C,Hor L I,Lo C F. 2015. The opportunistic marine pathogen Vibrio arahaemolyticus becomes virulent by acquiring a plasmid that expresses a deadly toxin[J]. Proceedings of the National Academy of Sciences of the United States of America,112(34):10798-10803. doi:10.1073/pnas.1503129112.
Lopatek M,Wieczorek K,Osek J. 2018. Antimicrobial resistance,virulence factors,and genetic profiles of Vibrio parahaemolyticus from seafood[J]. Applied and Environmental Microbiology,84(16):e00537-18. doi:10.1128/AEM.00537-18.
Matsuo T,Hayashi K,Morita Y,Koterasawa M,Ogawa W,Mizushima T,Tsuchiya T,Kuroda T. 2007. VmeAB,an RND-type multidrug efflux transporter in Vibrio parahaemolyticus[J]. Microbiology,153(12):4129-4137. doi:10. 1099/mic.0.2007/009597-0.
Miranda C D,Kehrenberg C,Ulep C,Schwarz S,Roberts M C. 2003. Diversity of tetracycline resistance genes in bacteria from Chilean salmon farms[J]. Antimicrobial Agents and Chemotherapy,47(3):883-888. doi:10.1128/aac.47.3. 883-888.2003.
Mohamad N,Amal M N A,Saad M Z,Yasin I S M,Zulkiply N A,Mustafa M,Nasruddin N S. 2019. Virulence-associa-ted genes and antibiotic resistance patterns of Vibrio spp. isolated from cultured marine fishes in Malaysia[J]. BMC Veterinary Research,15(1):176. doi:10.1186/s12917-019- 1907-8.
Ng L K,Martin,Alfa M,Mulvey M. 2001. Multiplex PCR for the detection of tetracycline resistant genes[J]. Mole-cular and Cellular Probes,15(4):209-215. doi:10.1006/mcpr.2001.0363.
Pei R T,Kim S C,Carlson K H,Pruden A. 2006. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Research,40(12):2427-2435. doi:10. 1016/j.watres.2006.04.017.
Rodríguez-Blanco A,Lemos M L,Osorio C R. 2012. Integra-ting conjugative elements as vectors of antibiotic,mercuy,and quaternary ammonium compound resistance in marine aquaculture environments[J]. Antimicrobial Agents and Chemitherapy,56(5):2619-2626. doi:10.1128/AAC. 05997-11.
(責任编辑 兰宗宝)
